2015 年我国部分地区2.1d 新基因亚型猪瘟病毒的分子流行病学分析
冯丽苹,张洪亮,刘春晓,冷超粮,陈家锃,李 真,彭金美,王 倩,白 云,张武超,安同庆,蔡雪辉,童光志,田志军
(1.中国农业科学院哈尔滨兽医研究所兽医生物技术国家重点实验室,黑龙江哈尔滨 150001;2.南阳师范学院,河南南阳 473061;3.中国农业科学院上海兽医研究所,上海 200241)
猪瘟(Classical swine fever,CSF)是由猪瘟病毒(CSFV)引起的一种猪的高度接触性传染病,CSFV属于黄病毒科瘟病毒属成员,为单链RNA 病毒,基因组全长约为12.3 kb。目前,CSFV 被分为3 个基因型(1、2 和3 基因型)10 个亚型(1.1、1.2、1.3、2.1、2.2、2.3、3.1、3.2、3.3、3.4)[1],2.1 亚型被进一步分为2.1a 和2.1b[2],近几年还出现了2.1c 亚型[3]和1.4 亚型[4]。我国存在4 个亚型(1.1、2.1、2.2和2.3),上世纪九十年代后2.1b 亚型CSFV 在我国占主导地位。CSF 为危害我国养猪业的一个重要疫病,被世界动物卫生组织(OIE)列为必须上报的动物传染病之一[5]。
我国对CSF 的防控主要采取兔化弱毒疫苗(C株)强制免疫进行该病的防控,并取得了良好的效果,C 株疫苗免疫原性好,对我国2.1b 基因亚型病毒株也具有良好的交叉保护效果[2-3]。但2014 年在我国部分猪场出现了2.1d 新基因亚型的CSFV[6],为了进一步了解2.1d 亚型CSFV 在我国的流行现状,本研究对2015 年在我国部分地区检测到的CSFV 进行分子流行病学分析,为CSF 的有效防控提供了流行病学数据。
1 材料和方法
1.1 临床样品采集及处理 在4 个省份(山东、吉林、黑龙江、河北)的疑似感染CSFV 的猪场采集病猪肺、脾、肾、血清共28 份样品。将组织研磨均匀,取一部分用于提取RNA,其余的病料样品置于-80 ℃保存,用于后续研究分析。
1.2 主要试剂 RNA 提取试剂盒RNeasy plus Mini Kit 购于Qiagen 公司;AMV 反转录酶、LA Taq DNA 聚合酶、dNTPs 和pMD18-T 载体等购自TaKaRa公司。
1.3 E2 基因的扩增及测序 利用RNA 提取试剂盒提取组织匀浆和血清中的总RNA。经反转录制备cDNA。根据已发表的CSFV zj0801(FJ529205)的E2基因序列设计扩增引物(5'-GTAAATATGTGTGTGTT AGACCAGA-3'/5'-GTGTGGGTAATTGAGTTCCCTAT CA-3')。PCR 反应程序为,94 ℃5 min;94 ℃30 s、54.8 ℃30 s、72 ℃1.5 min,共35 个循环;72 ℃10 min。对PCR 产物用1 %的琼脂糖凝胶电泳鉴定。将目的片段用Gel Extraction Kit(OMEGA,USA)回收。纯化的PCR 产物克隆到pMD18-T 载体中。重组克隆由库美生物进行测序。
1.4 遗传进化分析 将阳性样品的E2 基因全长序列进行测序,利用MEGA5.1 的Neighbor-joining method 进行进化树分析。将样品序列结果与171 株参考株的E2 全长(1 119 bp)进行遗传进化分析。
1.5 E2 基因和推导氨基酸分析 利用DNAstar 软件将阳性样品的E2 基因的核苷酸和氨基酸序列与shimen、HCLV、2.1b 国内流行株及2.1d 自身进行比对。
2 结果
2.1 CSFV 检测结果 2014 年8 月起,一些经CSF兔化弱毒疫苗免疫的猪场出现疑似CSF,表现为发热、厌食、体重减轻、皮肤黄萎病等,死亡率为5 %~50 %。表1 为我们2014 年~2015 年检测到的CSFV 阳性样品信息,其中2015 年在4 个省检测到14 份阳性样品。
2.2 遗传进化树分析 进化树分析选取了119 个E2 全序列,134 个E2 部分序列[6]。CSFV 分为3 个基因型(1、2 和3),基因型2 进一步分为2.1 亚型、2.2 亚型和2.3 亚型。2.1 亚型包括2.1a、2.1b、2.1c和2.1d(图1)。2015 年检测到的14 个CSFV 病毒株有11 株(SDQZ150414、SDZC150416、SDQZ150319、SDLK150320、HLJSH150609、SDZC150514、SDZC 150601、HB150528、HB150309、JL150418、NK 150425)属于2.1d 新亚型。其余的3 个病毒株(SDZC150526、SDHS10150129、SDHS9150129)属于2.1b 亚型。E2 全序列与E2 部分序列的遗传进化树分析都显示相同的结果。
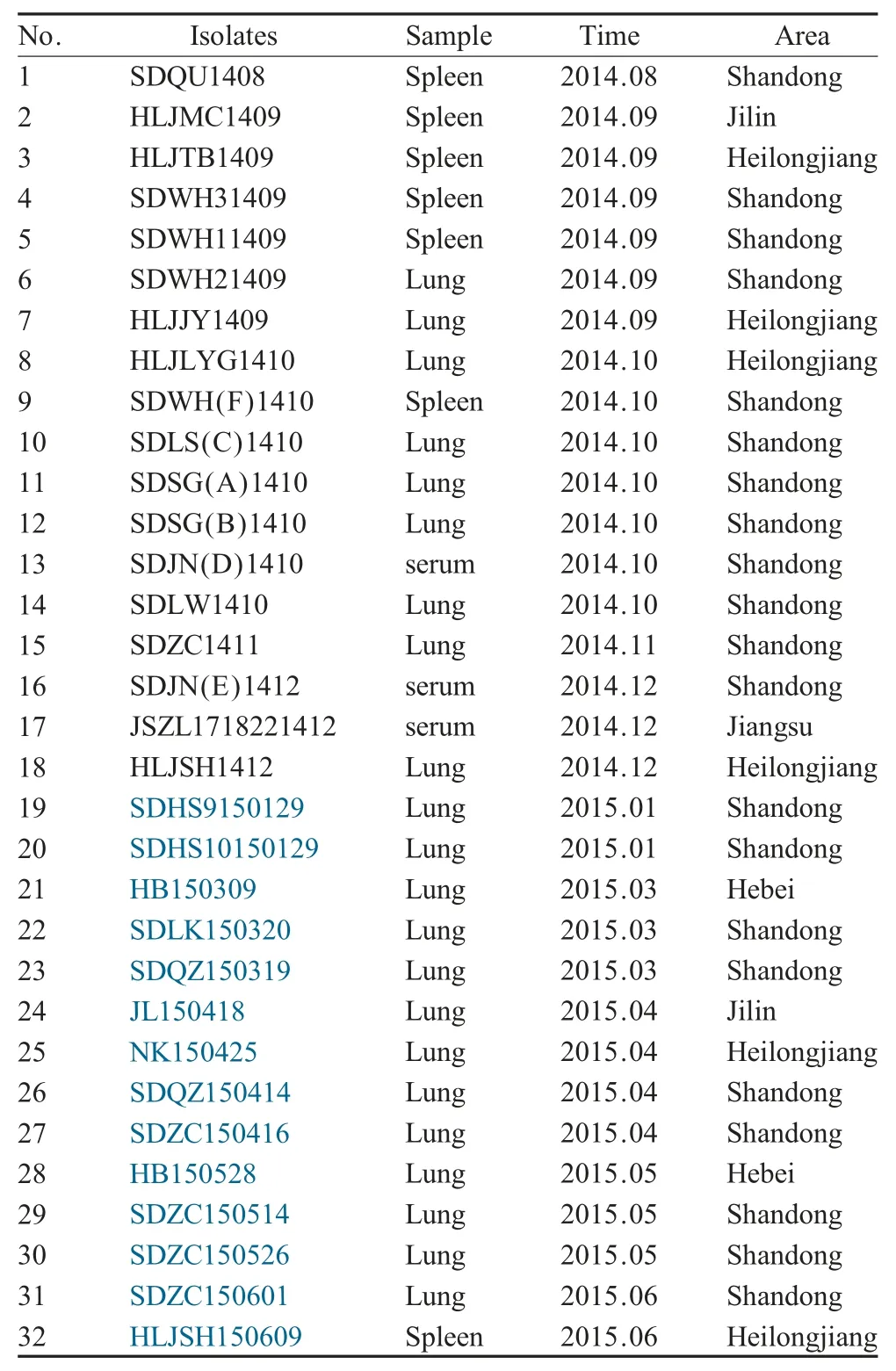
表1 2014 年~2015 年CSFV 阳性样品信息Table 1 Information of CSFVs isolated during 2014-2015
2.3 E2 的核苷酸和氨基酸序列分析 2015 年14个新分离病毒株的E2 基因全长有1119 个核苷酸,编码373 个氨基酸。14 个病毒株中11 个属于2.1d新基因亚型,它们与CSFV 代表株Shimen、HCLV和2.1b 基因亚型毒株的核苷酸同源性分别为82.8 %~84.1 %、81.1 %~82.4 %和92.6 %~97.1 %,氨基酸同源性分别为89.8 %~91.2 %、88.2 %~89.8 %和94.1 %~98.9%(表2),其余3 个病毒株属于2.1b亚型。这些2.1d 亚型新分离株在E2 基因R31、S34、K303和A3314 个氨基酸上具有相同的分子特征(图2)。
3 讨论
CSF 是一种高度接触性、急性传染病,以高发病率、高死亡率、全身各脏器出血、高热稽留为主要特征,曾对我国养猪业造成严重经济损失。自从上个世纪五十年代猪瘟兔化弱毒疫苗在我国的广泛应用,CSF 对我国养猪业的危害已显著降低[7-8],但CSF 在我国并没有根除,并随着时间不断演变,流行株从1.1 亚型为主逐渐演化到2.1 亚型为主[8-10]。近些年尽管分离到了野毒株,然而分离到的病毒株数量少,分子特征不明显,而且可能需要一年甚至几年的时间[11-12]。测序和遗传进化分析是了解CSFV流行的重要工具之一,同时也为CSF 的防控提供了分子流行病学的数据。目前,用于CSFV 分型的基因区域主要有5'NTR、E2、NS5B,应用最广泛的是E2 和NS5B 基因区。基于E2 和NS5B 基因,Lowings 等将1945~1994 年间14 个国家的CSFV 分成了2 个基因型和5 个基因亚型[13]。目前,CSFV 分为3 个基因型和10 个基因亚型,2.1 亚型进一步分为2 个基因亚型(2.1a 和2.1b),随后Postel 等报道了1.4 亚型[4]。近年来,我国主要流行2.1b 亚型,Jiang 等首次报道了我国2.1c 亚型的出现。2014 年我国一些猪场出现疑似CSF 症状,造成不同程度的损失[6],检测证实这些疫情是由CSFV 引起,通过序列比对和遗传进化分析(基于E2 全长和E2 部分序列),结果表明检测到的CSFV 和2014 年的一些病毒株形成一个新2.1d 基因亚型,2.1d 亚型病毒株间具有相同的分子特征[6]。
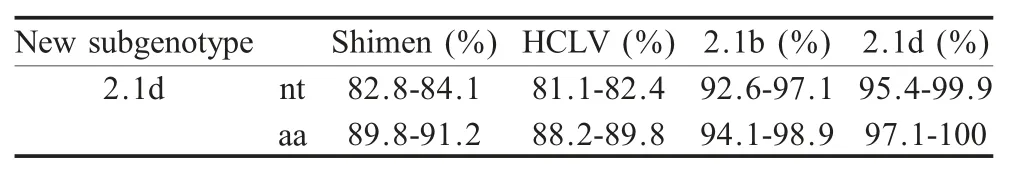
表2 2.1d 新亚型的E2 基因与CSFV 参考株同源性比对结果Table 2 The nucleotides and amino acids of E2 gene between 2.1d new subgenotype and shimen,HCLV,2.1b subgenotype and themselves
为进一步了解2.1d 亚型CSFV 在我国的流行现状,本研究中,我们对2015 年在我国部分地区检测到的CSFV 进行分子流行病学分析,研究结果与我们2014 年CSFV 流行病学研究结果一致,表明2.1d亚型病毒株已成为我国现阶段CSFV 的主要流行株。由于在不到一年内仅我们实验室就在我国5 个省20多个猪场检测到2.1d 亚型毒株,因此该病毒株可能在我国已广泛出现,但2.1d 亚型病毒株在我国的具体流行状况和危害程度还需要进一步验证,并尽快开展有关2.1d 亚型病毒株的致病性及现有疫苗对其保护效果的研究,更有效地控制CSFV 的流行。
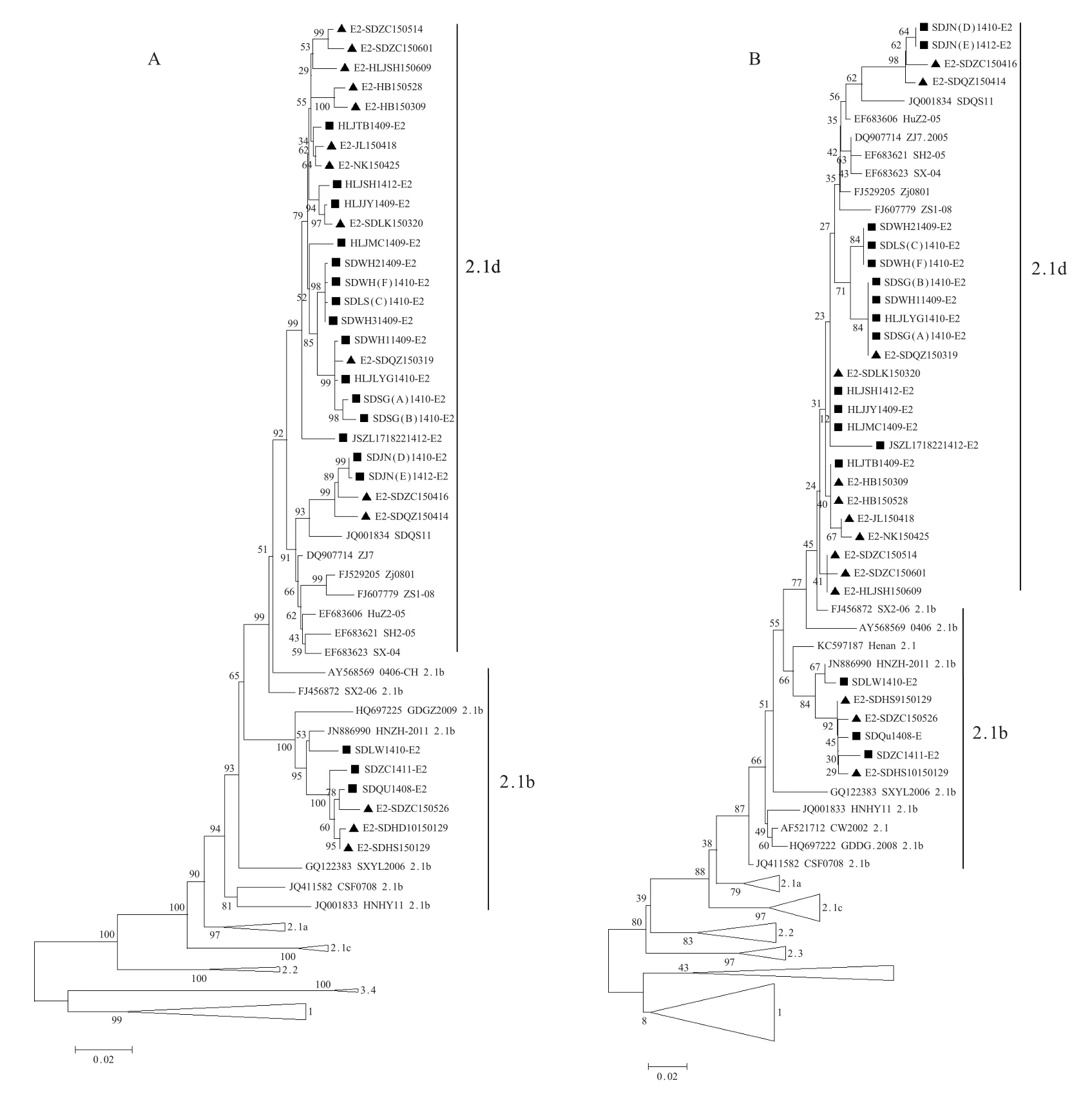
图1 2014~2015 年32 株新分离CSFV 与参考病毒株的遗传进化树Fig.1 Phylogenetic tree based on 32 new isolates of CSFVs and reference strains

图2 32 株新分离毒株和19 株参考毒株的E2 基因的氨基酸比对结果Fig.2 Alignments of 32 new isolates and other 19 reference CSFVs based on E2 amino acid sequence
[1]Paton a D J,McGoldricka A,Greiser-Wilkeb I,et al.Genetic typing of classical swine fever virus[J].Vet Microbiol,2000,73:137-157.
[2]Pan C H,Jong M H,Huang T S,et al.Phylogenetic analysis of classical swine fever virus in Taiwan[J].Arch Virol,2005,150:1101-1119.
[3]Jiang Da-liang,Gong Wen-jie,Li Run-cheng,et al.Phylogenetic analysis using E2 gene of classical swine fever virus reveals a new subgenotype in China[J].Infect Genetics Evol,2013,17:231-238.
[4]Postel A,Schmeiser S,Perera C L,et al.Classical swine fever virus isolates from Cuba form a new subgenotype 1.4[J].Vet Microbiol,2013,161(3-4):334-8.
[5]Edwards S,Fukusho Akio,Lefèvrec P C,et al.Classical swine fever:the globai situation[J].Vet Microbiol,2000,73(2):103-119.
[6]Zhang Hong-liang,Leng Chao-liang,Feng Li-ping,et al.A new subgenotype 2.1d isolates of classical swine fever virus in China,2014[J].Infect Genet Evol,2015,34:94-105.
[7]仇华吉,童光志,沈荣显.猪瘟兔化弱毒疫苗-半个世纪的回顾[J].中国农业科学,2005,8(8):1675-1685.
[8]Luo Yu-zi,Li Su,Sun Yuan,et al.Classical swine fever in China[J].Vet Microbiol,2014,172:1-6.
[9]Tu Chang-chun,Lu Zong-ji,Li Hong-wei,et al.Phylogenetic comparison of classical swine fever virus in China[J].Virus Res,2001,81:29-37.
[10]吕宗吉,涂长春,余兴龙,等.我国猪瘟的流行病学现状分析[J].中国预防兽医学报,2001,23:300-303.
[11]彭志成,龚文杰,吕宗吉,等.广东省猪瘟病毒流行毒株遗传多样性分析[J].中国兽医学报,2014,6,34:894-903.
[12]Luo Ting-rong,Liao Su-huan,Wu Xian-shi,et al.Phylogenetic analysis of the E2 gene of classical swine fever virus from the Guangxi Province of southern China[J].Virus Genes,2011,42:347-354.
[13]Paul L,Georgina I,Jennifer N,et al.Classical swine fever virus diversity and evolution[J].J Gen Virol,1996,77(6):1311-1321.

