高粱SbNAC0584基因克隆与表达分析
祖祎祎,伊巴代提•喀迪尔,孙清鹏,潘金豹,卢敏
北京农学院植物科学技术学院,北京102206
高粱SbNAC0584基因克隆与表达分析
祖祎祎,伊巴代提•喀迪尔,孙清鹏,潘金豹,卢敏*
北京农学院植物科学技术学院,北京102206
摘要:NAC转录因子是植物体特有且在调控植物生长发育、抵抗逆境胁迫等方面具有重要生物功能的转录调控因子。本研究从高粱中克隆得到一个受逆境胁迫诱导表达的NAC家族基因,系统进化分析结果表明该基因与玉米Zma NAC0584亲缘关系最近,故将其命名为SbNAC0584;SbNAC0584基因全长929 bp,编码290个氨基酸,氨基酸序列比对结果表明SbNAC0584蛋白N-末端具有典型的NAC保守结构域,C-末端为序列高度多样性的转录调控结构域。采用实时荧光定量PCR技术分析SbNAC0584基因对不同非生物逆境胁迫的应答模式,结果表明SbNAC0584受NaCl、脱水、PEG胁迫和植物激素ABA诱导表达上调,推测SbNAC0584在高粱非生物胁迫应答过程中发挥重要作用;组织特异性表达分析结果表明SbNAC0584在高粱旗叶和根中表达量相对较高。本研究为深入研究SbNAC0584的抗逆生物学功能奠定基础。
关键词:高粱;转录因子;克隆;非生物胁迫;表达
植物受到干旱、盐碱和冷等非生物逆境胁迫损伤,其正常的生长发育将受到影响造成粮食减产。植物在长期进化过程中形成了有效的生理生化和分子作用机制来抵抗外界不良环境[1]。植物抗逆信号转导途径是涉及多基因共同作用的复杂调控体系,其中转录调控因子通过调控胁迫相关基因的表达来参与应答逆境胁迫。
NAC转录因子是植物体内广泛存在且特有的一类转录调控因子[2]。NAC转录因子编码蛋白的N-末端具有序列高度保守、参与DNA结合的NAC结构域。蛋白C-末端为序列多样性的转录激活或转录抑制结构域[3]。基于氨基酸序列一致性又将NAC结构域细分为5个相对保守的子结构域。NAC蛋白在调控植物生长发育、抵抗生物和非生物逆境胁迫等过程中发挥重要作用[4]。近年来,随着测序技术的发展和植物基因组测序的完成,使得在植物全基因组水平上挖掘NAC转录因子家族成员成为可能[5-7]。水稻、拟南芥等模式植物中挖掘得到诸多受干旱、盐碱、冷等非生物逆境胁迫和机械损伤、病原等生物逆境胁迫诱导表达显著变化的NAC转录因子编码基因。通过病毒诱导基因沉默(VIGS)技术研究发现,小麦TaNAC1作为负调控因子在抵抗小麦条锈病等方面发挥重要作用[8]。水稻SNAC1基因通过调控下游靶基因的表达提高了转基因水稻的抗逆性,田间测产实验结果表明干旱胁迫没有造成SNAC1过表达转基因水稻的减产[9]。NAC转录因子在依赖于ABA和不依赖于ABA的抗逆信号转导途径中均发挥重要作用,前人研究结果表明在依赖于ABA的信号途径中存在OsNAC5、OsNAC052和SNAC2(OsNAC6)等NAC家族成员均在提高转基因植株抗逆性方面具有重要的生物学功能[10-12]。其中水稻OsNAC5通过与OsNAC6和SNAC1蛋白相互作用从而发挥其转录调控作用,过表达OsNAC5能够显著提高转基因水稻的抗旱和耐盐性。近年来,研究发现包括鹰嘴豆NAC家族成员CarNAC3、CarNAC6[13,14],小麦TaNAC67、TaNAC2在内等诸多NAC基因均参与植物非生物逆境胁迫应答[15,16],但多数NAC基因的抗逆生物学功能特别是在重要耐旱作物高粱中参与植物逆境胁迫应答的相关研究较少。本研究从高粱耐旱自交系中克隆得到一个逆境胁迫应答基因SbNAC0584,对其进行序列比对和系统进化分析,并进一步分析其在不同非生物逆境胁迫及ABA处理下的诱导表达模式,明确其组织表达特异性,本研究为深入探明SbNAC0584的抗逆生物学功能奠定基础。
1 材料与方法
1.1植物材料与处理
选用的实验材料为高粱耐旱自交系“XGL-1”,是由原中国农业科学院品资所选育,种子由本实验室保存。选取饱满的高粱自交系“XGL-1”种子,分别用75%乙醇和0.5%次氯酸钠对种子进行消毒,再用无菌水反复冲洗4~5遍,将种子种植于装有蛭石和营养土混合花盆中(4~5株/盆),室温(28℃)放置培养间(16 h光照/8 h黑暗)进行培养,待高粱长至三叶期时,配制Hoagland溶液,分别对高粱幼苗进行20%PEG、200 mM NaCl、脱水和100µM ABA (Sigma)处理,在材料处理同时对植株根部进行通气,防止根系无氧呼吸,并在各处理0、1、3、6、12和24 h分别取植株地上和地下部分立即放置于液氮冷冻保存。
同时将高粱耐旱自交系“XGL-1”种植与北京农学院实验地,分别取高粱植株的根系、气生根、叶片、穗、茎、旗叶6个不同组织进行基因的组织特异性表达分析,取材后立即放置液氮冷冻,-80℃保存。
1.2总RNA提取与第一链cDNA的合成
将低温保存的样品取出,在液氮中迅速研磨,直至样品成粉末状。主要参照TRIzol(Invitrogen)试剂盒提供的实验方法提取高粱各样品总RNA。电泳鉴定RNA的质量,并用NanoDrop2000紫外分光光度计测定所提取RNA的浓度,放置于-80℃长期保存。高粱第一链cDNA的合成:用RNase free DNase I(Fermentas)消化样品中的基因组DNA,并参照Thermo Scientific Revert Aid First Strand cDNA Synthesis Kit (#K1622)试剂盒说明书反转录合成高粱第一链cDNA,将原液放置于-20℃长期保存。
1.3SbNAC0584基因全长cDNA的克隆与序列分析
高粱基因组数据库(http://genome.jgi-psf.org/)查询高粱参考基因组序列,设计特异性引物FP (5'–AACATAGCGAGGGAGCCA - 3')和RP(5' - CAGTTGAGCTGAATCCGTCC - 3'),以各处理混合的高粱cDNA为模板,PCR扩增SbNAC0584基因全长cDNA。扩增体系如下:ddH2O 9.5 μL,2×Trans Taq High Fidelity(HiFi)PCR SuperMix 12.5 μL,Forward primer 1 μL,Reverse primer 1 μL,cDNA 1 μL。PCR扩增程序如下:95℃预变性5 min;95℃变性40 s;56℃退火30 s,72℃延伸2 min,32个循环后72℃延伸40 min。经1 %的琼脂糖凝胶电泳检测后切胶回收,连接pMD18–T克隆载体(TaKaRa,大连),转化大肠杆菌TOP10,挑取单菌落进行菌裂PCR检测,选择PCR检测为阳性的克隆进行测序。
利用DNAMAN(Version 5.0)和ClustalX(Version 1.83)软件进行基因序列比对。应用Contig Express(version 6.0; Invitrogen, Carlsbad, CA)软件进行测序序列的拼接比对,采用MEGA4(version 4)软件neighbor-joining(NJ)计算方法对SbNAC0584编码蛋白及已报道的NAC家族成员进行氨基酸序列比对和系统进化分析(brootstrap=1000)。
1.4实时荧光定量PCR分析
在基因3’末端非保守区设计针对SbNAC0584基因的特异性荧光定量(quantitative real-time PC R, qRT-PCR ) PCR引物:(FP:5’- ATGGAATCGGAGGCGTCG-3’,RP:5’- ATGTTGCCAAAGA AGTCGGC-3’),扩增片段长度251 bp。以高粱18S rRNA(FP:5’-GGCTCGAAGACGATCAGAT ACC-3’,RP:5’-TCGGCATCGTTTATGGTT-3’)为内参基因对样品cDNA模板量进行校正,采用S YBR®Premix EX TaqTM染料和ABI7300 (Applied Biosystems, Foster City,美国)荧光定量PCR仪进行定量PCR分析,实时荧光定量PCR反应程序:94℃1 min , 94℃15 s, 56℃15 s, 72℃30 s,扩增40个循环,通过采用2-ΔΔCt法[17]分析基因的表达水平,胁迫诱导表达分析以0 h的相对表达量为1,组织表达特异性以中茎的表达量为参照(相对表达倍数为1),实验进行三次生物重复,取平均值,计算标准差。表达倍数超过2倍认定为上调表达。
2 结果与分析
2.1SbNAC0584基因全长cDNA的克隆
如图1所示,根据高粱参考序列设计引物,以高粱耐旱自交系“XGL-1”为材料,RT-PCR扩增获得SbNAC0584基因全长cDNA(GenBank登录号为KR809886),测序结果表明SbNAC0584基因全长929 bp,开放阅读框(ORF)全长873 bp,共编码290个氨基酸。
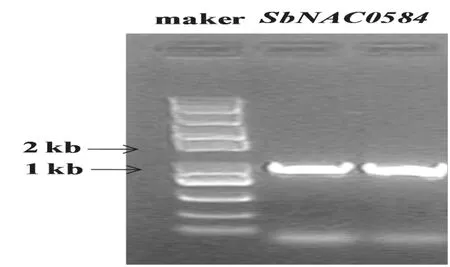
图1 SbNAC0584全长cDNA扩增Fig.1 PCR amplification of the full-length cDNA of SbNAC0584
2.2氨基酸序列比对与系统进化分析
将拟南芥、小麦、大豆、水稻等植物中报道的已知功能的NAC转录因子家族基因与高粱SbNAC0584蛋白序列进行多序列比对,结果表明SbNAC0584与包括拟南芥ATAF1、ATAF2、CUC1、NAM、水稻OsNAC052、大豆GmNAC20和小麦TaNAC69在内的诸多参与非生物逆境胁迫应答的NAC家族蛋白序列特征具有高度相似性。如图2所示,SbNAC0584蛋白的N-末端具有典型的且序列高度保守的NAC结构(NAC-domain),基于氨基酸序列一致性又可将该结构域分为5个子结构域(A-E),其中B、D子结构域主要参与DNA特异结合,而C-末端则是序列高度多样性的转录调控结构域,发挥其转录激活或转录抑制功能。

图2 SbNAC0584与已知功能NAC家族的蛋白多序列比对Fig.2 Analysis on the sequence SbNAC0584 and some reported NAC proteins
为了进一步研究高粱SbNAC0584蛋白与其他NAC转录因子之间的亲缘关系,将高粱SbNAC0584蛋白与已报道的拟南芥ATAF1、CUC3、GRAB1、玉米ZmaNAC0584、ZmSNAC1、水稻OsNAC052、大豆GmNAC2、GmNAC6和小麦TaNAC67、TaNAC4、TaNAC4a、TaNAC1、TaNAC8a等进行系统进化分析,结果表明高粱SbNAC0584蛋白与玉米ZmaNAC0584、玉米Zma054594的氨基酸序列一致性分别为88.67 %和87.79 %(图3),由此判断高粱SbNAC0584是玉米ZmaNAC0584的同源基因,两者同为逆境胁迫应答相关的NAC家族成员。
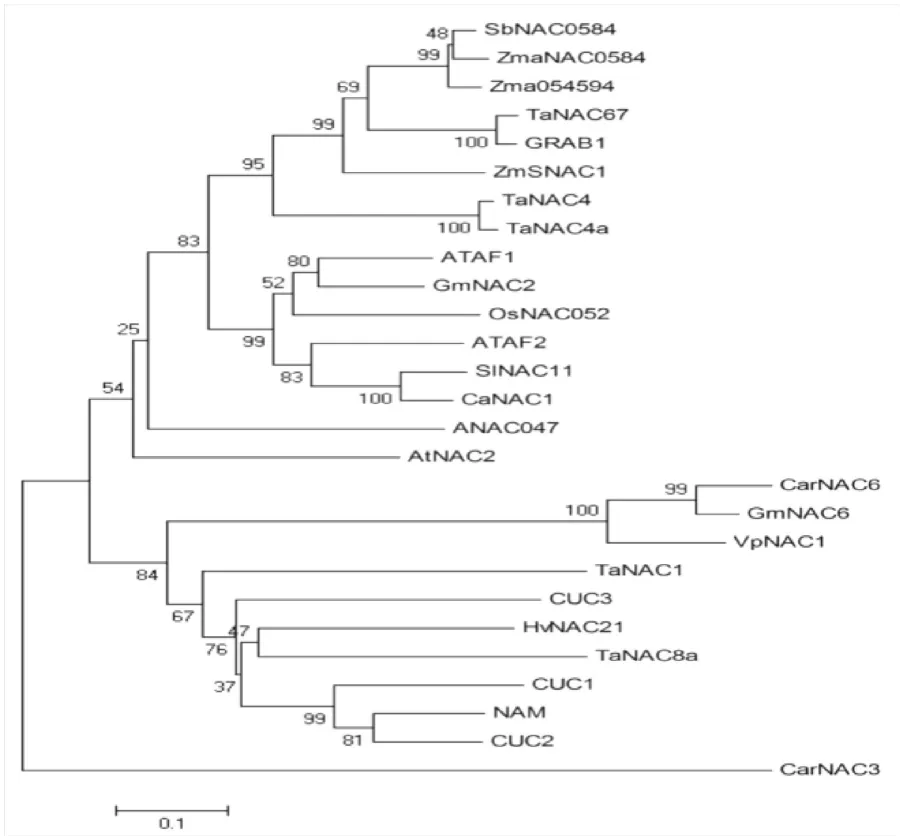
图3 SbNAC0584与已知功能的胁迫应答相关的NAC家族成员进化分析Fig.3 Phylogenetic relationship between SbNAC0584 and stress-responsive NAC proteins
2.3SbNAC0584基因胁迫诱导表达分析
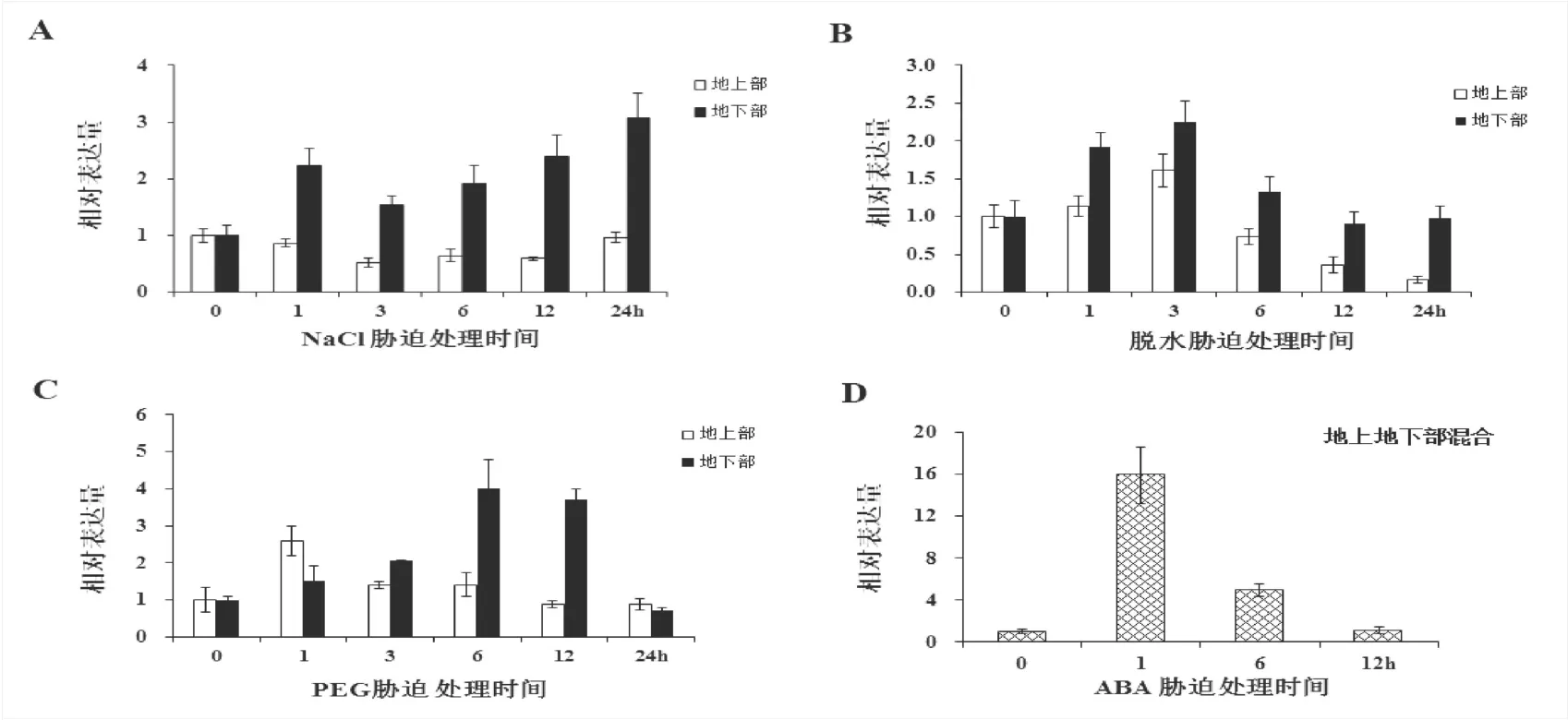
图4 200 mM NaCl、脱水、20 % PEG和100µM植物激素ABA胁迫下(A-D)SbNAC0584的相对表达量Fig.4ExpressionofSbNAC0584inSorghum,under200mMNaCl,dehydration,20%PEG,ABAstressconditions(A-D),respectively
如图3所示,系统进化分析结果表明SbNAC0584与诸多已知功能的NAC家族成员的亲缘关系较近,为了进一步研究SbNAC0584基因在非生物逆境胁迫和ABA胁迫下的诱导表达模式,分别对三叶期高粱自交系进行NaCl、脱水、PEG和植物激素ABA胁迫处理,分析SbNAC0584在不同处理下的表达水平。结果表明在NaCl胁迫下,SbNAC0584基因在高粱地下部诱导表达显著上调,至处理24 h时表达量达到最大(约3倍),而地上部表达量随胁迫处理时间变化没有显著差异。SbNAC0584 受20 % PEG胁迫处理诱导表达上调,在地下部处理3 h时上调表达2.05倍,处理6 h时表达量达到最大(约4倍),处理12 h时表达量开始降低(约3.7倍)。在脱水胁迫处理下,SbNAC0584在高粱地下部呈快速诱导表达上调模式,在处理3 h时达到最大(2.3倍)。同时,检测到SbNAC0584在转录水平上应答外源植物激素ABA胁迫,最高上调表达15倍。
2.4SbNAC0584基因组织特异性表达分析
如图5所示,qRT-PCR分析结果表明,SbNAC0584在检测的所有的组织(根系、气生根、叶片、穗、茎、旗叶)中均有表达,但在高粱气生根和旗叶中的表达量较其它组织相对较高,其次是在根中,在高粱茎和穗中的表达水平相对较低。
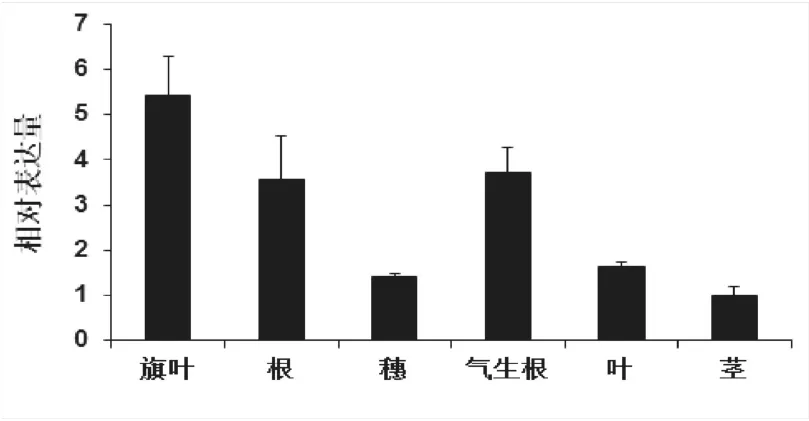
图5 SbNAC0584的组织特异性表达分析Fig.5 Tissue-specific expression of SbNAC0584 in sorghum
3 讨论
近年来,NAC基因作为植物体内最大的转录因子家族之一,因其在调控植物生长发育和抵抗非生物和生物逆境胁迫等方面发挥的重要功能受到研究者广泛关注[18]。目前,通过分子生物学手段挖掘到包括拟南芥、水稻和烟草等模式植物在内的诸多具有重要生物学功能的NAC转录因子家族成员,它们通过蛋白互作或调控下游靶基因的表达来调节植物生长发育进程和适应外界不良环境。然而在重要禾谷类作物高粱中NAC基因与抗逆相关的报道较少。
本研究从高粱耐旱自交系中克隆得到一个NAC家族基因,SbNAC0584基因cDNA全长929 bp,ORF全长873 bp,编码290个氨基酸。与拟南芥、水稻、小麦和大豆等作物中的NAC家族蛋白氨基酸序列特征相一致,SbNAC0584蛋白的N-末端具有高度保守的NAC-domain(约160个氨基酸),蛋白C-末端为序列多样性的转录调控结构域。基因结构的保守性、进化的相似性往往决定了基因功能的相关性。前人研究结果表明,过表达小麦胁迫诱导表达基因TaNAC67能够显著提高转基因小麦的抗旱性[15],玉米ZmaNAC0584和Zma054594受多种非生物逆境胁迫和植物激素ABA诱导表达显著上调[18],而高粱SbNAC0584作为与三者亲缘关系较近的基因,其受到脱水、NaCl、PEG胁迫诱导表达显著上调(图4),表明SbNAC0584很可能在高粱抵抗外界不良环境胁迫、提高植株抗逆性方法发挥重要作用。另一方面,本研究发现在植物激素ABA胁迫处理下,SbNAC0584基因表达显著上调,ABA作为重要的信号分子参与调控气孔开闭等植物的抗逆过程,表明高粱SbNAC0584蛋白可能依赖于ABA的抗逆信号转导途径中发挥其转录调控作用。
NAC转录因子基因在植物生长发育不同阶段不同组织中的表达量各不相同,例如在干旱胁迫处理下水稻SNAC1在植株根中和有保卫细胞组成的气孔中诱导表达,而在叶舌、雄穗和花丝组织的表达量没有显著变化[9]。拟南芥NAC1基因在植物根中表达水平最高,在茎和叶中表达水平相对较低。NAC家族基因的组织表达特异性表明其在植物体不同组织和器官中潜在的作用机制存在差异。
SbNAC0584蛋白在高粱气生根和旗叶中的相对表达量较高,玉米ZmaNAC0584和Zma054594的组织表达分析结果同样证明这两个基因在植物根中的表达量相对较高,而植物根系是其响应并适应外界不良环境的关键部位,其生理特性和渗透调节能力与作物的抗逆性之间有紧密关联。
本研究克隆高粱SbNAC0584基因全长cDNA,对其进行序列和系统进化分析,解析其在非生物逆境胁迫和植物激素ABA处理下的诱导表达模式和组织表达特异性,为进一步深入研究SbNAC0584抗逆相关生物学功能和潜在的分子作用机制奠定基础。
4 结论
克隆高粱NAC转录因子家族基因SbNAC0584,该基因与诸多逆境胁迫应答相关的NAC基因亲缘关系较近,在气生根和旗叶中表达量相对较高,且受非生物逆境胁迫NaCl、脱水、PEG和植物激素ABA诱导表达上调,表明SbNAC0584可能在依赖于ABA的信号途径中参与应答非生物逆境胁迫。
参考文献
[1] Qin F, Shinozaki K, Yamaguchi-Shinozaki K. Achievements and challenges in understanding plant abiotic stress responses and tolerance [J]. Plant Cell Physiol, 2011,52(9):1569–1582
[2] Yamaguchi-Shinozaki K, Shinozaki K. Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses [J]. Annu Rev Plant Biol, 2006,57:781–803
[3] Olsen A N, Ernst H A, Leggio L L, et al. NAC transcription factors: structurally distinct, functionally diverse [J]. Trends Plant Sci, 2005,10(2):79–87
[4] Nakashima K, Takasaki H, Mizoi J, et al. NAC transcription factors in plant abiotic stress responses [J]. BBA Gene Regul Mech, 2012,1819(2):97–103
[5] Nuruzzaman M, Manimekalai R, Sharoni A M, et al. Genome-wide analysis of NAC transcription factor family in rice [J]. Gene, 2010,465(1–2):30–44
[6] Su H Y, Zhang S Z, Yin Y L, et al. Genome-wide analysis of NAM-ATAF1, 2-CUC2 transcription factor family in Solanum lycopersicum [J]. Plant Bioche Biot, 2015,24(2):176–183
[7] Puranik S, Sahu P P, Mandal S N, et al. Comprehensive genome-wide survey, genomic constitution and expression profiling of the NAC transcription factor family in foxtail millet (Setaria italica L.) [J]. Plos One, 2013,8(5):e64594
[8] Wang F T, Lin R M, Feng J, et al. TaNAC1 acts as a negative regulator of stripe rust resistance in wheat, enhances susceptibility to Pseudomonas syringae, and promotes lateral root development in transgenic Arabidopsis thaliana [J]. Front Plant Sci, 2015,6:108
[9] Hu H H, Dai M Q, Yao J L, et al. Overexpressing a NAM,ATAF, and CUC (NAC) transcription factor enhances drought resistance and salt tolerance in rice [J]. Proc Natl Acad Sci USA, 2006,103(35):12987–12992
[10] Gao F, Xiong AS, Peng R H , et al. OsNAC52, a rice NAC transcription factor, potentially responds to ABAand confers drought tolerance in transgenic plants [J]. Plant Cell Tiss Organ Cult, 2010,100(3):255–262
[11] Hu H H, You J, Fang Y J, et al. Characterization of transcription factor gene SNAC2 conferring cold and salt tolerance in rice [J]. Plant Mol Biol, 2008,67(1–2):169–181
[12] Song S Y, Chen Y, Chen J, et al. Physiological mechanisms underlying OsNAC5-dependent tolerance of rice plants to abiotic stress [J]. Planta, 2011,234(2):331–345
[13] Movahedi A, Zhang J, Gao P, et al. Expression of the chickpea CarNAC3 gene enhances salinityand drought tolerance in transgenic poplars [J]. Plant Cell, 2015,120(1):141–154
[14] Movahedi A, Zhang J, Yin T, et al. Functional Analysis of Two Orthologous NAC Genes, CarNAC3, and CarNAC6 from Cicerarietinum,InvolvedinAbioticStressesinPoplar[J].PlantMolBiolRep,2015:1–13
[15] Mao X G, Chen S S, Li A, et al. Novel NAC transcription factor TaNAC67 confers enhanced multi-abiotic stress tolerances in Arabidopsis [J].Plos One, 2014,9(1):e84359
[16] Mao X G, Zhang H Y, Qian X Y, et al. TaNAC2, a NAC-type wheat transcription factor conferring enhanced multiple abiotic stress tolerances in Arabidopsis [J]. Exp Bot, 2012,63(8):2933–2946
[17] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCtmethod [J]. Methods, 2001,25(4):402–408
[18] Lu M, Sun Q P, Zhang D F, et al. Identification of 7 stress-related NAC transcription factor membersin maize (Zea mays L.) andcharacterization of theexpressionpatternof thesegenes[J]. Biochem Bioph Res Co, 2015,462(2):144–150
The Analysis on the Cloning and Expression of SbNAC0584 Gene in Sorghum bicolor L.
ZU Yi-yi, Kadier•Yibadaiti, SUN Qing-peng, PAN Jin-bao, LU Min*
College of Plant Science and Technology, Beijing University of Agriculture, Beijing 102206, China
Abstract:NAC proteins are plant-specific transcription factors that play important role in abiotic stress adaptations and tolerance, as well as in the plant processes in development. In this study, by searching against the plant genome databases, a stress induced NAC member was isolated from sorghum. It was designed as SbNAC0584 for its close relationship with maize ZmaNAC0584. The full-length cDNA of SbNAC0584 was 929 bp that encoded 290 amino acids. Sequence analysis indicated that a NAC conserved domain was localized in the N-terminal region of the SbNAC0584 protein. A highly diverse sequence was also observed in the C terminus. Transcription analyses were carried out to determine the effects of various abiotic stresses and of the phytohormone ABA on the expression of the SbNAC0584. SbNAC0584 was significantly induced by NaCl, dehydration, PEG and ABA treatments. We speculated that it might be involved in the response to abiotic stresses. The spatial expression pattern of SbNAC0584 revealed that higher transcripts existed in the root and flag leaf of sorghum plants than those in other tissues. This information provides a background for the further functional study of SbNAC0584.
Keywords:Sorghum bicolor L.; transcription factor; cloning; abiotic stress; expression
*通讯作者:Author for correspondence. E-mail:lumin.sdau@gmail.com
作者简介:祖祎祎、伊巴代提•喀迪尔,女,硕士研究生,从事作物遗传育种研究. E-mail:13691112388@163.com
基金项目:北京市教委科研计划面上项目(KM201510020003)
收稿日期:2015-05-23修回日期: 2015-06-23
中图法分类号:Q781
文献标识码:A
文章编号:1000-2324(2015)04-0497-06

