三峡库区5个鲢群体遗传变异的微卫星分析
庞美霞俞小牧童金苟
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
三峡库区5个鲢群体遗传变异的微卫星分析
庞美霞1,2俞小牧1童金苟1
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
研究利用 10个高度多态的微卫星标记对三峡水库秭归、巫山、云阳、忠县、木洞等 5个库区鲢(Hypophthalmichthys molitrix)的野生群体进行了遗传多样性分析。检测到161个等位基因, 群体共有等位基因84个, 每个微卫星位点的等位基因数7—29不等。平均观测杂合度Ho为0.784—0.846, 平均期望杂合度He为0.828—0.847, 平均多态信息含量PIC为0.797—0.817。Fst值为–0.001—0.009, 表明5个鲢群体间没有遗传分化。Hardy-Weinberg平衡检验表明巫山、云阳、木洞群体在一些位点上偏离遗传平衡。Bottleneck分析显示长江三峡库区江段的鲢群体可能在历史上经历了遗传瓶颈。5个群体间的遗传相似系数为0.891—0.950, 遗传距离为 0.050—0.115, 根据 Nei’s遗传距离所绘制的聚类图, 表明鲢群体间的遗传距离与其地理距离基本一致。贝叶斯分析结果也证实三峡库区 5个鲢群体可视为一个类群。尽管没有检测到遗传分化, 数据清晰地表明三峡库区的鲢群体仍有很高的遗传多样性, 研究结果为三峡地区和长江上游的鲢种质资源保护和种群评估提供了参考。
鲢; 微卫星; 野生群体; 遗传多样性; 三峡水库
鲢(Hypophthalmichthys molitrix)是我国“四大家鱼”之一, 广泛分布于我国长江、黑龙江、珠江等各大水系, 是我国重要的经济鱼类。长江流域是我国鲢天然资源的主要产地, 其养殖性能明显优于珠江、黑龙江等其他水系[1]。然而由于长期的过度捕捞、水体污染、水利建设等一系列人为因素导致近年来长江水系自然环境剧烈变化[2], 从而引起包括鲢在内的“四大家鱼”资源量的严重衰退[3]。群体遗传分析是合理评估鱼类种群遗传多样性和种群分化的有效手段。过去已有一些针对长江流域鲢野生群体遗传多样性分析的报道[4—8], 但主要集中在长江中下游地区, 有关长江中上游地区的相关报道少见[6]。长江三峡水库已经建成, 针对这一河流型水库中鲢野生种群的遗传结构分析方面的资料还非常缺乏,这种状况不利于鱼类资源的评估和合理利用。
微卫星标记是一种以1—6 bp的核苷酸序列为核心序列的简单重复序列[9], 由于其具有共显性遗传、数量多且分布广泛均匀、多态信息含量高及便于检测等诸多优点, 已被广泛应用于群体遗传分析[10,11]。本研究采用本实验室开发的4—5核苷酸重复的10个多态性鲢微卫星标记对来自三峡水库从库首到库尾的5个库区(秭归、巫山、云阳、忠县、木洞)的鲢样本进行分析, 旨在掌握三峡库区以及长江上游鲢野生群体的遗传多样性和遗传结构现状, 为长江流域“四大家鱼”种质遗传资源的保护和利用提供理论依据。
1 材料与方法
1.1 试验材料及DNA提取
本实验所分析的 5个野生鲢群体样本采集于2012—2013年, 采样点位于湖北省秭归(ZG)、重庆市巫山(WS)、云阳(YY)、忠县(ZX)和木洞镇(MD),样本总数227尾(表1、图1)。剪取每尾鱼的少量鳍条用无水酒精保存, 基因组 DNA采用经典的酚-氯仿法[12]提取。
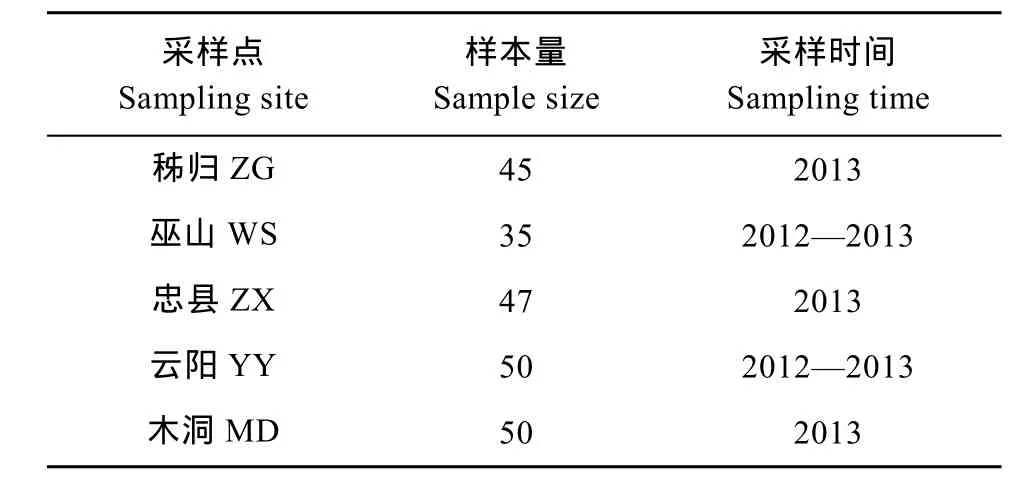
表1 三峡库区5个鲢群体样本的基本信息Tab. 1 The basic information of five silver carp populations in the Three Gorges Reservoir
1.2 标记选取及PCR扩增
在本研究中使用的标记均来自于本实验室开发的鲢微卫星标记。为了能够清晰地分型便于数据统计, 本文所选取的 10个鲢微卫星标记[13]均是 4—5碱基重复的高度多态性位点(表2)。根据Li-Cor 4300 DNA 分析系统的检测要求, 在合成每一对微卫星引物时, 将一段19碱基( 5′-CACGACGTTGTAAAA CGAC-3′)的接头序列添加到每一个微卫星位点的正向引物序列 5′端[14]。在PCR进行扩增时, 带有接头序列的微卫星上游引物既可与目的片段结合, 又可与 5′端带有荧光接头的 IR-M13F引物[M13 Forward(-29) IRDye 700 Primer 或M13 Forward (-29) IRDye 800 Primer] (LI-COR Biosciences, Lincoln, NE, USA) 结合, 从而把IR-M13F 带入到扩增反应中, 使得扩增的目的片段末端带有荧光标记从而通过 4300 DNA 分析仪检测并成像[15]。 本文所用的微卫星引物和荧光标记引物均由上海生工生物工程有限公司合成。
PCR反应总体积为12.5 µL, 反应混合物包含以下组分: 30—50 ng模板DNA, 0.4 U Taq DNA聚合酶, 1.25 µL 10×PCR buffer, 0.4 µL dNTP (2.5 mmol/L), 0.4 µL正反向混合引物(各 2.5 µmol/L), 0.3 µL IR-M13F引物, 10 µL灭菌去离子水。PCR扩增条件如下: 94℃预变性5min; 扩增35个循环, 每个循环94°C变性35s, 48—60℃退火35s(退火温度见表2), 72℃延伸40s; 72℃终延伸10min。
1.3 微卫星分型及数据统计
在 PCR扩增结束后, 在含有扩增产物的薄壁管中加入 7 µL变性剂( 98%去离子甲酰胺, pH 8.0, 10 mmol/L EDTA, 0.25%溴酚蓝), 95℃变性10min后立即将置于冰水中冷却。变性产物在 Li-Cor 4300 DNA分析仪中进行电泳检测并自动成像。MS-TOOLS软件用于计算观测杂合度(Ho)、期望杂合度(He)、等位基因数(Na)等多样性参数, 以及计算多态信息含量(PIC)。PopGene 32软件计算各个群体间的Nei’s遗传距离[16]和相似系数。并根据Nei’s遗传距离用 MEGA 4软件[17]对 5个鲢群体进行聚类分析。Arlequin 3.1软件[18]计算各群体间的分化系数 Fst, 同时用于进行各群体的哈迪-温伯格平衡(Hardy-Weinberg equilibrium)检验、连锁不平衡(Linkage disequilibrium)检验。进行多重比较时, 概率的显著性水平需要进行 Bonferroni校正[19]。利用分子变异分析(AMOVA)方法[20]计算群体遗传结构。根据各位点等位基因频率, 使用BOTTLENECK3.4软件[21]进行瓶颈效应分析。基于无限等位基因模型(Infinite allele model, IAM), 采用符号检验(Sign test)分析杂合度过剩情况, 依此推测群体数量动态变化。利用Structure 2.2[22]软件中的贝叶斯方法混合模型来构建群体遗传结构图。

图1 三峡水库鲢群体采样点分布示意图Fig. 1 Distribution of sampling sites for silver carps in the Three Gorges Reservoir
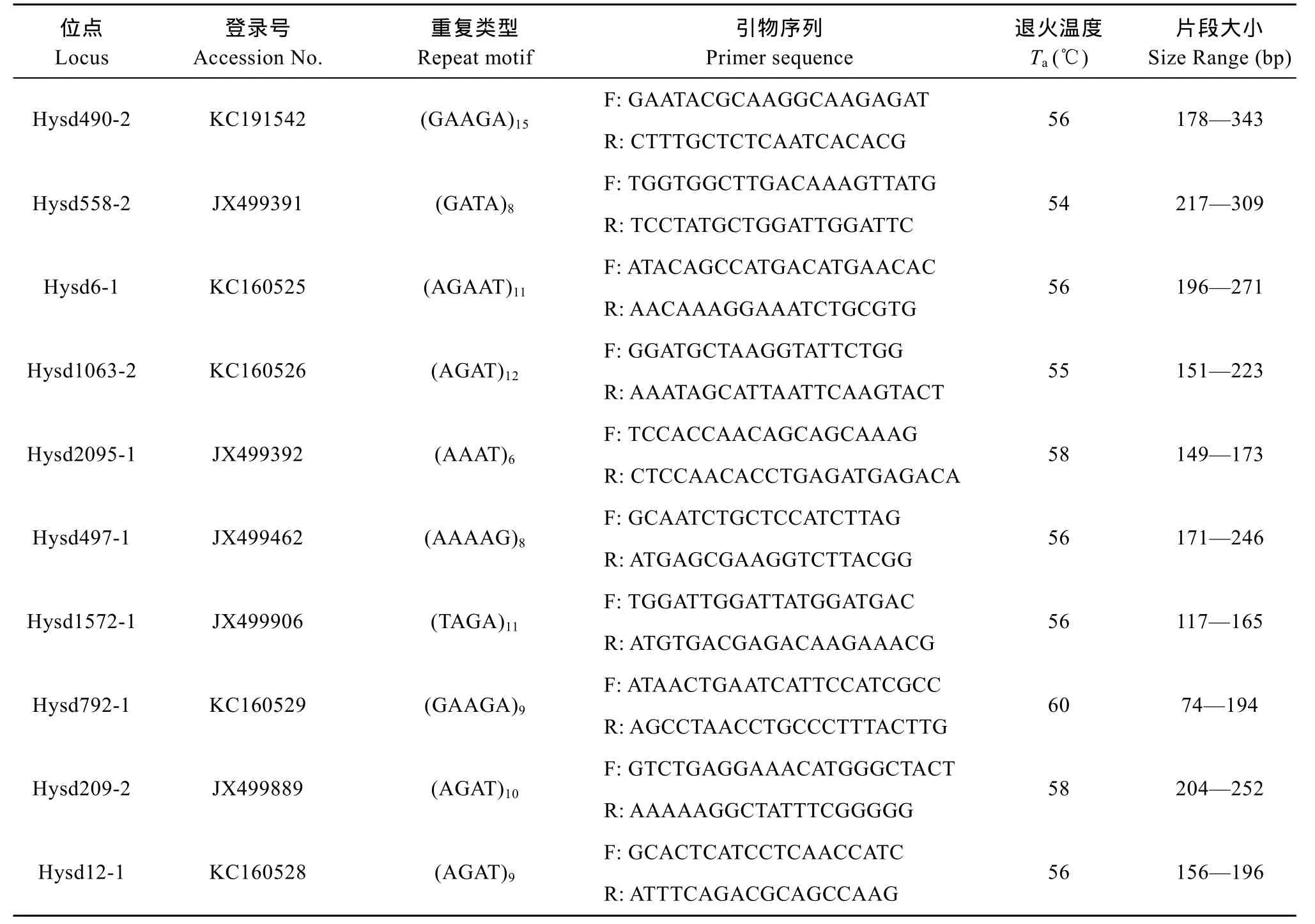
表2 本研究所用鲢微卫星标记的基本信息Tab. 2 The microsatellite markers of silver carps used in this study
2 结果
2.1 鲢各群体的遗传多样性特征及群体动态分析
5个鲢群体各位点的等位基因数(Na)、期望杂合度(He)、观测杂合度(Ho)以及多态信息含量(PIC)统计结果见表3。在10个微卫星位点上共检测到161个等位基因, 其中ZG、WS、YY、ZX、MD群体分别测出127、107、122、135和123个等位基因, 5个鲢群体共有等位基因84个, 并且ZG、WS、YY、ZX、MD群体分别有4、3、5、5和2个特殊等位基因。每个微卫星位点的等位基因数7—29不等。ZX群体的平均Na最高(13.5), 巫山群体最低(10.7)。平均 Ho、He和 PIC分别为 0.784—0.846、0.828—0.847和0.797—0.817, 其中WS群体Ho最低, ZX群体Ho最高; YY群体He和PIC在所有群体中均最低,而ZG群体He和PIC在所有群体中均最高。
对各个鲢群体进行哈迪-温伯格平衡检验并用Bonferroni校正后发现, WS群体在 Hysd792-1、Hysd490-2、Hysd2095-1、Hysd209-2、Hysd497-1 位点, YY群体在 Hysd2095-1位点, MD群体在Hysd490-2、Hysd1063-2 位点, 在 0.05置信水平上偏离哈迪-温伯格平衡。而连锁不平衡分析结果显示,所有10个微卫星标记在5个鲢群体均不存在连锁不平衡。遗传瓶颈效应分析结果(表3)显示, 在IAM模型下, 除 ZX群体外其他群体均表现出杂合过度(P<0.05), 表明这些群体近期可能经历了瓶颈效应。
2.2 群体间的遗传分化

5个鲢群体的遗传分化分析结果表明, 群体间的Fst值(表4)为–0.001—0.009, 群体两两间Fst差异均不显著(P>0.05), 表明他们没有遗传分化。AMOVA分析结果显示群体间的遗传变异仅占0.47% , 而群体内的遗传变异占99.53% 。对群体内的各个体进行混合模型分析结果显示, 当K值(理论群)取2时, 5个鲢群体遗传结构高度一致; 当K值取3—5时, WS群体少部分个体遗传结构相对独立, 5个鲢群体遗传结构基本一致。鲢各个群体间的遗传距离和相似系数如表 5所示。Nei氏遗传距离和相似系数分别为 0.050—0.115、0.891—0.950, 以 ZX群体和MD群体之间的遗传距离最小, 而MD群体与WS群体之间的遗传距离最大。
依据计算出来的Nei氏遗传距离(表5), 5个野生鲢群体的聚类分析结果(图2)显示, 邻接树中共有两大分枝, 其中ZG和WS聚为一枝, 其余3个群体聚为另一大分枝。这个聚类结果基本与群体间的地理距离关系一致。
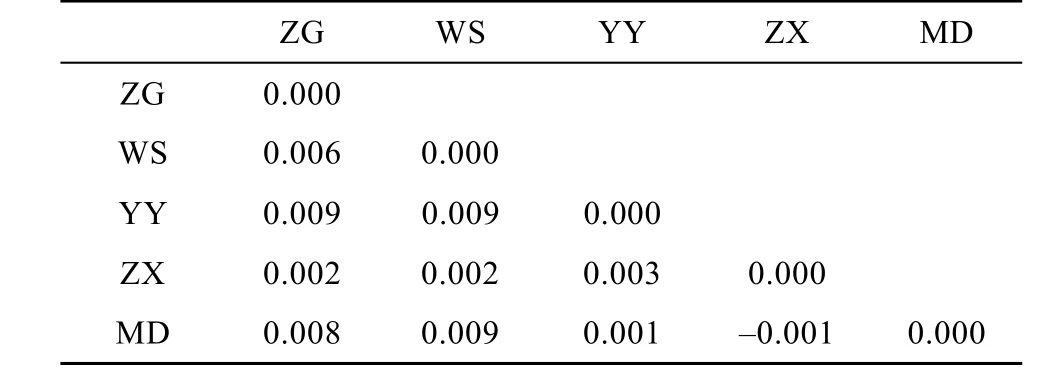
表4 三峡库区5个鲢群体两两间Fst值Tab. 4 Pairwise Fstvalues of five silver carp populations in the Three Gorges Reservoir
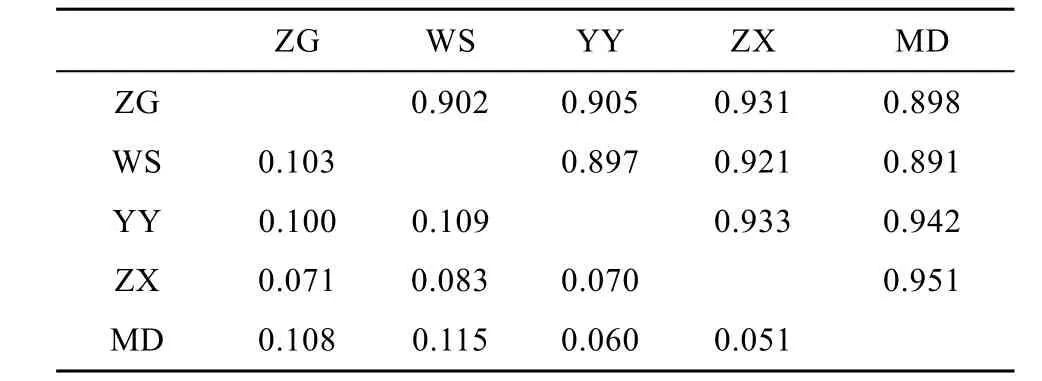
表5 五个鲢群体两两间Nei遗传一致性(对角线上方)和遗传距离(对角线下方)Tab. 5 Nei’s genetic identity (above diagonal) and genetic distance (below diagonal) among five silver carp populations
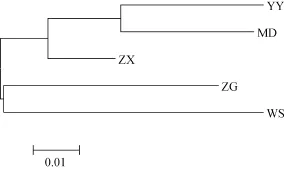
图2 依据Nei氏遗传距离构建的三峡库区鲢5个鲢群体间的邻接树Fig. 2 Neighbour-Joining tree of five silver carp populations in the Three Gorges Reservoir of the Yangtze River based on Nei’s genetic distance
3 讨论
3.1 微卫星标记的适用性
微卫星标记具有共显性遗传、数量多且在基因组中分布广泛、多态信息含量高及便于检测等诸多特点, 是物种种群遗传分析的最常用分子遗传标记。微卫星标记的多态性来源于微卫星核心重复序列重复次数的变化[23], 与其他诸如RAPD、AFLP、SNP等标记相比, 微卫星标记具有更高的分辨率。本研究采用的10个鲢微卫星标记, 均是4—5碱基重复序列。有研究表明, 相比于二核苷酸重复的微卫星标记, 多核苷酸重复的微卫星标记的扩增稳定性更高, 能忠实地反映DNA序列遗传本质[24,25]。在长江三峡库区 5个野生鲢群体中, 每个微卫星位点的等位基因数从7—29不等(表3), 表明这10个微卫星均为较高多态性的座位。平均多态信息含量(PIC)是衡量等位基因片段多态性的理想指标, 当PIC>0.5时该位点表现为高度多态, 当 0.25<PIC<0.5时该位点表现为中度多态, 当PIC<0.25 时该位点表现为多态性低下[26]。在本研究中使用的10个微卫星标记中, 除了 Hysd2095-1为中度多态性以外,其余标记的PIC值均大于0.7为高度多态(表3), 这说明本文所选用的微卫星位点是鲢野生群体遗传多样性研究的理想分子遗传标记。
3.2 鲢群体遗传多样性及群体动态分析
基因杂合度表示群体在某座位杂合子的比例。观测杂合度(Ho)和期望杂合度(He)是反映群体遗传变异的两个重要指标。杂合度越大变异越大, 对环境适应能力越强[7]。本研究发现 5个鲢野生群体的平均等位基因为 10.7—13.5, 平均有效等位基因数Ne为 6.6—7.5, 平均观测杂合度Ho为0.784—0.846,平均期望杂合度He为 0.828—0.847, 平均多态信息含量PIC为0.797—0.817(表3), 这说明三峡库区鲢野生群体遗传多样性仍在很高水平, 对长江“四大家鱼”种质资源保护和恢复有积极意义。关于长江流域鲢群体遗传多样性分析, 迄今已有一些报道。朱晓东等[5]利用30个微卫星标记对长江中下游5个鲢群体进行遗传多样性分析, 发现这 5个鲢群体的平均有效等位基因数 Ne为 2.444—2.633, 平均观察杂合度 Ho为 0.323—0.351,平均期望杂合度 He为0.442—0.470,平均多态信息含量 PIC 为 0.406—0.428。王长忠等[6]对长江中上游万州和监利鲢群体的遗传多样性分析显示, 两个鲢群体的平均有效等位基因数Ne分别为4.107和3.395;平均观测杂合度Ho为0.834和0.775,平均期望杂合度He为0.713和0.623; 两群体平均多态信息含量 PIC为 0.617。由于本研究采用的微卫星标记绝大部分为高度多态性标记, 而且本文采用更加灵敏的荧光标记基因分型手段, 因此Ne比过去的报道要高的多。本研究中Ho、He、PIC等遗传多样性参数也远高于朱晓东等关于长江中下游 5个鲢群体的报道, 而与王长忠等[6]对于长江中上游2个鲢群体的报道较为接近。 这可能在一定程度上表明, 长江中上游相较于长江中下游鲢群体的遗传多样性更为丰富。
本文在所检测的微卫星位点中, WS群体在5个位点、YY群体在1个位点、MD群体在2个位点偏离哈迪-温伯格平衡。可见WS群体内有较多位点的基因型频率发生了较大改变, 而这种改变可能与多种因素有关, 例如突变、选择、迁移和杂交等。当一个群体的基因位点出现杂合度过剩及不足的概率大致均等时, 表明这个群体处于突变-漂移平衡的状态[27]。而当群体经历瓶颈效应则会表现出杂合度过剩的现象, 因此杂合度过剩可以衡量群体数量下降的瞬间效应[28]。本研究采用 IAM 模型符号检验发现, ZG、WS、YY、MD 4个群体显示出显著杂合子过剩(表 3), 说明以上 4 个群体可能在近期经历了不同程度的瓶颈效应, 群体数量发生一定程度的下降。在长江三峡水库建成运行的条件下, 库区过度人工捕捞等因素尤其应该引起重视, 以免对鲢等“四大家鱼”的遗传资源造成不可逆的破坏。
3.3 鲢群体遗传分化
基因分化系数是衡量群体间等位基因的遗传分化程度的重要参数。当 Fst为 0—0.05 时, 群体间遗传分化很弱; 0.05—0.15 时, 遗传分化中等; 0.15—0.25时, 遗传分化较大;大于0.25时, 表示群体间分化极大[29]。在 本研究中各群体间的 Fst值均小于0.05(表4), 表明三峡库区5个鲢野生群体间几乎没有分化。AMOVA分析结果也支持这一结果: 群体间的遗传变异只占0.47% , 而群体内的遗传变异高达 99.53% , 表明三峡库区鲢的遗传变异基本来自于群体内部。利用 structure软件中混合模型, 根据设定的分群数(K)进行群体遗传结构研究, K=2—5时均显示 5个鲢群体间遗传分化不明显, 基本可划分为一个类群。而ZG、WS、YY、ZX、MD 5个群体各有4、3、5、5和2个特殊等位基因, 这些特殊基因可能为 5个群体间将来出现遗传分化提供方向。过去的一些研究报道认为长江流域中各鲢群体或江段之间存在一定程度的遗传分化。如王淞等[7]对4 个鲢群体进行了mtDNA D-loop的PCR-RFLP分析, 结果显示长江干流的监利和瑞昌两个群体与长沙及宁河群体间有一定的分化; 朱晓东等[5]对长江中下游5个鲢群体进行了微卫星分析发现5个鲢群体间存在中等程度的分化; 而王长忠等[6]对长江中上游万州和监利两个鲢群体进行了微卫星分析认为万州和监利鲢群体间存在显著遗传分化, 应隶属于不同的种群。本研究认为三峡库区的5个鲢群体间没有遗传分化, 可以看成一个天然群体。这一结果也与郭稳杰[13]对长江中上游9个鲢群体的微卫星分析结果类似。郭稳杰[13]认为除湖口外, 其他群体间只呈现出较弱的群体分化。本文所研究的三峡库区的 5个采样点, 尽管有少数采样点在长江支流,但整体来说这5个群体之间的地理距离都不是太大,目前也没有明显的地理隔离, 故我们认为这些水域的鲢群体之间进行了较频繁的基因交流。
根据Nei氏遗传距离(表5)所绘制的5个野生鲢群体的邻接树(图2)显示, YY与MD的遗传距离最小, 而与 WS最大, 表明群体间的遗传距离与地理距离之间有一定的关联性。但也并非完全地理位置越远的群体间遗传分化程度越大, 例如 YY在地理位置上与ZX和WS更近, 结果显示YY与MD的聚类关系反而更近, 这可能是由于 YY部分位点偏离哈迪温伯格平衡, 导致部分基因型缺失, 从而在一定程度上影响了其遗传多样性; 而YY与WS的聚类关系反而最远, 可能是由于 WS独特的地理环境导致WS群体与其他群体间的基因交流受到一定影响; 也有可能是由于 WS群体受到较大的捕捞压力导致基因型频率受到影响。
遗传多样性是种内个体之间或群体内不同个体的遗传变异总和, 是生物适应环境的基础, 同时种内的遗传变异程度又决定其进化的潜势[30]。对一个物种来说, 遗传多样性越高, 则其对生存环境变化所产生的适应能力越强, 进化的潜力也越大。由于过去对长江上游尤其是三峡库区鲢野生群体的遗传多样性和遗传结构的现状了解不多, 人类对该地区鲢、鳙等经济鱼类的种群动态和种质资源评价方面长期缺乏群体遗传学分析数据。本研究的结果证实长江三峡库区范围内的鲢基本可看作一个天然群体,也在一定程度上给出了该地区鲢群体仍有较高的遗传多样性的事实。本研究分析结果也提示三峡地区可能多个鲢群体曾发生过遗传瓶颈效事件, 这也应引起鱼类保护生物学工作者的注意。本文对于长江上游以及整个长江流域的鲢野生资源保护或增殖放流效果评估等研究有积极作用。
[1] Li S F, Wu L Z, Wang Q, et al. Changjiang River, Zhujiang River, Heilongjiang River Silver Carp, Bighead Carp, Grass Carp Germplasm Resource Research [M]. Shanghai: Shanghai Science & Technology Press. 1990, 25—50 [李思发, 吴力钊,王强, 等. 长江、珠江、黑龙江鲢、鳙、草鱼种质资源研究.上海: 上海科学技术出版社. 1990, 25—50]
[2] Zhang M Y, Xu D P, Liu K, et al. Analysis of genetic diversity among silver the artificially released populations of carps in the lower reach of Yangtze River with microsatellite markers [J]. Acta Agriculturae Universitatis Jiangxiensis, 2012, 34(1): 141—146 [张敏莹, 徐东坡, 刘凯, 等. 长江下游放流鲢群体遗传多样性的微卫星标记分析. 江西农业大学学报, 2012, 34 (1): 141—146]
[3] Liu S P, Chen D Q, Duan X B, et al. Monitoring of the four famous Chinese Carps resources in the middle and upper reaches of the Yangtze River [J]. Resources and Environment in the Yangtze Basin, 2004, 13(2): 183—186 [刘绍平,陈大庆, 段辛斌, 等. 长江中上游四大家鱼资源监测与渔业管理. 长江流域资源与环境, 2004, 13(2): 183—186]
[4] Zhang S M, Deng H, Wang D Q, et al. Population structure and genetic diversity of Silver carp and Grass carp from populations of Yangtze River system revealed by RAPD [J]. Acta Hydrobiologica Sinica, 2001, 25(4): 324—330 [张四明,邓怀, 汪登强, 等. 长江水系鲢和草鱼遗传结构及变异性的 RAPD 研究. 水生生物学报, 2001, 25(4): 324—330]
[5] Zhu X D, Geng B, Li J, et al. Analysis of genetic diversity among silver carp populations in the middle and lower Yangtze River using thirty microsa-tellite markers [J]. Hereditas (Beijing), 2007, 29(6): 705—713 [朱晓东, 耿波,李娇, 等. 利用 30 个微卫星标记分析长江中下游鲢群体的遗传多样性. 遗传, 2007, 29(6): 705—713]
[6] Wang C Z, Liang H W, Zou G W, et al. Genetic variation analysis of two silver carp populations in the middle and upper Yangtze River by microsatellite [J]. Hereditas (Beijing), 2008, 30(10): 1341—1348 [王长忠, 梁宏伟, 邹桂伟, 等. 长江中上游两个鲢群体遗传变异的微卫星分析.遗传, 2008, 30(10): 1341—1348]
[7] Ji C H, Gu J J, Mao R X, et al. Analysis of genetic diversity among wild silver carp (Hypophthamlichthys molitrix) populations in the Yangtze, Heilongjiang and Pearl Rivers using microsatellite markers [J]. Journal of fisheries of China, 2009, 33(3): 354—371 [姬长虹, 谷晶晶, 毛瑞鑫,等. 长江、珠江、黑龙江水系野生鲢遗传多样性的微卫星分析. 水产学报, 2009, 33(3): 354—371]
[8] Wang S, Cao X X, Tanniguchi N. PCR-RFLP analysis on mtDNA D-loop region of four populations of silver carp (Hypophthalmichthys molitrix) [J]. Freshwater Fisheries, 2010, 40(4): 3—15 [王淞, 曹晓霞, 谷口顺彦, 等. 4 个群体鲢 mtDNA D-loop的 PCR-RFLP 分析.淡水渔业, 2010, 40(4): 3—15]
[9] Tautz D, Renz M. Simple sequences are ubiquitous repetitive components of eukaryotic genomes [J]. Nucleic Acids Research, 1984, 12(10): 4127—4138
[10] Liu L, Liu H Q, Guo Y S, et al. Isolation of microsatellite markers in the common snapping turtle (Chelydra serpentina L.) and its application in genetic diversity analysis in a cultured population [J]. Acta Hydrobiologica Sinica, 2013, 37(4): 669—677 [刘丽, 刘海情, 郭昱嵩, 等. 蛇鳄龟微卫星标记的开发及一个养殖群体的遗传多样性分析. 水生生物学报, 2013, 37(4): 669—677]
[11] Wang J J, Tong J G, Zhang Y G, et al. Study on the genetic diversity of two wild populations of Megalobrama pellegrini (teleostei, cyprinidae) [J]. Acta Hydrobiologica Sinica, 2014, 38(5): 975—979[王瑾瑾, 童金苟, 张耀光, 等. 厚颌鲂两个野生群体遗传多样性分析. 水生生物学报, 2014, 38(5): 975—979]
[12] Sambrook J, Russell D W. Molecular Cloning: A Laboratory Manual [M]. NewYork: Cold Spring Harbor Laboratory Press, 2001
[13] Guo W J. Studies on population genetic structure and comparative genome mapping in silver carp [D]. Thesisi for Doctor of Science. Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan, 2013 [郭稳杰. 鲢群体遗传结构及比较基因组作图研究. 博士学位论文. 中国科学院水生生物研究所, 武汉, 2013]
[14] Wang Y W, Samuels T D, Wu Y Q. Development of 1030 genomic SSR markers in switchgrass [J]. Theoretical and Applied Genetics, 2011, 122(4): 677—686
[15] Zhang J R, Wei C L. Establishment a SSR-PCR System of Tea Plant Based on the 4300 DNA Analysis System [J]. Journal of Tea Science, 2014, 34(5): 481—488 [张洁茹, 韦朝领. 基于 4300 DNA 分析系统的茶树 SSR 发掘方法优化与建立. 茶叶科学, 2014, 34(5): 481—488]
[16] Nei M, Li W H. Mathematical model for studying genetic variation in terms of restriction endonucleases [J]. Proceedings of the National Academy of Sciences, 1979, 76(10): 5269—5273
[17] Tamura k, Dudley J, Nei M, et al. MEGA4: Molecular evolutionary genetics aualysisi (MEGA) sofeware version 4.0 [J]. Molecular Biology & Evolution, 2007, 24(8): 1596—1599
[18] Excoffier L, Laval G, Schneider S. Arlequin(version 3.1): An integrated software package for population genetics dada analysis [J]. Evolutionary Bioinformatics Onlion, 2006, 1(1): 47—50
[19] Rice W. Analyzing tables of statistical tests [J]. Evolution, 1989, 43(1): 223—225
[20] Excoffier L, Smouse P E, Quattro J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to Human mitochondrial DNA restriction data [J]. Genetics, 1992, 131(2): 479—491
[21] Maruyama T, Fuerst P A. Population bottlenecks and nonequilibrium models in population genetics. II. Number of alleles in a small population that was formed by a recent bottleneck [J]. Genetics, 1985, 111(3): 675—689
[22] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data [J]. Genetics, 2000, 155(2): 945—959
[23] Liu M, Yu D H, Huang G J. Isolation and screening of microsatellite DNA markers from silver-lip pearl oyster Pinctada maxima [J]. Marine Science, 2010, 34(8): 1—5 [柳明, 喻达辉, 黄桂菊. 大珠母贝微卫星 DNA标记的分离与筛选. 海洋科学, 2010, 34(8): 1—5]
[24] Kijas J M H, Thomas M R, Fowler J C S, et al. Integration of trinucleotide microsatellites into a linkage map of Citrus [J]. Theoretical and Applied Genetics, 1997, 94(5): 701—706
[25] Gastier J M, Pulido J G, Sunden S, et al. Survey of trinucleotide repeats in the human genome: assessment of their utility as genetic markers [J]. Human Molecular Genetics, 1995, 4(10): 1829—1836
[26] Nei M. Genetic distance between populations [J]. American Naturalist, 1972, 106(949): 283—292
[27] Cornuet J M, Luikart G. Description and power analysis of two tests for detecting recent population bottlenecks from allele frequency data [J]. Genetics, 1996, 144(4): 2001—2014
[28] Nei M, Maruyama T, Chakraborty R. The bottleneck effect and genetic variability in populations [J]. Evolution, 1975, 29(1): 1—10
[29] Wright S. Evolution and the Genetics of Population Variability Within and Among Natural Population [M]. Chicago: University of Chicago Press, 1978, 4
[30] Chen J M. Studies on Conservation Genetics of the Rare and Endangered Aquatic plant Caldesia grandis (Alismataceae) [D]. Thesis for Doctor of Science. Wuhan University, Wuhan. 2005 [陈进明. 珍稀濒危水生植物宽叶泽苔草(泽泻科)的保护遗传学研究. 博士学位论文,武汉大学, 武汉. 2005]
THE MICROSATELLITE ANALYSIS OF GENETIC DIVERSITY OF FIVE SILVER CARP POPULATIONS IN THE THREE GORGES RESERVOIR OF THE YANGTZE RIVER
PANG Mei-Xia1,2, YU Xiao-Mu1and TONG Jin-Gou1
(1. State Key Laboratory of Freshwater Ecology and Biotechnology, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
In this study we employed ten microsatellite markers to analyze the genetic diversity of five silver carp populations including Zigui, Wushan, Yunyang, Zhongxian and Mudong in the Three Gorges Reservoir of the Yangtze River. A total of 161 different alleles were detected, and 84 of them were common in all 5 populations. The number of alleles per locus ranged from 7 to 29. In the five populations, the average values of observed and expected heterozygosity ranged from 0.784 to 0.846 and 0.828 to 0.847 respectively, and the mean PIC was 0.797 to 0.817. The Fstvalues were between –0.001 and 0.009, which indicated that the genetic differentiation was not significant. Chi-square test was used to analyze the genotypes based on the Hardy-Weinberg equilibrium, and the P value indicated that some loci in three populations deviated from the HWE. The bottleneck analysis revealed that the silver carp populations from the Three Gorges Reservoir of the Yangtze River might have undergone a recent genetic bottleneck. The genetic similarity coefficient of the five populations was between 0.891 and 0.950, and the genetic distance of the populations was between 0.050 and 0.115. We also constructed the phylogenetic tree of the five silver carp populations based on Nei’s standard genetic distance, and the results showed that genetic distances between the populations were consistent with their geographical distances. The Bayesian analysis also suggested that the silver carp samples from the 5 locations of the Three Gorges Reservoir could be characterized as a single population. Although we did not observe the genetic differentiation, silver carp populations in the Three Gorges Reservoir displayed high genetic diversity. Our study provided valuable information on the genetic resources of silver carps in the Three Gorges Reservoir and in the upstream of the Yangtze River, which should help develop appropriate policies on the conservation and utilization of silver carps.
Silver carp (Hypophthalmichys molitrix); Microsatellite; Wild population; Genetic diversity; The Three Gorges Reservoir
Q346+.5
A
1000-3207(2015)05-0869-08
10.7541/2015.115
2015-02-12;
2015-03-26
中国长江三峡集团公司科研项目(CT12-08-01); 淡水生态与生物技术国家重点实验室自主课题(2011FBZ20)资助
庞美霞(1989—), 女, 湖南长沙人; 博士研究生; 主要从事鱼类遗传学研究。E-mail: pang1mei2xia3@163.com
童金苟, E-mail: jgtong@ihb.ac.cn

