三峡库区鲌属鱼类线粒体COⅠ基因遗传多样性的初步分析
王 丹程庆武杨镇宇连玉喜叶少文刘家寿李钟杰罗相忠邹桂伟
(1. 中国水产科学研究院长江水产研究所, 中国水产科学研究院淡水鱼类育种与健康养殖重点实验室 武汉 430223;2. 武汉市江夏区国营牛山湖渔场, 武汉 430205; 3. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室,武汉 430072; 4. 中国科学院大学, 北京 100049)
三峡库区鲌属鱼类线粒体COⅠ基因遗传多样性的初步分析
王 丹1程庆武2杨镇宇1连玉喜3,4叶少文3刘家寿3李钟杰3罗相忠1邹桂伟1
(1. 中国水产科学研究院长江水产研究所, 中国水产科学研究院淡水鱼类育种与健康养殖重点实验室 武汉 430223;2. 武汉市江夏区国营牛山湖渔场, 武汉 430205; 3. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室,武汉 430072; 4. 中国科学院大学, 北京 100049)
鲌属鱼类(Culter spp.)隶属于鲤科(Cyprinidae)、 鲌亚科(Cultrinae), 是东亚特有鲤科鱼类的一个重要组成部分,广泛分布于中国、越南、朝鲜和俄罗斯, 在我国除青藏高原外, 其他各地均有分布[1], 该属大部分物种都是重要的经济鱼类(如 常见的翘嘴 鲌C. alburnus、 蒙古 鲌C. mongolicus、 达氏 鲌C. dabryi等), 同时它们在维持水域生态平衡中也发挥着重要作用。近年来, 由于过度捕捞、栖息地改变、水质污染等人为因素影响, 保护 鲌属鱼类的种质资源刻不容缓, 已有学者对 鲌属不同地理种群的遗传结构进行了调查评估[2,3], 但主要集中在长江中下游流域,针对长江上游地区的相关研究少有报道。
线粒体DNA具有分子小、拷贝数多、编码效率高、母系遗传、进化速率快等特点[4], CO Ⅰ(Cytochrome Oxidase subunit Ⅰ)基因是位于线粒体DNA的蛋白质编码基因, 能够对大部分物种进行有效鉴定, 并且存在显著的序列变异, 因而广泛应用于物种鉴定、群体遗传学等研究中[5—9]。彭居俐等[5]用COⅠ 基因序列探讨了 鲌属鱼类种间的系统发育关系; 张大莉等[6]用 COⅠ基因序列研究大口黑鲈Micropterus salmoides的遗传多样性, 认为大口黑鲈佛罗里达亚种的遗传多样性高于北方亚种。
在三峡水库蓄水后, 库区水位升高、水流减缓、泥沙沉积、饵料生物组成变化, 这种条件一般适宜于喜缓流水或静水生活的鱼类(例 如适应范围较广的 鲌亚科鱼类), 香溪河[10]、大宁河[11]、小江[12]等库区典型支流的渔获物调查结果也表明翘嘴 鲌、蒙古 鲌等在现阶段鱼类群落中属于常见种或优势种, 目前有关三峡水库 鲌属野生种群的的遗传结构分析资料还非常缺乏。本研究从CO Ⅰ基因水平上评估三峡库区几个典型支流的 鲌属鱼类遗传多样性, 分析不同种群的遗传结构差异, 以期为合理利用和保护三峡库区 鲌属鱼类自然种质资源提供理论依据和科学参考。
1 材料与方法
1.1 研究区域
本研究的鱼类采样地点包括三峡水库湖北省秭归县境内的百岁溪、香溪河和童庄河, 重庆市巫山县境内的大宁河, 以及重庆市云阳县境内的小江水域(图1); 其中百岁溪距离三峡大坝约7 km, 香溪河和童庄河距离大坝约34 km,大宁河和小江河距离大坝分别约124 km和252 km。
1.2 鱼类样品采集
三峡库区 鲌属鱼类样品采集于2013年3—8月进行,沿河岸调查渔民渔获物情况, 所用网具主要为三层刺网,其规格为网长100 m或150 m, 网高4—20 m, 网目大小3—12 cm 。 鲌属种类鉴定依照《中国动物志》[1], 调查期间采集到的 鲌属鱼类包括达氏 鲌、蒙古 鲌和翘嘴 鲌, 现场剪下胸鳍条放入 95%乙醇浸泡保存。样品采集信息见表1, 其中达氏 鲌在大宁河和小江渔获物中的出现率和相对丰度均很低, 因而造成样品不足, 蒙古 鲌在香溪河以及翘嘴 鲌在百岁溪和大宁河也存在同样情况, 鲌属鱼类样品采集分布情况较大程度上与它们在各研究区域的资源量现状相符合。
1.3 基因组DNA提取与检测
基因组 DNA提取采用经典的酚-氯仿法[13]。基因组DNA提取后用0.8%琼脂糖凝胶电泳检测其质量, 用紫外分光光度计测定DNA浓度, 置于4℃保存备用。
1.4 PCR扩增及测序
PCR扩增的目的片段是 COⅠ基因靠近 5′末端的长度为 943 bp左右的序列, 其扩增引物为 L5996-COⅠ(5′-CACAAAGACATTGGCACCCT-3′)和 H6855-COⅠ(5′-AGTCAGCTGAAKACTTTTAC-3′)[5]。

图1 采样地点分布图Fig. 1 Geographic map of the sampling sites in the Three Gorges Reservoir

表1 三峡水库鲌属鱼类样品采集信息Tab. 1 Sampling information for Culter spp. in the Three Gorges Reservoir
PCR反应条件如下: 94℃预变性 5min; 循环 35次,包括94℃变性35s, 退火温度50 ℃ ,退火时间35s, 72℃延伸50s; 最后72℃终延伸10min。
PCR反应体系: 总体积25 μL, 包括大约20 ng总DNA,上、下游引物各1 μL (5 μmol/L), 1 μLdNTP (10 mmol/L), 0.6 U Taq聚合酶(TIANGEN), 2.5 μL缓冲液(含 Mg2+1.5 mmol/L)。
每次PCR反应设立空白对照。扩增产物在0.8%的琼脂糖凝胶上进行电泳以检测有无扩增产物; 电泳后凝胶采用溴化乙锭进行染色, 紫外凝胶扫描系统扫描记录;PCR产物克隆后测序。引物合成和目的片段测序工作由武汉擎科生物技术有限公司完成。
1.5 数据处理与分析
利用 ContigExp软件对获得的测序序列进行片段拼接和序列分析; MEGA6软件计算组内遗传距离, 分析序列的碱基组成[14]; DnaSP 软件计算单倍型多样性(Haplotype diversity, Hd)和核苷酸多态性(Nucleotide diversity, Pi)[15]。
2 结果
2.1 鲌属三种鱼类COⅠ序列特征及遗传多样性
通过PCR 扩增得到三峡库区翘嘴 鲌 、蒙古 鲌、达氏鲌的共53个COⅠ扩增带。获得CO Ⅰ基因靠5′端一段长度约为943 bp的片段, 编码314氨基酸。在943个排列位点中, 变异位点 208个, 约占 22.1%; 简约信息位点(Parsimony informative sites) 71 个, 单一多态位点(Singleton variable sites) 137个。单倍型共有42个, 每个群体的单倍型和独有单倍型数见表2。
三峡库区三种 鲌鱼的单倍型多样性为 0.9869±0.007,核苷酸多态性为 0.0301±0.0042。单倍型多样性范围为0.8667—1, 核苷酸多态性范围为 0.0022—0.0715(表 2),达氏 鲌群体遗传多样性高低排序为大宁河>童庄河>香溪河, 小江蒙古 鲌群体的遗传多样性大于香溪河群体, 翘嘴 鲌小江群体遗传多样性显著大于香溪河群体。
2.2 鲌属三种鱼类碱基组成
如表3所示, 三峡库区 鲌属三种鱼类的T、C、A、G碱基平均含量分别为 28.4%、26.7%、26.3%、18.6%, 其中A+T含量(54.7%)明显高于G+C含量(45.3%)。G+C含量排列顺序由高到低依次为蒙古 鲌(46.3%)、 翘嘴鲌(45.2%)和 达氏 鲌(45%)。
2.3 鲌属三种鱼类的遗传距离
三峡库区三种 鲌属鱼类两两比较的种间平均遗传距离见表4, 蒙古 鲌 和达氏 鲌的种间距离最大(0.0458), 蒙古 鲌和翘嘴 鲌的种间距离最小(0.0378), 鲌属三种鱼类的平均遗传距离为0.0314。
由表4和5可知, 达氏 鲌的种内平均遗传距离为0.0116, 种内遗传距离最大为 0.0412, 发生在童庄河和大宁河间; 最小种内遗传距离发生在小江和香溪河间, 其值为0.0020 。翘嘴 鲌的种内平均遗传距离为0.0230, 其中遗传距离最大为 0.1515, 发生在大宁河和香溪河群体间;最小遗传距离为0.0095, 发生在小江和香溪河群体间。蒙古 鲌的种内平均遗传距离为 0.0230, 小江和香溪河群体间遗传距离为0.0029。
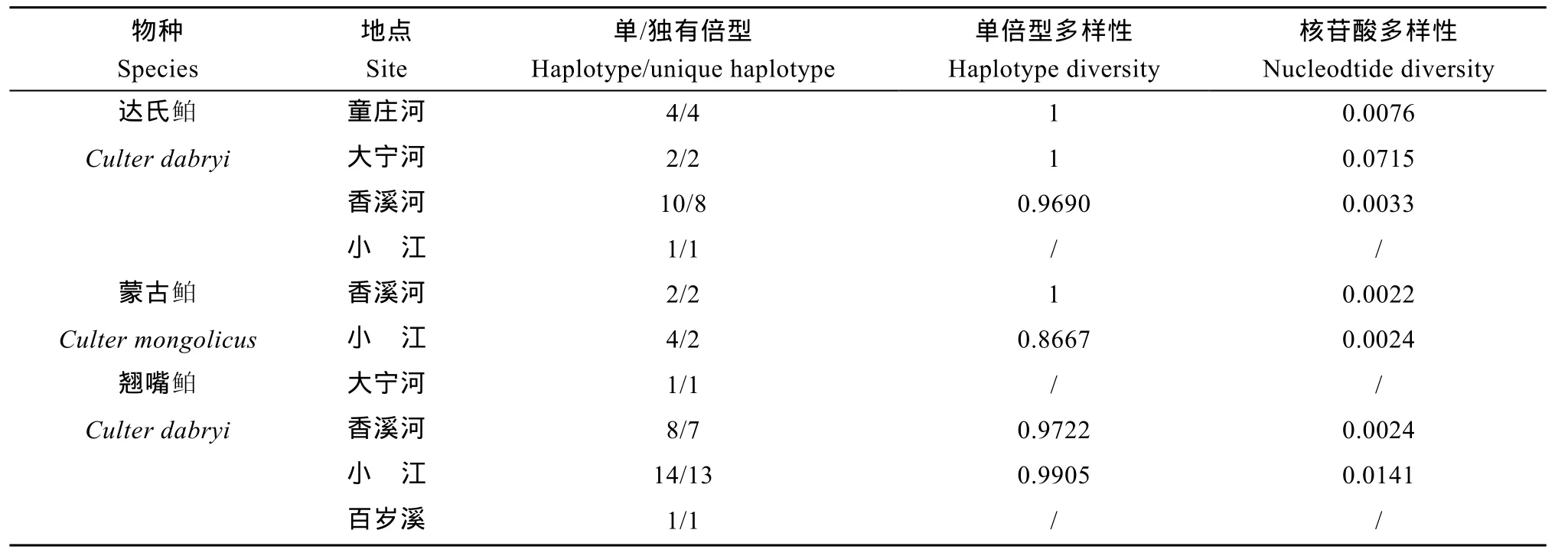
表2 三峡库区鲌属三种鱼类的单倍型多样性和核苷酸多样性Tab. 2 Haplotype diversity and nucleotide diversity of Culter spp. in the Three Gorges Reservoir

表3 三峡库区三种鲌属鱼类COⅠ碱基组成Tab. 3 Composition of nucleotide of Culter spp. in the Three Gorges Reservoir

表4 三峡库区三种鲌属鱼类两两比对的种间平均遗传距离和种内遗传距离Tab. 4 Genetic distance within species and pairwise distance between Culter spp. in the Three Gorges Reservoir
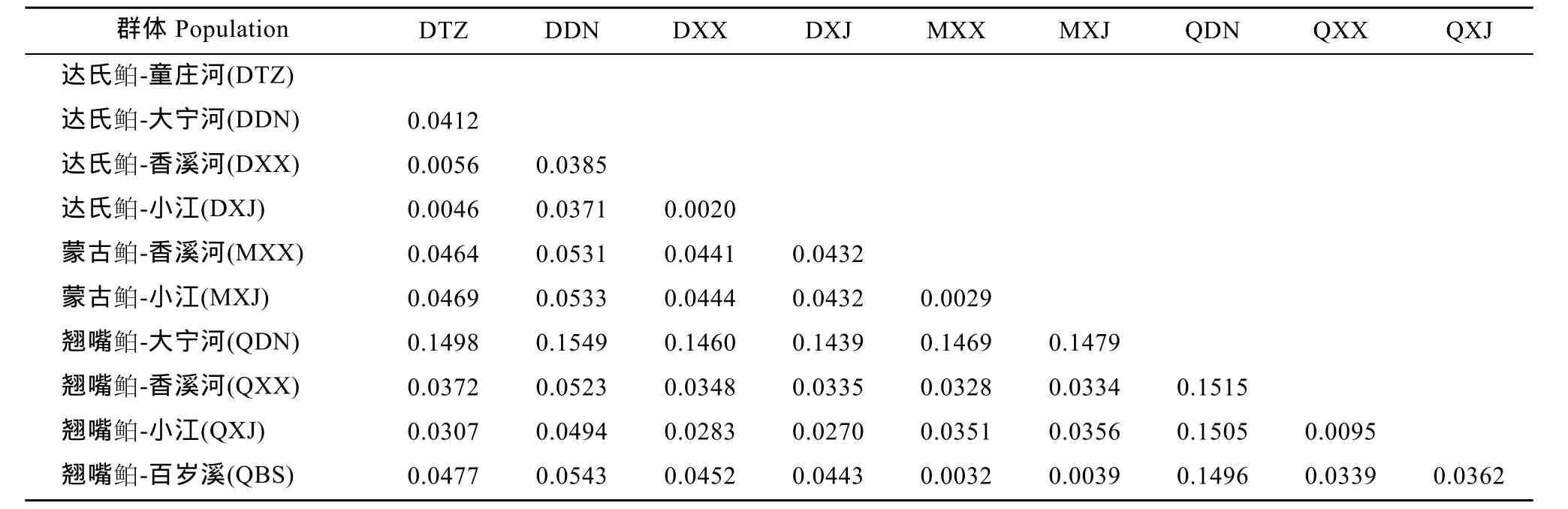
表5 三峡库区三种鲌属鱼类组间遗传距离Tab. 5 The genetic distance between groups of Culter spp. in Three Gorges Reservoir
3 讨论
鲌属鱼类是东亚特有鲤科鱼类中较为重要的一个类群, 经历了相同的短时间物种暴发过程。作为一群快速分化的物种, 其基因序列的变异度相对较小[16]。在本研究中,三峡库区53 条 鲌属鱼类线粒体DNA序列中变异位点208个, 约占22.1%。从碱基组成上来看, T、C、A、G各个碱基所占百分比分别为28.4%、26.7%、26.3%和18.6%。平均 G+C含量为 45.3%, 这与彭居俐等[5]的研究结果(45.6%)相似, 表现出明显的A+T偏倚性。核苷酸碱基构成的偏倚是线粒体蛋白质编码基因的一个显著特征, 与脊椎动物线粒体DNA的特点是一致的[17]。
从单倍型数据来看, 本研究共测序 53个样品, 单倍型数目高达 42个, 各群体的单倍型数目很高, 拥有较多的独有单倍型, 仅少数个体共享单倍型, 反映了三峡库区 鲌属鱼类整体比较保守。检测到独有单倍型最多的 3个群体分别是翘嘴 鲌小江群体、达氏 鲌香溪河群体和翘嘴鲌香溪河群体。可能香溪河和小江地区的 鲌属鱼类具有更高的遗传多样性。
种间和种内遗传距离的大小是进行物种鉴别的主要标准。Hebert等[4]对动物界11个门、13320个物种的研究结果显示, 种内遗传距离大多数在 1% 以下, 很少超过2%。比较群体间的遗传距离, 三峡库区 鲌属三种鱼类平均遗传距离为 0.0314, 显著大于种间鉴定的最小遗传距离2% 。达氏 鲌 、蒙古 鲌 和翘嘴 鲌的种内平均遗传距离分别是0.0116、0.0031和0.0230, 种间遗传距离大于种内平均遗传距离, 均符合Hebert等[4]的结论。
单倍型多样度(Hd)和核苷酸多样度(Pi)是评价一个群体mtDNA变异程度的主要指标, H和Pi值越大, 表明群体的遗传多样性越丰富。核苷酸多态性考虑各种mtDNA单倍型在群体中所占的比例, 反映的群体mtDNA多态程度更为精确。本研究结果表明, 基于线粒体 COⅠ基因序列, 鲌
三峡库区的 属鱼类具有很高的核苷酸多态性0.0301 (Pi>0.005)和较高的单倍型多样性 0.9869(Hd>0.8),这一结果高于鲑科鱼类[8]和细鳞斜颌鲴 Xenocypris microlepis[9]。
综上所述, 鲌三峡库区 属鱼类具有较高的遗传多样性, 不同地理群体享有较多的特有单倍型, 可能蕴藏着较大的进化潜能和更丰富的种质资源, 后续研究将考虑增加样品数量和采集点, 鲌全面评估三峡库区 属鱼类的种质状况和遗传结构。
[1] Chen Y Y, Fauna Sinica·Osteichthyes·Cypriniformes II [M]. Beijing: Sciences Press. 1998, 185—197 [陈宜瑜. 中国动物志·硬骨鱼纲·鲤形目(中卷). 北京: 科学出版社. 1998,185—197]
[2] Wang W, Chen L Q, Yu N, et al. Assessment of genetic diversity of topmouth culter Culter alburnus populations by CO Ⅱ sequences [J].Journal of Dalian Fisheries University, 2008, 23(5): 403—408 [王伟, 陈立侨, 禹娜, 等. 应用COⅡ基因部分序列分析翘嘴 鲌群体的遗传多样性. 大连水产学院学报, 2008, 23(5): 403—408]
[3] Yang T Y, Chen H X, Liu X Q, et al. Genetic diversity of Erythroculter ilishaeformis from Danjiangkou Reservoir revealed by RAPD and ISSR analysis [J]. Oceanologia et Limnologia Sinica, 2008, 39(3): 240—244 [杨太有, 陈宏喜,刘向奇, 等. 丹江口水库翘嘴鲌(Erythroculter ilishaeformis)遗传多样性的 RAPD和 ISSR分析. 海洋与湖沼, 2008, 39(3): 240—244]
[4] Hebert P D N, Ratnasingham S, deWaard U R. Barcoding animal life: cytochrome c oxidsse subunit 1 divergences among closely related species [J]. Proceeding of the Royal Society of London B: Biological Science, 2003, 270(suppl. 1): 96—99
[5] Peng J L, Wang X Z, Wang D, et al. Application of DNA barcoding based on the mitochondrial CO I gene sequences in classification of Culter (Pisces: Cyprinidae) [J]. Acta Hydrobiologyica Sinica, 2009, 33(2): 271—276 [彭居丽, 王绪祯, 王丁, 等. 基于线粒体COⅠ基因序列的DNA条形码在鲤科鲌 属鱼类物种鉴定中的应用. 水生生物学报, 2009, 33(2): 271—276]
[6] Zhang D L, Dong S, Bai J J, et al. Sequence analysis of mtDNA COⅠ region in northern and Florida subspecies of largemouth bass Micropterus salmoides [J]. Journal of Dalian Fisheries University, 2014, 29(3): 212—216 [张大莉,董仕, 白俊杰, 等. 大口黑鲈北方亚种和佛罗里达亚种mtDNA COⅠ序列的分析. 大连海洋大学学报, 2014, 29(3): 212—216]
[7] Liu S F, Chen L L, Dai F Q, et al. Application of DNA barcoding gene COⅠ for classifying family Sciaenidae [J]. Oceanologia et Limnologia Sinica, 2010, 41(2): 223—232 [柳淑芳, 陈亮亮, 戴芳群, 等, 基于线粒体 COⅠ基因的DNA 条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用. 海洋与湖沼, 2010, 41(2): 223—232]
[8] Li F G, Zou S M. Genetic diversity analysis of Salmo truttafario based on COⅠ and COⅡ [J]. Journal of Anhui Agricultural Sciences, 2014, 42(23): 7710—7713 [李福贵, 邹曙明. 亚东鲑线粒体COⅠ与COⅡ基因遗传多样性分析.安徽农业科学, 2014, 42(23): 7710—7713]
[9] Qiao D L, He X M, Wei C B, et al. Genetic variation of mtDNA COⅡ gene in three populations of Xenocypris microlepis [J]. Freshwater Fisheries, 2011, 41(5): 17—21 [乔德亮, 何晓梅, 韦传宝, 等. 细鳞斜颌鲴三个群体线粒体COⅡ基因的遗传变异. 淡水渔业, 2011, 41(5): 17—21]
[10] Zhao S S, Ye S W, Xie S G, et al. The current situation of fishery resources and fisheries management strategies in the Xiangxi River of The Three Gorges Reservoir [J]. Acta Hydrobiologica Sinica, 2015, 39(5): 973—982 [赵莎莎, 叶少文, 谢松光, 等. 三峡水库香溪河鱼类资源现状及渔业管理. 水生生物学报, 2015, 39(5): 973—982]
[11] Yang F, Yao W Z, Deng H T, et al. The current situation of fish resources in the Daning River after the impoundment of the Three Gorges Reservoir [J]. Freshwater Fisheries, 2013, 43(4): 51—57 [杨峰, 姚维志, 邓华堂, 等. 三峡库区蓄水后大宁河鱼类资源现状研究. 淡水渔业, 2013, 43(4): 51—57]
[12] Li B, Jiang X, Wang Z, et al. The current situation of fishery resources in the Xiaojiang River of the Three Gorges Reservoir [J]. Freshwater Fisheries, 2011, 41(6): 37—42 [李斌,江星, 王志坚, 等. 三峡库区蓄水后小江鱼类资源现状.淡水渔业, 2011, 41(6): 37—42]
[13] Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning: a Laboratory Manual [M]. NewYork: Cold Spring Harbor Laboratory Press. 1989
[14] Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0 [J]. Molecular Biology and Evolution, 2013, 30: 2725—2729
[15] Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data [J]. Bioinformatics, 2009, 25: 1451—1452
[16] He S P, Liu H, Chen Y Y, et al. Molecular phylogenetic relationships of Eastern Asian Cyprinidse (Pisces: Cypriniformes) inferred from cytochrome b sequences [J]. Science in China, 2004, 47: 130—138
[17] Xiao W H, Zhang Y P. Genetics and evolution of mitochondrial DNA in fish [J]. Acta Hydrobiologica Sinica, 2000, 24(4): 384—391 [肖武汉, 张亚平. 鱼类线粒体DNA 的遗传与进化. 水生生物学报, 2000, 24(4): 384—391]
PRELIMINARY ANALYSIS ON THE MTDNA COⅠ GENETIC DIVERSITY OF CULTER SPP. (CYPRINIDAE) IN THE THREE GORGES RESERVOIR
WANG Dan1, CHENG Qing-Wu2, YANG Zhen-Yu1, LIAN Yu-Xi3,4, YE Shao-Wen3, LIU Jia-Shou3, LI Zhong-Jie3, LUO Xiang-Zhong1and ZHOU Gui-Wei1
(1. Yangtze River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Wuhan 430223, China; 2. NiuShan Lake Farm of Jiangxia District, Wuhan 430205, China; 3. State Key Laboratory of Freshwater Ecology and Biotechnology, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 3. University of Chinese Academy of Sciences, Beijing 100049, China)
三峡水库; 鲌属; COⅠ基因; 遗传多样性
Three Gorge Reservoir; Culter spp.; CO Ⅰgene; Genetic Diversity
Q347
A
1000-3207(2015)05-1054-05
10.7541/2015.138
2015-03-26;
2015-05-05
中央级公益性科研院所基本科研业务费专项资金(2015JBFM39); 淡水生态与生物技术国家重点实验室(2014FB16);农业部淡水水产生物技术与遗传育种重点实验室(FBB2014-02); 农业部热带亚热带水产资源利用与养殖重点实验室开放课题资助
王丹(1980—), 女, 湖北武汉人; 博士; 助理研究员; 研究方向为鱼类遗传学。E-mail: wangdan@yfi.ac.cn
邹桂伟, E-mail: zougw@yfi.ac.cn

