时间维信息的4D多发性硬化损伤区域分割方法
詹天明,詹永照,冯 灿
(1.江苏大学计算机科学与通信工程学院,江苏镇江212013;2.北方信息控制集团有限公司,江苏南京211153)
时间维信息的4D多发性硬化损伤区域分割方法
詹天明1,詹永照1,冯 灿2
(1.江苏大学计算机科学与通信工程学院,江苏镇江212013;2.北方信息控制集团有限公司,江苏南京211153)
为了解决3D脑核磁共振图像分割方法无法准确分割4D多发性硬化损伤区域的问题,结合4D数据中每个时间点的信息并根据相邻时间点图像的相关性构造动态先验改进邻域驱动的水平集模型,提出一种基于时间维信息的4D多发性硬化损伤区域的分割模型.通过交替迭代和最速下降法求该模型的最小化问题得到4D多发性硬化损伤区域的分割算法.为验证该模型,对临床4D数据的分割效果进行测试,并与3D分割方法所得结果进行比较.结果表明:与3D图像分割方法相比,所提出的4D多发性硬化损伤区域的分割方法由于增加了时间维上的约束,得到的分割结果更加准确.这为后续的病灶定量化分析提供了有效的帮助.
多发性硬化;损伤区域;时间维;水平集;分割
多发性硬化损伤是一种慢性、炎症性、脱髓鞘的中枢神经系统疾病,可引起各种症状,包括感觉改变、视觉障碍、肌肉无力、忧郁、协调与讲话困难、严重的疲劳、认知障碍、平衡障碍、体热和疼痛等,严重的可以导致活动性障碍和疾病.核磁共振图像为研究该疾病的病因和病理以及评判治疗效果提供了有效的依据.对核磁共振图像进行处理可以为准确测量以及定量分析多发性硬化损伤区域特别是活动区域提供有效的依据.因此,需要利用图像分割方法[1-2]对核磁共振图像进行分割以提取出损伤区域以及活动区域来分析多发性硬化区域损伤的特点以及其随时间的变化情况.
目前,基于核磁共振图像的多发性硬化损伤区域的分割方法已经有很多.文献[3]将这些方法分为基于监督学习的分割方法和非监督的分割方法.其中,基于监督学习的方法[4-6]通过对训练集中已分割出的多发性硬化损伤区域的学习提取出多发性硬化损伤区域的共性特征,并利用这一特征对其他数据进行分割.而非监督的分割方法[7-9]无需对训练集中的结果进行学习,直接根据待分割图像中多发性硬化损伤区域的某些特点进行分割.这两类方法虽然都能在一定程度上较好地分割多发性硬化损伤区域,但缺点也较明显.其中基于监督学习的分割方法对训练集的选择非常重要,训练集中的数据要包含各种不同特征的多发性硬化损伤区域.此外,训练集中的多发性硬化损伤区域大都是通过手动分割而得到的,这一个工作非常耗时,且不同专家的手动分割结果的差别也较大.而非监督的方法对图像的质量要求很高,且需要专家提供一些先验信息.
上述这些多发性硬化损伤区域的分割方法都只是对3D脑核磁共振图像进行分割,其分割结果只能分析多发性硬化损伤区域的大小以及灰度等特点.但检测不同时间点上多发性硬化损伤区域的变化即活动区域也是临床分析中的一个关键点.图1为2个时间点上的FLAIR(fluid attenuated inversion recovery)序列图像.

图1 2个时间点上的FLAIR序列图像
图1b中的多发性硬化区域明显较图1a中的硬化区域有了显著的变化.如果用上述方法分别从这2个时间点的图像中分割出多发性硬化损伤区域,再用2个时间点的分割结果的差得出活动区域以及病灶变化情况会使得测量结果与真实结果偏差较大[10].为此,应充分利用时间维上的信息来指导分割从而得到准确的活动区域的测量结果.有些学者对已经配准好的2组三维数据进行非刚性配准再计算出2个时间点的图像的非刚性形变场,从而得到由于多发性硬化损伤的影响而导致的脑组织的变化情况[10-12].但灰度的变化会影响形变,因此该类方法无法确保能捕获活动区域的复杂特性.
针对上述问题,文中提出一种基于时间维信息的4D多发性硬化损伤区域的分割方法.首先对2个时间点的数据进行单独分割为后续整体分割提供初始值.再根据第2个时间点的数据的分割结果相结合构造动态先验提出4D多发性硬化损伤区域的水平集分割模型.该模型由数据拟合项和时间维约束项构成.数据拟合项是采用RSF(region scalable fitting)模型[13],而时间维约束项则由相邻时间点的图像灰度差和水平集函数构造而成.
1 4D多发性硬化损伤区域分割方法
为检测活动的多发性硬化损伤区域,需要对多个时间点的数据进行分割.如果用3D的多发性硬化损伤区域分割方法分别分割各个时间点的数据,分割结果并不准确,从而影响后续的活动区域的检测结果.因此需要对4D的数据进行整体的分割,提高分割精度.文中先对每个时间点的数据单独进行分割为后续整体分割提供初始化,再提出基于时间维信息的4D多发性硬化损伤区域的分割模型,通过能量极小化的方法得到4D的分割结果.最后根据4D的分割结果检测出活动区域.3D的分割方法可以为后续4D的分割方法提供有效的初始化,而4D分割方法则充分利用时间维的信息,提高了分割精度.
1.1 预处理
设I={I(t1),I(t2),…,I(tN)}是4D FLAIR序列图像,I(tj)是同一个样本在第j个时间点扫描得到的3D FLAIR序列图像,文中N=2.由于成像的参数不同以及扫描时的位置不同,为了后续准确的4D分割,需对这批图像进行预处理.首先利用刚性配准的方法将后续时间点的3D图像与第1个时间点的3D图像进行配准.然后,利用BET(brain extraction tool)方法将第1个时间点的脑壳剔除,并建立模板将后续时间点的脑图像中的脑壳去除.最后利用全局归一化的方法调整所有时间点的图像灰度,使得各个时间点的图像灰度范围较为接近.在试验中,以第2个时间点的灰度分布为基准,将第1个时间点的灰度往标准的灰度分布上进行匹配,为后续构造时间维的约束提供了有效的帮助.
1.2 每个时间点数据的独立分割
为了给4D多发性硬化损伤区域的分割方法提供先验知识并提高分割效率,先在每个时间点的3D图像中分别分割出多发性硬化损伤区域,其分割结果作为4D分割方法的初始输入值.具体做法如下:首先利用参数化分割方法[14]对FLAIR序列图像进行分割和去偏移场,利用其分割结果构造ROI(region of interesting).再用异常检测方法计算ROI中不服从脑组织分布的异常区域.最后以异常区域构造初始水平集,利用RSF方法在感兴趣区域内演化曲线得到每个时间点的多发性硬化区域的最终分割结果.这种多发性硬化损伤的分割流程只对每个时间点的数据独立进行分割,并没有考虑时间维上图像之间的相关性,这会导致在某些非活动区域的分割结果在各个时间点上存在一定的差别,从而影响后续的统计测量以及活动区域检测的结果.因此需要充分利用时间维上的图像相关性,对时间维上的分割结果进行相关约束.
1.3 4D多发性硬化损伤区域的分割模型
为了充分利用时间维上的信息,提高分割精度减少分割误差,提出基于时间维信息的4D多发性硬化损伤区域的水平集分割模型.该模型是用3D水平集分割模型作为数据拟合项体现了各个时间点图像的各自特点,并增加了各向异性的时间维的水平集约束项.该约束项使得在图像相似性较大的区域保持水平集有相同的演化方式,而在图像相似性较小的区域降低该约束项的约束能力,各个时间点的水平集按照各自图像的特点分别进行演化.这样既可以保证不变的区域在时间上具有相同的分割结果,又可以在活动区域保持各自变化的特点.
1.3.1 数据拟合项
文中的数据拟合项是通过在ROI中定义RSF模型[13]并将其扩展到4D框架,其总能量方程为


式中:J(t)为第t个时间点去完偏移场后的图像;φ(t)为第t个时间点的水平集函数;{φ(t)>0}和{φ(t)<0}分别为第t个时间点的感兴趣区域R(t)被曲线C(t)划分的2个区域;K为高斯核函数;H为Heaviside函数;γ和μ为非负常数.式(1)中的第1项和第2项成为区域调节项;第3项是曲线长度项,其目的是使曲线在演化过程中保持光滑的特点;第4项是水平集惩罚项.
1.3.2 时间维约束项
与4D脑组织分割的时间维约束项不同,多发性硬化损伤区域的时间维约束项既要保持在硬化不变区域内保持分割结果的连续性又要保证在硬化变化区域内分割结果变化的特点.为此利用2个时间点的灰度差以及水平集函数构造各向异性的时间维约束项:


图2 D(Jt(y))的图例
1.3.3 能量方程及求解
根据以上分析,由数据拟合项和时间维约束项构成的4D多发性硬化损伤区域分割的总能量为

式中α是非负参数.该总能量方程不但体现了每个时间点的数据的各自特点,而且还通过各向异性的时间维约束项根据病灶区域变化的特点约束分割结果在时间维的变化.
由变分原理,f1(x,t)和f2(x,t)的最优表达式为

根据欧拉定理,关于第t个时间点的水平集φ(t)极小化能量E可利用梯度下降法求解,第1个时间点的梯度下降流方程为

第2个时间点的梯度下降流方程为

式中:δε为Dirac函数;e1(x,t)和e2(x,t)记为
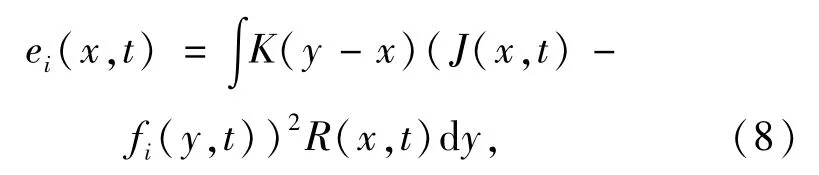
式中K为核函数.从式(6)-(7)可以看出当相邻2个时间点的图像的灰度值较接近时,D(Jt)的值近似为1,若H(φ(1))与H(φ(2))不同,时间约束项将对水平集的演化起到驱动作用,使得相邻2个时间点的水平集在图像灰度比较接近的地方保持一致(具体分析见图4).而当相邻2个时间点的图像灰度值相差较大时,D(Jt)的值近似为0,时间约束项则不起作用,这样可以保证水平集在图像灰度不同的地方按照各自的情况演化.
下面给出整个能量极小化过程.算法:4D多发性硬化损伤区域分割方法,步骤如下:①初始化水平集φ0;②更新f1(x,t),f2(x,t);③由式(8)更新e1(t),e2(t);④由式(6)和(7)更新φk+1(t);⑤判断是否收敛,若收敛停止迭代,否则转步骤②.
2 试验结果与分析
本节所有的临床数据来自ACCORD-MIND数据集,该数据集共有6组4D数据,每组数据有2个时间点.本节试验的参数选择如下:在水平集演化中,参数λ1,λ2的值为1使2个区域公平地推进曲线的演化.影响时间维约束项的约束能力的2个参数是D(Jt)中的k以及α.为了保证图像灰度相似的地方D(Jt)值较大,而图像灰度不同的地方D(Jt)值较小,文中选取k=1 000.相应地,灰度差在[0,30]范围对应的D(Jt)值的范围为[0.4,1.0],这样可以保证灰度差小于30的像素点都会受到时间维约束项的约束,从而允许预处理中的归一化存在一定的误差.影响时间约束项作用的另一个参数是α,为了使时间维约束项真正起到约束作用,其量级需与数据拟合项的量级一致,通过试验发现α的值可在[500,1 000]中选取,值越大时间维的约束能力就越强,2个时间点的分割结果就越相似.文中取α的值为500.
图3显示了4D多发性硬化损伤区域的分割方法的细节以及与3D分割方法的区别.
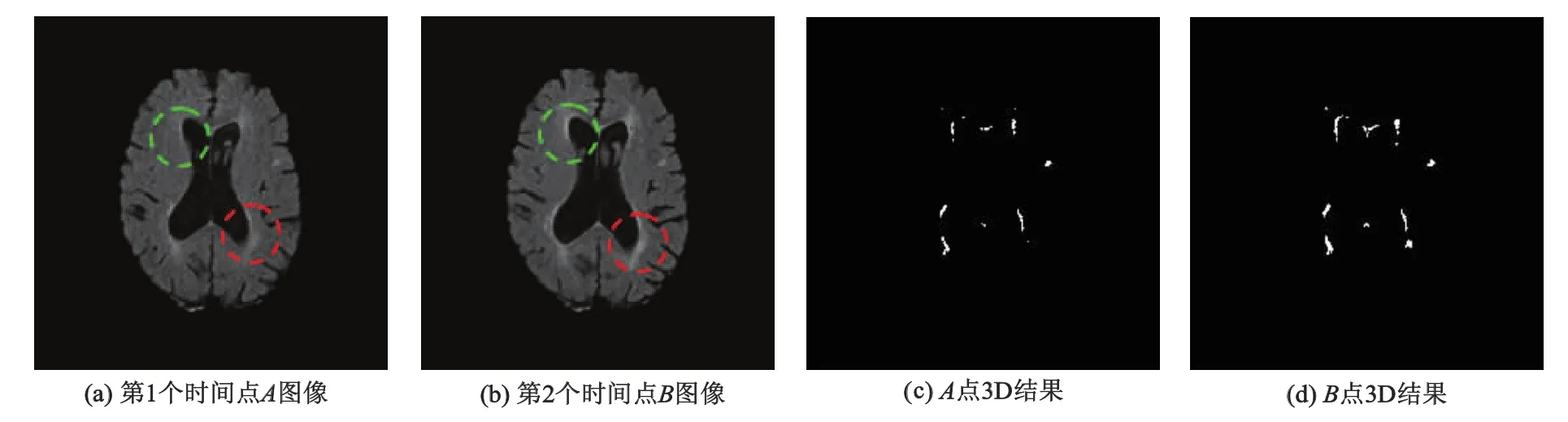

图3 4D多发性硬化损伤区域的分割细节以及与3D方法的区别
图3a,3b是2个相邻时间点的FLAIR图像.图3c,3d是3D多发性硬化损伤区域分割方法的分割结果.从图3c,3d可以看出,该方法可以检测到灰度较亮的多发性硬化损伤区域的边界,但对灰度值不是特别明显的区域(如图中红色和绿色标记处的区域处)分割结果不好.图3e,3 f分别显示了水平集在演化时式(6)中的第1项(数据拟合项)和最后一项(时间约束项)的值.可以看出图中红色以及绿色区域在数据拟合项中的值较小,因此3D分割方法得到的结果并不完整.而在时间约束项中,这2个区域的值较高.因此时间约束项起到驱动水平集向这2个区域的边界运动,从而得到更完整的分割结果.图3g,3h显示的是4D多发性硬化损伤区域分割方法得到的结果,从图3g中可见,与3D分割方法得到的结果相比,4D分割方法通过在时间维上建立了约束,可以更好地捕捉在某个时间点灰度不是特别明显的多发性硬化损伤区域.
为了定量分析和比较文中提出的4D多发性硬化损伤区域分割方法的分割效果,分别使用文中方法对仿真数据以及临床数据进行分割以检测活动区域.所有数据都有2个时间点的3D FLAIR序列图像以及T1序列图像,且都含有活动区域以及不变区域.图4是对仿真图像的试验结果.

图4 仿真图像的试验结果
图4a和4b显示了相邻2个时间点的FLAIR序列图像.其中,标记为1和2的区域的体积逐渐增加而标记为3的区域的体积并未变化.图4c-4e分别为文中提出的4D多发性硬化损伤区域分割方法的分割结果及体积变化图像.从结果中可以看出4D方法得到的结果与事实相符.这说明4D方法能较真实地体现多发性损伤区域的变化情况,减少了诊断误差.图4f-4h显示的是3D多发性硬化损伤区域分割方法得到的结果,图4e和图4h是2个时间点的分割结果的体积变化图.可以看出,由于未对时间维上的变化加约束条件,标记为3的区域的体积也存在变化,这与仿真数据不符,导致后续对体积变化的分析和统计产生误差,最终影响诊断结果.由此可见,在时间维上增加文中提出的各向异性的约束项,不但保证了2个时间点的图像中未变化区域的分割结果一致,而且也能突出存在变化的区域的分割结果符合变化的特点.
图5显示的是真实图像的3D分割方法与4D分割方法的结果比较图像.
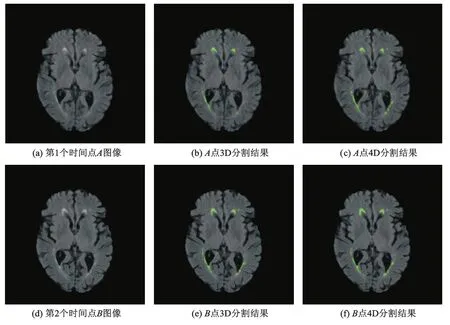
图5 3D分割方法和4D分割方法的分割结果比较
图5a和图5d显示的是2个时间点的原始图像,图5b和图5e显示的是用3D多发性硬化损伤分割方法得到的分割结果,图5c和图5 f是文中提出的4D多发性硬化损伤分割方法得到的分割结果.从图中可以看出3D多发性硬化损伤分割方法可以检测出灰度呈高亮度的病变区域,但对灰度值不高的病变区域的分割效果不是很好.而4D多发性硬化损伤分割方法因除了利用灰度信息外还增加了时间维上的信息,在自身灰度值不高但在相邻时间点上图像较相似的病变区域,时间维的约束项将起到推动曲线演化的作用,从而捕获边界.从图5c和图5 f的4D方法分割结果中可以看出文中提出的方法与3D分割方法相比具有更好的分割结果.
3 结 论
1)所提出的基于时间维信息的4D多发性硬化损伤区域的分割方法不仅适用于2个时间点的数据分割还适用于多个时间点的数据分割.
2)文中构造的时间维约束项具有各向异性的特点:在相邻时间点图像中灰度差异较大时不对纵向分割结果进行约束,而在灰度差较小的区域严格保持分割结果的连续性.
3)在临床数据上的测试结果显示了4D多发性硬化损伤区域分割方法较3D分割方法更具优越性,但4D多发性硬化损伤区域分割方法对配准和灰度归一化有很高的要求.
4)4D多发性硬化损伤区域分割是一个非常新的研究方向,目前公开发表的4D分割方法较少,尚未有成熟的软件或者源代码来进行对比试验,也没有公开数据集和分割金标准来测试文中提出的方法.以后笔者将对公开数据集进行定量分析来验证文中方法的有效性.
(
)
[1]宋余庆,王春红,陈健美,等.基于高斯混合密度模型的医学图像聚类方法[J].江苏大学学报:自然科学版,2009,30(3):293-296. Song Yuqing,Wang Chunhong,Chen Jianmei,et al. Clustering of medical image based on Gaussian mixture densitymodel[J].Journal of Jiangsu University:Natural Science Edition,2009,30(3):293-296.(in Chinese)
[2]刘 哲,宋余庆,刘雅婧.基于核模糊聚类的变分水平集医学图像分割[J].江苏大学学报:自然科学版,2014,35(6):693-698. Liu Zhe,Song Yuqing,Liu Yajing.Variational level set medical image segmentation based on kernel fuzzy clus-tering[J].Journal of Jiangsu University:Natural Science Edition,2014,35(6):693-698.(in Chinese)
[3]García-Lorenzo Daniel,Francis Simon,Narayanan Sridar,et al.Review of automatic segmentationmethods of multiple sclerosis white matter lesions on conventional magnetic resonance imaging[J].Medical Image Analysis,2013,17(1):1-18.
[4]Mortazavi D,Kouzani A Z,Soltanian-Zadeh H.Segmentation ofmultiple sclerosis lesions in MR images:a review[J].Neuroradiology,2012,54(4):299-320.
[5]Tomas-Fernandez X,Warfield SK.A new classifier feature space for an improved multiple sclerosis lesion segmentation[C]∥Proceedings of the2011 8th IEEE International Symposium on Biomedical Imaging:From Nano to Macro.Chicago:IEEE Computer Society,2011,1492-1495.
[6]Bijar A,Khanloo M M,Benavent A P,etal.Segmentation ofMS lesions using entropy-based EM algorithm and Markov random fields[J].Journal of Biomedical Science and Engineering,2011,(4):552-561.
[7]Gelineau-Morel R,Tomassini V,Jenkinson M,et al. The effectof hypointensewhitematter lesions on automated gray matter segmentation in multiple sclerosis[J]. Human Brain Mapping,2012,33(12):2802-2814.
[8]García-Lorenzo D,Prima S,Arnold D L,et al. Trimmed-likelihood estimation for focal lesions and tissue segmentation in multisequence MRI for multiple sclerosis[J].IEEE Transactions on Medical Imaging,2011,30(8):1455-1467.
[9]Steenwijk M D,Pouwels P JW,Daams M,etal.Accuratewhitematter lesion segmentation by k nearest neighbor classification with tissue type priors(kNN-TTPs)[J].NeuroImage:Clinical,2013,3:462-469.
[10]SormaniM P,Calabrese M,Signori A,et al.Modeling the distribution of new MRI cortical lesions in multiple sclerosis longitudinal studies[J].PLoS One,2011,6(10):e26712.
[11]Vrenken H,Jenkinson M,Horsfield M A,et al. Recommendations to improve imaging and analysis of brain lesion load and atrophy in longitudinal studies of multiple sclerosis[J].Journal of Neurology,2012,260(10):2458-2471.
[12]Roy S,Carass A,Shiee N,et al.Longitudinal intensity normalization in the presence of multiple sclerosis lesions[C]∥Proceedings of the 2013 IEEE 10th International Symposium on Biomedical Imaging.Piscataway:IEEE Computer Society,2013:1384-1387.
[13]Li CM,Kao C Y,Gore JC,et al.Minimization of region-scalable fitting energy for image segmentation[J]. IEEE Transactionson Image Processing,2008,17(10):1940-1949.
[14]LiChunming,Huang Rui,Ding Zhaohua,etal.A level setmethod for image segmentation in the presence of intensity inhomogeneities with application to MRI[J]. IEEE Transactions on Image Processing,2011,20(7):2007-2016.
(责任编辑 梁家峰)
4D MS lesion segmentation method based on temporal information
Zhan Tianming1,Zhan Yongzhao1,Feng Can2
(1.School of Computer Science and Communication Engineering,Jiangsu University,Zhenjiang,Jiangsu 212013,China;2.North Information Control Group Co.,Ltd.,Nanjing,Jiangsu 211153,China)
To solve the problem that 3D brain magnetic resonance(MR)image segmentation method could not accurately segment4Dmultiple sclerosis(MS)lesion,integrating the information of each time point in 4D data,a 4D MS lesion segmentation method was proposed by building a dynamic prior information from adjacent time point data to improve the level setmethod driven by the local information. The 4D MS lesion segmentation algorithm was obtained by solving the minimum of the proposed model based on the alternative iteration algorithm and the steepest descentmethod.To validate the effect of the method,the real clinical4D data were tested and compared with the results obtained by 3D segmentation method.The results show that the proposed 4D MS lesion segmentation method can introduce the temporal information constraint to obtain more accurate segmentation results and realize efficiently quantitative analysis of the following lesions.
multiple sclerosis;lesion;temporal;level set;segmentation
TP391.4
A
1671-7775(2015)05-0566-07
詹天明,詹永照,冯 灿.时间维信息的4D多发性硬化损伤区域分割方法[J].江苏大学学报:自然科学版,2015,36(5):566-572.
10.3969/j.issn.1671-7775.2015.05.013
2014-11-26
中国教育部博士点基金(博导类)资助项目(20133219110029);中国博士后基金资助项目(2014M561587);江苏省博士后基金资助项目(1402094C);江苏大学高级人才基金资助项目(14JDG041)
詹天明(1984—),男,江苏高邮人,博士(ztm@ujs.edu.cn),主要从事图像处理与分析研究.
詹永照(1962—),男,福建尤溪人,教授,博士生导师(yzzhan@ujs.edu.cn),主要从事模式识别研究.

