东方百合查尔酮异构酶基因LhCHI的克隆及表达1)
窦晓莹 郎利新 包放 孔滢 尚宏忠 白锦荣 王乃彦
(北京市辐射中心,北京,100875) (北京师范大学)
东方百合查尔酮异构酶基因LhCHI的克隆及表达1)
窦晓莹 郎利新 包放 孔滢 尚宏忠 白锦荣 王乃彦
(北京市辐射中心,北京,100875) (北京师范大学)
利用RT-PCR结合RACE的方法从东方百合‘索邦’花被片中克隆了查尔酮异构酶(CHI)基因,命名为LhCHI(GenBank登录号为KJ784468)。该基因开放阅读框702 bp,编码233个氨基酸,预测该蛋白相对分子质量25 KD,等电点(pI)为4.7。同源比对和系统进化分析表明,LhCHI基因编码的氨基酸序列具有查尔酮异构酶典型的催化活性保守位点,与百合科郁金香(Tulipafosteriana)查尔酮异构酶序列一致性为83.3%。半定量PCR和荧光实时定量PCR分析结果表明,LhCHI基因在百合的根、茎、叶片、鳞茎、开放花被片、花药以及柱头中均有表达,花器官中相对表达量较高,花发育后期的柱头、花柱、花被片等组织中LhCHI基因表达水平普遍高于花发育早期。
百合;查尔酮异构酶;LhCHI基因
The geneLhCHIencoding chalcone isomerase (CHI) involved into flavonoids synthesis was cloned from inner tepals of Oriental hybrid lily ‘Sorbonne’ (LhCHI, GenBank accession No. KJ784468). The cDNA ofLhCHIwas obtained by RT-PCR and Rapid Amplification of cDNA Ends techniques. The open reading frame ofLhCHIwas 702 bp in length, encoding a protein polypeptide of 233 amino acids with a predicted molecular weight of 25 kD and a theoreticalpI of 4.7. The deduced amino acid sequence ofLhCHIshared 83.3% identity with CHI fromTulipafosteriana, and contained typical conserved catalytic chalcone elements. Quantitative real-time PCR analyzed the expression profiles ofLhCHIin different tissues.LhCHIwas constitutively expressed in root, stem, leaf, bulb, tepal, anther, style and stigma with particularly high expression in flowers. The expression level ofLhCHIin later stage flowers is generally higher than that in the early stage flowers.
百合(Liliumspp.)花色丰富、花型硕大,是重要的商品化切花。在百合育种中,花色改良具有十分重要的意义。花青素苷(anthocyanidin)是大部分植物花器官的主要显色物质,由类黄酮代谢分支途径合成[1-2]。近年来,科学家们通过对百合类黄酮代谢途径结构类基因和调节类基因的克隆和研究,对百合花青素合成途径的分子调节机制已经有了一定的了解[3]。在结构类基因中,目前已分离出类黄酮代谢早期关键基因查尔酮合酶(CHS)[4]、黄烷酮-3-羟基化酶(F3H)、类黄酮-3’-羟基化酶(F3’H)、二氢黄酮醇-4-还原酶(DFR)、花青素合成酶(ANS)等基因[5-6]。在调节类基因中,Nakatsuka et al.[7]从亚洲百合中分离出了basic-helix-loop-helix(bHLH)型转录因子LhbHLH1和LhbHLH2,发现LhbHLH2与LhDFR的表达相关。R2R3-MYB型转录因子LhMYB12的转录水平影响着花被片中色素物质的积累水平[8],对花青素合成途径早期和晚期的相关结构类基因的转录有调节作用[6]。LhMYB12的等位基因LhMYB12-Lat控制着百合花被片斑点中色素的形成[9]。
查尔酮异构酶(chalcone isomerase,CHI)是类黄酮合成早期关键酶,广泛分布于高等植物中,和查尔酮合酶(CHS)共同调控类黄酮代谢途径[10]。很多植物中的查尔酮异构酶都已经得到分离,如矮牵牛[11]、玉米[12]、水稻[13]、水母雪莲[14]、葡萄[15]。发现查尔酮异构酶在果实发育、花器官中色素积累、叶片对抗紫外损伤等方面都发挥着重要作用[14-19]。目前,百合中类黄酮代谢途径部分结构类关键基因已得到分离和研究,但还未见百合查尔酮异构酶的基因序列及其功能研究的相关报道。
东方百合‘索邦’(Oriental hybrid lily ‘Sorbonne’)花被片主体呈粉色,其色素类物质主要为花青素苷,转录组学分析表明,‘索邦’花器官中的颜色受到类黄酮代谢途径的调节[3,20]。本试验以‘索邦’作为研究材料,利用RACE技术在其开放的内轮花被片中分离了查尔酮异构酶编码区序列,分析查尔酮异构酶基因在‘索邦’各组织中的表达模式以及在花发育过程中的表达规律,以期为开展百合花色改良的分子育种奠定基础,同时为百合花色形成机理的研究提供参考资料。
1 材料与方法
供试百合品种‘索邦’购自荷兰,栽植于北京市辐射中心种质资源圃,分别于2013年5、6、7月份采集不同发育时期的根、鳞茎、茎生叶、茎、开放花的内外轮花被片、花药、柱头等组织,液氮速冻后保存于-80 ℃备用。百合花发育分为9个时期:St.1~5期和花朵开放后的1~4期。St.1~5期基于花被片着色或花形态变化进行划分,St.1期,完全未着色的花蕾;St.2期,花被片内侧开始出现斑点;St.3期,开始着色的花蕾;St.4期,完全着色未开放的花蕾;St.5期,完全开放的花。本研究取百合花发育的St.1~5期的内外轮花被片、柱头、花柱和花药,液氮速冻后保存于-80 ℃。以上材料用于基因克隆、RT-PCR和qRT-PCR分析所需的RNA提取,其中qRT-PCR用不同批次材料重复3次。
RNA提取及cDNA合成:参照文献[21]中所用的CTAB法提取百合各组织中的总RNA,琼脂糖凝胶电泳检测所提取RNA的完整性,并用NanoDrop微量紫外分光光度计(Nanodrop Technologies Inc,Delaware,USA)检测样品纯度和浓度。
用ReverTra qPCR RT Master Mix gDNA remover(TOYOBO公司)试剂盒对RNA进行反转录,获得的cDNA进行PCR扩增或存于-20 ℃。
LhCHI基因克隆:从GenBank下载已登录的水稻(Oryzasativa,Os03g0819600)、郁金香(Tulipafosteriana,AGJ50586.1)、矮牵牛(Petunia×hybrida,P11651.1)、拟南芥(Arabidopsisthaliana,AEE79342.1)和葡萄(Vitislabrusca,ACS36662.1)的CHI同源蛋白序列进行比对,保守区设计简并引物CHI-1和CHI-2(表1),以反转录的‘索邦’花被片cDNA为模板进行PCR扩增。扩增条件为94 ℃预变性3 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min。电泳检测后回收与预期片段大小一致的条带,连接到克隆载体pEASY-Blunt Cloning Vector(TransGen Biontech公司)后转化感受态细胞(Trans1-T1 Phage Resistant Chemically Competent Cell,TransGen Biontech公司),经氨苄青霉素抗性标记筛选阳性克隆后交送Invitrogen公司进行测序。根据测序结果和SMARTer RACE cDNA Amplication说明书中引物设计原则在序列上游设计特异性引物LhCHI-GSP3-1、LhCHI-GSP3-2(表1);序列下游设计特异性引物LhCHI-GSP5-1、LhCHI-GSP5-2(表1)。按照SMARTer RACE cDNA Amplication试剂盒操作步骤进行RNA反转录以及3’、5’末端cDNA的扩增,电泳检测后回收清晰且特异的条带,连接pEASY-Blunt载体,转化感受态细胞,通过氨苄青霉素抗性标记筛选阳性克隆进行测序。将测序片段进行拼接,设计引物LhCHI-F和LhCHI-R(表1),用KOD-Plus-Neo聚合酶(TOYOBO公司)扩增LhCHI基因cDNA全长,对扩增结果进行测序。本研究中引物合成及基因测序均委托Invitrogen公司进行。
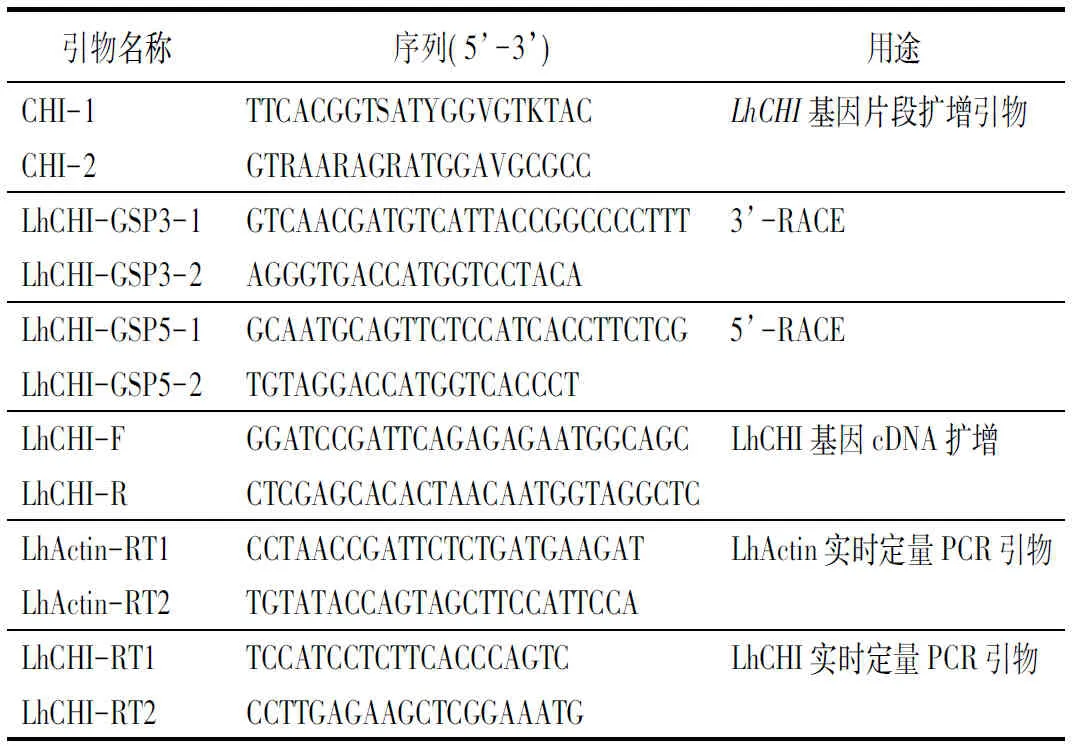
表1 PCR引物序列
LhCHI基因生物信息学分析:将获得的LhCHI基因序列在NCBI的ORF finder预测开放阅读框,并在http://blast.ncbi.nlm.nih.gov/Blast上进行序列比对和同源基因查询,应用DNAMAN 8软件对序列一致性进行分析,使用MEGA 6.0软件建立CHI蛋白系统进化树;通过http://www.cbs.dtu.dk/services/SignalP/分析蛋白质信号肽结构;通过http://www.cbs.dtu.dk/services/TMHMM/分析蛋白跨膜域。
LhCHI基因表达模式分析:以百合LhActin基因(基因登录号:AB438963)作为内参基因,设计特异性引物LhActin-RT1、LhActin-RT2(表1);根据LhCHI基因cDNA序列设计特异性引物LhCHI-RT1、LhCHI-RT2(表1)。半定量RT-PCR以‘索邦’根、茎、叶、鳞茎、花被片、花药、柱头RNA反转录的cDNA为模板,扩增体系为10 μL 2×EasyTaq®PCR Super Mix,上、下游引物各0.5 μL,cDNA 1 μL,无菌水8.5 μL;扩增程序为95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸20 s,35个循环,结果通过琼脂糖凝胶电泳进行调整和检测。qRT-PCR以‘索邦’根、茎、叶、鳞茎、花被片、花药、花柱、柱头等组织RNA反转录的cDNA为模板,参照KOD SYBR qPCR Mix(TOYOBO公司)试剂盒说明书进行操作,通过ABI7500荧光定量PCR仪检测LhCHI基因在各组织中的相对表达量,每个样品设3个重复,每轮扩增采用不同批次样品,重复3次。应用Origin 8.0和Microsoft Excel 2010软件对数据进行计算和处理,采用2-ΔΔCt法(Forkmann and Dangelmayr,1980)分析基因的相对表达情况。
2 结果与分析
2.1 LhCHI基因克隆与序列分析
以‘索邦’开放内轮花被片RNA反转录的cDNA为模版,利用简并引物CHI-1和CHI-2扩增得到一个长度为321 bp的中间片段(图1A),之后用RACE法分别获得447 bp的5’末端以及635 bp的3’末端片段(图1B),序列拼接后在NCBI的ORF finder预测开放阅读框,设计特异性引物LhCHI-F和LhCHI-R扩增编码区(图1C),得到702 bp的ORF序列,推测其编码的蛋白由233个氨基酸残基组成(图2),等电点4.7,相对分子质量25 KD。经TMHMM预测该蛋白无跨膜域,SignalP 4.1分析显示该蛋白无信号肽。将该基因命名为LhCHI(Liliumhybrid chalcone isomerase),GenBank登录号KJ784468。
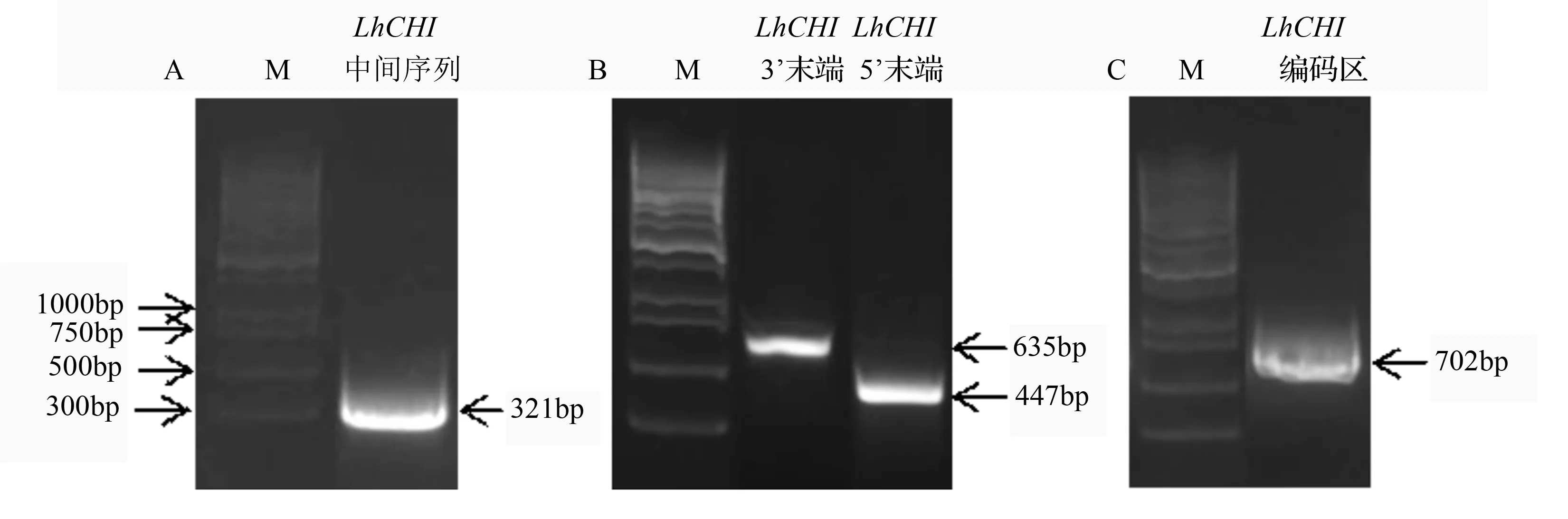
图1 LhCHI基因中间序列、3’末端、5’末端及基因编码区扩增结果

黑色方框中ATG为起始密码子,方框中TGA(*)为终止密码子。
2.2 LhCHI蛋白序列分析
通过DNAMAN软件将百合LhCHI基因编码的氨基酸序列与其他植物查尔酮异构酶序列进行比对,发现百合LhCHI氨基酸序列与同为百合科的郁金香ThCHI序列一致性最高,达到83.3%,与水稻、矮牵牛、拟南芥、葡萄中查尔酮异构酶序列相似性分别为61.8%、64.1%、63.4%和64.6%(图3),它们都具有保守的查尔酮催化结构域。在百合LhCHI氨基酸序列中,属于查尔酮异构酶催化活性保守位点氨基酸分别为Thr48、Tyr106、Asn113和Ser190(图3“*”号标记的氨基酸)。

进行序列比对的蛋白名称、所属物种及基因登陆号:LhCHI.百合(Lilium‘Sorbonne’),KJ784468;TfCHI.郁金香(Tulipafosteriana),AGJ50586.1;OsCHI.水稻(Oryzabrachyantha),Os03g0819600;PhCHI-A.矮牵牛(Petunia×hybrida),P11650.1;AtCHI.拟南芥(Arabidopsisthalianna),AT3G55120;VvCHI1葡萄(Vitisvinifera),A5ANT9.1。带*的氨基酸代表位于活性中心的保守氨基酸位点。
图3 LhCHI与其他植物CHI氨基酸序列比对图
将LhCHI与NCBI检索到的其他物种CHI蛋白进行聚类分析并构建系统发育树,结果表明(图4),LhCHI与郁金香TfCHI遗传距离最近,其次为美洲油棕及大花美人蕉。由系统进化树可以看出,单双子叶植物中的CHI蛋白在进化上分成了两个并列的分支,百合中的LhCHI蛋白与单子叶植物中的CHI蛋白亲缘关系较近。
2.3 LhCHI基因表达模式分析
通过半定量RT-PCR分析LhCHI基因在不同组织的表达情况,结果如图5所示,LhCHI基因在百合根、茎、茎生叶、鳞茎、开放花被片、花药、花柱、柱头中均有表达,但表达程度不同,在叶片、开放花被片、花柱及柱头中较高,实时定量qRT-PCR分析结果与RT-PCR结果基本一致,各组织对应的相对表达量数值分别为根中5.31、茎中1.00、叶片中54.7、鳞茎中3.91、开放的花被片中263.1、花药中4.56、花柱中146.02、柱头中186.97。说明LhCHI基因在开放花被片中表达水平最高,其次是柱头、花柱、叶片、根、花药、鳞茎和茎。

标尺代表遗传距离;各节点处数字代表从1000次重复计算得到的bootstrap值。

图5 RT-PCR分析LhCHI基因在‘索邦’不同组织中的表达模式
‘索邦’花发育的St.1~5期,内外花被片颜色、花药颜色以及柱头颜色都逐渐加深,说明色素类物质积累也在逐渐增多,文中通过qRT-PCR分析了LhCHI基因在花发育不同阶段和组织中的相对表达量。在‘索邦’花蕾的内轮花被片和外轮花被片中,LhCHI基因表达量在St.2期开始显著升高,在St.4期达到顶峰,St.5期略有下降,在St.2~5期外轮花被片LhCHI基因表达量略高于内轮花被片(表2);在花柱中,St.1~3期LhCHI基因的表达水平没有明显差异,St.4期开始升高,其相对表达量约为前3期平均值的4倍,到St.5期达到最高;在柱头中,LhCHI基因表达量从St.3期开始上升,到St.5期达到最高;在花药中,St.1~5期LhCHI基因的表达量一直处于较低的水平,并没有随着花药成熟出现明显变化(表2)。
由此可见,LhCHI基因主要在百合花器官发育后期的花被片、花柱、柱头等组织中表达,并受到生长发育时期的影响。
3 结束语
CHI是类黄酮合成途径早期的关键酶,根据催化反应底物的不同,将查尔酮异构酶分成两类,Type I和Type II型[22]。Type I型查尔酮异构酶普遍存在于大部分植物中,而Type II型查尔酮异构酶主要存在于豆科植物中,这两类CHI均具有保守的查尔酮催化结构域[10]。通过对苜蓿中II型CHI蛋白结构研究发现,在它的活性位点上有4个非常重要的保守氨基酸残基(Thr48、Tyr106、Asn113、Thr190)[23-24]。本研究通过分析LhCHI的氨基酸序列发现,LhCHI具有与苜蓿II型CHI蛋白同样的Thr48、Tyr106、Asn113保守氨基酸残基,而Thr190在豆科植物中较为保守,在百合、郁金香、水稻、拟南芥和葡萄中该位点变为Ser190。
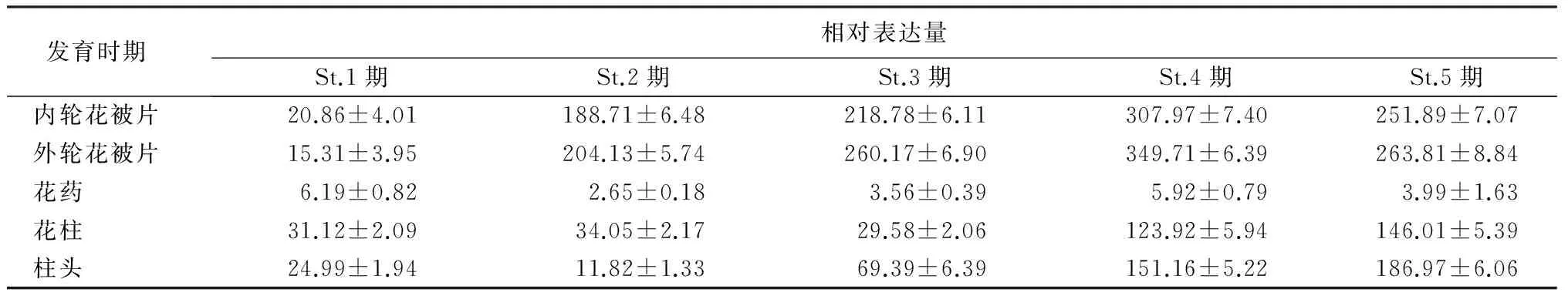
表2 LhCHI基因在St.1~5期内轮花被片、外轮花被片、花药、花柱及柱头中的相对表达量
CHI基因的cDNA序列首次通过抗血清方法从菜豆中克隆[10,25],之后通过同源克隆的方法在多种植物中分离得到了CHI基因。本试验从百合中获得LhCHI基因,推测的LhCHI蛋白与其他植物的CHI高度一致,构建系统进化树结果显示,百合LhCHI与单子叶的郁金香、美洲油棕、玉米、水仙等聚为一类,而矮牵牛、烟草、葡萄等双子叶植物CHI蛋白聚为一类。这表明单子叶植物和双子叶植物CHI蛋白聚为两大类,其中LhCHI与单子叶植物亲缘关系更近,CHI在单、双子叶之间存在明显区别,因此,推测CHI基因可能发生在单子叶与双子叶植物的进化之后。
文中通过qRT-PCR分析发现百合LhCHI基因在花被片、花柱、柱头等组织中表达水平明显高于其他组织,在花蕾未着色的St.1期已有表达,在花即将开放或完全开放时表达量达到最高。在花被片中,LhCHI基因的表达水平没有在完全开放的St.5期达到最高,而是在开放前的St.4期达到最高,推测原因可能由于LhCHI基因位于类黄酮代谢早期,在St.4期时,LhCHI基因转录水平的积累已足够推动下游反应进行,故到St.5期时转录水平开始略有下降,但并不影响花青素合成下游基因的表达,因此,开放花瓣花色素物质的积累并没有随着LhCHI基因表达水平的下降而减少。已有研究表明,CHI基因的表达水平与植物花色密切相关,如矮牵牛中CHI-A表达水平下降会使花粉由黄色变为绿色[26];在烟草中抑制CHI基因的表达会使花粉和花瓣颜色变淡[18],CHI基因与植物花色的关系可能是LhCHI基因在花器官中高表达的原因。但在St.1~5期的花药中LhCHI基因表达量较低,说明在百合中该基因可能并不主要在花药中发挥功能。在叶片中检测到LhCHI基因的表达水平高于根、茎等其他营养组织,有研究证明,日光和紫外线UV-A会诱导CHI基因的表达,百合LhCHI基因在叶片中的高表达可能是由于叶片需要合成类黄酮以抵抗紫外线辐射[10]。
花色是影响百合观赏性状的重要因素,传统杂交育种培育新花色存在着周期长和远缘杂交不亲和等弊端,基因工程技术为培育新花色品种提供了新的途径。通过基因工程改变CHI基因的表达以改变植物类黄酮物质积累的研究已有大量报道,抑制康乃馨和仙客来中CHI基因的表达会使花瓣中积累大量的查尔酮,花朵变为黄色[27-28],将洋葱中CHI基因失活,植株中高水平积累查尔酮,导致出现黄色球茎[10,29]。本试验克隆了LhCHI基因并研究了其表达模式,为继续通过转基因等手段研究该基因在百合花色形成过程中的作用奠定了基础。
[1] 朱满兰,王亮生,张会金,等.耐寒睡莲花瓣中花青素苷组成及其与花色的关系[J].植物学报,2012,47(5):437-453.
[2] Lai Yunsong, Li Huanxiu, Yamagishi M. A review of target gene specificity of flavonoid R2R3-MYB transcription factors and a discussion of factors contributing to the target gene selectivity[J]. Frontiers in Biology,2013,8(6):577-598.
[3] Zhang Mingfang, Jiang Lingmin, Zhang Dongmei, et al. De novo transcriptome characterization ofLilium‘Sorbonne’ and key enzymes related to the flavonoid biosynthesis[J]. Molecular Genetics and Genomics,2015,290:399-412.
[4] 杨丽,刘雅莉,王跃进,等.百合查尔酮合成酶(CHS)基因的克隆与分析[J].西北植物学报,2006,26(5):933-936.
[5] Yamagishi M. Oriental hybrid lily Sorbonne homologue of LhMYB12 regulates anthocyanin biosyntheses in flower tepals and tepal spots[J]. Molecular Breeding,2011,28(3):381-389.
[6] Lai YunSong, Shimoyamada Y, Nakayama M, et al. Pigment accumulation and transcription of LhMYB12 and anthocyanin biosynthesis genes during flower development in theAsiatichybridlily (Liliumspp.)[J]. Plant Science,2012,193:136-147.
[7] Nakatsuka A, Yamagishi M, Nakano M, et al. Light-induced expression of basic helix-loop-helix genes involved in anthocyanin biosynthesis in flowers and leaves ofAsiatichybridlily[J]. Scientia Horticulturae,2009,121(1):84-91.
[8] Yamagishi M, Yoshida Y, Nakayama M. The transcription factor LhMYB12 determines anthocyanin pigmentation in the tepals ofAsiatichybridlilies (Liliumspp.) and regulates pigment quantity[J]. Molecular Breeding,2012,30(2):913-925.
[9] Yamagishi M, Toda S, Tasaki K. The novel allele of the LhMYB12 gene is involved in splatter-type spot formation on the flower tepals ofAsiatichybridlilies (Liliumspp.)[J]. New Phytologist,2014,201(3):1009-1020.
[10] 张越,刘学英,王效忠,等.植物查尔酮异构酶基因(CHI)研究进展[J].基因组学与应用生物学,2011,30:1043-1050.
[11] Van Tunen AJ, Koes RE, Spelt CE, et al. Cloning of the two chalcone flavanone isomerase genes fromPetuniahybrida: coordinate, light-regulated and differential expression of flavonoid genes[J]. The EMBO Journal,1988,7(5):1257-1263.
[12] Grotewold E, Peterson T. Isolation and characterization of a maize gene encoding chalcone flavonone isomerase[J]. Molecular and General Genetics MGG,1994,242(1):1-8.
[13] Druka A, Kudrna D, Rostoks N, et al. Chalcone isomerase gene from rice (Oryzasativa) and barley (Hordeumvulgare): physical, genetic and mutation mapping[J]. Gene,2003,302(1):171-178.
[14] Li Fengxia, Jin Zhiping, Qu Wenquan, et al. Cloning of a cDNA encoding theSaussureamedusachalcone isomerase and its expression in transgenic tobacco[J]. Plant Physiology and Biochemistry,2006,44(7):455-461.
[15] 周军,姚泉洪,彭日荷,等.巨峰葡萄查尔酮异构酶基因克隆及表达分析[J].西北植物学报,2009,29(9):1723-1729.
[16] McKhann H I, Paiva N L, Dixon R A, et al. Chalcone synthase transcripts are detected in alfalfa root hairs following inoculation with wild-typeRhizobiummeliloti[J]. Molecular Plant-Microbe Interactions,1997,10(1):50-58.
[17] Muir S R, Collins G J, Robinson S, et al. Overexpression of petunia chalcone isomerase in tomato results in fruit containing increased levels of flavonols[J]. Nature Biotechnology,2001,19(5):470-474.
[18] Nishihara M, Nakatsuka T, Yamamura S. Flavonoid components and flower color change in transgenic tobacco plants by suppression of chalcone isomerase gene[J]. FEBS Letters,2005,579(27):6074-6078.
[19] 赵昶灵,郭维明,陈俊愉.植物花色形成及其调控机理[J].植物学通报,2005,22(1):70-81.
[20] Banba H. Pigments of lily flowers I. Survey of anthocyanin[J]. J Jpn Sci Hort Sci,1967,36(4):61-65.
[21] Gambino G, Perrone I, Gribaudo I. A rapid and effective method for RNA extraction from different tissues of grapevine and other woody plants[J]. Phytochemical Analysis,2008,19(6):520-525.
[22] Shimada N, Aoki T, Sato S, et al. A cluster of genes encodes the two types of chalcone isomerase involved in the biosynthesis of general flavonoids and legume-specific 5-deoxy (iso) flavonoids inLotusjaponicus[J]. Plant Physiology,2003,131(3):941-951.
[23] Jez J M, Bowman M E, Dixon R A, et al. Structure and mechanism of the evolutionarily unique plant enzyme chalcone isomerase[J]. Nature Structural & Molecular Biology,2000,7(9):786-791.
[24] Jez J M, Noel J P. Reaction mechanism of chalcone isomerase pH dependence, diffusion control, and product binding differences[J]. Journal of Biological Chemistry,2002,277(2):1361-1369.
[25] Mehdy M C, Lamb C J. Chalcone isomerase cDNA cloning and mRNA induction by fungal elicitor, wounding and infection[J]. The EMBO Journal,1987,6(6):1527.
[26] Van Tunen A J, Mur L A, Recourt K, et al. Regulation and manipulation of flavonoid gene expression in anthers of petunia: the molecular basis of the Po mutation[J]. Plant Cell,1991,3(1):39-48.
[27] Forkmann G, Dangelmayr B. Genetic control of chalcone isomerase activity in flowers ofDianthuscaryophyllus[J]. Biochemical Genetics,1980,18(5/6):519-527.
[28] Miyajima I, Maehara T, Kage T, et al. Identification of the main agent causing yellow color of yellow-flowered cyclamen mutant[J]. Journal of the Japanese Society for Horticultural Science (Japan),1991,60(2):409-414.
[29] Kim S, Jones R, Yoo K S, et al. Gold color in onions (Alliumcepa): a natural mutation of the chalcone isomerase gene resulting in a premature stop codon[J]. Mol Genet Genomics,2004,272(4):411-419.
Cloning and Expression Analysis of Chalcone Isomerase GeneLhCHIin Oriental Hybrid Lily (Liliumspp.)//
Dou Xiaoying, Lang Lixin, Bao Fang, Kong Ying, Shang Hongzhong, Bai Jinrong
(Beijing Radiation Center, Beijing 100875, P. R. China); Wang Naiyan(Beijing Normal University)//Journal of Northeast Forestry University,2015,43(9):6-11,17.
Lilium; Chalcone isomerase;LhCHI
窦晓莹,女,1983年4月生,北京市辐射中心,助理研究员。E-mail:douxiaoying@126.com。
白锦荣,北京市辐射中心,副研究员。E-mail:bjr301@126.com。
2015年4月22日。
Q946.2;Q949.71+8.23
1)“十二五”农村领域国家科技计划课题(2013BAD01B0706)、北京市科学技术研究院创新团队计划(IG201404N)、北京市科学技术研究院青年骨干计划(201524)。
责任编辑:任 俐。

