信号放大策略在电化学生物传感器中的应用
陈颖
(西南大学化学化工学院,重庆400715)
信号放大策略在电化学生物传感器中的应用
陈颖
(西南大学化学化工学院,重庆400715)
近年来,快速、简单、灵敏的生物分子检测在临床诊断、食品分析、生物恐怖主义的防御和环境监测等方面变得日益重要。电化学生物传感器由于具有简单、灵敏、成本低并可广泛运用于不同领域的固有优势而受到越来越多的关注。为了实现高灵敏的生物检测,不同信号放大方法被用于传感器的构建中。该文简单介绍了电化学生物传感器的基本原理,并重点概括了广泛运用于电化学生物传感器中的信号放大策略。
电化学;生物检测;信号放大策略
0 引言
电化学生物传感器是将生物分子作为目标物识别敏感元件固定于电极表面,生物分子间的特异性识别作用通过电极进行信号转换,以电阻、电位、电流或电容等物理量形式作为特征检测信号输出,从而完成对目标物的定性或者定量分析任务的一类装置。依据传感器输出特征物理信号所产生方式的不同,电化学生物传感器一般可归为生物催化型和亲和型传感器两个大类:前者感受器部位元件采用能特异性识别目标分子的酶、细胞或组织等并催化底物生成电活性物质;后者则是基于生物识别元件与目标分子间的特异性结合作用,例如抗原-抗体、适体-目标分子、生物素-亲和素之间及DNA碱基互补配对等相互作用。电化学生物传感器性能的好坏可以通过传感器的动力学线性范围、检测限、重现性、选择性及响应时间等参数来评估。
1 信号放大策略在电化学生物传感器中的应用
临床诊断中对痕量及超痕量生物分子检测的要求使得科研工作者致力于提高传感器灵敏度的研究,随着纳米技术的飞速发展以及多种生物技术的兴起,学科间进一步交叉渗透融合,各种信号放大方法相继出现并成功运用于生物分析,电化学生物传感器的灵敏度不断提高,甚至达到单分子检测水平。下面就各类近年来广泛运用的信号放大策略分别作简要介绍。
1.1 酶催化放大技术
酶(enzyme)是一类由生命体产生的具有生理调节功能的蛋白质,参与机体代谢及各种转化反应,特定的酶只对特定底物发生高效的催化作用,酶的这种专一性在一定程度上确保了传感器的选择性,其高效性也对灵敏度有所贡献。在生物分析中,通常将酶作为标记物间接指示目标分子浓度,常用标记酶有碱性磷酸酯酶(ALP)、辣根过氧化物酶(HRP)及葡萄糖氧化酶(GOD)等。此外,酶催化法一般要与纳米材料放大技术联用,借助其大比表面积实现酶的大量固载,并通过其优良的导电性促进电子传递,进一步提高酶对底物的转化率,以达到增强灵敏度的目的。根据酶的催化作用所产生变化的区别,酶催化放大信号主要通过以下两种途径:酶催化产物沉积法和多酶催化底物循环增强法。
酶催化产物沉积法是指酶催化形成不溶于水的有机物附着于电极表面,使得传感界面性质发生改变,通过电化学阻抗谱、循环伏安法、计时电位法等技术进行检测的一类分析方法。Akter等[1]使用碳纳米管固载HRP作为二抗标记物,一抗固定在碳纳米管/纳米金复合材料表面,通过夹心免疫法捕获目标抗原继而结合二抗,二抗标记物HRP催化底物4-氯-1萘酚生成难溶性沉淀附着于电极表面,阻碍电子传输,电流信号减小,实现对前列腺抗原PSA的高灵敏分析,并成功运用于人血清实际样品的检测(图1)。Wang等[2]同样利用HRP对底物的催化作用,由于底物转化与过氧化氢浓度相关,通过生成不同量有机产物对CdS量子点的电致化学发光淬灭作用的差别构建了过氧化氢传感器。

图1 基于多酶催化的PSA免疫传感器原理示意图[1]Fig.1 Schematic illustrations of the multiple-HRP strategy-based PSA immunosensor[1]
将酶作为信号标记物,通过检测酶在催化反应中产生的催化电流也可指示目标分子的浓度。Caruana等[3]在微电极上电沉积一层高聚物充当电子媒介并固定捕获探针,与标记了HRP的目标序列互补杂交后,检测HRP对H2O2的电化学还原催化电流即可实现对特异性DNA的分析。研究表明通过多种酶催化电活性物质的氧化还原循环体系可以有效增强电流响应信号[4-6],也就是之前提到的多酶催化底物循环增强法,该方法可以显著提高传感器的灵敏度。Xiang等[7]通过ALP和辅酶对底物的催化产生增强的氧化还原循环电流信号构建凝血酶传感器(图2)。
1.2 纳米材料放大技术
酶放大技术受到酶活性等因素影响,一般稳定性差且成本高,因此研究不同类型的功能化纳米材料(如金属纳米材料、量子点、碳纳米材料、磁性纳米材料、高分子聚合物等)在生物传感器中的运用近年来备受关注。由于其优良的生物相容性、高的比表面积、良好的化学稳定性、催化性能及导电性等,纳米材料能大大提高传感器的分析性能,有效放大检测信号,并具有稳定识别探针或生物传感界面的作用。
纳米材料可以作为电极材料使用,在通过不同分析原理构建高性能电化学传感平台以检测目标分子中发挥着重要的作用。Yang等[8]通过一步电化学法合成了石墨烯/聚黄尿酸纳米复合材料作为DNA捕获探针固载材料,用电化学阻抗法实现了高灵敏DNA检测。他们还合成了磺化聚苯胺/石墨烯复合材料,作为一种新型电极材料,通过其直接电化学达到检测DNA[9]的目的。与此类似的策略也报道用于构建基于水溶性电活性染料偶氮红功能化石墨烯的免标记电化学DNA生物传感器[10]。此外,其他材料,例如汞膜/碳纳米管[11]或生物兼容性好的纳米结构的氧化镁/壳聚糖膜[12]等也用作先进电极材料构建高性能电化学生物传感器。
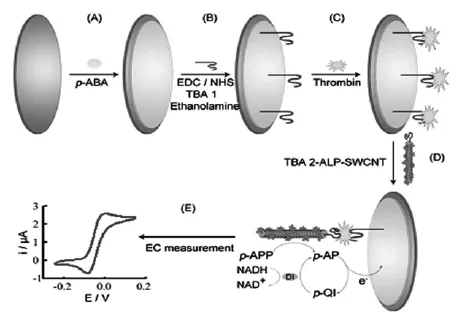
图2 双重信号放大凝血酶传感器原理示意图[7]Fig.2 Illustration of the dual amplifed protocol for ultrasensitive thrombin detection[7]

图3 协同放大信号的DNA传感器原理示意图[13]Fig.3 Schematic illustrations of the cooperative amplifcation-based genosensor[13]
以金纳米颗粒和碳纳米材料为代表的各种纳米材料作为大量酶、寡核苷酸及氧化还原探针等信号分子的优良载体广泛使用。Qiu等[13]采用纳米金为信号分子载体,发夹形DNA为捕获探针,该探针茎部含有限制性内切酶(EcoRI)特异性识别位点。如图3所示,由于EcoRI酶的高效高保真性,只有在目标物不存在时才会在发夹形捕获探针茎部的识别位点发生催化剪切反应,降低背景信号。目标物存在时可以通过杂交打开发夹形捕获探针,引起剪切位点变形,迫使生物素标记远离电极表面,通过生物素-链霉亲和素特异性结合作用捕获修饰了大量二茂铁(Fc)信号探针的纳米金颗粒以产生信号。此外,Fc可以通过与经EcoRI酶处理过的捕获探针残基杂交拉近与电极表面的距离,促进界面电子传递,进一步增强信号。该传感器实现了检测限低至zeptomole级的超灵敏分析,且具有7个数量级的宽动态范围。Wang等[14]在纳米金表面修饰了两种DNA信号探针:一种与目标序列互补,另一种非互补,该非互补探针的存在可以降低目标DNA和修饰于纳米金上的互补探针之间的交叉反应,从而提高该夹心DNA传感器的灵敏度。
新近发展的一些纳米材料可通过自身大量的信号物质直接用作电活性标记物,结合有效测定纳米标记物的方法,可以轻松构建超灵敏的电化学生物分析策略。较为典型的代表是量子点,这种准零维纳米材料具有优越的光学和电化学特性,可直接检测其荧光或电致化学发光信号,也可通过酸溶解后检测其金属离子的伏安峰,因此被广泛运用于光学和电化学生物分析中。早期的研究工作中,一般将量子点作为单个信号标记物直接修饰生物分子[15-17], 灵敏度有限,而后发展的使用碳纳米管、石墨烯等载体原位生成或共价键合量子点的方法[18-20]可以有效降低ECL发光电位且提高信号分子固载量,该复合物在传感器构建过程中充当电极材料或信号标记物显著降低了分析检测限。Jie等[21]合成了一种树枝状的量子点纳米簇,借助碳纳米管/纳米金的协同放大作用和金磁纳米易于分离富集的特点,通过夹心模式构建传感器可显著放大ECL和电化学信号,分别实现了对DNA的ECL分析和癌细胞的电化学分析(图4)。
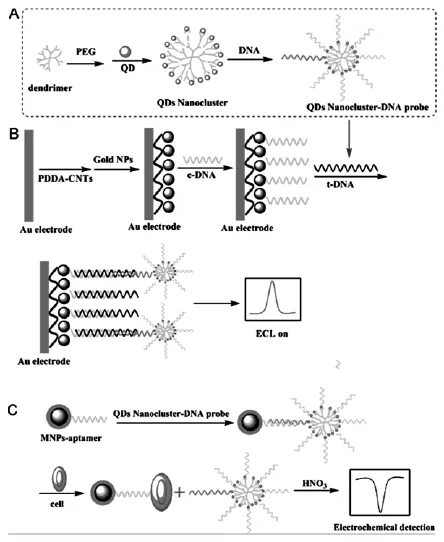
图4 (A)树枝状QDs纳米簇-DNA复合物的制备,(B)ECL DNA传感器构建原理示意图,和(C)基于金磁纳米的电化学癌细胞传感器原理示意图[21]Fig.4 (A)Preparation of the dendrimer nanoclusters/QDs-DNA probe,(B)fabrication of the ECL biosensor for DNA detection,and(C)electrochemical detection of cancer cells based on the Fe3O4@Au-aptamer[21]
使用具有酶催化活性的纳米材料也是电化学生物传感器中放大信号的新方法。Zhang等[22]构建了一个基于纳米金自催化生长为导电桥梁放大检测DNA杂交的高灵敏带隙-电化学生物传感器。如图5所示,目标DNA存在时,与捕获探针杂交形成dsDNA,由于静电作用不能吸附到纳米金表面,此时的纳米金具有葡萄糖氧化模拟酶的催化活性,纳米金在葡萄糖和氯金酸底液中经自催化作用增大半径尺寸,致使纳米金最终生长形成金膜,导电性显著提高。相反,没有目标物存在时,单链DNA捕获探针通过核苷酸碱基-金相互作用吸附于纳米金表面,使其催化活性完全钝化。

图5 基于纳米金自催化生长作为导电桥梁的带隙-电化学DNA传感器原理示意图[22]Fig.5 Schematic diagram of the gap-electrical biosensor strategy based on self-catalytic growth of unmodifed AuNPs as conductive bridges for DNA hybridization detection[22]
归纳而言,纳米材料主要通过四种方式实现对目标分子的放大分析:(1)利用纳米材料构建电极传感界面具有的优越导电性及生物相容性,降低电化学反应能垒,增强响应信号;(2)利用纳米材料的大比表面积增加电活性物质、催化活性酶或仿酶(模拟酶)的固载量,从而放大响应信号;(3)利用纳米材料自身产生的特征峰电流或电致化学发光信号实现对目标物的放大检测;(4)利用纳米材料自身催化活性及导电性促进电子传递的速率,进而增强检测信号。
1.3 DNA等温扩增放大技术
聚合酶链式反应 (Polymerase Chain Reaction, PCR)是一种高效率的体外核酸扩增技术,具有快速、灵敏和特异性高等特点,广泛应用于生物学领域,但由于需要昂贵的仪器设备提供温度循环以及专业的技术人员,因此检测成本较高。随着分子生物学和生物化学的迅猛发展,一些在等温条件下的新型核酸扩增技术相继出现,该类技术的关键是不需要进行变温操作,在恒温条件下即可实现DNA扩增,在一定程度上克服了PCR的缺点。DNA等温扩增技术的典型代表是发明于1998 年 的 滚 环 扩 增 技 术 (rolling circle amplification,RCA),该技术以病原生物体滚环复制为模型,实现了恒温条件下以环状DNA为模板短单链DNA为引物的核酸快速复制,产生一段含有大量重复序列单元的长单链DNA,该重复单元与模板DNA互补。由于RCA具有操作简单、快速、高效、灵敏度高、特异性高和可定量分析等优点已经广泛用于临床和微生物分子诊断领域。Cheng等利用RCA的信号放大作用,采用夹心免疫模式构建了血管内皮生长因子(VEGF)传感器[23],二抗上标记生物素,通过亲和素结合生物素标记的引物,在模板DNA和原料脱氧核苷三磷酸(dNTP)存在下发生RCA反应产生长单链,继而与大量修饰互补短DNA链的量子点结合,最后检测量子点的溶出峰实现对VEGF的高灵敏分析(图6)。
2000年,Notomi等建立了环介导等温扩增法(loop-mediated isothermal amplification,LAMP),该方法使用一种具有自动链置换活性的Bst DNA聚合酶,通过两条特异的外部引物和两条特异的内部引物,在等温(60~65℃)的条件下进行靶序列特异性扩增,且具有特异、灵敏、快速、简便等特点[24]。一般来说,通过比较LAMP扩增反应中产生的白色焦磷酸镁沉淀的混浊度,或加入染料可实现对初始靶序列的定量分析[25]。最近,LAMP扩增反应产物也可用电化学方法检测,实现对生化分子的检测。Sun等[26]在离子液体修饰的基底电极上固定特异性单链捕获探针构造了一个电化学DNA生物传感器,使用电化学杂交指示剂亚甲基蓝 (MB)检测基于杂交的LAMP扩增子。Nakamura等[27]发展了一种使用染料 Hoechst 33258作为杂交指示剂检测LAMP产物的电化学DNA芯片传感器。利用适体和相应目标分子的关系转换可以建立基于LAMP放大的非DNA分析平台。Xie等[28]报道了LAMP放大的电化学方法用于赫曲霉毒素A(OTA)的检测。如图7所示,首先将OTA适体的互补链自组装于纳米金修饰玻碳电极表面,己硫醇封闭后与OTA适体杂交形成部分互补的DNA双链体,没有目标物时,OTA适体可以进行LAMP扩增反应生成大量双链结合反应底液中的MB,致使游离MB减少,DPV检测信号较小;当目标物OTA出现时,它会与其适体形成络合物从而将适体从电极表面带走,LAMP反应的引物减少,扩增产物减少,反应液中游离的MB相应增加,DPV检测信号增大,以此进行定量分析,实验表明该传感器对OTA检测限为0.3 pmol/L。
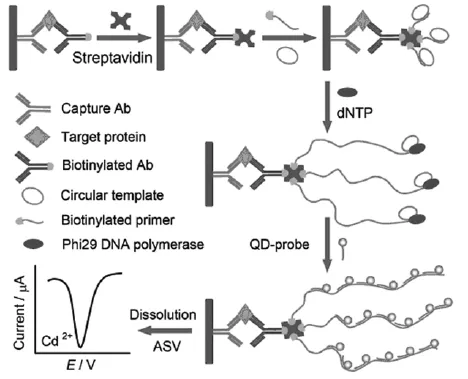
图6 基于RCA放大的VEGF传感器原理示意图[23]Fig.6 Schematic representation of the VEGF sensor with RCA for amplification[23]
2006年,Willner课题组提出了一种内切酶放大的链置换 DNA 等 温扩增 法(strand displacement amplification,SDA)[29-30]。该方法依赖于目标物沿着模板链聚合形成核酸内切酶可识别位点,经内切酶切割产生3′-OH缺口,继而再次被聚合酶识别沿着3′端到5′端方向聚合,置换出之前的扩增产物并再次形成可识别切割位点,如此进行“切刻-聚合-置换”循环反应,游离出大量聚合剪切的单链DNA产物,从而实现DNA扩增。该方法具有扩增效率高且操作简单的优点,具有生化分析方面的应用潜力[31-32]。Yang等[33]结合SDA和杂交链式反应 (hybridization chain reaction,HCR)的双重信号放大能力设计了一种电化学检测miRNA的方法。如图8所示,目标miRNA存在时,可以沿着模板DNA序列聚合形成限制性内切酶识别位点和扩增产物,被内切酶切割后再次形成聚合酶识别缺口并聚合置换出之前形成的扩增产物,如此在聚合酶和内切酶交替作用下产生大量扩增DNA产物,该扩增产物作为HCR的引发剂促使形成大量含环状富C序列的双链DNA,并以此为DNA模版原位生成银纳米簇产生电化学信号。

图7 LAMP放大的电化学方法用于OTA检测原理示意图[28]Fig.7 Schematic illustration of OTA detection principle using electrochemical method based on LAMP[28]

图8 基于SDR和HCR放大的电化学方法用于miRNA检测原理示意图[33]Fig.8 Schematic illustration of miRNA detection principle based on SDR and HCR Amplifcations[33]
1.4 DNA自组装放大技术
自组装是指基本结构单元,如分子、纳米粒子、生物大分子或微米尺度物质,在一定条件下依赖分子间非共价作用力(氢键、范德华力、静电力、疏水作用力、π-π堆积作用等)自发地形成一种稳定有序结构的过程。DNA自组装是一种严格遵循碱基互补配对原则的自下而上(bottom-up)的分子自发组装技术,区别于活细胞中DNA作为遗传信息载体的功能,此处的DNA充当构建纳米结构材料基元的角色。通过对碱基序列进行合理设计,可以实现DNA纳米结构的精密控制,并作为信号放大手段运用于电化学生物传感器。Pierce等[34]提出的杂交链式反应(hybridization chain reaction,HCR),其实质是一种链置换驱动的DNA自组装,该反应在等温无酶条件下进行,通过一段单链寡核苷酸引发两条分别含有粘性末端的发夹形单体交叠杂交组装成一段延展的带有缺口的双链DNA。HCR组装特异性高,相当于DNA扩增,但却不需要聚合酶等酶的参与,操作简单。此外,产物易于与双链指示剂结合或设计带有功能化侧链的双链产物而达到放大检测信号的目的,目前相关研究非常热门。

图9 基于四面体DNA纳米结构和HCR放大的电化学DNA检测原理示意图[35]Fig.9 Schematic illustration of electrochemical DNA detection based on tetrahedral DNA nanostructure and HCR amplification[35]
Ge等[35]展示了一个结合HCR放大作用和四面体DNA纳米结构探针的超灵敏miRNA检测平台。在该方案中,三维四面体DNA纳米结构作为固定捕获探针的支架更容易接近目标miRNA而增加反应性,由于发夹形单体修饰了生物素,通过HCR形成的触须状DNA纳米线便含有大量生物素,可以捕获大量亲和素修饰的催化酶,从而有效放大检测信号(图9)。实验表明,四面体DNA纳米结构和HCR产物的协同效应可以大大提高检测DNA和miRNA的灵敏度。事实上,大多数报道的电化学生物传感器都是与其他放大技术联用构建的。Zhuang等[36]结合HCR放大与银纳米探针采用电化学方法实现了高灵敏的DNA检测。由于目标物引发发夹形单体自组装形成长的DNA纳米结构,使得大量银纳米探针不需要银增强底物和生物活性的酶就可以直接固定其上,在施加的电位下纳米银产生强电化学信号从而进行定量分析(图10)。在优化的实验条件下,目标传感器对人类免疫缺陷病毒(HIV)特异性DNA序列的检测表现出良好的电化学性能,检测限低至0.5 fmol/L。通过单链DNA间的交叠组装也可以形成类似于HCR产物的超夹心DNA纳米结构。Xia等[37]采用MB修饰DNA探针分子与目标物进行级联杂交形成超夹心结构实现信号放大,报道了一种电化学高灵敏DNA检测法(图11)。Yin等[38]也通过超夹心DNA组装放大方法,向生成的长双链DNA中嵌入双链指示剂MB,分别实现了对DNA和三磷酸腺苷(ATP)的检测。

图10 基于HCR放大和银纳米探针的电化学DNA检测原理示意图[36]Fig.10 Schematic illustration of electrochemical DNA detection principle based on HCR amplification and silver nanotags[36]

图11 电化学超夹心DNA检测原理示意图[37]Fig.11 Schematic illustration of the electrochemical DNA supersandwich assay[37]
1.5 目标物循环放大技术
目标物循环一般是利用核酸切酶的剪切作用,将被捕获的目标物释放游离出来,并参与和未作用DNA探针的下一轮结合-剪切过程,如此循环,使得单个目标分子不断重复利用,即目标分子与DNA探针按照1:n比例反应,达到n倍放大检测信号的效果,是近几年来发展迅速的一种信号放大技术,在高灵敏生物分析中广泛使用[39-42]。根据对DNA底物作用方式和切割位置的不同,核酸切酶主要分为限制性内切酶及核酸外切酶。
限制性内切酶是可以识别特异的碱基序列并在识别位点或其周围进行切割的一类酶,可分为识别具回文序列的双链DNA并对两条DNA均剪切的一类,和识别特异性双链但只剪切其中一条单链的一类。Chen等[43]将包含一段富G序列的DNA捕获探针通过金巯键组装于电极表面,目标DNA与其杂交产生后限制性内切酶识别位点,在限制性内切酶催化剪切作用下释放出目标DNA,游离出的目标物又能参与下一个杂交-剪切过程,如此循环,使得电极表面固定的捕获探针大量转化为仅保留富G序列的短DNA链,易于与卟啉铁(hemin)结合形成hemin/G四倍体DNA酶,可以催化过氧化氢介导的四甲基联苯胺的氧化,从而产生增强的催化电流信号,达到检测目的(图12)。Zhou等[44]也利用限制性内切酶辅助目标DNA循环在电极表面形成大量hemin/G四倍体DNA酶,该DNA酶通过催化H2O2的还原竞争共反应试剂,从而淬灭电极材料石墨烯/量子点复合物的ECL发射,实现了DNA的高灵敏分析。限制性内切酶能否发挥作用依赖于特异性识别位点的形成,这要求目标DNA具有相应的特殊序列,限制了其适用范围,为了解决这一问题,可向体系中引入一条辅助探针,通过捕获探针、辅助探针及目标DNA间的相互杂交形成Y型结构[45-46],酶切位点位于Y结构上的辅助探针/捕获探针双链分枝,从而扩大可检测DNA对象,具有普适性。Bai等[47]结合纳米材料与酶辅助目标物循环放大技术的优点,设计了一种电化学适体传感器用于内毒素(LSP)的超灵敏检测。如图13所示,首先通过LPS结合固定在金磁纳米表面的适体,置换出之前杂交好的互补链DNA1作为间接目标物检测,释放的DNA1和辅助探针共同被发夹形捕获探针捕获形成Y型结构和识别位点,在限制性内切酶作用下促使DNA1循环利用产生大量被剪短的能与DNA2杂交的DNA片段,通过DNA2上修饰的甲苯胺蓝-石墨烯(Tb-Gra)复合物产生增强的电化学检测信号。当LPS不存在时,电极表面组装的捕获探针呈发夹形闭合构型,不能与DNA2杂交,因此背景信号较低。结果表明,该传感器对LPS检测限为8.7 fg/mL,远远低于之前文献报道。
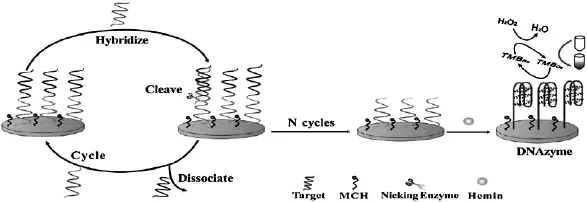
图12 基于酶辅助目标物循环电化学DNA检测原理示意图[43]Fig.12 Schematic illustration of the electrochemical DNA assay based on nuclease-assisted target recycling[43]

图13 电化学适体传感器原理示意图[47]Fig.13 Schematic illustration of the electrochemical aptasensor[47]
核酸外切酶是指能够从分子链的末端顺次水解磷酸二酯键生成单核苷酸的酶,作用底物可以是双链DNA也可以是单链DNA,作用方向可以沿3′→5′端,也可以沿5′→3′端。由于该类酶不要求特殊的目标碱基序列,因此在基于酶辅助目标物循环检测方案的构建上具有较强的灵活性,常用的有核酸外切酶III(Exo III)、外切酶I(Exo I)和λ外切酶(λ Exo)等[48-51]。Wu等[52]设计了一种基于Exo III辅助目标物循环的电化学免标记法用于检测DNA。传感界面的构建是通过自组装巯基DNA捕获探针和巯基己醇混合单分子膜制备的,当没有目标DNA时,通过电极表面DNA的负电性磷酸骨架吸附大量正电性电活性分子六氨合钌(RuHex)产生较强的电化学还原峰,当有目标物时,可以与DNA捕获探针杂交形成平末端的双链DNA结构,Exo III沿3′→5′方向逐步催化去除捕获探针的单核苷酸而释放出目标DNA,释放出的DNA可与其他未被剪切的捕获探针杂交致使捕获探针的催化降解,如此循环,单个目标分子就能导致n个捕获探针被催化剪切,减少RuHex的静电吸附量,电化学信号相应减小并以此为定量依据(图14)。

图14 通过Exo III辅助目标物循环的免标记电化学DNA传感器原理示意图[52]Fig.14 Schematic illustration of the label-free electrochemical DNA sensor via Exo III-aided target DNA recycling for sequence-specifc detection of DNA[52]
此外,通过聚合酶辅助引物扩增的链置换反应也能实现目标物的循环。该方法的基本原理是:首先将DNA模板设计成发夹形构象以钝化其与引物DNA的结合位点,通过适体-目标物特异性结合或者DNA杂交打开发夹形DNA模板,裸露出可结合引物位点,与引物DNA杂交后在聚合酶和脱氧核苷三磷酸 (dNTP)作用下进行DNA扩增反应,产生延展的DNA产物能置换出之前与模板DNA结合的目标物,被释放的目标物可以再次结合钝化的DNA模板,发生下一轮聚合-置换反应,以此达到目标物重复利用的目的[53]。Cheng等[54]将包含VEGF适体的发夹形捕获探针组装于金电极表面,目标物VEGF与其结合打开发夹结构暴露出引物DNA的互补序列继而与引物杂交,在聚合酶和dNTP作用下以捕获探针为模板发生引物扩增反应,形成与捕获探针完全互补序列置换出VEGF,释放的目标物参与下一个循环的聚合-置换反应,最后使得电极表面的发夹形捕获探针与引物扩增产物形成完全互补双链体结构,该引物DNA上标记有生物素,可结合亲和素修饰的磷酸酶(ST-AP),产生增强的酶催化信号(图15)。Zhang等[55]通过目标DNA打开发夹形捕获探针诱导循环的引物扩增反应,产生大量扩增产物与发夹形捕获探针形成双链杂交结构,使原本靠近电极表面的光敏剂远离电极,光电流信号减小从而实现了目标DNA的灵敏分析(图16)。

图15 基于循环的目标物诱导引物扩增的电化学适体传感器原理示意图[54]Fig.15 Schematic presence of electrochemical aptasensor based on cyclic target-induced primer extension[54]

图16 基于等温链置换反应的光电化学DNA传感器原理示意图[55]Fig.16 Principle of the photoelectrochemical DNA biosensor based on isothermal circular strand-displacement polymerization reaction[55]
[1]Akter R,Rahman M A,Rhee C K.Amplified electrochemical detection of a cancer biomarker by enhanced precipitation using horseradish peroxidase attached on carbon nanotubes[J].Analytical Chemistry,2012,84 (15):6407-6415.
[2]Wang J,Zhao W W,Tian C Y,et al.Highly efficient quenching ofelectrochemiluminescence from CdS nanocrystal film based on biocatalytic deposition[J].Talanta,2012,89:422-426.
[3]Caruana D J,Adam H.Enzyme-amplified amperometricdetection of hybridization and of a single base pair mutation in an 18-base oligonucleotide on a 7-μm-diameter microelectrode[J].Journal of the American Chemical Society,1999,121(4):769-774.
[4]Kwon S J,Yang H,Jo K,et al.An electrochemical immunosensor using p-aminophenol redox cycling by NADH on a self-assembled monolayer and ferrocenemodified Au electrodes[J].Analyst,2008,133(11): 1599-1604.
[5]Limoges B,Marchal D,Mavré F,et al.High amplification rates from the association of two enzymes confined within a nanometric layer immobilized on an electrode:modeling and illustrating example[J].Journal of the American Chemical Society,2006,128(18):6014-6015.
[6]Limoges B,Marchal D,Mavré F,et al.Theory and practice of enzyme bioaffinity electrodes.Chemical,enzymatic,and electrochemical amplification of in situ product detection[J].Journal of the American Chemical Society, 2008,130(23):7276-7285.
[7]Xiang Y,Zhang Y Y,Qian X Q,et al.Ultrasensitive aptamer-based protein detection via a dual amplifed biocatalytic strategy [J].Biosensors and Bioelectronics, 2010,25(11):2539-2542.
[8]Yang T,Li Q,Meng L,et al.Synchronous electrosynthesis of poly(xanthurenic acid)-reduced graphene oxide nanocomposite for highly sensitive impedimetric detection of DNA[J].ACS Applied Materials Interfaces,2013, 5(9):3495-3499.
[9]Yang T,Meng L,Wang X,et al.Direct electrochemical DNA detection originated from the self-Redox signal of sulfonated polyaniline enhanced by graphene oxide in neutral solution[J].ACS Applied Materials Interfaces, 2013,5(21):10889-10894.
[10]Guo Y X,Guo Y J,Dong C.Ultrasensitive and label-free electrochemical DNA biosensor based on water-soluble electroactive dye azophloxine-functionalized graphene nanosheets[J].Electrochimica Acta,2013,113:69-76.
[11]Serpi C,Voulgaropoulos A,Girousi S.Use of mercury film glassy carbon electrode modified with multiwalled carbon nanotubes in electrochemical analysis of DNA[J]. Electroanalysis,2013,25(5):1256-1262.
[12]Patel M K,Ali M A,Zafaryab M,et al.Biocompatible nanostructured magnesium oxide-chitosan platform for genosensing application[J].Biosensors and Bioelectronics,2013,45:181-188.
[13]Qiu L,Qiu L,Wu Z S,et al.Cooperative amplificationbased electrochemical sensor for the zeptomole detection of nucleic acids[J].Analytical Chemistry,2013,85(17): 8225-8231.
[14]Wang Z J,Zhang J,Zhu C F,et al.Amplified detection of femtomolar DNA based on a one-to-few recognition reaction between DNA-Au conjugate and target DNA[J]. Nanoscale,2014,6:3110-3115.
[15]Huang H P,Zhu J J.DNA aptamer-based QDs electrochemiluminescence biosensor for the detection of thrombin[J].Biosensors and Bioelectronics,2009,25(4): 927-930.
[16]Huang H P,Li J J,Tan Y L,et al.Quantum dot-based DNA hybridization by electrochemiluminescence and anodic stripping voltammetry[J].Analyst,2010,135(7): 1773-1778.
[17]Huang H P,Jie G F,Cui R J,et al.DNA aptamer-based detection of lysozyme by an electrochemiluminescence assay coupled to quantum dots[J].Electrochemistry Communications,2009,11(4):816-818.
[18]Cheng L X,Liu X,Lei J P,et al.Low-Potential Electrochemiluminescent sensing based on surface unpassivation of CdTe quantum dots and competition of analyte cation to stabilizer[J].Analytical Chemistry,2010,82(8): 3359-3364.
[19]Wang T,Zhang S Y,Mao C J,et al.Enhanced electrochemiluminescence of CdSe quantum dots composited with graphene oxide and chitosan for sensitive sensor[J]. Biosensors and Bioelectronics,2012,31(1):369-375.
[20]Tian C Y,Zhao W W,Wang J,et al.Amplified quenching of electrochemiluminescence from CdS sensitized TiO2nanotubes by CdTe-carbon nanotube composite for detection of prostate protein antigen in serum[J].Analyst, 2012,137(13),3070-3075.
[21]Jie G F,Zhang J,Jie G X,et al.A novel quantum dot nanocluster as versatile probe for electrochemiluminescence and electrochemical assays of DNA and cancer cells[J].Biosensors and Bioelectronics,2014,52:69-75.
[22]Zhang J,Nie H,Wu Z,et al.Self-catalytic growth of unmodified gold nanoparticles as conductive bridges mediated gap-electrical signal transduction for DNA hybridization detection[J].Analytical Chemistry,2014,86 (2):1178-1185.
[23]Cheng W,Yan F,Ding L,et al.Cascade signal amplification strategy for subattomolar protein detection by rolling circle amplifcation and quantum dots tagging[J].Ana-lytical Chemistry,2010,82(8):3337-3342.
[24]Notomi T,Okayama H,Masubuchi H,et al.Loop-mediated isothermal amplification of DNA[J].Nucleic Acids Research,2000,28(12):E63.
[25]Zhang X Z,Lowe S B,Gooding J J.Brief review of monitoring methods for loop-mediated isothermal amplifcation(LAMP)[J].Biosensors and Bioelectronics,2014,61: 491-499.
[26]Sun W,Qin P,Gao H,et al.Electrochemical DNA biosensor based on chitosan/nano-V2O5/MWCNTs composite film modified carbon ionic liquid electrode and its application to the LAMP product of Yersinia enterocolitica gene sequence[J].Biosensors and Bioelectronics, 2010,25(6):1264-1270.
[27]Nakamura N,Ito K,Takahashi M,et al.Detection of six single-nucleotide polymorphisms associated with rheumatoid arthritis by a loop-mediated isothermal amplification method and an electrochemical DNA chip[J]. Analytical Chemistry,2007,79(24):9484-9493.
[28]Xie S B,Chai Y Q,Yuan Y L,et al.Development of an electrochemical method for Ochratoxin A detection based on aptamer and loop-mediated isothermal amplification [J].Biosensors and Bioelectronics,2014,55:324-329.
[29]Weizmann Y,Beissenhirtz M K,Cheglakov Z,et al.A virus spotlighted by an autonomous DNA machine[J]. Angewandte Chemie International Edition,2006,45(44): 7384-7388.
[30]Beissenhirtz M K,Elnathan R,Weizmann Y,et al.The aggregation of Au Nanoparticles by an Autonomous DNA machine detects viruses[J].Small,2007,3(3):375-379.
[31]Liu Y Q,Zhang M,Yin B C,et al.Attomolar ultrasensitive microRNA detection by DNA-scaffolded silvernanocluster probe based on isothermal amplifcation[J]. Analytical Chemistry,2012,84(12):5165-5169.
[32]Zhang M,Liu Y Q,Yu C Y,et al.Multiplexed detection of microRNAs by tuning DNA-scaffolded silver nanoclusters[J].Analyst,2013,138(17):4812-4817.
[33]Yang C,Shi K,Dou B,et al.In Situ DNA-templated synthesis of silver nanoclusters for ultrasensitive and labelfree electrochemical detection of microRNA [J].ACS Applied Materials&Interfaces,2015,7(2):1188-1193.
[34]Dirks R M,Pierce N A.Triggered amplification by hybridization chain reaction[J].Proceedings of the National Academy of Sciences of the United States of America, 2004,101(43),15275-15278.
[35]Ge Z,Lin M,Wang P,et al.Hybridization chain reaction amplifcation of microRNA detection with a tetrahedral DNA nanostructure-based electrochemical biosensor[J]. Analytical Chemistry,2014,86(4):2124-2130.
[36]Zhuang J Y,Fu L B,Xu M D,et al.Sensitive electrochemical monitoring of nucleic acids coupling DNA nanostructures with hybridization chain reaction[J]. Analytica Chimica Acta,2013,783:17-23.
[37]Xia F,White R J,Zuo X,et al.An electrochemical supersandwich assay for sensitive and selective DNA detection in complex matrices[J].Journal of the American Chemical Society,2010,132:14346-14348.
[38]Yin B C,Guan Y M,Ye B C.An ultrasensitive electrochemical DNA sensor based on the ssDNA-assisted cascade of hybridization reaction[J].Chemical Communications,2012,48(35):4208-4210.
[39]Zhu Z,Gao F L,Lei J P,et al.A Competitive strategy coupled with endonuclease-assisted target recycling for DNA detection using silver-nanoparticle-tagged carbon nanospheres as labels[J].Chemistry-A European Journal,2012,18(43),13871-13876.
[40]Chen J H,Zhang J,Li J,et al.An ultrahighly sensitive and selective electrochemical DNA sensor via nicking endonuclease assisted current change amplifcation[J]. Chemical Communications,2010,46(32):5939-5941.
[41]Zhou H,Zhang Y Y,Liu J,et al.Efficient quenching of electrochemiluminescence from K-doped grapheme-CdS:Eu NCs by G-quadruplex-hemin and target recycling-assisted amplification forultrasensitive DNA biosensing[J].Chemical Communications,2013,49(22): 2246-2248.
[42]Guo Y S,Jia X P,Zhang S S.DNA cycle amplifcation device on magnetic microbeads for determination of thrombin based on graphene oxide enhancing signal-on electrochemiluminescence[J].Chemical Communications,2011,47(2):725-727.
[43]Chen J,Zhang J,Guo Y,et al.An ultrasensitive electrochemical biosensor for detection of DNA species related to oral cancer based on nuclease-assisted target recycling and amplification of DNAzyme[J].Chemical Communications,2011,47(28):8004-8006.
[44]Zhou H,Zhang Y Y,Liu J,et al.Efficient quenching of electrochemiluminescence from K-doped graphene -CdS:Eu NCs by G-quadruplex-hemin and target recycling-assisted amplification for ultrasensitive DNA biosensing[J].Chemical Communications,2013,49(22): 2246-2248.
[45]Nakayama S,Yan L,Sintim H O.Junction probes-sequence specific detection of nucleic acids via template enhanced hybridization processes[J].Journal of the American Chemical Society,2008,130(38):12560-12561.
[46]Kong R M,Zhang X B,Zhang L L,et al.Molecular beacon-based junction probes for efficient detection of nucleic acids via a true target-triggered enzymatic recycling amplification[J].Analytical Chemistry,2010,83 (1):14-17.
[47]Bai L J,Chai Y Q,Pu X Y,et al.A signal-on electrochemical aptasensor for ultrasensitive detection of endotoxin using three-way DNA junction-aided enzymatic recycling and graphene nanohybrid for amplification[J]. Nanoscale,2014,6(5):2902-2908.
[48]Cui L,Ke G,Wang C,et al.A cyclic enzymatic amplifcation method for sensitive and selective detection of nucleic acids[J].Analyst,2010,135(8):2069-2073.
[49]Zuo X,Xia F,Xiao Y,et al.Sensitive and selective amplifed fluorescence DNA detection based on exonuclease III-aided target recycling[J].Journal of the American Chemical Society,2010,132(6):1816-1818.
[50]Wu Z,Zhen Z,Jiang J H,et al.Terminal protection of small-molecule-linked DNA for sensitive electrochemical detection of protein binding via selective carbon nanotube assembly[J].Journal of the American Chemical Society,2009,131(34):12325-12332.
[51]Hsieh K,Xiao Y,Tom Soh H.Electrochemical DNA detection via exonuclease and target-catalyzed transformation of surface-bound probes[J].Langmuir,2010,26 (12):10392-10396.
[52]Wu D,Yin B C,Ye B C.A label-free electrochemical DNA sensor based on exonuclease III-aided target recycling strategy for sequence-specific detection of femtomolar DNA[J].Biosensors and Bioelectronics,2011,28 (1):232-238.
[53]Guo Q,Yang X,Wang K,et al.Sensitive fluorescence detection of nucleic acids based on isothermal circular strand-displacement polymerization reaction[J].Nucleic Acids Research,2009,37(3):e20.
[54]Cheng W,Ding S,Li Q,et al.A simple electrochemical aptasensor for ultrasensitive protein detection using cyclic target-induced primer extension[J].Biosensors and Bioelectronics,2012,36(1):12-17.
[55]Zhang X,Xu Y,Zhao Y,et al.A new photoelectrochemical biosensors based on DNA conformational changes and isothermal circular strand-displacement polymerization reaction[J].Biosensors and Bioelectronics,2013,39 (1):338-341.
The application of signal amplification strategies in electrochemical biosensors
Chen Ying
(School of Chemistry and Chemical Engineering,Southwest University,Chongqing 400715,China)
Rapid,simple and sensitive determination of biomolecules has become increasingly important in clinical diagnosis,food analysis and bioterrorism/environmental monitoring over the past few years.Electrochemical biosensor has gained increasing interest due to its inherent advantages such as simplicity,sensitivity and low cost in cooperation with the comprehensive applications in different fields.Various signal amplification methods have been reported to achieve high sensitivity for biomolecules determination.In this article,we briefly introduce the fundamentals of the electrochemical biosensor,and emphatically summarize the popular signal amplification strategies applied in electrochemical biosensors.
electrochemistry;biodetection;signal amplification strategy

