小分子靶标核酸适配体研究进展
王红旗, 张 玲, 刘冬梅, 王 敏,葛艳静, 周 玲, 刘继红*
(1.河南省农业科学院 农业质量标准与检测技术研究所,河南 郑州,450002;2.河南省粮食质量安全与检测重点实验室,河南 郑州450002;3.农业部农产品质量安全风险评估实验室(郑州),河南 郑州450002)
小分子靶标核酸适配体研究进展
王红旗1,2,3, 张 玲1,2,3, 刘冬梅1,2,3, 王 敏1,2,3,葛艳静1,2,3, 周 玲1,2,3, 刘继红*1,2,3
(1.河南省农业科学院 农业质量标准与检测技术研究所,河南 郑州,450002;2.河南省粮食质量安全与检测重点实验室,河南 郑州450002;3.农业部农产品质量安全风险评估实验室(郑州),河南 郑州450002)
核酸适配体是能够与靶分子高亲和力、高特异性结合的单链寡核苷酸。目前,在生物传感、疾病诊断和治疗等方面,以核酸适配体作为分子识别元件已经开发了许多具有应用可行性的各种检测方法。尽管核酸适配体具有这些方面的应用可能性,但是目前能够结合小分子的核酸适配体非常少。小分子具有多种多样的生物功能以及临床、商业价值,因此针对小分子靶标展开研究,开发新型、实用的分子识别探针用于检测这些小分子具有重要的研究意义。作者主要介绍了针对小分子靶标核酸适配体开发所面临的技术挑战,以及在生物传感和食品安全应用方面所存在的机遇。
核酸适配体;小分子;食品安全;生物传感;指数富集配基系统进化技术
1 核酸适配体与指数富集配基系统进化技术(SELEX)
1.1 核酸适配体与SELEX技术
20多年前,研究人员利用人工合成的方法获得了一段能够特异性结合靶标分子的RNA片段,即“核酸适配体”,从而彻底革新了人们对分子识别事件的传统认识[1-2]。核酸适配体既可以是RNA也可以是DNA,广泛应用在食品安全检测中作为危害因子的特异性分子识别元件,在物质分离富集过程中作为能够与靶目标发生特异性结合的配基以及在医学上用于临床诊断和治疗。相对于传统的分子识别元抗体来说,核酸适配体具有自身的优势[3]:靶标范围非常广泛——从金属离子、有机小分子、蛋白质到病毒甚至细胞;靶标没有免疫原性或免疫原性低,可以是毒性靶标;筛选过程不依赖生物体,无需在生理条件下进行;可体外人工合成——成本低廉、批次间差异小、纯度高、化学稳定性和热稳定性高;易于化学修饰和标记。
相对于其他人工合成的分子识别元件如分子印迹聚合物(Molecular Imprinted Polymers,MIPs)来说,核酸适配体选择性更高,因此其在生物传感和食品安全应用方面显示出广阔的应用前景。当然,核酸适配体也不是完美无瑕的,与抗体或MIPs不同的是,核酸适配体的三级结构与检测方法所采用的分析条件紧密相关,在血液中很容易降解;与抗体相比,核酸适配体的化学多样性略显不足。然而,目前这些问题部分已经得以解决,比如可以通过化学修饰来提高核酸适配体的核酸酶耐受性或增加可供选择的核苷酸的多样性等。
指数富集配基系统进化技术(Systematic Evolution Of Ligands by Exponential Enrichment,SELEX技术)[3]最早报道于20世纪60年代。随着现代生物技术的发展,聚合酶链反应(PCR)技术、反转录酶的提取技术,长片段核酸序列的固相化学合成技术等技术的出现,该技术通过与这些现代生物技术的联合被广泛应用。虽然有报道称一些研究小组对SELEX筛选过程进行了适当的改进,但是SELEX技术的大致流程依然保持不变,大致分为结合、分离、洗脱、放大四个步骤,见图1。首先通过化学合成构建一个约含有1015个不同序列的单链寡核苷酸文库,将靶标与文库一起孵育以便于靶标与相应的核酸充分结合,然后通过分离程序将结合与未结合的核酸分离,接着利用洗脱程序将结合的核酸洗脱,最后以这些洗脱的核酸为模板进行PCR扩增并用于下一轮筛选。通过不断地的结合、分离、洗脱与扩增,一些与靶标不结合或亲和力较小以及中亲和力的核酸相继被淘汰,而具有高亲和力的核酸适配体逐渐被富集起来。
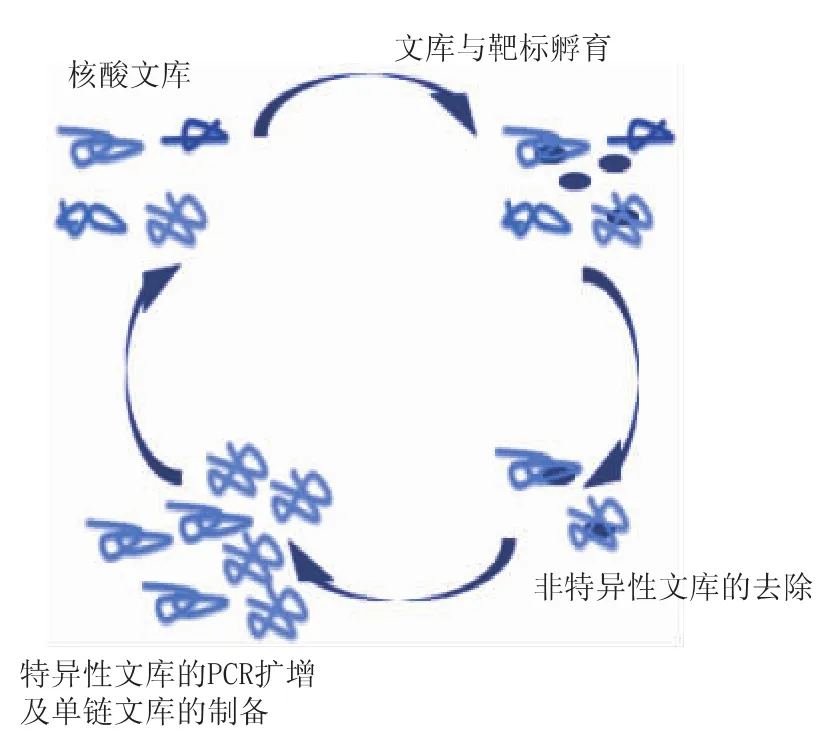
图1 SELEX筛选过程示意图Fig.1 Schematic of SELEX
SELEX技术最大的优点在于其筛选过程中结合条件的多样性以及文库设计的灵活性。最早的SELEX技术改进是引入了负相或反相筛选步骤,目的是消除筛选过程中与固相载体或结构相似性化合物有亲和力的非特异性结合序列。目前SELEX技术的改进主要包括改变筛选压力、筛选平台、筛选文库以及实现过程的自动化等。文库的改进包括引入已知功能的固定序列或者增加文库结构的多样性,其中文库的多样性可以通过初始文库的设计或者突变PCR来实现。无论哪种改进方式,最终目的都是为了获得亲和力有所改善的核酸适配体或者简化SELEX的筛选过程。
1.2 识别小分子的核酸适配体
目前,小分子靶标核酸适配体的数量不到核酸适配体总数的1/4,见图2。

图2 各类靶标的核酸适配体所占的比例Fig.2 Ratio of all kinds of aptamers
蛋白质和细胞相对分子质量较大的靶标具有较多的功能基团和结构单元,通过特定的核酸序列与靶标之间的氢键、静电及疏水相互作用筛选成功的几率较大[5]。在发现蛋白质或细胞的核酸适配体更容易筛选出来后,针对小分子靶标核酸适配体筛选工作变得越来越少,但是筛选小分子靶标核酸适配体仍然具有很大的吸引力,因为小分子靶标在许多生物过程中扮演着重要的角色。小分子靶标可以是毒素或致癌物等有毒有害物质,也可以是药物或营养成分等有益物质。生物分析方面,小分子作为细胞信号传导分子、色素或作为防御机制的一部分具有重要的实际检测价值,而核酸适配体因易快速制备、经济适用、尺寸较小、通用性强,在该领域具有巨大的应用前景。但是,相关的小分子靶标要么是生理上微量存在的,要么是受血液或体液中非特异性化合物严重污染的,而已知的相关小分子靶标的亲和力处于微摩尔至纳摩尔的居多,还不足够强,因此这类基于核酸适配体的小分子靶标分析方法大多需要通过有效的信号放大途径或工具,如采用纳米金、量子点、磁性微球以及各种酶等提高测定方法的灵敏度。在农产品及食品质量安全领域,针对真菌毒素、重金属离子、农药残留等危害因子,结合纳米材料在光学、电学等方面表现出的优良特性,开发了许多基于核酸适配体的高灵敏、高特异性快速检测技术,然而目前这些方法主要是针对基质比较简单的实际样品,且多是单一靶标的检测,有待进一步提高检测方法的抗复杂基质干扰的能力以及提高方法的检测通量。在医药领域,核酸适配体具有很多潜在的优势,如易于微型化的构建、快速灵敏、特异性高、成本低廉,在新药和药物输送系统的发展中可以作为诊断治疗工具和生物传感探针,目前这方面的应用瓶颈在于感兴趣靶标核酸适配体的筛选以及核酸适配体的半衰期。表1列出了已被表征过的能够识别小分子靶标DNA核酸适配体。
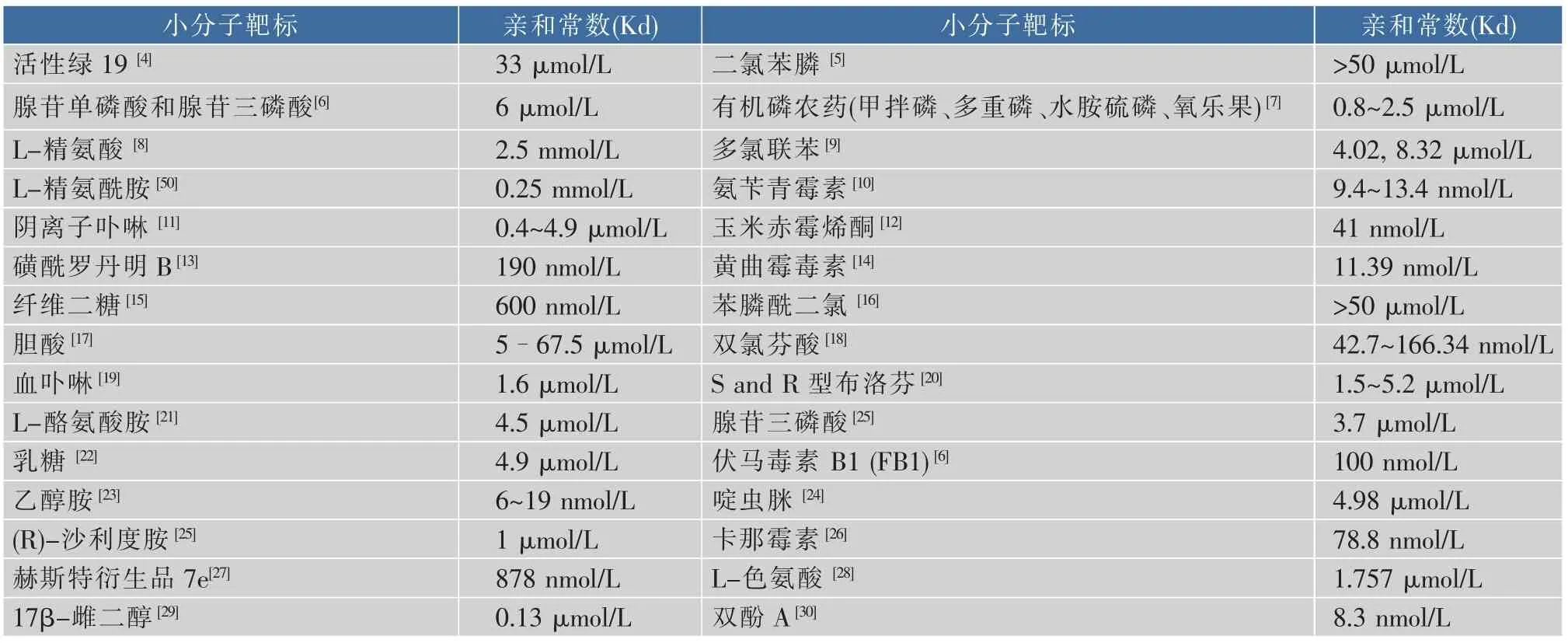
表1 文献中已经报道的小分子靶标DNA核酸适配体(截止到2014年)Table 1 DNA aptamers for small molecule target reported in the open literature(upto 2014)
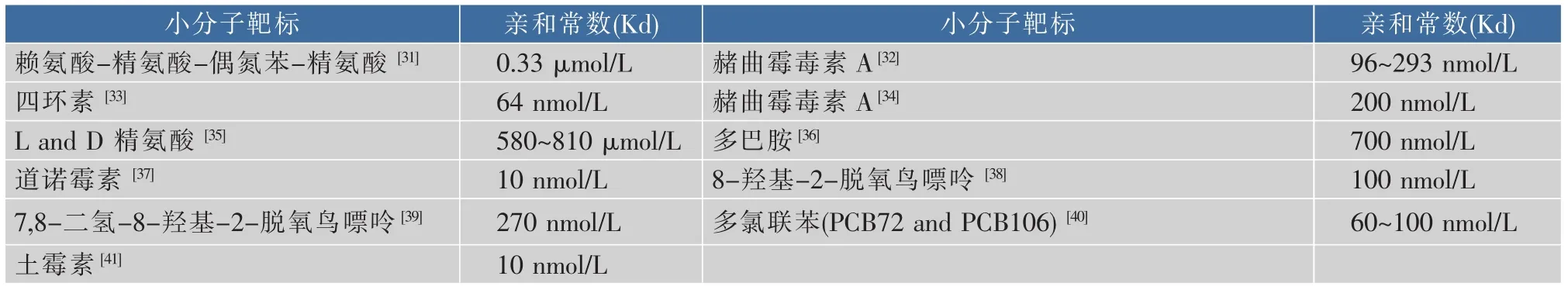
续表1
2 小分子靶标核酸适配体筛选的挑战
2.1 小分子靶标核酸适配体筛选的理论挑战
虽然小分子靶标核酸适配体的数量比较少,但相关研究却最彻底、最广泛。据过去10年文献报道的次数,ATP核酸适配体仅次于凝血酶核酸适配体位居第二,可卡因和茶碱的核酸适配体分别位居第五和第七。茶碱RNA核酸适配体是第一例具有极高选择性的核酸适配体,与茶碱的亲和力比跟咖啡因(分子结构上与茶碱只有一个甲基的不同)的亲和力强10 000倍,且与茶碱的抗体相比选择性提高了10倍[42]。普遍认为小分子靶标核酸适配体的亲和力较弱,位于低微摩尔到中微摩尔之间,难于满足大多数传感器应用的要求。Carothers等[43]研究表明,靶标的相对分子质量与核酸适配体的亲和力成正比例关系,该研究结果与其他小组的研究结果一致。然而,茶碱(相对分子质量180)是个例外;最近新筛选到的一些小分子靶标核酸适配体的Kd值也低至纳摩尔,如双酚A[45]和土霉素[46]。另外,“天然适配体”核糖开关也具有很强的亲和力,如鸟嘌呤核糖开关的Kd值为5 nmol/L,维生素B1焦磷酸酯的核糖开关的Kd值在皮摩尔范围 。据此推断:靶标含有的可旋转化学键越少,其相应的核酸适配体的亲和力越高。
20世纪90年代,研究者们曾鼓吹SELEX技术简单易行、成本低廉、成功率高,促进了一批有关筛选的研究见诸报道。其实SELEX筛选技术相当费时费力,成功率不足30%[47]。此外,几乎每一个核酸适配体的实际应用都申请了专利,一定程度上限制了核酸适配体筛选的创新。目前,很少有课题组在开发新型小分子靶标核酸适配体上投入过多的时间和资金,特别是考虑到在筛选过程中可能要面临的技术挑战。
2.2 小分子靶标核酸适配体筛选的技术挑战
2.2.1 靶标的固定 分离步骤是SELEX筛选的关键。对于蛋白质和细胞等大分子靶标,可以分别采用硝酸纤维素膜和离心、荧光激活细胞分选术(FACS)以及对贴壁细胞进行温和洗涤等方式实现结合与不结合文库的分离,不需要对靶标进行化学修饰,有利于获得自由态靶标的核酸适配体。然而这种经典的筛选方法却不适用于小分子靶标。小分子靶标核酸适配体筛选的首要困难在于靶标在磁珠、聚合物微球、琼脂糖等固相基质上的固定。早期的筛选主要基于商品化的固定有靶标的琼脂糖基质或者采取人工偶联的方法。偶联的前提是小分子靶标上必须有可利用的功能团,这种要求往往难以满足。即使在偶联可以实现的情况下,大量的柱填装料会带来比较严重的文库非特异性吸附问题。另外,对小分子靶标进行化学修饰会导致筛选到能够结合基质或链接臂的核酸适配体的几率大大增加。如果筛选到的核酸适配体的亲和力部分来自与固相基质或者化学修饰基团,那么其实际应用功效则会大大降低。据报道,罗丹明的核酸适配体与自由靶标的结合弱于与固相基质上的靶标[48]。
2.2.2 亲和力的测定 (Kd) 亲和力的测定是制约小分子靶标核酸适配体快速发展的重要因素。传统的Kd值测定方法必须固定核酸适配体或者靶标,通过不断增加另一组分的浓度进行滴定从而产生结合曲线。常用的Kd值测定方法见表2,其中可有效测定小分子靶标核酸适配体Kd值的方法相当少。基于靶标-核酸适配体复合物尺寸动态变化的分离模式检测技术和基于表面质量敏感型的检测方法,一般需要将靶标或者核酸适配体固定在表面上。当用于小分子靶标时候,针对将核酸适配体在表面固定进行测定的情况,这类方法的灵敏度面临考验,因为小分子靶标结合引起的总体质量变化比较小;将小分子靶标固定在表面上进行测定,又会再一次面临化学修饰影响亲和力的问题。基于构象变换原理的检测方法虽然可以用于小分子,但不是一种通用的检测方法,因为并非所有的核酸适配体在与小分子靶标结合时都能发生可量测的构象变换[49]。其他的Kd值测定方法通常要求靶标自身具有荧光或紫外吸收特性,然而实际应用中许多小分子靶标不具备这个特性。新近开发的一些检测方法如自动化微芯片电泳技术和原子力光谱技术能够部分克服以上所述难题,但依然缺乏一种通用的小分子靶标核酸适配体亲和力测定技术。

表2 核酸适配体Kd的测定方法Table 2 Methods for determining Kd of aptamer
3 小分子靶标核酸适配体的机遇
由于小分子核酸适配体的筛选比较困难,目前已经报道的小分子靶标核酸适配体非常少。然而在生物传感和化学生物学领域,小分子靶标核酸适配体却有着广阔的应用前景。各种天然或人工合成的化合物大都属于小分子,包括氨基酸、甾族化合物、糖类化合物和核苷酸等,既可以用作治疗的药物、染料、辅酶因子、生理代谢物以及神经递质等各种有益物质,又可以充当各种有害物质如环境污染物、食品掺杂物、致癌物以及滥用药物等。随着小分子生物功能认识的不断深入,化学合成小分子药物数量逐渐增多,环境和食品污染物监控需求不断增加,现在比过去更加迫切需要有效的小分子检测/监测工具或手段。目前已经报道的基于核酸适配体的小分子检测方法大都是针对ATP、可卡因或者茶碱三种明星靶标,包括电化学、荧光、比色等检测方法。这些方法的通用性仍需要其他小分子靶标的验证。如表1所示,许多核酸适配体序列可以用于小分子生物传感检测方法的开发,其中一些小分子靶标与环境监测、农产品以及食品质量安全、医疗诊断密切相关。为此,我们应该加快推进基于核酸适配体的生物传感技术从概念型走向实用型并用于实际样品的分析应用。
Li等[67]报道了一种著名的通用型“结构开关型”小分子靶标生物传感检测方法,该方法利用了核酸适配体固有的结合特性:既可以结合同源靶标又可以与其互补链杂交,核酸双链转换成靶标-适配体复合物的同时伴随构象变换,从而将特异性的分子识别事件转换成可量测的信号(光、电、热、质量等)。如果核酸适配体与小分子靶标结合时能够发生较大的构象变化,那么这种检测策略就相当吸引人,因为它可以避开其他检测方法在小分子检测方面所面临的诸多困难。不管是DNA还是RNA核酸适配体 ,无论是在溶液中还是在界面上 ,都可以采用这种检测策略。目前该检测策略已经用于ATP、茶碱、可卡因、组氨酸、OTA、L-精氨酸、络氨酸、GTP和精氨酸等小分子的检测。最近有报道将核酸适配体与其他功能序列偶联嵌合的检测技术,小分子靶标核酸适配体尤其适合用于偶联嵌合序列的制备。如将纺锤型核糖酶的一个茎环序列用ATP核酸适配体序列替代,从而可以用ATP来调控核糖酶的活性 ;孔雀石绿(MG)核酸适配体与核黄素单核苷酸核酸(FMN)适配体构成的偶联体,可以用来检测FMN。类似的设计还用于ATP和茶碱传感器的研发。
小分子靶标核酸适配体还是一种重要的研究工具,可用于阐明一些具有重要生理作用的小分子调节关键细胞调控回路的作用。例如,我们可以利用小分子靶标核酸适配体干扰细胞内的生理重要性小分子,从而进一步提高对生物体系的认识。Marletta等利用亚铁血红素RNA和DNA核酸适配体在细胞内进行表达,利用反馈抑制系统实现了亚铁血红素大肠杆菌生物合成的预期调控。小分子靶标核酸适配体还对体内疾病治疗药物的开发起着一定的主导作用。最近有报道称多巴胺的DNA核酸适配体可用于精神分裂症潜伏期的动物的治疗。
4 展望
小分子靶标核酸适配体在筛选和应用的过程中遭遇了一系列的理论或技术上的挑战,严重制约了其进一步研究和商业化的进程。然而,我们仍需继续努力不断拓展小分子靶标核酸适配体的数量和范围。目前,其研发和应用的瓶颈在于自由态靶标筛选策略的开发及Kd值的测定技术:开发不需要对小分子靶标进行固定或化学修饰的SELEX筛选技术;开发新的真正意义上的Kd值测定方法。理想情况下,Kd值的测定应该在核酸适配体与靶标在溶液中自由结合的情况下进行,以便消除基质引起的非特异性结合。新近出现的核酸适配体数据库(http://aptamerbase.semanticscience.org/)包含大量的核酸适配体筛选条件和结合常数测定方法的信息可供查询。另外,基于核酸适配体的检测新方法的开发也需要进一步拓宽模型靶标的范围,不能仅仅局限于几种明星靶标,应该进一步挖掘已经筛选到核酸适配体的大量未曾开发过的新靶标资源。目前,这种转变趋势已经出现,大量的基于赭曲霉毒素A(OTA)的核酸适配体生物传感器已经报道,如固相亲和净化柱和生物传感器,并在实际的样品检测条件下进行了相应的检测性能评估。可以预见,随着代谢组学、药物开发以及合成生物学等领域的快速发展,小分子靶标核酸适配体的应用很快也将呈现迅猛增长趋势。
参考文献:
[1]Tuerk C,Gold L.Systemic evolution of ligands by exponential enrichment:RNA ligands to bacteriophage T4 DNA polymerase [J].Science,1990,249(4968):505-510.
[2]Ellington A D,Szostak J W.In vitro selection of RNA molecules that bind specific ligands[J].Nature,1990,346(6287):818-822.
[3]Jayasena S D.Aptamers:an emerging class of molecules that rival antibodies in diagnostics[J].Clinical Chemistry,1999,45(9):1628-1650.
[4]Ellington A D,Szostak J W.Selection in vitro of single-stranded DNA molecules that fold into specific ligandbinding structures [J].Nature,1992,355(6363):850-852.
[5]Renaud A,Faverie De La,Hamon F,et al.Nucleic acids targeted to drugs:SELEX against a quadruplex ligand[J].Biochimie,2011,93(8):1357-1367.
[6]EHuizenga D,Szostak W.A DNA aptamer that binds adenosine and ATP[J].Biochemistry,1995,34(2):656-665.
[7]Wang L,Liu X,Zhang Q et al,Selection of DNA aptamers that bind to four organophosphorus pesticides[J].Biotechnology Letters,2012,34(5):869-874.
[8]Harada K,Frankel A D.Identification of two novel arginine binding DNAs[J].EMBO Journal,1995,14(23):5798-5811.
[9]Xu S,Yuan H,Chen S,et al.Selection of DNA aptamers against polychlorinated biphenyls as potential biorecognition elements for environmental analysis[J].Analytical Biochemistry,2012,423(2):195-201.
[10]Song K M,Jeong E,Jeon W,et al.Aptasensor for ampicillin using gold nanoparticle based dual fluorescence-colorimetric methods[J].Analytical and Bioanalytical Chemistry,2012,402(6):2153-2161.
[11]Li Y,Geye C R r,Sen D.Recognition of anionic porphyrins by DNA aptamers[J].Biochemistry,1996,35(21):6911-6922.
[12]Chen Xiujuan,Huang Yukun,Duan Nuo,et al.Selection and identification of ssDNA aptamers recognizing zearalenone[J]. Analytical and Bioanalytical Chemistry,2013,405(20):6573-6581.
[13]Wilson C,J W Szostak.Isolation of a fluorophorespecific DNA aptamer with weak redox activity[J].Chemistry and Biology,1998,5(11):609-617.
[14]Ma Xiaoyuan,Wang Wenfeng,Chen Xiujuan,et al.Selection,identification,and application of Aflatoxin B1 aptamer[J]. Eoropean Food Research and Technology,2014,238(6):919-925.
[15]Yang Q,Goldstein I J,Mei H-Y,et al.DNA ligands that bind tightly and selectively to cellobiose[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95(10):5462-5467.
[16]Renaud A,Faverie De La,Hamon F,et al.Nucleic acids targeted to drugs:SELEX against a quadruplex ligand[J].Biochimie,2011,93(8):1357-1367.
[17]Kato T,Takemura T,Yano K,et al.In vitro selection of DNA aptamers which bind to cholic acid[J].Biochimica et Biophysica Acta Gene Structure and Expression,2000,1493(1-2):12-18.
[18]Joeng C B,Niazi J H,S J,et al.ssDNA aptamers that recognize diclofenac and 2-anilinophenylacetic acid[J].Bioorganic and Medicinal Chemistry,2009,17(15):5380-5387.
[19]Okazawa A,Maeda H,Fukusaki E,et al.In vitro selection of hematoporphyrin binding DNA aptamers[J].Bioorganic and Medicinal Chemistry Letters,2000,10(23):2653-2656.
[20]Kim Y S,Hyun C J,Kim I A,et al.Isolation and characterization of enantioselective DNA aptamers for ibuprofen[J].Bioorganic and Medicinal Chemistry,2010,18(10):3467-3473.
[21]Vianini E,Palumbo M,Gatto B.In vitro selection of DNA aptamers that bind L-tyrosinamide[J].Bioorganic and Medicinal Chemistry,2001,9(10):2543-2548.
[22]Masud M M,Kuwahara M,Ozaki H,et al.Sialyllactose-binding modified DNA aptamer bearing additional functionality by SELEX[J].Bioorganic and Medicinal Chemistry,2004,12(5):1111-1120,
[23]Mann D,Reinemann C,Stoltenburg R,et al.In vitro selection of DNA aptamers binding ethanolamine[J].Biochemical and Biophysical Research Communications,2005,338(4):1928-1934,
[24]He J,Liu Y,Fan M,et al.Isolation and identification of the DNA aptamer target to acetamiprid[J].Journal of Agricultural and Food Chemistry,2011,59(5):1582-1586.
[25]Shoji A,Kuwahara M,Ozaki H,et al.Modified DNA aptamer that binds the(R)-isomer of a thalidomide derivative with high enantioselectivity[J].Journal of the American Chemical Society,2007,129(5):1456-1464.
[26]Song K M,Cho M,Jo H,et al.Gold nanoparticle-based colorimetric detection of kanamycin using a DNA aptamer[J].Analytical Biochemistry,2011,415(2):175-181.
[27]Sando S,Narita A,Aoyama Y.Light-up Hoechst-DNA aptamer pair:generation of an aptamer-selective fluorophore from a conventional DNA-staining dye[J].Chem Bio Chem,2007,8(15):1795-1803.
[28]Yang X,Bing T,Mei H,et al.Characterization and application of a DNA aptamer binding to l-tryptophan[J].Analyst,2011,136(3):577-585.
[29]Kim Y S,Jung H S,Matsuura T,et al.Electrochemical detection of 17β-estradiol using DNA aptamer immobilized gold electrode chip[J].Biosensors and Bioelectronics,2007,22(11):2525-2531.
[30]Jo M,Ahn J Y,Lee J,et al.Development of singlestranded DNA aptamers for specific bisphenol a detection[J]. Oligonucleotides,2011,21(2):85-91.
[31]Hayashi G,Hagihara M,Dohno C,et al.Photoregulation of a peptide-RNA interaction on a gold surface[J].Journal of the American Chemical Society,2007,129(28):8678-8679.
[32]Barthelmebs L,Jonca J,Hayat A et al.Enzyme-Linked Aptamer Assays(ELAAs),based on a competition format for a rapid and sensitive detection of Ochratoxin A in wine[J].Food Control,2011,22(5):737-743.
[33]Niazi J H,Lee S J,M B Gu.Single-stranded DNA aptamers specific for antibiotics tetracyclines[J].Bioorganic and Medicinal Chemistry,2008,16(15):7245-7253.
[34]Cruz-Aguado J A,Penner G.Determination of ochratoxin A with a DNA aptamer[J].Journal of Agricultural and Food Chemistry,2008,56(22):10456-10461.
[35]Ohsawa K,Kasamatsu T,Nagashima J I,et al.Argininemodified DNA aptamers that show enantioselective recognition of the dicarboxylic acid moiety of glutamic acid[J].Analytical Sciences,2008,24(1):167-172.
[36]Walsh R,DeRosa M C.Retention of function in the DNA homolog of the RNA dopamine aptamer[J].Biochemical and Biophysical Research Communications,2009,388(4):732-735.
[37]Wochner A,Menger M,Orgel D,et al.A DNA aptamer with high affinity and specificity for therapeutic anthracyclines[J]. Analytical Biochemistry,2008,373(1):34-42.
[38]Miyachi Y,Shimizu N,Ogino C,et al.Selection of a DNA aptamer that binds 8-OHdG using GMP-agarose[J].Bioorganic and Medicinal Chemistry Letters,2009,19(13):3619-3622.
[39]Rink S M,Shen J C,Loeb L A.Creation of RNA molecules that recognize the oxidative lesion 7,8-dihydro-8-hydroxy-2-deoxyguanosine(8-oxodG)in DNA[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95(20):11619-11624.
[40]Mehta J,Rouah-Martin E,Van Dorst B,et al.Selection and characterization of PCB-binding DNA aptamers[J].Analytical Chemistry,2012,84(3):1669-1676.
[41]Niazi J H,Lee S J,Kim Y S,et al.ssDNA aptamers that selectively bind oxytetracycline [J].Bioorg Med Chem,2008,16:1254-1261.
[42]Jenison R D,Gill S C,A Pardi,et al.Highresolution molecular discrimination by RNA[J].Science,1994,263(5152):1425-1429. [43]Carothers J M,Goler J A,Kapoor Y,et al.Selecting RNA aptamers for synthetic biology:investigating magnesium dependence and predicting binding affinity[J].Nucleic Acids Research,2010,38(8):2736-2747.
[44]Pfeffer P,H Gohlke.Drug Score RNA—knowledge based scoring function to predict RNA—Ligand interactions[J].Journal of Chemical Information and Modeling,2007,47(5):1868-1876.
[45]M Jo,Ahn J Y,Lee J,et al.Development of singlestranded DNA aptamers for specific bisphenol a detection[J]. Oligonucleotides,2011,21(2):85-91.
[46]Niazi J H,Lee S J,Kim Y S,et al.Development of single-stranded DNA aptamers for specific bisphenol A detection[J].Bioorg Med Chem,2008,16:1254-1261.
[47]Gold L,Ayers D,Bertino J,et al.Aptamer-based multiplexed proteomic technology for biomarker discovery[J].PLoS One,2010,5(12):15004.
[48]Wilson C,J W Szostak.Isolation of a fluorophorespecific DNA aptamer with weak redox activity[J].Chemistry and Biology,1998,5(11):609-617.
[49]White R J,Rowe A A,Plaxco K W,et al.Re-engineering aptamers to support reagentless,self-reporting electrochemical sensors [J].Analyst,2010,135(3):589-594.
[50]Flinders J,DeFina S C,Brackett DM,et al.Recognition of planar and nonplanar ligands in the malachite green—RNA aptamer complex[J].Chem Bio Chem,2004,5(1):62-72.
[51]Cruz-Aguado J A,Penner G,Fluorescence polarization based displacement assay for the determination of small molecules with aptamers[J].Analytical Chemistry,2008,80(22):8853-8855.
[52]Guedin A,Lacroix L,Mergny J L,et al.Thermal melting studies of ligand DNA interactions[J].Methods in Molecular Biology,2010,613:25-35.
[53]Lin P,Chen R,Lee C,et al.Studies of the binding mechanism between aptamers and thrombin by circular dichroism,surface plasmon resonance and isothermal titration calorimetry[J].Colloids and Surfaces B,2011,88:552-558.
[54]Yang C,Lates V,Prieto-Sim'on B,et al.Aptamer-DNAzyme hairpins for biosensing of Ochratoxin A [J].Biosensors and Bioelectronics,2012,32:208-212.
[55]Y Sultan,R Walsh,C Monreal,et al.Preparation of functional aptamer films using layer-by-layer selfassembly[J]. Biomacromolecules,2009,10(5):1149-1154.
[56]Deng Q,German I,Buchanan D,et al.Retention and separation of adenosine and analogues by affinity chromatography with an aptamer stationary phase[J].Analytical Chemistry,2001,73(22):5415-5421.
[57]Hu J,Easley C.A simple and rapid approach for measurement of dissociation constants of DNA aptamers against proteins and small molecules via automated microchip electrophoresis[J].Analyst,2011,136(17):3461-3468.
[58]Drabovich A P,Berezovski M,Okhonin V,et al.Selection of smart aptamers by methods of kinetic capillary electrophoresis[J]. Analytical Chemistry,2006,78(9):3171-3178.
[59]Bao J,Krylova S M,Reinstein O,et al.Label-free solution-based kinetic study of aptamer small molecule interactions by kinetic capillary electrophoresis with UV detection revealing how kinetics control equilibrium[J].Analytical Chemistry,2011,83:8387-8390.
[60]Turgeon R T,Fonslow B R,M Jing,et al.Measuring aptamer equilibria using gradient micro free flow electrophoresis[J]. Analytical Chemistry,2010,82(9):3636-3641.
[61]Hall B,S Arshad,K Seo,et al.In vitro selection of RNA aptamers to a protein target by filter immobilization[J].Current Protocols in Molecular Biology,2009,88:2431-24327.
[62]Baaske P,Wienken C J,Reineck P,et al.Optical thermophoresis for quantifying the buffer dependence of aptamer binding[J]. Angewandte Chemie,2010,49(12):2238-2241.
[63]Potty A S R,Kourentzi K,H Fang,et al.Biophysical characterization of DNA aptamer interactions with vascular endothelial growth factor[J].Biopolymers,2009,91(20):145-156.
[64]Girolamo A De,McKeague M,Miller J D,et al.Determination of ochratoxin A in wheat after clean-up through a DNA aptamer-based solid phase extraction column[J].Food Chemistry,2011,127(3):1378-1384.
[65]Regulski E E,Breaker R R.In-line probing analysis of riboswitches[J].Methods in Molecular Biology,2008,419:53-67.
[66]McManus S A,Y Li.Multiple occurrences of an efficient self-phosphorylating deoxyribozyme motif[J].Biochemistry,2007,46(8):2198-2204.
[67]Nutiu R,Li Y.Structure-switching signaling aptamers[J].Journal of the American Chemical Society,2003,125(16):4771-4778.
Research Progress of Aptamer for Small Molecule Target
WANG Hongqi1,2,3, ZHANG Ling1,2,3, LIU Dongmei1,2,3, WANG Min1,2,3,
GE Yanjing1,2,3, ZHOU Ling1,2,3, LIU Jihong*1,2,3
(1.Institute of Quality Standards and Testing Teclmology for Agro-Products,Henan Academy of Agricultural Sciences,Zhengzhou 450002,China;2.Henan Key Laboratory of Grain Quality and Safety and Testing,Zhengzhou 450002,China;3.Laboratory of Quality&Safety Risk Assessment for Agro-Products(Zhengzhou),Ministry of Agriculture,Zhengzhou 450002,China)
Aptamer as a single-stranded oligonucleotide can bind to target with high affinity and selectivity.Although many viable approaches for biosensing,diagnostics,and therapeutics have emerged,relatively few aptamers binding to small molecules exist.Small molecules are very important targets with diverse biological functions and clinical and co mMercial uses.Therefore,developing novel and effective molecular recognition probes for these compounds are greatly interesting.This paper will highlight various challenges during aptamer development relative to smallmolecule targets,as well as opportunities for their application in biosensing and food safety.
aptamer,small molecule,food safety,biosensor,SELEX
Q527
A
1673—1689(2015)08—0790—09
2014-07-28
国家自然科学基金项目(21305031);国家公益性行业(农业)项目(201203094);河南省科技攻关项目(122102310048)。
王红旗(1979—),男,河南郑州人,理学博士,副研究员,主要从事农产品质量安全检测技术方面的研究。E-mail:huda2000@126.com
*通信作者:刘继红(1976—),女,河南郑州人,理学博士,副研究员,主要从事农产品质量安全检测技术及农产品风险评估方面的研究。E-mail:ljha3100@163.com

