运用逆向疫苗学对沙眼衣原体疫苗候选蛋白的初步筛选
王 伟,张 强,王海梅,郭 普,钟政荣(蚌埠医学院第一附属医院检验科,蚌埠 233004)
沙眼衣原体(Chlamydia trachomatis)是一类在细胞内寄生的微生物,全球感染沙眼衣原体的人数多以亿计[1,2],仅治疗所需的费用就高达数十亿美元[2]。在沙眼衣原体的 18 个血清型中[3],沙眼生物型A、B、Ba、C血清型可感染眼结膜,居于致盲病因的首位,而D-K血清型可以引起生殖道感染或泌尿系统感染,是目前导致男性不育或女性不孕的重要原因之一[4,5]。虽然抗生素对沙眼衣原体的感染有较好的疗效,但因沙眼衣原体的感染常为隐性感染,致使绝大多数患者在感染早期不能及时就诊[6]。临床治疗中大规模使用抗生素还可能造成耐药株的出现。因此,使用疫苗进行预防是最为合理有效的防治措施[7]。2000年,Pizza革命性地创造了一种新的研究策略,首先应用于B型脑膜炎奈瑟菌的疫苗研究中,由于与传统疫苗的研究策略刚好相反,因此被称为逆向疫苗学。逆向疫苗学与传统疫苗学研究的不同之处在于需要计算机的辅助,不需要体外培养病原菌。而是通过对某一病原体的基因组测序分析,筛选出抗原决定簇,通过高通量技术,在短期内完成大量候选抗原的克隆、表达和纯化,鉴定出具有一定免疫原性特征的蛋白质,为一些不能用传统疫苗研究方法获得有效疫苗的疑难疫病的防控开辟了一条新的途径。本研究应用逆向疫苗学理论,在沙眼衣原体全基因组测序完成的基础上,整合生物信息学分析、比较基因组数据、基因表达丰度分析和免疫蛋白质组学来初步筛选针对沙眼衣原体疫苗的候选抗原,现将结果报告如下。
1 材料与方法
1.1 生物信息学分析、预测编码表面暴露蛋白的基因
基因组数据为GenBank中全基因组测序菌株L2b/UCH-1/proctitis株,应用 P-CLASSIFIER[8]预测沙眼衣原体蛋白质的亚细胞定位。应用SignalP 3.0[9]预测沙眼衣原体蛋白质中的信号肽。沙眼衣原体蛋白质中的α-穿膜螺旋结构用TMHMM预测[10]。BOMP[11]用来预测沙眼衣原体中的β-桶型外膜蛋白。脂蛋白用 SpLiP 预测[12]。BLASTP[13]程序用来比对沙眼衣原体不同型别之间以及沙眼衣原体和人之间的同源蛋白质。
1.2 实时定量PCR
选择生长状态良好的衣原体(Hep-2细胞系和沙眼衣原体菌株由蚌埠医学院科研中心保存),使用Trizol一步法抽提总RNA,并将mRNA反转录成cDNA。实时定量 PCR检测 CTLon_0133、CTLon_0242、CTLon_0288、CTLon_0395等蛋白编码基因的表达量,内参使用16srDNA序列,引物见表1。反应条件:95℃ 30 s,95℃ 5 s,54℃ 30 s共35个循环。

表1 扩增基因引物序列Table 1 The primer sequences and PCR conditions
1.3 蛋白表达纯化
cDNA核酸片断克隆进pET28b(+)质粒载体,转化入感受态DH5α细胞,IPTG浓度为1 mmol/L诱导表达。经可溶性鉴定后,冰上超声破碎细胞后,在非变性条件下,终浓度为0.05%Triton X-100,抽提蛋白。NTA树脂层析,洗涤并浓缩。加入等体积2×SDS上样缓冲液进行聚丙烯酰胺电泳,80 V,350 mA,半干转移至PVDF膜上,2 h后,加入相应的抗体稀释液,4℃过夜。TBS/0.5%Tween20洗膜3-4次后,每次10 min,再加入l∶2 000稀释的抗HRP抗体,37℃孵育1 h,TBS/0.5%Tween20充分洗膜3-4次后,每次致少10 min,用ECL显色系统检测相应信号并分析。
1.4 免疫动物
每组蛋白免疫4只BALB/c小鼠,初次免疫后第10天和第20天分别加强免疫两次,采用皮下多点注射,注射剂量为10 μg/只,第一次注射用弗氏完全佐剂1∶1混合,第二、三次注射用弗氏不完全佐剂1∶1混合,到第35天全部小鼠眼球取血,处死。
1.5 ELISA法检测抗体特异性
按标准ELISA法,利用纯化的重组蛋白包被96孔板过夜,用PBS洗涤后BSA封闭,每孔加200倍稀释的100 μl血清,室温结合30 min;PBS洗板3次,加碱性磷酸酶AP标记IgG抗体,室温结合1 h;PBS洗板 3次,加底物 pNPP,室温反应 15 min,NaOH中止反应,405 nm波长测吸光度值。实验中各检测样品重复3孔,以样品孔OD吸光度值≥阴性对照平均值+3×阴性对照标准差者为阳性。
1.6 凝集试验检测抗体效价
用无菌生理盐水在无菌96孔板上稀释待测血清为1∶25-1∶3 200,每排各孔中加入稀释血清100 μl,最后一孔用生理盐水作阴性对照。将培养好的生长良好的原体混悬液加入各排孔内,每孔100 μl,混匀后置37℃孵育2 h。用吸管取各孔中反应悬液滴于清洁载玻片上,盖上盖玻片,置于显微镜下观察结果。若凝集成团,呈蜘蛛状或原体溶解成颗粒状或蝌蚪状则为阳性。记录下出现阳性凝集的血清最高稀释度,血清抗体效价判断标准:能与抗原发生凝集反应(++)的血清最高稀释度为抗体最终效价。
2 结果
2.1 生物信息学分析编码表面暴露蛋白基因的预测结果
沙眼衣原体基因组中有890个开放读码框(ORFs),应用P-CLASSIFIER预测216个蛋白质为表面暴露蛋白;同时应用相应的生物信息学软件预测,在890个ORFs中有224个蛋白质具有信号肽,261个蛋白质预测具有不超过4个穿膜结构的α-螺旋结构,89个蛋白质预测具有β-桶型结构,63个蛋白质为脂蛋白。P-CLASSIFIER预测表面暴露蛋白和具有以上4个特征性结构之一的蛋白质的交集数目是126个。应用BLASTP计算出衣原体不同型别株蛋白质以及和人蛋白质之间的相似性(cutoff值,不同血清衣原体相似性>70%且E值≤e×10-10;衣原体株和人蛋白质E值≤e×10-10)。结果显示,在两个血清型衣原体间有672个同源蛋白质,与人蛋白质之间相似的蛋白质数目为68个。在126个蛋白质的数据集中保留衣原体间的同源蛋白质,并且去除掉和人之间相似的蛋白质后,最终得到73个蛋白质。这73个蛋白编码基因为生物信息学分析的最终结果。通过比对,我们发现预测结果包括已经证实的外膜蛋白如:MOMP等,这也证实我们预测方法的可行性。
2.2 实时定量PCR
从73个基因中挑取30个未曾鉴定过的基因进行实时定量 PCR检测,对表达量进行分析。与16srDNA相比,这30个基因表达量为其0.2-5.4倍不等。我们选取了其中表达最高的4个基因,分别为 CTLon_0133,CTLon_0242,CTLon_0288,CTLon_0395,分析结果见图1。
2.3 Western blot检测结果
cDNA核酸片段克隆进pET28b(+)质粒载体,转化入感受态DH5α细胞,表达的蛋白经抽提、洗涤并浓缩后,CTLon_0133,CTLon_0242,CTLon_0288,CTLon_0395四种高表达基因的Western blot检测结果(见图2)。

图1 实时定量PCR检测候选基因表达结果Figure 1 Expression of candidate genes by real-time quantitative PCR
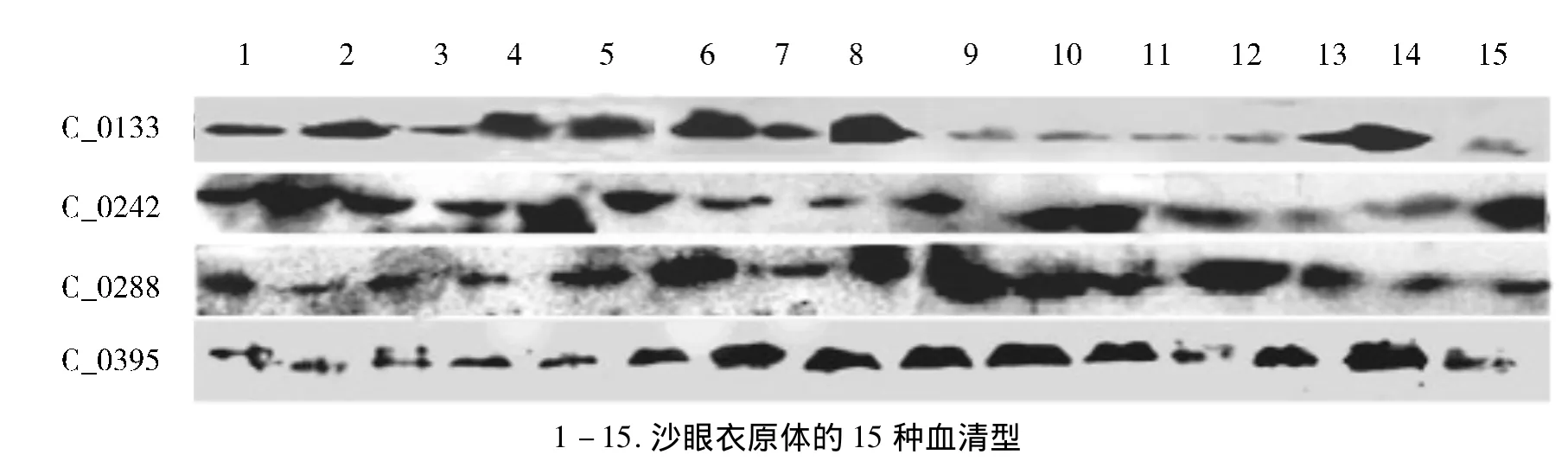
图2 四种高表达基因蛋白表达纯化后在15种血清型中Western blot检测结果Figure 2 Western blot analysis of the expression of CTLon_0133,CTLon_0242,CTLon_0288,CTLon_0395 proteins in 15 serotypes
2.4 免疫动物血清效价鉴定结果
对 CTLon_0133,CTLon_0242,CTLon_0288,CTLon_0395构建质粒在DH5α工程菌中表达并纯化,免疫BALB/c小鼠。血清特异性检测及其效价分析结果见表2,3。凝集实验结果表明,C_0133、C_0242、C_0288和C_0395的抗体效价分别为1∶800,1∶400,1∶1 600 和1∶400,C_0288 蛋白产生的抗体效价较高。
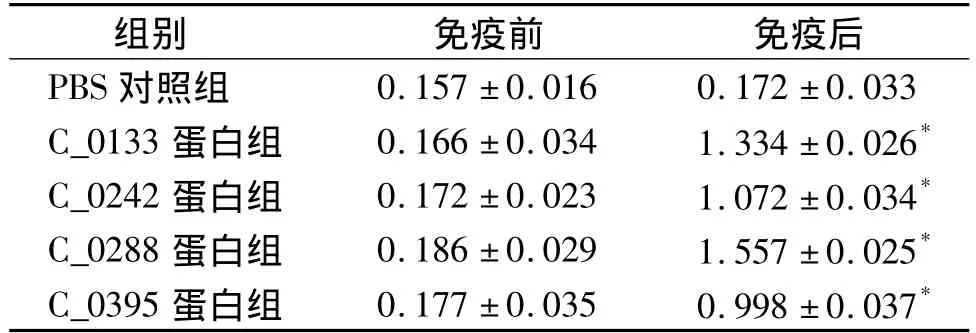
表2 CTLon_0133,CTLon_0242,CTLon_0288,CTLon_0395抗体特异性检测结果Table 2 Assessment of antibody specificity by ELISA for CTLon_0133,CTLon_0242
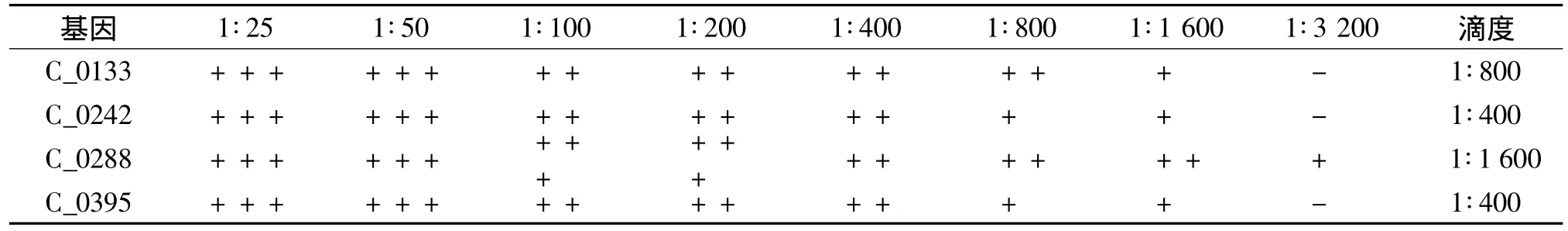
表3 CTLon_0133,CTLon_0242,CTLon_0288,CTLon_0395 抗体效价测定结果Table 3 Assessment of antibody titers by ELISA for CTLon_0133,CTLon_0242
3 讨论
现在使用的衣原体疫苗为全菌体或者外膜蛋白疫苗[14-17]。由于这些疫苗存在以下问题:一是不同型别之间缺乏交叉保护,保护范围较窄;二是保护时间短,需要多次免疫才能维持保护作用,尤其是不能引起机体长期的免疫保护以及对不同血清型衣原体缺乏交叉保护作用,因此衣原体的亚单位疫苗成为研究的重点,一些疫苗候选分子如MOMP等相继被研究鉴定[18],虽然在一些动物实验中具有较好的免疫保护效果,但至今没有合适的衣原体亚单位疫苗进入临床,因此需要新的研究思路和方法进行衣原体疫苗研究。
随着200多种微生物的基因组测序相继完成,对微生物的研究已经进入后基因组时代即功能基因组时代。许多致病菌全基因组信息的获得以及生物信息学的发展使得疫苗学的研发进入了一个新的时代。现在可以应用逆向疫苗学的方法,从一个微生物的基因序列开始筛选出疫苗候选基因。逆向疫苗学首先应用于B群脑膜炎奈瑟菌[19],现在已经在多种微生物中应用,如肺炎链球菌、钩端螺旋体,无乳链球菌、金黄色葡萄球菌和肺炎衣原体等[20]。逆向疫苗学研究的策略和方法主要有生物信息学、基因组学、转录组学和蛋白质组学等。生物信息学分析是逆向疫苗学研究的第一个重要策略。衣原体的蛋白质具有5种亚细胞定位,分别为胞质、内膜、周质、外膜以及细胞外分泌蛋白质。表面暴露蛋白质是指位于内膜、周质、外膜和细胞外的蛋白质,因为它们更易于与抗体识别,从而可能会引起保护性免疫反应,因此表面暴露蛋白是合适的疫苗候选分子。现在已经有许多生物信息学软件根据蛋白质的序列分析预测它们在微生物中的亚细胞定位情况[8,21,22]。逆向疫苗学研究的第二个重要策略是应用芯片技术分析进行基因的转录组学研究。荧光信号值高于中位值以上的基因代表着其在基因组中的转录超过5-10个mRNA拷贝[23]。这些高表达的基因可以作为疫苗的候选基因。此外,蛋白质组学等方法也可以用于逆向疫苗学筛选合适的疫苗候选分子。联合使用这些研究方法,可以最终筛选鉴定到编码潜在的疫苗抗原的基因。
在本课题的研究中,我们首先应用P-CLASSIFIER程序预测分析衣原体L2b/UCH-1株中所有蛋白质的亚细胞定位,P-CLASSIFIER是一个基于蛋白质氨基酸序列以及多重支持向量机(support vector machine,SVM)的预测蛋白质亚细胞定位的程序[8]。此外,我们又应用相应的生物信息学程序预测衣原体蛋白质中具有α-穿膜螺旋、β-桶形结构、信号肽以及脂蛋白等4种特征结构的蛋白质。根据PCLASSIFIER程序预测分析的结果,衣原体中同时为表面暴露蛋白质而且具有上述4种特征结构(α-穿膜螺旋、β-桶形结构、信号肽以及脂蛋白)之一的蛋白质作为疫苗候选分子的初选。同时,去除掉那些具有4个以上α-穿膜螺旋的蛋白质,这是因为具有4个以上α-穿膜螺旋的蛋白质会完全包埋在细胞膜内而不容易接触到抗体,而且因为它们难以在大肠埃希菌中进行表达。通过BLAST程序,我们保留在衣原体L2b/UCH-1株基因组中的保守基因并去除衣原体L2b/UCH-1株与人同源的蛋白质,因为衣原体L2b/UCH-1株中与人同源的蛋白质有可能会引起自身免疫反应。最终,我们通过生物信息学分析,衣原体疫苗候选基因的范围缩小到73个基因。
为了初步验证我们预测结果的可靠性,我们挑选出表达量较高的四个基因,进行体外表达纯化,并免疫BALB/c小鼠。通过体外抗体滴度实验验证,CTLon_0288表达的蛋白免疫小鼠后产生了滴度较高的抗体。
本研究通过综合生物信息学的方法,成功预测出沙眼衣原体疫苗候选基因。通过实时定量PCR挑取其中表达量相对较高的基因进行分析,并进行了免疫动物和抗体效价的测定,证实逆向疫苗学理论在沙眼衣原体疫苗的抗原筛选工作中是可行的,为下一步寻找有效的候选抗原打下了良好的基础。
[1]Hafner LM.Pathogenesis of fallopian tube damage caused by Chlamydia trachomatis infections[J].Contraception,2015,92(2):108-115.
[2]Lewis DA,Latif AS,Ndowa F.WHO global strategy for the prevention and control of sexually transmitted infections:time for action[J].Sex Transm Infect,2007,83(7):508-509.
[3]Banks J,Schachter J.Serotyping of Chlamydia:antibodies to lymphogranuloma venereum strains compared by microimmunofluorescence and neutralization tests[J].Infect Immun,1978,20(3):864-866.
[4]Chudzicka-Struqata I,Karpinski TM,Zeidler A,et al.Occurrence of Chlamydia trachomatis infections in infertile women in Poland[J].Ginekol Pol,2014,85(11):843-846.
[5]Peipert JF.Clinical practice.Genital chlamydial infections[J].N Engl J Med,2003,349(25):2424-2430.
[6]李伟,张宁洁,余平.Toll7/8/9及其激动剂在抗衣原体感染免疫应答中的作用[C].重庆:第八届全国免疫学学术大会论文集,2012:313-314.
[7]Farris CM,Morrison RP.Vaccination against Chlamydia genital infection utilizing the murine C.muridarum model[J].Infect Immun,2011,79(3):986-996.
[8]Wang J,Sung WK,Krishnan A,et al.Protein subcellular localization prediction for Gram-negative bacteria using amino acid subalphabets and a combination of multiple support vector machines[J].BMC Bioinformatics,2005,13(6):174.
[9]Bendtsen JD,Nielsen H,von Heijne G,et al.Improved prediction of signal peptides:SignalP 3.0[J].J Mol Biol,2004,340(4):783-795.
[10]Krogh A,Larsson B,von Heijne G,et al.Predicting transmembrane protein topology with a hidden Markov model:application to complete genomes[J].J Mol Biol,2001,305(3):567-580.
[11]Berven FS,Flikka K,Jensen HB,et al.BOMP:a program to predict integral beta-barrel outer membrane proteins encoded within genomes of Gram-negative bacteria[J].Nucl Acid Res,2004,32:W394-399.
[12]Setubal JC,Reis M,Matsunaga J,et al.Lipoprotein computational prediction in spirochaetal genomes[J].Microbiology,2006,152(Pt 1):113-121.
[13]Altschul SF,Madden TL,Schaffer AA,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucl Acid Res,1997,25(17):3389-3402.
[14]Martinez R,Perez A,Quinones Mdel C,et al.Efficacy and safety of a vaccine against human leptospirosis in Cuba[J].Rev Panam Salud Publica,2004,15(4):249-255.
[15]Yan Y,Chen Y,Liou W,et al.An evaluation of the serological and epidemiological effects of the outer envelope vaccine to leptospira[J].J Chin Med Assoc,2003,66(4):224-230.
[16]Rey-Ladino J,Ross AG,Cripps AW.Immunity,immunopathology,and human vaccine development against sexually transmitted Chlamydia trachomatis[J].Hum Vaccin Immunother,2014,10(9):2664-2673.
[17]Vasilevsky S,Greub G,Nardelli-Haefliger D,et al.Genital Chlamydia trachomatis:understanding the roles of innate and adaptive immunity in vaccine research[J].Clin Microbiol Rev,2014,27(2):346-370.
[18]Zuerner R,Haake D,Adler B,et al.Technological advances in the molecular biology of Leptospira[J].J Mol Microbiol Biotechnol,2000,2(4):455-462.
[19]Pizza M,Scarlato V,Masignani V,et al.Identification of vaccine candidates against serogroup B meningococcus by whole-genome sequencing[J].Science,2000,287(5459):1816-1820.
[20]Serruto D,Adu-Bobie J,Capecchi B,et al.Biotechnology and vaccines:application of functional genomics to Neisseria meningitidis and other bacterial pathogens[J].J Biotechnol,2004,113(1-3):15-32.
[21]Gardy JL,Laird MR,Chen F,et al.PSORTb v.2.0:expanded prediction of bacterial protein subcellular localization and insights gained from comparative proteome analysis[J].Bioinformatics,2005,21(5):617-623.
[22]Yu CS,Lin CJ,Hwang JK.Predicting subcellular localization of proteins for Gram-negative bacteria by support vector machines based on n-peptide compositions[J].Protein Sci,2004,13(5):1402-1406.
[23]Allan E,Wren BW.Genes to genetic immunization:identification of bacterial vaccine candidates[J].Methods,2003,31(3):193-198.

