淋球菌rpsE基因突变与大观霉素耐药性的研究
朱文 蒋法兴 苏晓红 乐文静 王娜
·论著·
淋球菌rpsE基因突变与大观霉素耐药性的研究
朱文 蒋法兴 苏晓红 乐文静 王娜
目的探讨淋球菌rpsE基因突变与淋球菌大观霉素耐药的相关性。方法对临床分离的4株大观霉素耐药的淋球菌株(MIC128 μg/ml、256 μg/ml)的rpsE基因进行PCR扩增测序分析,寻找可能的突变位点,通过DNA转化技术将含有突变基因的细菌基因组DNA转化入敏感的淋球菌株,检测转化成功的淋球菌的MIC并进行PCR扩增测序,分析发现的突变位点与淋球菌大观霉素耐药的相关性。结果4株大观霉素耐药的菌株均发现rpsE基因A70C(Thr24Pro)突变,而16S rRNA大观霉素耐药决定区(SRDR)未发现任何突变;大观霉素敏感菌株的16S rRNA和rpsE基因均未发现突变。转化后获得的大观霉素耐药淋球菌中亦发现了同样的rpsE基因突变。结论rpsE基因单点突变与淋球菌对大观霉素耐药相关。
淋病奈瑟球菌;基因,rpsE;点突变;大观霉素;抗药性
淋球菌是最常见的性传播感染的病原体之一。由于至今仍没有研制出淋球菌疫苗,及时诊断和早期抗生素治疗淋病患者是目前控制淋球菌传播的有效方法。我国目前仅推荐头抱曲松与大观霉素用于治疗淋球菌感染,头抱曲松可能会出现过敏反应并且已分离出对头抱曲松耐药的淋球菌株[1],使得大观霉素对淋病的治疗更为重要。随着大观霉素的广泛应用,大观霉素耐药淋球菌也相继出现。苏晓红等[2]于2003年报告了大观霉素治疗淋病失败3例原因分析,其中有2例为大观霉素耐药菌株所致。研究表明,rpsE基因编码的核糖体5S蛋白位于大观霉素结合部位附近,该基因发生突变可能导致大观霉素耐药菌株的形成。本研究对南京地区和合肥地区分离的4株大观霉素耐药淋球菌(MIC≥128 μg/ml)的16S rRNA大观霉素耐药决定区(spectinomycin resistance-determining region,SRDR)和 rpsE 基因进行PCR扩增、测序分析,并将含有突变基因的淋球菌基因组DNA转入敏感的淋球菌株,探讨rpsE基因单点突变与淋球菌对大观霉素耐药的相关性。
资料与方法
一、资料
1.试验菌株(表1):4株大观霉素耐药的淋球菌和2株大观霉素敏感的淋球菌。其中2株大观霉素耐药的菌株(06-3,06-5)和2株大观霉素敏感的(12-11358,11-8679)收集于2004—2012年中国医学科学院皮肤病研究所性病门诊中有症状的淋病患者,另外2株(G1,G2)收集于2007—2013年安徽省立医院皮肤性病科门诊。所有菌株均经过革兰染色显微镜镜检、氧化酶实验、糖发酵实验鉴定为淋球菌。菌株于-70℃保存于20%甘油肉汤(TSB-Gly)备用。
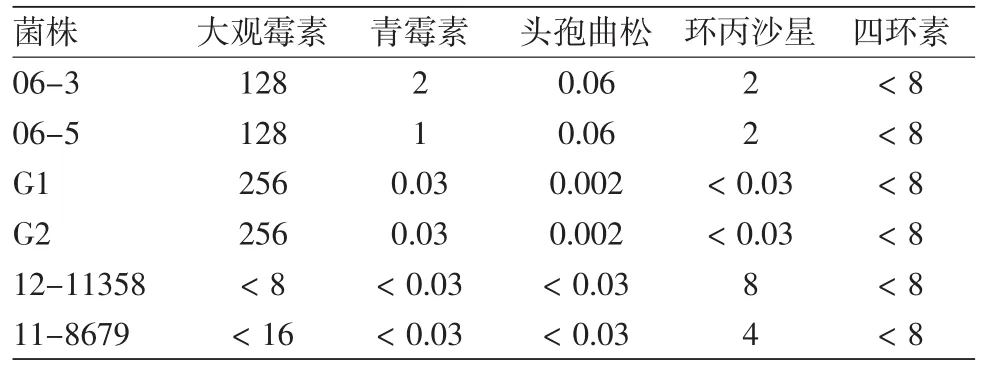
表1 临床分离淋球菌对抗生素的MIC μg/ml
2.培养基与培养条件:①固体培养基:淋球菌分离培养基购自珠海银科医学工程公司。后续的琼脂稀释法MIC实验使用的是GC巧克力培养基(GC琼脂基础18.5 g+血红蛋白粉5.5 g)溶于500 ml体积双蒸水中,20 min,121℃高压灭菌后冷却至50℃时加入增菌剂Vitox Supplement 10 ml,4℃保存,1周内用完。淋球菌均在37℃,5%CO2环境下培养18~24 h;②液体培养基:淋球菌的转化实验使用淋球菌液体培养基(15 g示蛋白胨,4 g K2HPO4,1 g KH2PO4,1 g NaCl)溶于 1 L 双蒸水中,20 min,121 ℃高压灭菌后冷却至50℃时加入增菌补充液Ⅰ10 ml和补充液Ⅱ1 ml。补充液Ⅰ:葡萄糖40 g,谷氨酰胺1 g,硫胺素焦磷酸2 mg溶于100 ml体积双蒸水中,补充液Ⅱ:Fe(NO3)3-9H2O,50 mg溶于100 ml双蒸水中,两种补充液均在生物安全柜通过0.2 μm滤菌器消毒灭菌,-20℃保存备用。
二、方法
1.药物敏感性实验:实验药物均购于美国Sigma公司。淋球菌的药物敏感性试验采用琼脂稀释法,以倍比稀释法制备含不同浓度大观霉素的琼脂培养基(8~512 μg/ml)。以淋球菌ATCC49226作为药敏质控菌株,以美国临床和实验室标准协会(CLSI)推荐的标准判断结果,即:MIC≥ 128 μg/ml为耐药,MIC 64 μg/ml为中度敏感,MIC ≤ 32 μg/ml为敏感。青霉素、头抱曲松、环丙沙星、四环素的药敏试验结果见表1。均由中国医学科学院皮肤病研究所提供。
2.细菌DNA的提取:淋球菌基因组DNA的提取采用BuccalAmpTMDNA试剂盒(美国Epicentre BioTechnologies公司),提取的DNA于4℃保存用于PCR反应,提取的G1菌的基因组DNA用于转化实验。
3.PCR扩增rpsE全基因片段:上游引物为5'-TGAGAAGGCTAAAGCAGCAGGTGT-3',下游引物为 5'-CGTGCACATGCACGATGAGATTCA-3',扩增产物为包括rpsE全基因在内的727 bp片段;扩增16SrRNA基因的大观霉素耐药决定区(SRDR)相关片段:采用Galimand等[3]和基于淋球菌全基因组序列FA1090(AE004969)设计的引物,上游引物为5'-CTTACCTGGTCTTGACA-3',下游引物为 5'-CGATTACTAGCGATTCC-3',扩增产物为16SrRNA基因上包括SRDR在内的373 bp片段;PCR反应总体系 50 μl,其中 Taq 酶 1.5 U,dNTP(各 2.5 mmol/L)4 μl,模板 DNA 2 μl,引物各 1 μl(0.4 μmol/L)。反应条件:94℃预变性3 min,94℃变性30 s,55℃退火30 s,72℃延伸1 min共30个循环,最后72℃延伸5 min。以双蒸水为模板的扩增体系作阴性对照,大观霉素耐药菌株WHOA作阳性对照。扩增产物经2%的琼脂糖凝胶电泳,紫外透射仪下观察结果,美国Eager eye图像分析系统扫描照相。PCR扩增产物测序分析委托上海生工生物工程技术服务有限公司完成。
4.DNA转化实验参照文献[4],主要步骤如下:①取20 μl供体菌G1的基因组DNA加入200 μl含有5 mmol/L MgSO4的GCBL中,涡旋5 s后37℃预热备用;②接种环刮取复苏20 h受体菌11-8679培养皿,上1/2的菌落悬浮于1 ml 37℃预热的GCBL中,涡旋备用;③涡旋后的受体菌液20 μl加入制备的DNA溶液中,37℃,15 min混合;④制备的全部转化液加入2 ml GCBL中,培养管半盖,37℃,5%CO2环境下孵育4 h;⑤孵育的菌液0.1 ml与37℃预热的GCBL 0.9 ml进行10倍系列稀释(10~10-6),分别移取上述稀释后的菌液100 μl涂布到含大观霉素的GC巧克力琼脂培养基以及无抗生素平板上,37℃,5%CO2环境下孵育48 h后进行菌落计数,转化实验重复3次,统计各个平板菌落数,计算转化率;与此同时我们用12-11358作为受体菌,重复上述转化实验。转化子均在128 μg/ml大观霉素平板上再次培养18~24 h,以确定转化成功,于-70℃保存,并提取转化子DNA、PCR扩增及测序。
结 果
1.琼脂稀释法大观霉素MIC实验:4株淋球菌为大观霉素耐药(MIC≥ 128 μg/ml),另两株为大观霉素敏感菌(MIC≤ 32 μg/ml)。见表2。
2.PCR产物测序结果序列比对:将测序结果与质控敏感菌株ATCC49226的16SrRNA和rpsE基因序列进行BLAST比对分析,应用DNAstar Lasergene.(version 7.1)软件进行核苷酸序列和氨基酸序列比对:发现4株大观霉素MIC≥128 μg/ml的淋球菌均发生rpsE基因的单点突变,即70位碱基A→C,其编码的核糖体5S蛋白的第24位氨基酸由苏氨酸转变为脯氨酸(图1)。而2株大观霉素MIC≤ 32 μg/ml的敏感菌株均未发生16SrRNA与rpsE基因的突变。
3.转化实验将含有rpsE突变基因A70C的淋球菌基因组DNA转化入受体菌(11-8679,12-113658)后,其对大观霉素的MIC值均增长到>128 μg/ml的耐药水平,在稀释度10-1大观霉素128 μg/ml的平板上分别有28.7±4.2个和13±4.6个菌落出现。PCR扩增转化子16SrRNA基因的大观霉素耐药决定区(SRDR)相关片段,与受体菌比较未发生任何突变;而转化子的rpsE基因发生单点突变,即70位碱基A→C。另外,对于两株受体菌(11-8679,12-113658)其转化率分别为1.1±0.4×10-6和1.5±0.5×10-6提示淋球菌对大观霉素耐药性在菌群中传播的风险是比较高的。
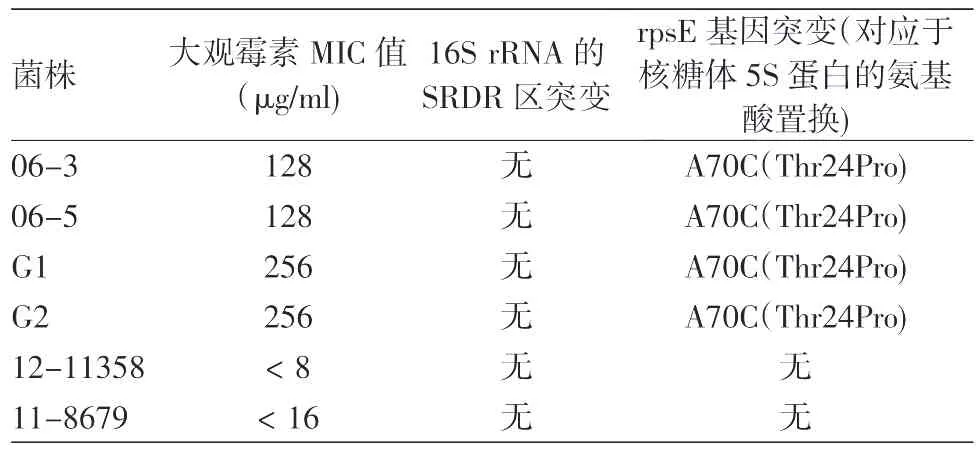
表2 临床分离淋球菌株的大观霉素MIC值与菌株的突变模式(以ATCC49226为对照)
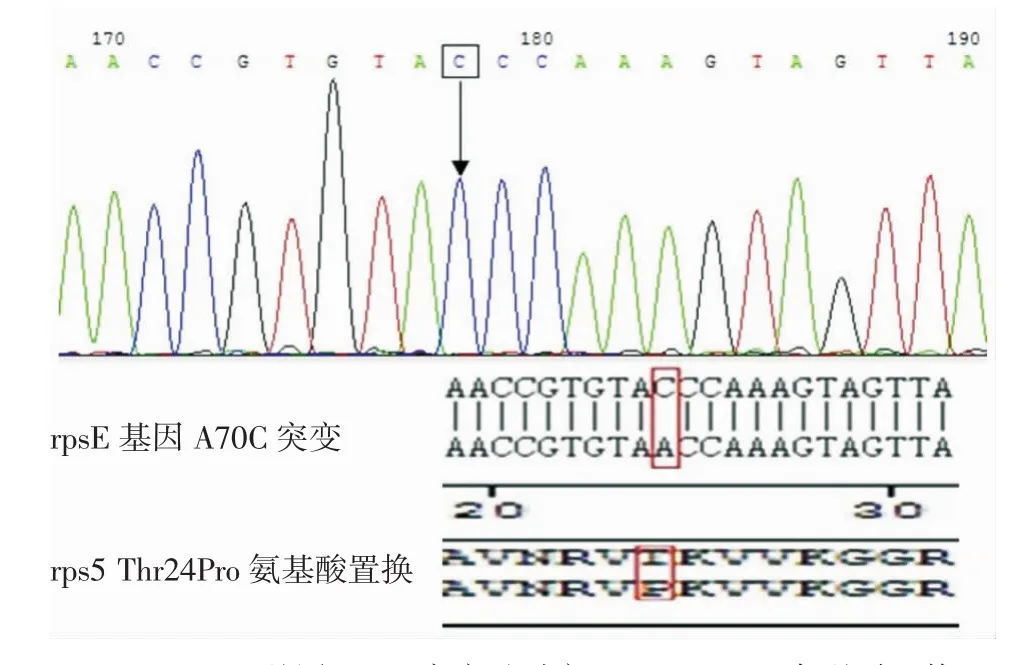
图1 rpsE基因A70C突变及对应rps5 Thr24Pro氨基酸置换
讨 论
大观霉素为链霉菌所产生的一种氨基环醇类抗生素,其抑菌的作用机制是与细菌30S核糖体亚基结合从而抑制蛋白质的合成,即大观霉素直接作用于16S rRNA,在多肽延长的过程中抑制延长因子G(EF-G)的催化和转位,阻止肽酰tRNA从A位点向P位点移动[5-6]。研究表明,大观霉素在16SrRNA上的作用位点位于靠近G1064-C1192的34螺旋上[7]。细菌对大观霉素耐药的机制:①腺苷酰转移酶将大观霉素腺苷酰化而灭活,未发现淋球菌对大观霉素耐药与腺苷酰转化酶灭活机制相关;②rpsE基因编码的核糖体5S蛋白中2螺旋的氨基酸置换;③16SrRNA的大观霉素结合区即螺旋34包含1063-1066和1190-1193的交联区域的基因突变[8-9]。在淋球菌中,16SrRNA基因的单位点突变C1192U已证实可导致高水平的大观霉素耐药[3,10]。Unemo等[11]的研究认为,淋球菌rpsE基因突变对应的核糖体5S蛋白的25位氨基酸的缺失和K26E的氨基酸置换可导致高水平的大观霉素耐药。虽然核糖体5S蛋白不在大观霉素结合区域,但它非常接近抗生素结合位(5A)[12],核糖体5S蛋白19~33环是RNA结合部位,也是与大观霉素耐药有关突变最频繁的区域[12],因此,核糖体5S蛋白上Thr24Pro氨基酸的置换可能会影响5S蛋白与16SrRNA 34螺旋的作用,从而参与淋球菌对大观霉素耐药性的形成。本研究发现,4株大观霉素MIC≥ 128 μg/ml的淋球菌显示出rpsE基因的单点突变(A70C),将含有rpsE突变基因A70C的淋球菌基因组DNA转化入受体菌后,转化子对大观霉素MIC增长到耐药水平,并且发生了同样的rpsE基因突变,该突变对应的核糖体5S蛋白氨基酸置换(Thr24Pro)导致了淋球菌对大观霉素耐药性的产生。
目前,淋球菌已对多种抗菌药物产生了不同程度的耐药性,且随着抗菌药物的长期使用及不合理使用,耐药性呈上升趋势,给淋病的治疗和预防带来困难。2011年7月,Ohnishi等[13]报道日本新发现的耐药菌株H041,该菌株对除大观霉素以外其他所有抗菌药物有耐药性。美国和挪威也相继发现了这种耐药菌株。到目前为止,尽管偶尔检出耐大观霉素的淋球菌[14],但对包括我国在内的大多数国家来说,大观霉素仍可作为治疗淋病的一线药物,同时应加强对淋球菌耐药机制的研究以应对不断严重的耐药形势。
[1]Tapsall JW,Ndowa F,Lewis DA,et al.Meeting the public health challenge of multidrug-and extensively drug-resistantNeisseria gonorrhoeae[J].Expert Rev Anti Infect Ther,2009,7(7):821-834.
[2]苏晓红,张传福,张津萍.大观霉素治疗淋病失败3例原因分析[J].中华皮肤科杂志,2003,36(5):285.
[3]Galimand M,Gerbaud G,Courvalin P.Spectinomycin resistance inNeisseria spp.due to mutations in 16S rRNA[J].Antimicrob Agents Chemother,2000,44(5):1365-1366.
[4]Dillard JP.Genetic manipulation ofNeisseria gonorrhoeae[J].Curr Protoc Microbiol,2011,Chapter 4:Unit4A.2.
[5]Carter AP,Clemons WM,Brodersen DE,et al.Functional insights from thestructureofthe 30S ribosomalsubunitand its interactions with antibiotics[J].Nature,2000,407(6802):340-348.
[6]Borovinskaya MA,Shoji S,Holton JM,et al.A steric block in translation caused by the antibiotic spectinomycin[J].ACS Chem Biol,2007,2(8):545-552.
[7]Moazed D,Noller HF.Interaction of antibiotics with functional sites in 16S ribosomal RNA[J].Nature,1987,327(6121):389-394.
[8]Sigmund CD,Ettayebi M,Morgan EA.Antibiotic resistance mutations in 16S and 23S ribosomal RNA genes ofEscherichiacoli[J].Nucleic Acids Res,1984,12(11):4653-4663.
[9]Johanson U,Hughes D.A new mutation in 16S rRNA ofEscherichia coliconferring spectinomycin resistance[J].Nucleic Acids Res,1995,23(3):464-466.
[10]Unemo M,Fasth O,Fredlund H,et al.Phenotypic and genetic characterization ofthe 2008 WHONeisseriagonorrhoeaereference strain panel intended for global quality assurance and quality control of gonococcal antimicrobial resistance surveillance for public health purposes[J].J Antimicrob Chemother,2009,63(6):1142-1151.
[11]Unemo M,Golparian D,Skogen V,et al.Neisseria gonorrhoeaestrain with high-level resistance to spectinomycin due to a novel resistance mechanism(mutated ribosomal protein S5)verified in Norway[J].Antimicrob Agents Chemother,2013,57(2):1057-1061.
[12]Wirmer J,Westhof E.Molecular contacts between antibiotics and the 30S ribosomal particle[J].Methods Enzymol,2006,415:180-202.
[13]Ohnishi M,Golparian D,Shimuta K,et al.IsNeisseria gonorrhoeaeinitiating a future era ofuntreatable gonorrhea?:detailed characterization of the first strain with high-level resistance to ceftriaxone[J].Antimicrob Agents Chemother,2011,55(7):3538-3545.
[14]叶顺章,王千秋,苏晓红,等.中国1993~2000年分离淋球菌的流行病学和细菌学特征.中华流行病学杂志,2003,24(2):119-122.
2013-10-21)
(本文编辑:吴晓初)
Mutations in the rpsE gene and spectinomycin resistance inNeisseria gonorrhoeae
Zhu Wen*,Jiang Faxing,Su Xiaohong,Le Wenjing,Wang Na.*Department of Dermatology,Anhui Provincial Hospital Affiliated to Anhui Medical University,Hefei 230001,China
s:Jiang Faxing,Email:jiangfxing@126.com;Su Xiaohong,Email:suxhong@yahoo.com
ObjectiveTo evaluate the relationship between spectinomycin resistance inNeisseria gonorrhoeaeand mutations in the rpsE gene.MethodsGenomic DNA was extracted from 4 clinical isolates ofNeisseria gonorrhoeaewith different levels of spectinomycin resistance.Then,PCR was performed to amplify the entire rpsE gene and the spectinomycin resistance-determining region(SRDR)in the 16S rRNA gene followed by direct sequencing.Two spectinomycin-sensitiveNeisseria gonorrhoeaestrains were transformed with the genomic DNA containing the mutant rpsE gene.Subsequently,the susceptibility of the transformants to spectinomycin was determined,and PCR was performed to amplify the rpsE and 16S rRNA genes in the transformants followed by sequencing.ResultsAll the 4 spectinomycin-resistantNeisseria gonorrhoeaestrains harbored an A70C transversion in the rpsE gene,but no abnormality in the SRDR of the 16S rRNA gene.No mutations were detected in the spectinomycin-sensitiveNeisseria gonorrhoeaestrains.The A70C transversion in the rpsE gene was also detected in the twoNeisseria gonorrhoeaetransformants with spectinomycin resistance.ConclusionThe A70C point mutation within the rpsE gene is associated with spectinomycin resistance inNeisseria gonorrhoeae.
Neisseria gonorrhoeae;Genes,rpsE;Point mutation;Spectinomycin;Drug resistance
10.3760/cma.j.issn.0412-4030.2014.09.001
安徽省自然科学基金(0904131430)
230001合肥,安徽医科大学附属省立医院皮肤科(朱文、蒋法兴、王娜);中国医学科学院北京协和医学院皮肤病研究所(苏晓红、乐文静)
蒋法兴,Email:jiangfxing@126.com;苏晓红,Email:suxhong@yahoo.com

