龙须菜RSAP分析及其SCAR标记的转化*
王津果,隋正红,周 伟,马金华,张 淑,常连鹏
(中国海洋大学海洋生物遗传学与育种教育部重点实验室,山东 青岛266003)
RSAP是由杜晓华于2006年首次提出的一种新型DNA分子标记技术,具有步骤简单、检测位点较多、成本较低等优势,未获得全基因组测序结果以前,在遗传多样性分析、连锁图谱构建和基因定位等方面具有一定的推广应用价值[1]。张明科[2]进一步研究了该技术的引物设计并优化了反应体系。乔利仙[3]与辛本华[4]先后将该技术应用到重楼属植物的遗传多样性分析与红藻门紫菜遗传多样性及种质鉴定方面。
目前,经青岛湛山湾野生种群选育而来的栽培品系981、07-2已在福建南部海区[5]、广东省汕头市南澳岛等地栽培多年[6],并在浙江、山东及辽宁沿海建立了龙须 菜 (Gracilariopsis lemaneiformis)栽 培 产 业[7]。栽培品系由于多年的无性繁殖而引起种质退化,野生资源由于沿岸改造与人为干扰,其分布面积逐渐减少。因此,为适应龙须菜产业化规模不断扩大的现状,如何保护野生资源的遗传多样性并使之可持续利用变得日益重要。本研究以湛山湾野生群体和栽培品系为材料,运用RSAP技术从基因组DNA水平上,分析等位基因及频率、遗传杂合度、多态信息含量等,旨在从分子水平上探讨龙须菜不同群体的的遗传差异,为龙须菜种质资源的保护和可持续利用以及遗传育种工作提供理论依据。
SCAR标记被广泛应用于一些形态上难以区分而且经济价值差异较大的种和品种鉴别及种质评价[8]。本研究通过分析RSAP指纹图谱,以期筛选获得龙须菜生产上的主要苗种来源981的特异条带,并将其转化为稳定可靠、特异性高的SCAR标记。通过利用特异的SCAR标记鉴定优良的龙须菜栽培品系981,可以防止或减少使用不合格品系给龙须菜生产造成的损失。
1 材料与方法
1.1 材料与处理
试验用龙须菜为2010年10月分别采自青岛胶州湾养殖海区和青岛湛山湾的981、07-2和湛山湾野生四分孢子体。将采集来的样品切片,置于显微镜下观察,在皮层细胞中分布有呈十字形分裂的四分孢子囊的藻株即为四分孢子体[9]。选取10株湛山湾野生四分孢子体与栽培品系981、07-2分别2个样品,在海水中用消毒毛刷除掉附着的杂藻和泥砂后,用蒸馏水冲洗1次。
1.2 试验方法
1.2.1 龙须菜基因组DNA的提取 采用天根植物基因组DNA提取试剂盒(TIANGEN BIOTECH,北京)提取龙须菜的基因组DNA,紫外分光仪测定浓度,1%琼脂糖电泳检测DNA分子的完整性。
1.2.2 RSAP分析 RSAP的分析参照杜晓华等[1]的方法并作了适当的改进。其中引物由上海生工技术有限公司合成,引物序列如表1。6个引物可分别与其它任何1个引物组合而得到15对引物,利用这15对引物对14个龙须菜样品DNA进行PCR扩增。20μL PCR反应体系中各反应物含量:2μL 10×PCR buffer,30ng 基 因 组 DNA,2.5mmol·L-1MgCl2,0.15 mmol·L-1dNTPs,0.4μmol·L-1引 物,2UTaq DNA聚合酶。PCR反应在杭州朗基96孔精品型PCR仪上进行,反应程序为:94℃变性5min,前5个循环为94℃变性1min,35℃退火1min,72℃延伸1min;接下来30个循环为:94℃变性1min,46℃退火1min,72℃延伸1min;最后72℃延伸5min。结束后产物进行4℃保存。
扩增产物通过6%的变性聚丙烯酰胺凝胶电泳(Polyacrylamide gel electrophoresis,PAGE)进 行 分析,银染[10]。

表1 RSAP分析与SCAR验证所用的引物Table 1Information of the primers used in RSAP analysis and SCAR verification
1.2.3 RSAP数据分析 人工观察统计电泳条带,以扩增清晰的带为标准进行标记分析,将电泳图谱中的每一条带的迁移位置记为一个位点,相同迁移位置的扩增带出现时记为1,缺失和模糊不清时记为0,构建0-1矩阵。运用PopGen[11]分析种群内的遗传多样性:观测到的等位基因数,Na、有效等位基因数,Ne、平均基因多样性指数,H以及Shannon多样性信息指数,I;种群间的遗传多样性:种群内总的基因多样性,Ht、种群内的基因多样性,Hs、种群间的基因分化度,Gst、基因流,Nm;计算出遗传距离,D,采用MEGA5.0软件按UPGMA法进行聚类。
1.2.4 981特异条带目的回收与测序 将筛选到的特异条带,按照 Poly-gel DNA extraction kit(OMEGA,上海)说明进行聚丙烯酰胺凝胶胶回收。以回收回来的DNA片段为模板,进行20μL×5的放大PCR,扩增程序参照RSAP的反应程序。放大PCR的产物经1.5%的琼脂糖电泳检测,100μL PCR产物琼脂糖电泳后,运用 TaKaRa Agarose Gel DNA Purification Kit按照说明书进行回收。
目的片段的连接按照pMDTM18-T Vector说明书进行连接。转化后平板培养,随机挑取10个阳性克隆进行目的片段检测。阳性克隆在1mL含Amp的LB培养基中培养4h,加终浓度20%甘油,经由上海生工3730测序仪进行单向测序。
1.2.5 SCAR引物的设计与验证 使用NCBI的Vec Screen对测序后的DNA片段在线去除载体序列,然后在GenBank里面比对是否有同源序列。此外,将整理过的序列载入软件Primer Premier 5.0,根据引物设计原则,在序列两端设计特异引物,并送上海生工合成。参照1.1提取981、07-2各2株和30株湛山湾野生四分孢子体的基因组DNA,确定SCAR引物的退火温度并进行验证PCR,程序如下:94℃变性5min;94℃变性1min,54℃(SCAR引物1)/65℃(SCAR引物2)退火1min,72℃延伸1min,30个循环;72℃延伸10min,4℃保存。1.5%琼脂糖凝胶电泳进行目的片段的检测。
2 结果与分析
2.1 RSAP扩增结果
15对引物在10株湛山湾野生四分孢子体、2个981样品、2个07-2样品共计14个样品中扩增出669个位点。龙须菜14个样品的RSAP聚丙烯酰胺凝胶电泳扩增条带情况见图1。

图1 龙须菜14个样品的RSAP聚丙烯酰胺凝胶电泳扩增结果图示Fig.1 RSAP amplification result of 14samples of G.lemaneiformis resolved by PAGE analysis
由图1可见,青岛湛山湾野生四分孢子体与栽培品系981、07-2的RSAP扩增片段大小在100~1 000bp,群体间差异条带少,群体内各个体间的差异条带更少。
2.2 龙须菜各群体内的RSAP分析
湛山湾野生和栽培品系981、07-2种群内的平均观测等位基因数(Na)、有效等位基因数(Ne)、平均基因多样性指数即平均杂合度(H)以及Shannon多样性信息指数(I)、多态性位点比率(P/%)见表2。

表2 各群体内的遗传多样性Table 2 The diversity of every group
由表2可以看出,各群体内多态性位点比率(P/%)都很低,最低的为栽培品系981,多态性比率不到0.2%,栽培品系07-2多态性位点比率最高,为0.9%,这反应了龙须菜各群体内部遗传变异水平是非常低的。各群体的有效等位基因数Ne在1.002 1~1.007 1之间,湛山湾野生群体与栽培品系有效等位基因数差别不大,有效等位基因数最大的是湛山湾野生群体,达到了1.007 1。各群体内的基因多样性指数H在0.001 2~0.003 7之间,该指数的值也是较低的。Shannon指数I,分布在0.001 8~0.005 4之间,最大的为栽培品系07-2。I值表示群体的基因变异程度,各群体的I值普遍很低,说明了各群体的基因变异程度低。各群体的有效等位基因数Ne和基因多样性指数H 以及Shannon指数I的变化趋势是一致的。
2.3 湛山湾野生与栽培品系981、07-2种群间RSAP分析
种群内总的基因多样性Ht、种群内的基因多样性Hs、种群间的基因分化度Gst、基因流Nm见表3。所有群体内总的基因多样性Ht为0.095 3,大于各群体内的基因多样性系数 Hs为(0.002 9),而湛山湾野生与2个栽培品系群体之间两两分析结果显示,任何2个群体的总基因多样性均小于0.01。由此可见,各群体间的基因多样性主要来自湛山湾的样品和栽培品系样品之间的差异。所有群体间的基因流为0.015 5,小于1,表现出很低的基因流动性,表明群体间几乎不存在基因流动。各群体间的基因分化度Gst也均大于0.5,说明在所有检测的样品中,遗传多样性多来自不同群体间的多样性。
湛山湾野生与2个栽培品系981、07-2群体个体间的遗传距离见表4,利用UPGMA方法对龙须菜野生群体与2个栽培品系进行聚类分析,相应的UPGMA聚类树见图2。

图2 龙须菜群内总的UPGMA法遗传聚类图Fig.2 The UPGMA dendrogrom of all the strains of G.lemaneiformis using genetic distance
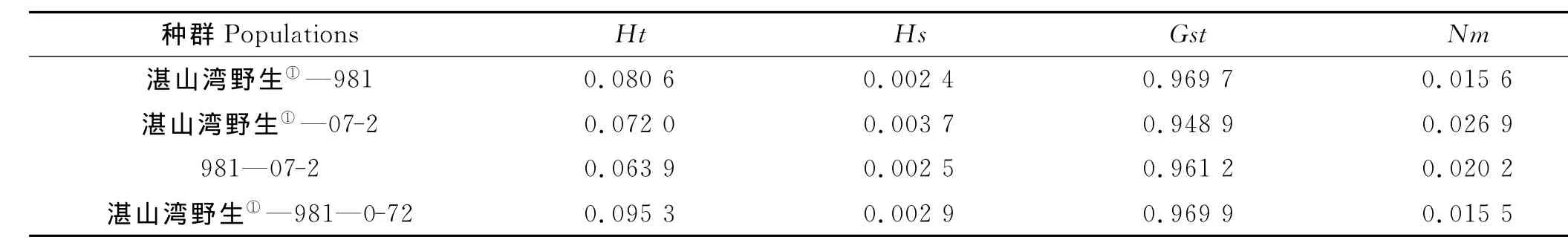
表3 种群间的基因多样性Table 3 Gene diversity of inter-groups of G.lemaneiformis

表4 14个个体间的遗传距离Table 4 Genetic distance between 14 G.lemaneiformis
由图2可以看出,1~10号样品为一个群体,11~12号样品为一个群体,13~14号样品为一个群体,与14个样品分属于3个不同群体相一致,1~10号为属于湛山湾野生群体,11~12号属于栽培品系981、13~14号属于栽培品系07-2。由聚类图可以看出,各群体间的遗传距离小,群体间遗传距离最长在0.17左右,而群体内的遗传距离均低于0.01。
2.4 目的片段的回收、测序以及SCAR引物设计与验证

图3 SCAR引物2对龙须菜新个体的验证PCR电泳图Fig.3 Agarose gel profile of G.lemaneiformis individuals amplified with the SCAR primer 2
将筛选得到的栽培品系981特异的片段回收测序后,得到大小分别为620bp、542bp的2条片段。在序列两端设计SCAR引物,引物信息见表1。SCAR引物2对981、07-2各3株和30株湛山湾野生四分孢子体共计36株龙须菜的扩增结果如图3,扩增片段大小为442bp,SCAR引物1扩增结果与SCAR引物2扩增结果一致,片段大小为616bp。结果表明,2对特异SCAR引物对981基因组DNA可以扩增出预期大小的特异条带,而在07-2和湛山湾野生四分孢子体中均无扩增条带。
3 讨论
RSAP技术的PCR扩增程序借鉴了SRAP[12]技术,引物的设计借鉴了RAPD[13]随机序列的模糊匹配原理。从操作步骤上讲,RSAP技术是AFLP[14]的极大简化;从基因组的扩增区域上讲,RSAP是对SRAP和TRAP[15]的有效补充。因为后两者主要针对单拷贝区扩增,而RSAP扩增的限制性位点则散布于整个基因组区(包括重复区和单拷贝区)。RSAP标记技术是一个操作简单、产率中等、可靠稳定、适用性较广的一种分子标记技术,已经在植物的多种研究中成功应用[1-4]。
采用1条RSAP引物,无论含6bp或含4bp的限制性位点识别序列的引物进行PCR扩增,均不能扩增或很少扩增出条带;而采用2条不同的引物组合进行PCR扩增时,可获得丰富的扩增谱带[1]。因此本试验中仅采用了由6条引物组合而成的15对引物。另外,在RSAP条带统计过程中,本研究只对条带的出现与否进行了统计,而未对条带的强弱进行分析,实际扩增条带的强弱在一定程度上也能反映DNA水平的变化,如拷贝数的多少等[16]。而在相关文献中,把扩增条带的强度差异作为区分不同样本的报道并不多见,因此本试验中进行RSAP条带统计时,也只将条带的有无作为是否多态性位点的标准。
RSAP结果分析表明,湛山湾二倍体野生群体与两个栽培品系981、07-2,各群体内的多态性比率都非常低,最低的栽培品系981的仅为0.15%,而湛山湾野生群体与栽培品系07-2的都低于1%,比Pang等[17]分析的青岛湛山湾二倍体的龙须菜多态性比率(14.7%)和丁弘叶等[18]研究所得的青岛湛山湾二倍体的龙须菜多态性比率(33.3%)低;同时各群体的有效等位基因数也很少,表示群体基因变异程度的Shannon指数I也非常低,湛山湾野生群体的仅为0.005 1,比Pang等[17]与丁弘叶等[18]的相应数值要低一个数量级甚至更多。以上数据都反映出本试验中各群体的基因变异程度非常低,这是由于所采用的分子标记技术RSAP是基于限制性内切酶酶切位点的多态性标记,基因发生突变的概率本身就很低,如果是特定位点的突变频率会更低,这就直接导致了在分析同一物种不同群体遗传多样性时会出现很低的情况。这也说明了同一物种的遗传多样性检测值与所采用的分子标记技术相关。
另外,群体间的RSAP分析结果显示,野生群体与两个栽培品系间的基因流为0.015 5,小于1,基因流动性很低,表明群体间几乎不存在基因流动。两两群体间的基因分化度Gst也均大于0.5接近1,说明各群体间的基因分化程度大。各群体间的遗传距离很小,遗传相似系数很大,尽管2个栽培品系981、07-2间遗传距离不大,但栽培品系与湛山湾野生群体间的遗传距离相对较大,而UPGMA聚类分析也明显将湛山湾野生群体与栽培品系981、07-2区分开来,表明野生群体与栽培品系间已产生一定的遗传隔阂。
本试验中RSAP分析的群体内及群体间遗传多样性系数均都很低,也是由于材料本身的原因:龙须菜产生的是不动精子,不能进行远距离受精;湛山湾二倍体野生群体中的个体均于2010年10月采自青岛湛山湾,时空的一致性导致了所采集个体几乎不与外来个体间发生杂交互换,缺乏基因的流动;栽培品系981最初是由湛山湾野生个体通过人工选择而来的,07-2则是在981基础上进一步经过人工诱变选育而来,在生产实践中一直都通过营养繁殖进行栽培,造成了龙须菜群体内由于缺乏等位基因的互换而遗传多样性低。针对龙须菜遗传多样性低的现状,一方面有必要通过保护其特殊生长环境保护龙须菜的野生资源,另一方面加快其遗传育种进度,同时建立种质库,进而更好地实现对现有龙须菜种群遗传资源的保护。
目前用SCAR标记进行农作物种质鉴定在国际上已普遍展开[19]。在国内藻类方面,SCAR标记已被广泛地应用于紫菜的种质鉴定[20-22],同时在龙须菜的种质鉴定方面也得到了应用[18]。栽培品系981生长快速,分支多,耐高温,速生抗逆,琼胶含量高且好,属主要的栽培品种,是从青岛湛山湾野生种群经过诱变选育而来[23],与栽培品系07-2、野生个体在外观上没有明显的区别,但在生长性状及生化特性上差异显著[24],而长期以来并没有很好的便捷的手段或方法区分。本试验中,在对龙须菜野生种群与栽培品系981、07-2进行RSAP遗传多样性分析过程中,发现6条栽培品系981的特异条带,并将其中两条带成功转化为981特异的SCAR标记。该SCAR标记将在生产上广泛应用的栽培品系981与其他龙须菜材料区分,保证栽培苗种的纯度,避免使用不合格苗种给龙须菜栽培产业造成损失,进而更有效地进行大规模的人工养殖和满足科研需要。
[1] 杜晓华,王得元,巩振辉.一种新型DNA标记技术—限制性位点扩增多态性(RSAP)的建立与优化 [J].西北农林科技大学学报:自然科学报,2006,34(9):45-50.
[2] 张明科,张鲁刚,巩振辉,等.RSAP标记技术新引物设计及其反应体系的优化 [J].西北农林科技大学学报:自然科学版,2009,37(2):148-154.
[3] 乔利仙,翁曼丽,孔凡娜,等.RSAP标记技术在紫菜遗传多样性检测及种质鉴定中的应用 [J].中国海洋大学学报:自然科学报,2007,37(6):951-956.
[4] 辛本华,田孟良,吴镔锣,等.重楼属植物遗传多样性的RSAP标记 [J].中国中药杂志,2011,36(24):3425-3427.
[5] 彭长连,温学,林植芳,等.龙须菜对海水氮磷富营养化的响应[J].植物生态学报,2007,31(3):505-512.
[6] 汤坤贤,游秀萍,林亚森,等.龙须菜对富营养化海水的生物修复[J].生态学报,2005,25(11):3044-3051.
[7] 张学成,费修绠,王广策,等.江蓠属海藻龙须菜的基础研究与大规模栽培 [J].中国海洋大学学报:自然科学版,2009,39(5):947-954.
[8] Mikhailova N A,Gracheva Y A,Backeljau T,et al.A potential species-specific molecular marker suggests interspecific hybridization between sibling species Littorina arcane and L.saxatilis(Mollusca,Caenogastropoda)in natural populations[J].Genetica,2009,137(3):333-340.
[9] 张学成,秦松,马家海,等.海藻遗传学 [M].北京:中国农业出版社,2004:236-241.
[10] 丁弘叶,隋正红,仲洁,等.青岛不同野生群体的单倍体和二倍体龙须菜的AFLP遗传多样性分析与比较 [J].中国海洋大学学报:自然科学版,2012,42(1-2):99-105.
[11] Yen F C,Yang R C,Boyle T.PopGene,the User Friendly Shareware or population genetic analysis[S].Edmonton,Canada:Molecular Biology and Biotechnology Center,University of Alberta,1997,10.
[12] Li G,Quiros C F.Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J].Theor Appl Genet,2001,103(2-3):455-461.
[13] Williams J G K,Kubelik A R,Livak K J,et al.DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J].Nucleic Acids Res,1990,18(22):653l-6535.
[14] Vos P,Hogers R,Bleeker M,et a1.AFLP:a new technique for DNA fingerprinting[J].Nucleic Acids Res,1995,23(21):4407-4414.
[15] Hu J G,Vick B A.Target region amplification polymorphism:a novel marker technique for plant genotyping[J].Plant Molecular Biology Reporter,2003,21(3):289-294.
[16] Wang X L,Yang Y X,Cong Y Z,et a1.DNA fingerprinting of Selected Laminaria (Phaeophyta)gametophytes by RAPD markers[J].Aquaculture,2004,238(1-4):143-153.
[17] Pang Q Q,Sui Z H,Kang K H,et al.Application of SSR and AFLP to the analysis of genetic diversity in Gracilariopsis lemanei formis(Rhodophyta)[J].Appl Phycol,2010,22(5):607-612.
[18] 丁弘叶.青岛野生种群不同倍性龙须菜AFLP遗传多样性分析以及与性别相关SCAR的标记开发与验证 [D].青岛:中国海洋大学,2011.
[19] Paran I,Michelmore R W.Development of reliable PCR-markers linked to downy mildew resistance genes in lettuce [J].Theor.Appl Genet,1993,85:985-993.
[20] 乔利仙.紫菜遗传多样性分析及其6-磷酸海藻糖合成酶基因转化水稻的研究 [D].青岛:中国海洋大学,2007.
[21] 石金锋,贾建航,金德敏,等.紫菜无性系特异SCAR标记的获得 [J].海洋学报,2003,25(1):128-131.
[22] 柳波,孙彬,金德敏,等.紫菜自由丝状SCAR标记的获得 [J].高技术通讯,2004,14(12):88-92.
[23] 张学成,费修绠.981龙须菜及其栽培技术介绍 [J].科学养鱼,2008(6):21-22.
[24] 孟琳,徐涤,陈伟洲,等.龙须菜新品系07-2的筛选及性状分析[J].中国海洋大学学报:自然科学版,2009,39(增刊):94-98.

