半滑舌鳎稚鱼脂肪分解关键酶基因的克隆及饲料中脂肪水平对其表达的调控❋
艾庆辉,袁禹惠,麦康森,李松林,徐 玮,张彦娇,周慧慧
(中国海洋大学农业部水产动物营养与饲料重点实验室,中国海洋大学海水养殖教育部重点实验室,山东青岛266003)
半滑舌鳎稚鱼脂肪分解关键酶基因的克隆及饲料中脂肪水平对其表达的调控❋
艾庆辉,袁禹惠,麦康森,李松林,徐 玮,张彦娇,周慧慧
(中国海洋大学农业部水产动物营养与饲料重点实验室,中国海洋大学海水养殖教育部重点实验室,山东青岛266003)
克隆了半滑舌鳎脂肪分解关键酶基因即激素敏感脂肪酶(Hormone-sensitive lipase,HSL)和脂肪甘油三酯脂肪酶(Adipose triglyceride lipase,ATGL)cDNA部分序列,并探讨了饲料中脂肪水平对半滑舌鳎稚鱼HSL及ATGL基因表达的影响。克隆所得到的HSL和ATGL片段长度分别为589bp和581bp,序列和系统进化分析表明半滑舌鳎的HSL和ATGL与大部分鱼类具有较高的同源性。采用不同水平的鱼油配制成5种不同脂肪水平(6.68%、9.84%、13.47%、17.89%和21.88%干物质)的等氮饲料,饲养35日龄半滑舌鳎稚鱼30 d,养殖过程中每天饱食投喂5次,养殖实验结束后采用实时荧光定量PCR分别测定各处理稚鱼内脏团的HSL及ATGL mRNA相对表达量。结果表明,高脂饲料组(21.88%)显著促进了半滑舌鳎稚鱼内脏团HSL基因的表达(P<0.05),但饲料脂肪水平对于半滑舌鳎稚鱼内脏团的ATGL表达量未产生显著影响(P>0.05)。
激素敏感脂肪酶;脂肪甘油三酯脂肪酶;基因克隆及表达;半滑舌鳎;脂肪水平
甘油三酯(TG)主要以脂滴形式储存在脂肪细胞的胞浆中,脂肪细胞脂滴内所储存的TG分解为游离脂肪酸(FFA)和甘油的过程,称为脂肪分解或脂肪动员[1]。调节脂肪分解的限速酶主要包括经典的激素敏感脂肪酶(Hormone-sensitive lipase,HSL)和2004年[2]才发现的脂肪甘油三酯脂肪酶(Adipose triglyceride lipase,ATGL)[2-4],ATGL也被称为含patatin样磷脂酶结构域酯酶2(Patatin-like phospholipase domaincontaining lipase 2,PNPLA2)、destnutrin或非钙依赖磷酸酯酶ζ(ζ-isoform of a calcium-independent phospholipase A2,iPLA2ζ)[5]。HSL和ATGL均主要分布于脂肪组织,但在其他组织也有少量表达[1,6]。HSL可以水解多种底物,包括甘油三酯(TG)、甘油二酯(DG)、单酰基甘油(MG)、胆固醇酯以及其他酯质。然而研究表明,在众多底物中,HSL对TG的水解能力最弱,而DG是其水解率最高的底物[7]。与HSL不同,ATGL具有高度的底物特异性,它在TG的第一步水解反应过程中发挥着关键作用,能特异性地将TG水解形成DG和FFA,但不能水解MG和胆固醇酯[2,4]。在许多哺乳动物上,已成功克隆到HSL和ATGL基因,并且许多研究表明HSL以及ATGL的转录水平受到激素水平、营养状态等调控[8-13]。目前,在多种鱼类上也已克隆到HSL和ATGL基因[13-18]。Han等[15]报道了高脂饲料促进了罗非鱼肝脏及肌肉中HSL基因表达、抑制了腹腔脂肪中HSL的表达。Wang等[13]和吉红等[18]的研究表明,饲料中的脂肪水平以及n-3长链多不饱和脂肪酸水平会显著影响ATGL的基因表达。但在鱼类上,饲料中营养素对于HSL和ATGL基因表达的调控研究还比较少,同时,在鱼类上对比HSL和ATGL受到饲料中脂肪水平调控的研究还未见报道。
半滑舌鳎(Cynoglossus semilaeυis)属鲽形目(Pleuronectiformes)、舌鳎科(Cynoglossidae)、舌鳎属(Cynoglossus),为我国近海底层的大型名贵经济鲆鲽鱼类[19-20]。目前,人工养殖的鱼类由于使用高脂及高植物油替代的饲料大多存在脂肪过量沉积的现象[21],然而脂肪过量沉积可能会导致鱼体产生脂肪肝等疾病[22],同时降低鱼肉品质[23],进而影响养殖鱼类的经济价值[13]。脂肪分解与脂肪沉积密切相关,因此,关于鱼类脂肪分解及其调控机制的研究,对指导解决养殖鱼类脂肪过量沉积问题具有重要的理论和实践意义。但是在鲆蝶鱼类上,关于营养素对脂肪分解关键酶基因表达的影响的研究还较少。此外,相比幼鱼和成鱼,稚鱼生长快,代谢旺盛,对于饲料中营养素的调控更为敏感,因此本实验采用舌鳎稚鱼作为研究对象,通过克隆得到HSL和ATGL基因的核心序列,并探究饲料中不同脂肪水平对HSL及ATGL基因表达的影响,为进一步探讨HSL和ATGL的作用机理及调控机制研究提供参考性资料。
1 材料与方法
1.1实验动物及养殖条件
实验用鱼为购自山东省海阳市黄海水产有限公司的35日龄的半滑舌鳎稚鱼,平均体重为(54±1)mg。养殖实验也在山东省海阳市黄海水产有限公司进行。实验开始时挑选外观正常、体格健壮和活力充足的鱼苗2 250尾,随机分成15组,每组150尾,分别放于循环水系统中。实验期间,水温(23±1)℃,p H=8.0±0.2,盐度30±3。
1.2内脏团中总RNA的提取及反转录
用Trizol试剂法提取半滑舌鳎稚鱼内脏团中的总RNA,并将提取的总RNA用DNase I(TaKaRa)处理去除RNA中可能存在的DNA污染。使用1.2%的琼脂糖凝胶电泳检测RNA的完整性,并采用NanoDrop
ND-1000(Wilmington,DE)确定RNA的质量和浓度。取1μg RNA按照PrimeScriptTMRT reagent Kit(Perfect Real Time)(TaKaRa)说明书完成cDNA第一链的合成,保存于-20℃。
1.3脂肪分解关键基因ATGL及HSL核心序列的克隆
根据NCBI Genebank中的斑马鱼Danio rerio(XM_ 005158028)、金头鲷Sparus aurata(EU254478)、斜带石斑鱼Epinephelus coioides(KF049203)以及人Homo sapiens(JF279441)、家牛Bos taurus(FJ897536)及大黄鱼Larimichthys crocea(HQ916211)等物种的核苷酸序列,利用Primer Premier 5.0设计HSL及ATGL的简并引物(见表1)。
PCR反应在Eppendorf Mastercycler gradient(Eppendorf,German)进行,反应条件为:预变性94℃5 min,1个循环;变性94℃30 s,引物退火51.5℃30 s,引物延伸72℃1 min,35个循环;72℃延伸10 min,1个循环;4℃保存。用1.2%的琼脂糖凝胶电泳检测和回收目的基因条带,然后将其连接到pEASY-T1载体(TransGen Biotech,China),转入到感受态细胞Escherichia coli TOP10中,经培养后涂布氨苄培养基。挑选5个阳性克隆测序(上海博尚),使用BLAST网页对测序结果进行比对分析(http://blast.ncbi. nlm.nih.gov/)。
1.4实时荧光定量PCR
以半滑舌鳎β-2微球蛋白(GenBank:FJ965561)基因作为内参基因。根据已获得的HSL、ATGL以及β-2微球蛋白序列,分别设计特异性引物(见表1)。反应体系为25μL体系:1μL特异性引物(上游引物和下游引物,10μmol/L)、1μL模板cDNA、12.5μL 2× SYBR Premix Ex TaqTM(Takara,Japan)以及9.5μL无菌水。反应条件为:95℃2 min;95℃10 s,58℃10 s,72℃20 s,40个循环。在每个PCR循环之后,通过溶解度曲线检验每个PCR反应是否只有1个PCR产物。通过制作浓度标准曲线验证目标序列与内参序列的扩增效率是否一致,扩增效率计算公式为E=10(-1/Slope)-1[24]。本实验中HSL、ATGL以及β-2微球蛋白的扩增效率分别为0.955 4,1.027 4和1.050 7。HSL以及ATGL的相对表达量采用2-ΔΔCt方法测定[25]。
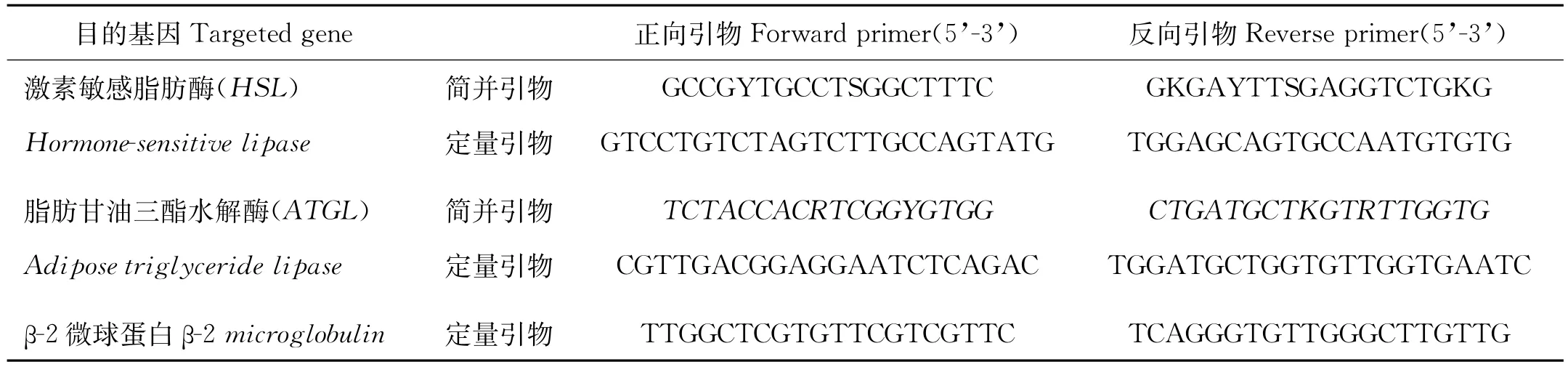
表1 本实验所使用的简并引物和定量引物序列Table 1 Degenerated primers and real-time quantitative PCR primers used in this experiment
1.5饲料中脂肪水平对目的基因表达调控的研究
以脱脂白鱼粉、酪蛋白、磷虾粉、鱿鱼粉以及水解鱼蛋白为主要蛋白源,以大豆卵磷脂和鱼油为主要脂肪源。通过调节鱼油水平设计5种不同脂肪水平(6.68%、9.84%、13.47%、17.89%和21.88%)的等氮微颗粒饲料(见表2),每个处理3个重复。每天人工投喂微颗粒饲料5次(6∶00、9∶00、15∶00、18∶00和 21∶00),饱食投喂,养殖周期30 d。实验结束后,每组随机选取6尾半滑舌鳎稚鱼进行解剖,分离内脏团于1.5 mL无RNase离心管中,并将离心管迅速放入液氮中,用于提取内脏团总RNA。
1.6序列及数据分析
应用DNAman进行拼接和推导氨基酸序列,在ClustalW多重比对网页上进行序列分析和比较,应用MEGA5软件N-J方法构建系统进化树;数据统计使用Windows SPSS 19.0进行单因素方差分析(ANOVA),显著水平为P<0.05,数据表示为“平均值±标准误”。
2 结果
2.1 ATGL及HSL核心序列克隆结果
PCR反应得到的序列,经过克隆测序,分别获得长为589bp的HSL(KJ459044)和581bp的ATGL(KJ459046),它们分别编码195和193个氨基酸(见图1和2)。同源性比对可以看出,克隆得到的HSL片段与斜带石斑鱼(Epinephelus coioides)HSL cDNA全序列(KF049203)相似性最高(99%),与金头鲷(Sparus aurata)HSL片段(EU254478)相似性91%,与牙鲆(Paralichthys oliυaceus)HSL片段(AB828673)相似性为88%;克隆得到的ATGL片段与大黄鱼(Larimichthys crocea)ATGL cDNA全序列(HQ916211)相似性84%,与鹌鹑(Coturnix coturnix)和人(Homo sapiens)ATGL cDNA全序列(GQ221783)相似性均为74%,与原鸡(Gallus gallus)和家鼠(Mus musculus)ATGL cDNA全序列(NM_001163689)相似性均为73%。此结果显示,人们用于RT-PCR的特异性引物序列正确,并克隆得到了实验所需要的HSL和ATGL基因。

表2 实验饲料配方及营养成分组成(干物质)Table 2 Formulation and proximate analysis of the experimental diets(dry weight)/%
2.2 ATGL和HSL氨基酸序列系统进化分析
应用MEGA5软件N-J方法分别构建了包括半滑舌鳎在内的十余种脊椎动物的ATGL及HSL氨基酸序列系统进化树(见图3和4)。结果显示:半滑舌鳎的HSL氨基酸序列与斜带石斑鱼、罗非鱼、金头鲷等大多数鱼类的HSL序列可以聚为一支,而后与人、鼠等哺乳动物的HSL序列聚为一支,草鱼的HSL氨基酸序列单独成为一支;半滑舌鳎的ATGL氨基酸序列与斑马宫丽鱼及罗非鱼聚为一支,再与大黄鱼聚为一支,最后与鸟类(鹌鹑、原鸡、绿头鸭)和哺乳动物(人、羊、猪、鼠兔)聚为一支,长腭泥鰕虎鱼的ATGL氨基酸序列单独成为一支。
2.3不同脂肪水平对ATGL及HSL基因表达的影响
随着饲料脂肪水平的升高,半滑舌鳎稚鱼内脏团HSL mRNA水平呈现升高的趋势,且21.88%脂肪水平组HSL表达量显著高于其他处理组(P<0.05),但其他各处理组之间差异不显著(P>0.05)(见图5)。与HSL有所不同,饲料脂肪水平对于半滑舌鳎稚鱼内脏团的ATGL表达量没有产生显著影响(P>0.05)(见图6)。

图1 半滑舌鳎HSL基因cDNA部分序列和翻译的氨基酸序列Fig.1 The partial nucleotide sequence and deduced amino acid sequence of cynoglossussemilaeυis HSL cDNA

图2 半滑舌鳎ATGL基因cDNA部分序列和翻译的氨基酸序列Fig.2 The partial nucleotide sequence and deduced amino acid sequence of cynoglossussemilaeυis ATGL cDNA

图3 根据NJ法构建的ATGL氨基酸序列系统树Fig.3 Phylogenetic tree showing the relationship of ATGL from cynoglossussemilaeυi s and other vertebrates on Neighbour-Joining method

图4 根据NJ法构建的HSL氨基酸序列系统树Fig.4 Phylogenetic tree showing the relationship of HSL from cynoglossussemilaeυi s and other vertebrates on Neighbour-Joining method

图5 饲料脂肪水平对半滑舌鳎稚鱼内脏团HSL基因表达的影响Fig.5 Effect of dietary lipid on HSL mRNA of larval cynoglossussemilaeυi s
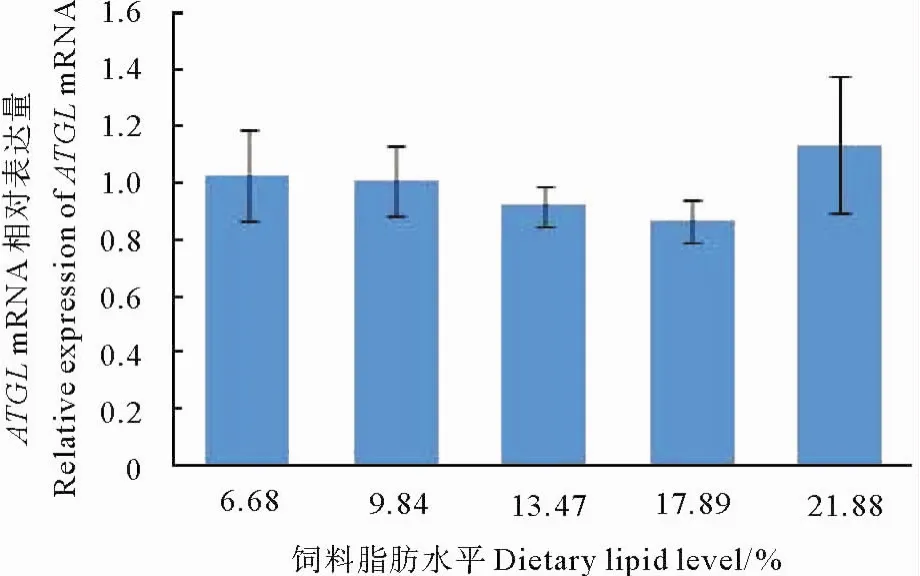
图6 饲料脂肪水平对半滑舌鳎稚鱼内脏团ATGL基因表达的影响Fig.6 Effect of dietary lipid on ATGL mRNA oflarval cynoglossussemilaeυi s
3 讨论
HSL基因最早在大鼠中得到克隆和研究,随后在人类、猪和小鼠中也检测到该基因的存在[26]。根据目前Genebank的记录,已经在斜带石斑鱼、罗非鱼、青鳉、黑棘鲷、金头鲷以及草鱼等鱼类中分离到部分或全部HSL cDNA序列。ATGL基因由Zimmermann等[2]筛选脂肪酶基因及蛋白数据库时发现,随后在小鼠脂肪组织中第一次验证并命名。目前在丽鲷、大黄鱼、斑马宫丽鱼、青鳉、红鳍东方鲀、罗非鱼及长腭泥鰕虎鱼等鱼类中已克隆到部分或全部ATGL序列。Blast结果显示,本实验克隆到的半滑舌鳎HSL和ATGL基因序列片段与大部分鱼类具有高度的相似性。此外,目前在虹鳟、河豚、青鳉、斑马鱼及牙鲆等鱼类上克隆到了2种不同的HSL cDNA[14,27],即HSL 1和HSL 2。Kittilson等[14]认为,由于亲缘关系相近物种进化的一致性,很可能所有硬骨鱼类都具有不止1个HSL转录本。因此,本实验虽然只克隆到一种HSL转录本,但半滑舌鳎也可能存在其他HSL转录本。
从分子进化树可以看出,大部分鱼类如半滑舌鳎、斜带石斑鱼、罗非鱼、青鳉、黑棘鲷及金头鲷的HSL可以聚为一类,哺乳动物如人、猩猩和家鼠等的HSL聚为一类,而草鱼的HSL单独聚为一类,说明草鱼的HSL在进化过程中较早分化出来,显示了该酶进化的复杂性。与HSL类似,大部分鱼类的ATGL可以聚为一类,哺乳动物和鸟类的ATGL分别聚为一类,但长腭泥鰕虎鱼的HSL分化较早、单独聚为一类。总体来看,半滑舌鳎HSL及ATGL氨基酸序列与大部分鱼类进化关系较近,与哺乳动物的进化关系较远,这与传统的形态学和生化特征分类进化地位基本一致。
对哺乳动物的很多研究发现,HSL和ATGL均可受到饲料中营养水平的调节[28]。在本实验中,高脂显著促进了半滑舌鳎稚鱼内脏团HSL基因的表达,这与Han等[15]对罗非鱼肝脏HSL基因表达研究的结果类似,说明在一定的高脂范围,鱼体会通过增加HSL基因的表达来缓解高脂造成的脂肪沉积。然而,在本实验中,半滑舌鳎稚鱼内脏团ATGL基因的表达量并没有受到饲料中脂肪水平的显著影响。Wang等[13]报道,高脂会显著降低大黄鱼ATGL基因的表达。吉红等[18]发现,草鱼摄食n-3 LC-PUFA饲料后,在第1周和第2周ATGL基因的表达水平显著高于对照组,但第3周后该基因的表达水平在处理组与对照组间无显著差异。这些研究结果的不一致可能与实验对象种类及规格、饲料所采用的油源以及养殖周期不尽相同有关。
ATGL和HSL均是脂肪组织甘油三酯动员的关键酶,但分别主要参与水解甘油三酯和甘油二酯[29]。HSL参与脂肪分解过程受到肾上腺素等促脂解激素的作用,其活性受到磷酸化和去磷酸化作用的调控[30]。ATGL发挥活性也受到激素的调节[29],在小鼠上的研究发现ATGL也可以发生磷酸化和去磷酸化,但与HSL不同的是,其磷酸化过程不由PKA通路介导[2]。本实验的对比结果显示,半滑舌鳎稚鱼饲喂不同脂肪水平的饲料后,HSL和ATGL在转录水平上的反馈调节并不一致,暗示调节HSL和ATGL基因表达的信号通路可能不同,而且半滑舌鳎稚鱼可能不只通过转录水平,还通过其他水平(例如蛋白水平和磷酸化水平)调节机体的脂肪分解代谢。
[1] 文秀英,卢坤,李若雪.脂肪甘油三酯脂肪酶,激素敏感脂肪酶与围脂滴蛋白的相关作用及机制[J].中国糖尿病杂志,2011,19(1):71-73.
[2] Zimmermann R,Strauss J G,Haemmerle G,et al.Fat mobilization in adipose tissue is promoted by adipose triglyceride lipase[J]. Science,2004,306(5700):1383-1386.
[3] 徐冲,徐国恒.脂肪组织甘油三酯水解酶参与脂肪分解调控[J].生理科学进展,2008,39(1):10-14.
[4] Zechner R,Kienesberger P C,Haemmerle G,et al.Adipose triglyceride lipase and the lipolytic catabolism of cellular fat stores[J].Journal of Lipid Research,2009,50(1):3-21.
[5] Kim J Y,Tillison K,Lee J H,et al.The adipose tissue triglyceride lipase ATGL/PNPLA2 is downregulated by insulin and TNF-α in 3T3-L1 adipocytes and is a target for transactivation by PPARγ[J].American Journal of Physiology-Endocrinology and Metabolism,2006,291(1):115-127.
[6] 胡深强,潘志雄,王继文.脂肪甘油三酯脂肪酶(ATGL)的生物学功能及调控机制[J].中国生物化学与分子生物学报,2011,27(8):721-727.
[7] Haemmerle G,Zimmermann R,Hayn M,et al.Hormone-sensitive lipase deficiency in mice causes diglyceride accumulation in adipose tissue,muscle,and testis[J].Journal of Biological Chemistry,2002,277(7):4806-4815.
[8] Holm C,Kirchgessner T G,Svenson K L,et al.Hormone-sensitive lipase:sequence,expression,and chromosomal localization to 19cent-q13.3[J].Science,1988,241(4872):1503-1506.
[9] Stenson Holst L,Langin D,Mulder H,et al.Molecular cloning,genomic organization,and expression of a testicular isoform of hormone-sensitive lipase[J].Genomics,1996,35(3):441-447.
[10] Stich V,Harant I,Glisezinski I D,et al.Adipose tissue lipolysis and hormone-sensitive lipase expression during very-low-calorie diet in obese female identical twins 1[J].The Journal of Clinical Endocrinology&Metabolism,1997,82(3):739-744.
[11] Kershaw E E,Hamm J K,Verhagen L A W,et al.Adipose triglyceride lipase function,regulation by insulin,and comparison with adiponutrin[J].Diabetes,2006,55(1):148-157.
[12] Kralisch S,Klein J,Lossner U,et al.Isoproterenol,TNFα,and insulin downregulate adipose triglyceride lipase in 3T3-L1 adipocytes[J].Molecular and cellular endocrinology,2005,240(1):43-49.
[13] Wang X,Wang Y,Li Y.Adipose triglyceride lipase(ATGL)clone,expression pattern,and regulation by different lipid sources and lipid levels in large yellow croaker(Pseudosciaena crocea R.)[J].Marine Biotechnology,2013,15(2):197-205.
[14] Kittilson J D,Reindl K M,Sheridan M A.Rainbow trout(Oncorhynchus mykiss)possess two hormone-sensitive lipase-encoding mRNAs that are differentially expressed and independently regulated by nutritional state[J].Comparative Biochemistry and Physiology Part A:Molecular&Integrative Physiology,2011,158(1):52-60.
[15] Han C Y,Wen X B,Zheng Q M,et al.Effects of dietary lipid levels on lipid deposition and activities of lipid metabolic enzymes in hybrid tilapia(Oreochromisniloticu s×O.aureus)[J].Journal of animal physiology and animal nutrition,2011,95(5):609-615.
[16] Cruz-Garcia L,Sánchez-Gurmaches J,Bouraoui L,et al.Changes in adipocyte cell size,gene expression of lipid metabolism markers,and lipolytic responses induced by dietary fish oil replacement in gilthead sea bream(Sparus aurata)[J].Comparative Biochemistry and Physiology Part A:Molecular&Integrative Physiology,2011,158(4):391-399.
[17] Alves Martins D,Rocha F,Martínez-Rodríguez G,et al.Teleost fish larvae adapt to dietary arachidonic acid supply through modulation of the expression of lipid metabolism and stress response genes[J].British Journal of Nutrition,2012,108(05):864-874.
[18] 吉红,黄吉芹,刘品.草鱼ATGL基因的表达及饲喂n-3 HUFAs对其的影响[J].水产学报,2012,36(5):732-739.
[19] Liu X,Xu Y,Ma A,et al.Effects of salinity,temperature,light rhythm and light intensity on embryonic development of Cynoglossus semilaeυis Gunther and its hatching technology optimization[J].Marine Fisheries Research,2003,25(6):1-6.
[20] 万瑞景,姜言伟,庄志猛.半滑舌鳎早期形态及发育特征[J].动物学报,2004,50(1):91-102.
[21] 刘振勇,谢友佺,林小金.大黄鱼肝脏病变组织病理学观察[J].海洋水产研究,2007,28(5):7-11.
[22] 曾端,麦康森,艾庆辉.脂肪肝病变大黄鱼肝脏脂肪酸组成,代谢酶活性及抗氧化能力的研究[J].中国海洋大学学报:自然科学版,2008,38(4):542-546.
[23] Duan Q,Zhong H,Si L,et al.Comparative analyses of biochemical composition in net cultured and wild Pseudosciaena crocea(Richardson)[J].J Zhejiang Ocean Univ,2000,19(2):125-128.
[24] Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod[J]. Methods,2001,25:402-408.
[25] Yao C L,Kong P,Wang Z Y,et al.Molecular cloning and expression of MyD88 in large yellow croaker,Pseudosciaena crocea[J].Fish&Shellfish Immunology,2009,26(2):249-255.
[26] 马晶晶,邵庆均,许梓荣,等.n-3高不饱和脂肪酸对黑鲷幼鱼生长及脂肪代谢的影响[J].水产学报,2009,33(4):639-649.
[27] Khieokhajonkhet A,Kaneko G,Ohara K,et al.Hormone-sensitive lipase in Japanese flounder Paralichthys olivaceus:the potential function of the inclinator muscle of fin as a lipid storage site[J].Fisheries Science,2014,80(2):341-351.
[28] Gaidhu M P,Anthony N M,Patel P,et al.Dysregulation of lipolysis and lipid metabolism in visceral and subcutaneous adipocytes by high-fat diet:role of ATGL,HSL,and AMPK[J].A-merican Journal of Physiology-Cell Physiology,2010,298(4):961-971.
[29] Zimmermann R,Lass A,Haemmerle G,et al.Fate of fat:the role of adipose triglyceride lipase in lipolysis[J].Biochimica et Biophysica Acta(BBA)-Molecular and Cell Biology of Lipids,2009,1791(6):494-500.
[30] Watt M J,Holmes A G,Pinnamaneni S K,et al.Regulation of HSL serine phosphorylation in skeletal muscle and adipose tissue[J].American Journal of Physiology-Endocrinology and Metabolism,2006,290(3):500-508.
Lipolysis Related Genes Clone and Regulation by Dietary Lipid Level in Half-Smooth Tongue Sole(Cynoglossus Semilaevis)Larvae
AI Qing-Hui,YUAN Yu-Hui,MAI Kang-Sen,LI Song-Lin,XU Wei,ZHANG Yan-Jiao,ZHOU Hui-Hui(The Key Laboratory of Aquaculture Nutrition and Feed,Ministry of Agriculture,The Key Laboratory of Mariculture,Ministry of Education,Ocean University of China,Qingdao 266003,China)
The lipolysis related key enzymes,namely hormone-sensitive lipase(HSL)and adipose triglyceride lipase(ATGL),were cloned and the effects of dietary lipid level on HSL and ATGL genes expression were investigated in half-smooth tongue sole(Cynoglossus semilaeυis)larvae.The obtained partial cDNA of HSL and ATGL were 589bp and 581bp,respectively,and they shared high identities with many other fish species.A 30 day feeding trial was conducted to investigate the effects of dietary lipid level on HSL and ATGL genes expression.Five isoproteic diets were formulated with graded levels of lipid ranging from 6.68%to 21.88%dry weight.Each diet was randomly allocated to triplicate groups of 150 larval tongue sole(35 DAH,(54±1)mg)and fish were fed five times daily to apparent satiation during the feeding experiment.The relative mRNA expression level was decided by real-time quantitative polymerase chain reaction(qPCR).Results showed that,high-lipid diet(21.88%)promoted HSL expression of larval visceral mass significantly(P<0.05),while dietary lipid level did not affect the ATGL mRNA expression significantly(P>0.05).
hormone-sensitive lipase;adipose triglyceride lipase;gene clone and expression;Cynoglossussemilaeυis;dietary lipid
Q785;S 917.4
A
1672-5174(2014)10-065-07
责任编辑 朱宝象
现代农业产业技术体系—鲆鲽类体系建设专项(2010GB23600673);国家科技支撑计划项目(2011BAD13B01)资助
2014-07-10;
2014-09-05
艾庆辉(1972-),男,教授,博导。E-mail:qhai@ouc.edu.cn

