甲烷氧化菌20Z利用Embden-Meyerhof-Parnas途径高效同化甲烷
崔金玉,姚陆,孙效乐,Marina G. Kalyuzhnaya,杨松,4
1 青岛农业大学生命科学学院 山东省应用真菌重点实验室,山东 青岛 266109
2 山东省农业科学院质量标准与检测试验研究所,山东 济南 250100
3 美国华盛顿大学微生物系,华盛顿州 西雅图 98105
4 天津大学系统生物工程教育部重点实验室,天津 300072
甲烷氧化菌20Z利用Embden-Meyerhof-Parnas途径高效同化甲烷
崔金玉1,姚陆1,孙效乐2,Marina G. Kalyuzhnaya3,杨松1,4
1 青岛农业大学生命科学学院 山东省应用真菌重点实验室,山东 青岛 266109
2 山东省农业科学院质量标准与检测试验研究所,山东 济南 250100
3 美国华盛顿大学微生物系,华盛顿州 西雅图 98105
4 天津大学系统生物工程教育部重点实验室,天津 300072
崔金玉, 姚陆, 孙效乐, 等. 甲烷氧化菌20Z利用Embden-Meyerhof-Parnas途径高效同化甲烷. 生物工程学报, 2014, 30(1): 43−54.
Cui JY, Yao L, Sun XL, et al. Highly efficient methane assimilation through Embden-Meyerhof-Parnas pathway in Methylomicrobium alcaliphilum 20Z. Chin J Biotech, 2014, 30(1): 43−54.
为了探究γ-变形菌纲 (Gammaproteobacteria) 甲烷氧化菌Methylomicrobium alcaliphilum 20Z的甲烷同化代谢过程。文中整合RNA-seq、LC-MS技术并结合13C标记策略对核酮糖单磷酸途径 (Ribulose monophosphate pathway) 及下游途径展开系统组学分析。M. alcaliphilum 20Z代谢物组定量分析表明Entner-Doudoroff (EDD) 途径的中间代谢物6-磷酸葡萄糖的浓度是(150.95±28.75) µmol/L,2-酮-3-脱氧-6-磷酸葡糖酸浓度低于质谱定量分析检测限,而Embden-Meyerhof-Parnas (EMP) 途径中果糖1,6-二磷酸、甘油醛-3-磷酸/二羟丙酮磷酸和磷酸烯醇式丙酮酸的浓度分别是 (1 142.02±302.88) µmol/L、(1 866.76±388.55) µmol/L和(3 067.57±898.13) µmol/L。通过EDD和EMP途径的代谢物13C同位素动态富集研究,进一步揭示3位标记丙酮酸丰度是1位标记丙酮酸丰度的4~6倍。最后,基因表达比较分析发现EMP途径的关键基因 (如:fbaA、tpiA、gap和pykA) 的表达水平 (RPKM) 分别是2 479.2、2 493.9、2 274.6和1 846.0,而EDD途径中基因 (如:pgi、eda和edd) 的RPKM仅是263.8、341.2和225.4。综合上述结果阐明EMP途径才是M. alcaliphilum 20Z进行甲烷同化的关键通路。EMP途径代谢功能的全新阐述不但改变对Gammaproteobacteria甲烷氧化菌甲烷同化模式的传统认知,而且为甲烷高效生物催化转化提供重要的理论基础。
甲烷氧化菌,甲烷同化途径,甲烷生物催化,13C标记代谢物组,转录物组
甲烷 (CH4) 是驱动全球气候变暖的重要温室气体,同时甲烷作为天然气和沼气的主要成分不但是高价值燃料资源,也是一碳化工的重要前体原料[1-2]。近年来,探讨如何有效实现甲烷催化转变成化学产品成为国内外化工领域关注的热点课题之一[3-4]。甲烷氧化菌是迄今为止发现的唯一能以甲烷作为生长碳源和能源的生物体。Gammaproteobacteria甲烷氧化菌利用核酮糖单磷酸途径 (Ribulose monophosphate pathway,RuMP) 同化代谢甲烷,是实现甲烷高效生物催化转化的最有前景微生物系[5-6]。然而除了单细胞蛋白和聚β-羟基丁酸(poly-β-hydroxybutyrrate)[7-8],目前基于甲烷的生物催化转化合成高附加值产品的成功研究还没有报道过。其中一个主要原因是甲烷氧化菌基因组和后基因组的信息缺少、代谢网络信息不完整,阻碍对甲烷氧化菌合理而有效的代谢工程改造。
甲烷氧化菌Methylomicrobium alcaliphilum 20Z是一株代表性的中性嗜盐、嗜碱Gammaproteobacteria菌,其生长速率较快且稳定,是具有广泛应用前景的甲烷催化系统[9-10]。2011年,M. alcaliphilum 20Z基因组测序诠释获得完成,为进一步从系统水平上阐明M. alcaliphilum 20Z的基因组功能、代谢网络调控机制及代谢工程改造奠定重要的理论基础[11]。传统生物化学和酶学研究一直认为Gammaproteobacteria甲烷氧化菌主要利用RuMP途径的Entner-Doudoroff pathway (EDD)分支途径进行甲烷氧化产物甲醛的同化吸收,而另一途径Embden-Meyerhof-Parnas (EMP) 是旁支代谢路径[5]。然而,最近基因组分析显示所有参与EMP途径代谢反应的基因都在M. alcaliphilum 20Z基因组中存在[11]。而且对Gammaproteobacteria嗜甲烷菌Methylococcus capsulatus Bath蛋白物组分析也表明EDD途径和EMP途径的催化酶在M. capsulatus Bath中适量表达[12]。考虑到RuMP途径中的EMP途径比EDD途径能更高效吸收利用甲烷[13],深入探究EMP途径在Gammaproteobacteria甲烷氧化菌中真正的代谢功能,就成为理解甲烷同化吸收过程和实现甲烷高效生物转化的关键所在。
本研究以阐述M. alcaliphilum 20Z的中心代谢网络特征为核心,利用RNA-seq、LC-MS技术并结合13C标记策略,深入探讨细胞的转录物和代谢物两个层次特征,揭示M. alcaliphilum 20Z中EMP途径是参与甲烷同化的重要代谢过程。本研究结果对代谢工程改造Gammaproteobacteria甲烷氧化菌高效转化甲烷合成高附加值产品具有十分重要的科学意义。
1 材料与方法
1.1 菌种
甲基营养杆菌Methylotrophic extorquens AM1和Methylomicrobium alcaliphilum 20Z由美国华盛顿大学Mary E.Lidstrom课题组赠送。
1.2 试剂
氨基酸、有机酸和糖磷酸等12C-标记标准品是分析级,购买于Sigma 公司(St. Louis, MO, USA)。乙醇从EMD Chemicals公司购买(Gibbstown, NJ, USA)。液相色谱使用是LC-MS级溶剂(Fisher Scientific, Fair Lawn, NJ, USA)。超纯水由Millipore Q Reference制备。13C标记甲醇 (99%) 和13C标记甲烷分别购买于Cambridge Isotope Laboratories公司 (Andover, MA, USA)和Sigma公司。
1.3 Methylotrophic extorquens AM1和Methylomicrobium alcaliphilum 20Z的培养
1.3.1 M. extorquens AM1分批培养和以13C标记甲醇为碳源的连续恒化培养
M. extorquens AM1在250 mL锥形瓶中进行液体分批培养,甲醇作为碳源,卡那霉素浓度是50 μg/mL,培养温度28 ℃,具体培养方法参见文献[14]。
M. extorquens AM1在1 L发酵罐(BioFlo110,New Brunswick Scientific, Edison, NJ, USA) 中连续恒化培养。连续培养的无机盐组分与分批培养相同,25 mmol/L13C标记甲醇作为碳源。培养过程中, 为除去空气中的CO2,将压缩空气通过3个串联的内含有1 L KOH (4 mol/L)溶液的锥形瓶。恒化培养稀释率D= 0.1 h–1, OD600=1.0±0.1,5个培养周期后收集细胞。
1.3.2 M. alcaliphilum 20Z的培养
M. alcaliphilum 20Z培养在改良NMS培养基上[10],含有3% NaCl,0.25g KNO3,pH为8.9,甲烷浓度是35% (V/V),摇床转速是250 r/min,培养温度是30 ℃,气相色谱火焰离子化检测器检测甲烷消耗浓度。
1.4 代谢物组的制备
1.4.113C标记内标代谢物的制备
10 mL13C标记M. extorquens AM1细胞培养液小心、快速转移至28 mL冷浸液中 (HEPES (70 mmol/L, pH 6.8),60%甲醇 (V/V) 溶液,–40 )℃,低温冷冻离心 (Dupont Sorvall RC5B, Waltham, MA, USA),弃去上清液,沉淀细胞颗粒重新溶解于同样的冷浸溶液中,并在10 000 r/min下再次离心6 min,接着提取13C标记内标代谢物。提取核苷酸、有机酸、氨基酸和糖磷酸的方法参见文献[15]。对于酰基辅酶,使用文献[15]中的酸提取方法。将所有13C标记的细胞提取物合并、稀释和分装在–80℃下贮存,待用。
1.4.2 M. alcaliphilum 20Z代谢物组的样品制备
6 mL (OD600=0.4±0.2) 的细胞,按照上述方法进行冷浸,接着在提取代谢物组前,将90 μL的13C标记内标代谢物快速添加到细胞中,代谢物组提取方法参考文献[15],所获样品溶解在100 μL的超纯水中待分析。
1.5 LC-MS分析代谢物组
LC-MS是在Waters (Milford, MA, USA) 系统上进行。LC-MS系统由1525 μ的高效液相色谱泵、2777C自动进样器及Quattro Micro API质谱仪(Micromass, Manchester, UK)组成。液相色谱分离采用HILIC模式,色谱柱是Luna NH2(250 mm×2 mm, 5 mμ, Phenomenex),具体方法参见文献[16],质谱扫描模式是多反应监测(MRM)。代谢物峰用Masslynx Quanlynx Applications Manager (version 4.1)软件进行分析。
1.6 标准曲线的建立
4 μL不同浓度的12C标记标准品添加到已制备好的36 μL13C标记内标代谢物中,最终12C标记标准品的浓度分别是200、500、1 000、2 500、5 000、10 000、25 000、50 000 nmol/L。以12C标准品峰面积和13C标记内标代谢物峰面积的比值作为纵坐标,以12C标准品浓度作为横坐标,构建标准曲线,进而计算拟合方程和R2,具体方法参见文献[15]。
1.713C标记动态代谢物组分析
M. alcaliphilum 20Z在指数生长中期(OD600=0.25±0.05) 转移到新培养瓶中,并快速添加相应浓度13CH4(纯度是99.9%),样品在时间点 (0、1、3、5、10、20、40 min) 进行收集、提取和保存,具体方法参见1.4。
1.8 转录物组分析
45 mL M. alcaliphilum 20Z培养液(OD600=0.30±0.05) 添加到5 mL停止液 (含5%苯酚) 中,总RNA/DNA的提取方法参考文献[17],RNeasy Mini kit 和MICROExpress kit 用来纯化RNA和富集mRNA,1% 琼脂糖凝胶电泳和Agilent 2100 Bioanalyzer分析提取的RNA质量。RNA-seq在美国华盛顿大学基因组测序中心完成,测序平台为Illumina高通量深度测序。测序读段 (Reads) 利用Burrows-Wheeler transform算法和M. alcaliphilum 20Z参考基因组比对和定位。利用Cufflinks软件计算在基因组上定位读段的数量即RPKM (Reads Per Kilobase of gene per Million mapped reads),RPKM是每百万读段中来自某一基因每千碱基长度的读段数目。RPKM=基因压读段计数×109/(基因长度×测序深度),具体分析方法参见文献[17]。
2 结果与分析
2.1 M. alca liphilum 20Z中心代谢网络的代谢物浓度定量
代谢物浓度影响代谢反应的方向和代谢反应的速率,因此准确定量代谢物是解析M. alcaliphilum 20Z代谢网络特征的重要元素,是和其他微生物系进行比较分析的基础。由于代谢物组样品成分复杂,代谢物在质谱离子源离子化过程中存在显著差异性,并且离子间存在抑制效应,因此13C标记内标代谢物 (13C-IS)的添加是矫正质谱分析和样品制备过程中出现误差的重要方法[15]。然而商业化13C-IS稀缺且价格昂贵,本研究首先利用甲基营养菌M. extorquens AM1生物合成13C-IS,进而将合成的13C-IS添加到M. alcaliphilum 20Z的代谢物提取液中,以提高M. alcaliphilum 20Z胞内代谢物浓度定量的准确性。选择M. extorquens AM1菌合成13C-IS是因为其代谢物组成分及代谢网络结构和M. alcaliphilum 20Z相似,而且M. extorquens AM1生长更快速。图1显示M. alcaliphilum 20Z代谢物组绝对定量流程。
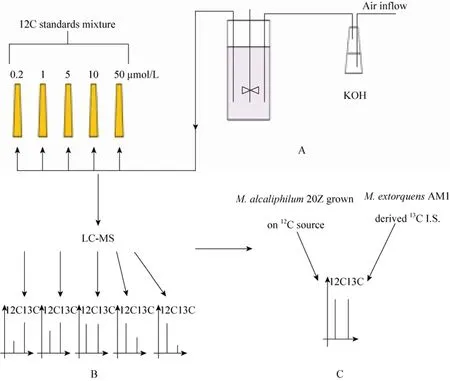
图1 甲烷氧化菌M. alcaliphilum 20Z的代谢物组绝对定量流程Fig. 1 Workflow of absolute quantitation of etabolome in M. alcaliphilum 20Z. A: bioreactor cultivation of M. extorquens AM1 grown on13C-methanol to obtain13C-IS; B: the development ofcalibration curve on LC-MS; C: the measurement of intracellular pool of M. alcaliphilum 20Z based on M. extorquens AM1 derived13C-IS.
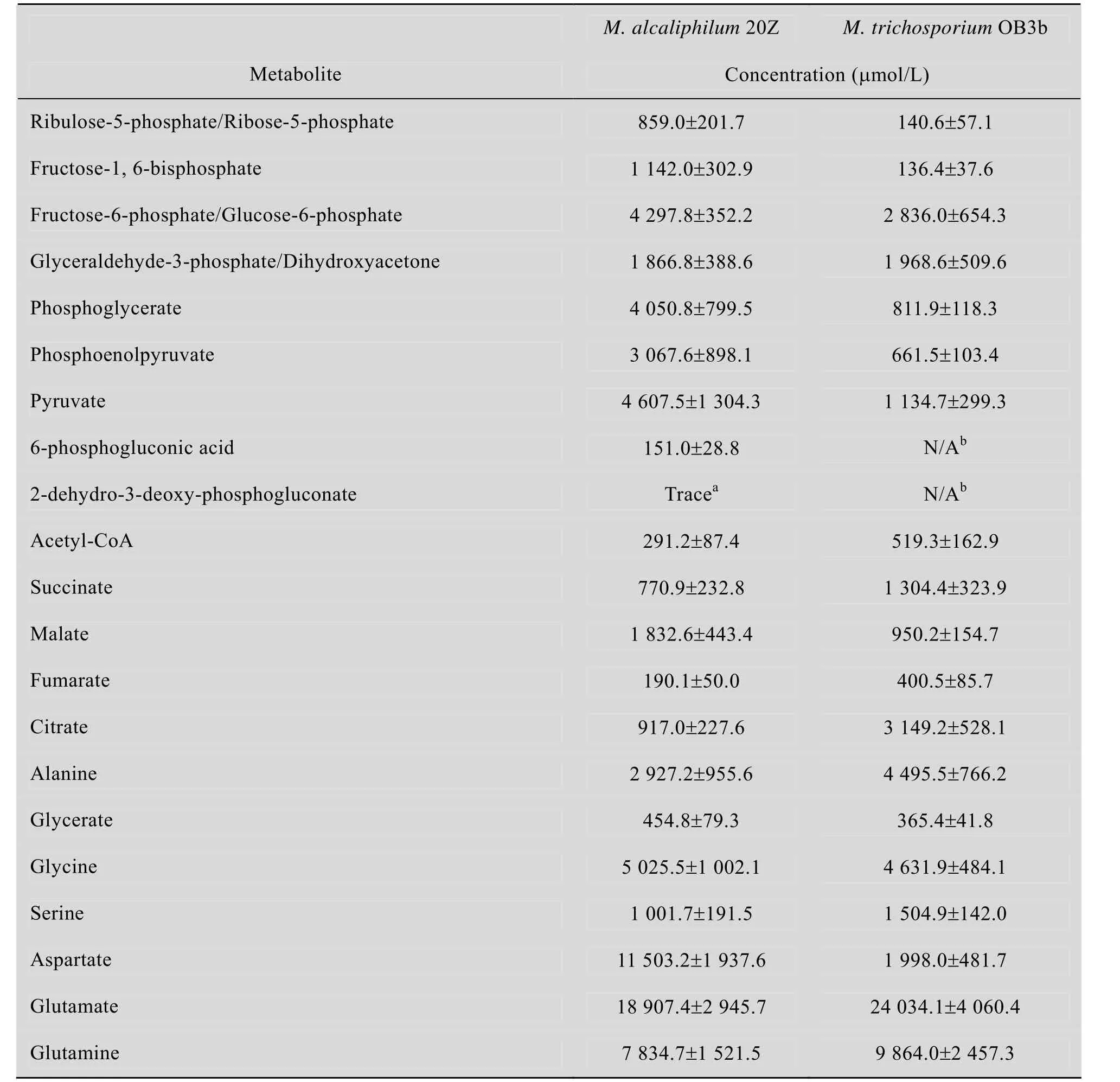
表1 M. alcaliphilum 20Z和M. trichosporium OB3b胞内中间代谢物浓度的比较分析Table 1 Comparison of intracellular pool of key metabolites between M. alc aliphilum 20Z and M. trichosporium OB3b
表1是M. alcaliphilum 20Z的24个中心代谢物浓度的定量结果。与我们前期研究报道α-变形菌纲 (Alphaproteobacteria) 甲烷氧化菌Methylosinus trichosporium OB3b的中心代谢物浓度相比较[18],丝氨酸循环途径 (Serine cycle) 的中间代谢物,如甘氨酸 (Glycine)、丝氨酸 (Serine)和甘油酸 (Glycerate)的浓度没有显著变化。在三羧酸 (Tricarxylic acid cycle,TCA) 循环途径中,由草酰乙酸 (Oxalaceticacid,OAA) 合成游离氨基酸天冬氨酸 (Aspartate),在M. alcaliphilum 20Z中天冬氨酸的浓度是M. trichosporium OB3b的5.8倍,该结果与M. alcaliphilum 20Z在高盐环境下生长积累的四氢甲基嘧啶羧酸关联[10]。在M. alcaliphilum 20Z中,果糖1,6-二磷酸(Fructose-1, 6-bisphosphate,FBP)、核酮糖5-磷酸 (Ribulose 5-phosphate,Ru5P)、磷酸烯醇式丙酮酸 (Phosphoenolpyruvate, PEP) 浓度分别是M. trichosporium OB3b的8.4倍、6.1倍和2.7倍,该结果与RuMP途径是M. alcaliphilum20Z进行甲烷同化吸收的主要代谢途径的观点相一致。此外,近来在M. extorquens AM1和M. trichosporium OB3b中分别发现新颖的乙醛酸回补途径 (Ethylmalonyl-CoA pathway,EMC途径) 的存在[18-19],然而在M. alcaliphilum20Z中未能检测到EMC途径的关键中间代谢物(如:甲基琥珀酸 (Methylsuccinic acid) 和3-羟丁酰- CoA (3-hydroxybutyryl-CoA)),该结果表明正常培养条件下,EMC途径在M. alcaliphilum 20Z中没有功能性激活。
传统研究认为EDD途径是RuMP途径的主要分支途径同化吸收甲烷氧化产物甲醛,而EMP途径仅是旁支途径[5]。然而,代谢物组定量分析却发现了相反结果。由表1可知,EDD途径的关键中间代谢物6PG和KDPG浓度很低(其中6PG浓度是(150.95±28.75) µmol/L,而KDPG浓度低于10 µmol/L不能准确定量)。EMP途径的关键中间代谢物浓度明显高于EDD途径的,其中FBP浓度是 (1 142.02±302.88) µmol/L, GAP/DHAP是(1 866.76±388.55) µmol/L,PEP是 (3 067.57±898.13) µmol/L。通过比较分析两个分支代谢途径的中间代谢物浓度,我们可以推测EMP途径很可能在甲烷同化吸收过程中起到更重要的作用。
2.2 EDD和EMP途径中间代谢物的13C同位素富集分析
由图2可见,在1 min时,果糖-6-磷酸/葡萄糖-6-磷酸 (Fructose-6-phosphate/Glucose-6-phosphate,F6P/G6P) 已经明显获得1个碳的13C富集,这表明甲烷快速通过核酮糖5-磷酸 (Ru5P)和甲醛的反应被吸收进入下游代谢途径。1个碳标记的F6P/G6P随着时序延长而相对丰度逐渐减少,而多碳标记的F6P/G6P相对丰度逐渐增加,这是由于磷酸戊糖途径的碳重排反应 (图3A)。EMP途径下游中间代谢物 (如:2-磷酸葡萄糖/3-磷酸葡萄糖 (Glucose-2-phosphate/ Glucose-3-phosphate, 2PG/3PG) 和PEP) 也同样地快速获得1碳、2碳和3碳的13C富集,而且2PG/3PG和PEP在40 min时的总13C富集丰度分别是54.9%和46.1%,这表明13C代谢流显著地通过EMP途径。由于6PG和KDPG浓度极低,不能清楚检测到13C富集。
Pyruvate是两个分支途径的共同终点代谢物,如果13C标记甲醛主要通过EMP途径同化吸收,将生成1个碳标记pyr在第3位 (即[3-13C1]-pyr),相反通过EDD途径将生成1个碳标记pyr在第1位 (即[1,13C1]-pyr) (图3A)。我们利用LC-MS/MS分析1个碳标记pyr的位置,发现在13C富集过程中,[3-13C1]-pyr的丰度高于[1,13C1]-pyr的约4−6倍 (图3B)。该结果清楚表明EMP途径实际上是更重要分支途径来进行甲烷的吸收代谢。
此外,我们也检测了丝氨酸循环途径的中间代谢物serine和glycine的13C富集规律,serine和glycine在0-40 min中13C富集丰度明显偏低 (图2)。
2.3 EDD和EMP途径基因表达水平的比较分析
为了进一步验证代谢物组和13C同位素动态富集研究的结论,我们比较分析了EDD和EMP途径相关基因的表达水平。由表2可见,在EDD途径中,参与催化反应的基因,例如基因pgi、 eda和edd的表达丰度 (即RPKM) 分别是263.8、341.2和225.4。反之,EMP途径的基因有明显更高的表达水平,例如基因fbaA、tpiA、gap和pykA的RPKM分别是2 479.2、2 493.9、2 274.6和1 846.0。因此整合转录物丰度、代谢物组浓度和13C标记分析的结果,我们认为EMP途径,而不是EDD途径才是M. alcaliphilum 20Z进行甲烷同化吸收的主要代谢通路 (图4)。
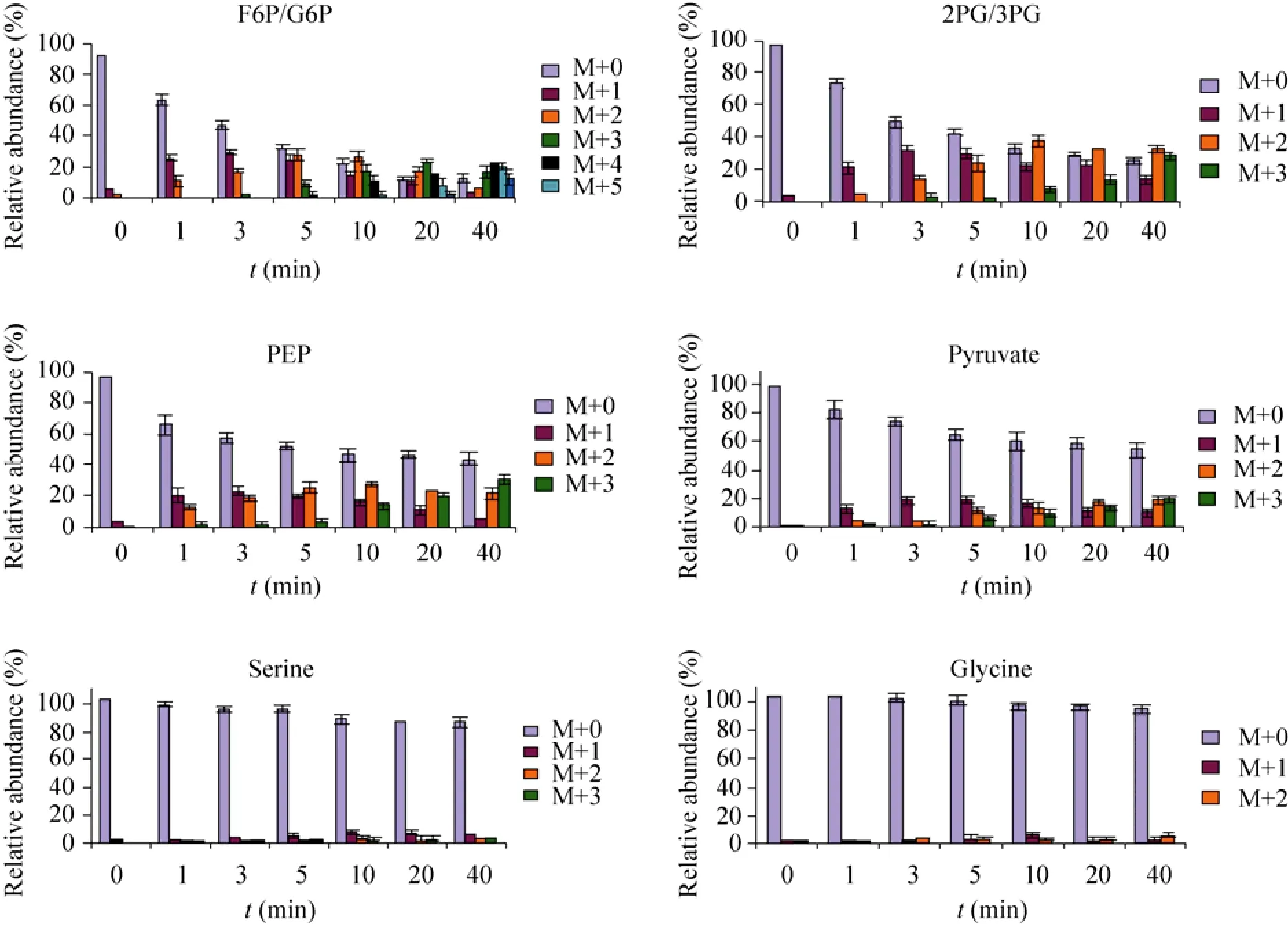
图 2 M. alcaliphilum 20Z胞内中间代谢物13C富集的变化规律Fig. 213C-incorporation in a time course in M. alcaliphilum 20Z. M+0 represents non-labeled, M+1 represents compound with one13C- label, M+2 represents compound with two13C-labels, etc. F6P/G6P: fructose-6-phosphate + glucose-6-phosphate; PEP: phosphoenolpyruvate; 2PG/3PG: 2-phosphoglycerate + 3-phosphoglycera.
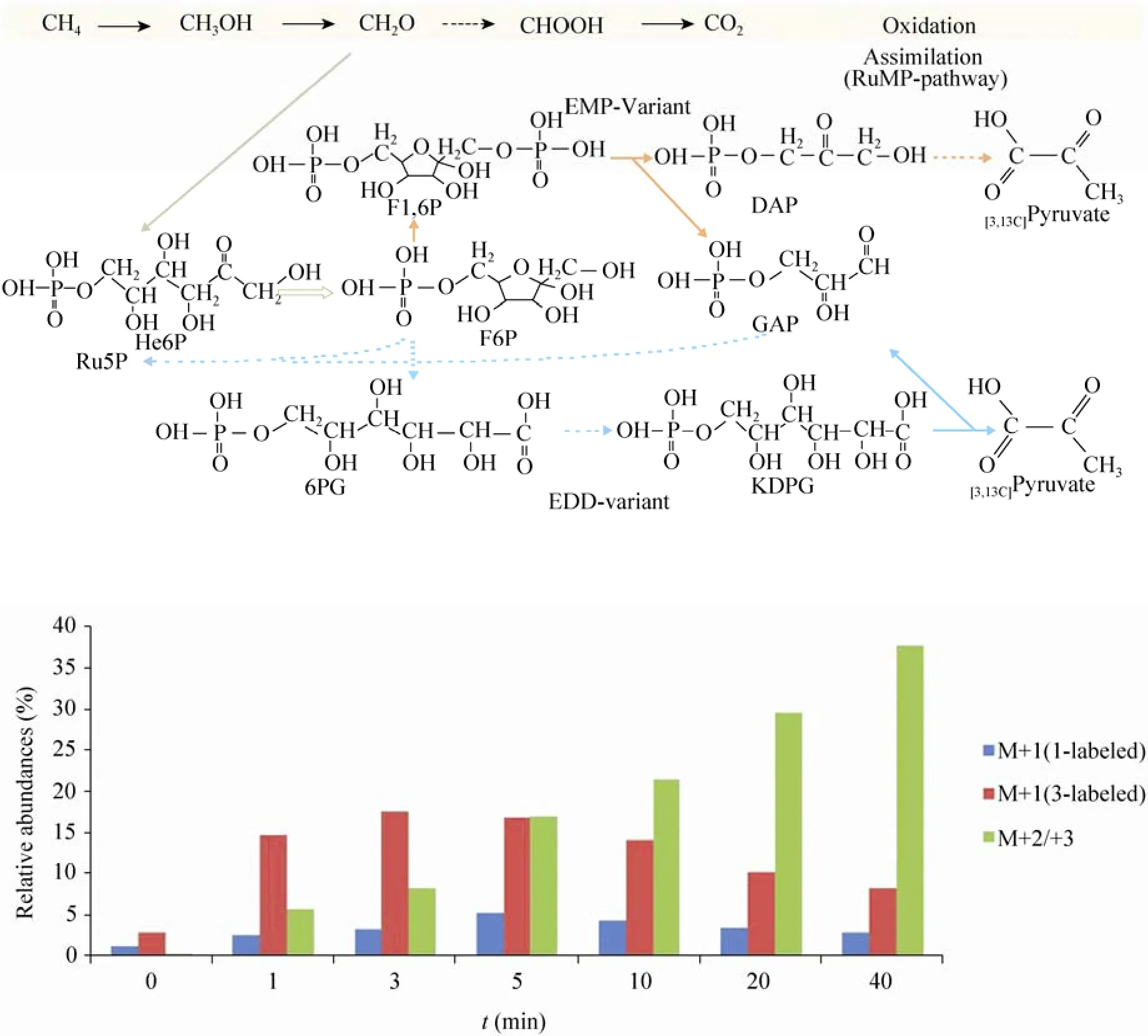
图3 丙酮酸13C示踪分析揭示EMP途径和EDD途径的甲烷同化代谢能力的差异性Fig. 3 Pyruvate as an indicator to distinguish between EMP pathway and EDD pathway. (A)Overview of methane oxidation and the ribulose-monophosphate pathway(RuMP) for formaldehyde assimilation. (B)13C-pyruvate labeling patterns in a time course.
3 讨论

图4 EMP途径是甲烷同化代谢关键通路Fig. 4 Aschematic model depicting EMP pathway is the major route for methane assimilation. The thickness of arrows indicates the level of gene expression. The color of box indicates the concentration of metabolites. Ru5P: ribulose-5-phosphate; He6P: hexulose-6-phosphate; F6P: fructose-6-phosphate; G6P: glucose-6-phosphate; 6PG: 6-phosphogluconate; KDPG: 2-keto-3-deoxy-6-phosphogluconate; GAP: glyceraldehyde-3-phosphate; FBP: fructose-1,6-bisphosphate; DHAP: dihydroxyacetone-phosphate; PEP: phosphoenolpyruvate. Gene predicted function: pmoCAB: particulate methane monooxygenase; mxaFI: PQQ-dependent methanol dehydrogenase; fadh: 2,4-dienoyl-CoA reductase; fdsACD: formate dehydrogenase; fdhA: formate dehydrogenase; hps: 3-hexulosephosphate synthase; hpi: phosphohexuloisomerase; pfk: fructose-2,6-biphosphatase; fba: fructose-bisphosphate aldolase; pykII: pyruvate kinaseII; fdhA: tungsten-containing NAD-dependent formate dehydrogenase; fdsACD: formate dehydrogenase.
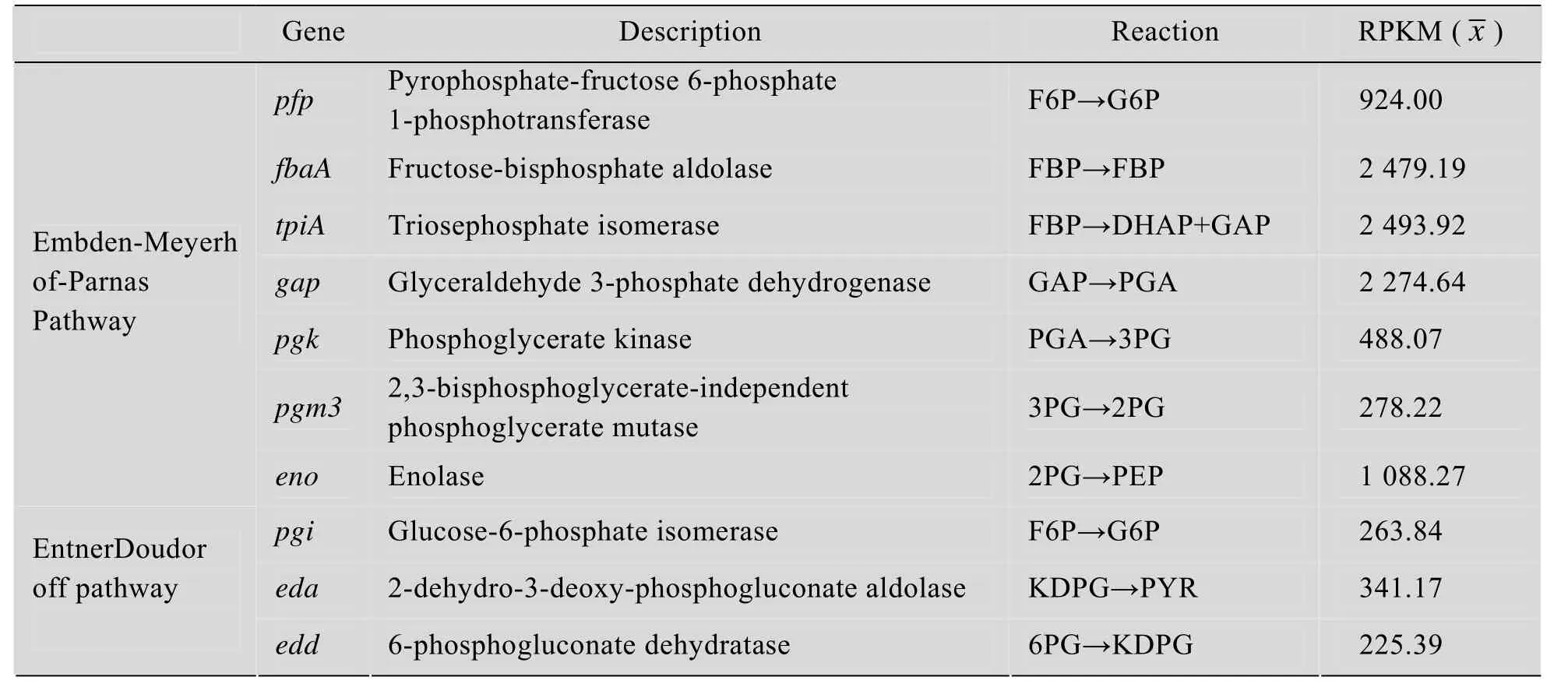
表 2 M. alcaliphilum 20Z的EDD和EMP途径基因表达水平比较分析Table 2 Gene expression profile of EDD and EMP pathways in M. alcaliphilum 20Z
目前对于甲烷氧化菌研究多涉及于生理、生化特征分析和重构代谢网络,以及甲烷单加氧化酶 (Methane monooxygenase enzyme) 催化机理与生物修复功能等的研究[3-5]。相比参与自然界碳循环的其他微生物系 (如:光合作用蓝细菌cyanobacterium Prochlorococcus marinus和甲烷菌Methanopyrus kandleri),甲烷氧化菌基因组研究起步较晚而后基因组研究几乎是空白[20],基因组和后基因组信息的缺少导致对甲烷氧化菌代谢工程调控困难和工业化放大限制。近年来Mary Lidstrom和Murrell Colin等课题组对M. alcaliphilum 20Z、M. trichosporium OB3b、Methylomicrobium buryatense strain 5G等不同生态环境的代表性甲烷氧化菌株进行基因组测序注释[11,21–22],这些前期工作为基因组功能挖掘和代谢调控机制阐明提供了重要的信息基础。M. alcaliphilum 20Z是从盐水湖中分离纯化的中度嗜盐和耐碱(pH7-11)的甲烷氧化菌。本研究工作整合LC-MS、RNA-seq和13C同位素示踪技术分别从代谢物组和转录物组两个层次系统研究M. alcaliphilum 20Z的中心代谢网络。甲烷被甲烷单加氧化酶氧化生成甲醇,进而氧化成甲醛,在3-磷酸己糖合成酶 (hps) 作用下,甲醛和Ru5P合成He6P,He6P在磷酸己糖异构酶(phi) 的作用下转化成F6P。通过EDD途径,F6P依次转化为G6P、6PG、KDPG和KDPG,最终KDPG分解生成GAP和丙酮酸;而在EMP途径中,F6P首先磷酸化生成FBP,进而分解生成DHAP和GAP,DHAP再进一步转化生成PEP和丙酮酸 (图4)。相对于Gammaproteobacteria甲烷氧化菌,Alphaproteobacteria甲烷氧化菌利用丝氨酸循环同化甲醛[5]。本研究的13C同位素富集结果显示丝氨酸循环途径的关键中间代谢物serine和glycine在40 min内13C同位素总富集丰度低于8.0%和7.0%,这进一步证实丝氨酸循环不是M. alcaliphilum 20Z进行甲烷吸收的主要途径。
传统研究认为RuMP 途径的EDD分支途径是同化甲醛主要途径,另一分支EMP是旁路途径。该结论的主要论据是EDD途径的两个关键酶6-磷酸葡萄糖脱水酶 (6-phosphogluconate dehydratase) 和 2-酮-3-脱氧-6-磷酸葡糖酸醛缩酶 (2-keto-3-deoxy-6-phosphogluconate aldolase)有相对更高的活性,而且EMP途径中pyruvate kinase酶活性不能在Gammaproteobacteria甲烷氧化菌中检测到[5,23]。然而我们研究表明,和EDD途径相比较,催化EMP途径关键酶的基因都有相对高丰度的表达,而且EMP途径的中间代谢物丰度明显高于EDD途径。更重要的是利用13C同位素标记技术,我们发现13C标记甲醛快速通过EMP途径被吸收,而且分析EDD和EMP途径的共同终点代谢物pyruvate的13C标记位置,我们更清楚证明高代谢通量通过EMP途径。
综合上述研究,我们提出正常培养条件下的Gammaproteobacteria型甲烷氧化菌
M. alcaliphilum 20Z实际是利用EMP途径而不是EDD途径进行甲烷同化吸收代谢的。由于EMP途径的多个中间代谢物可以作为合成包括C2 (如乙酰辅酶A)、C4 (如琥珀酸) 和C6 (如葡萄糖) 等的重要前体代谢物,这就为进一步利用合成生物学理念,改造M. alcaliphilum 20Z菌株,生产多种高附加值产品提供重要的基础理论。目前研究结果对探讨利用甲烷氧化菌加速甲烷减排的同时有效生物转化甲烷具有重要的科学意义。
REFERENCES
[1] Stolaroff JK, Bhattacharyya S, Smith CA, et al. Review of methane mitigation technologies with application to rapid release of methane from the Arctic. Environ Sci Technol, 2012, 46(12): 6455–6469.
[2] Liu C, Lu XH. Carbon reduction pattern in China: comparison of CCS and biomethane route. CIESC J, 2013, 64(1): 7–10 (in Chinese).刘畅, 陆小华. 我国碳减排模式探讨-CCS路线与生物甲烷路线的比较. 化工学报, 2013, 64(1): 7–10.
[3] Jiang H, Chen Y, Jiang P, et al. Methanotrophs: Multifunctional bacteria with promising applications in environmental bioengineering. Biochem Eng J, 2010, 49: 277–288.
[4] McMullin CL, Pierpont AW, Cundari TR. Complete methane-to-methanol catalytic cycle: a DFT study of oxygen atomtransfer from N2O to late-row (M=Ni, Cu, Zn) β-diketiminate CAH activationcatalysts. Polyhedron, 2013, 52: 945–956.
[5] Trotsenko YA, Murrell JC. Metabolic aspects of aerobic obligate methanotrophy. Adv Appl Microbiol, 2008, 63: 183–229.
[6] Schrader J, Schilling M, Holtmann D, et al. Methanol-based industrial biotechnology: current status and future perspectives of methylotrophic bacteria. Trends Biotechnol, 2009, 27(2): 107–115
[7] Bothe H, Møller Jensen K, Mergel A, et al. Heterotrophic bacteria growing in association with Methylococcus capsulatus (Bath) in a singlecellprotein production process. Appl Microbiol Biotechnol, 2002, 59(1): 33–39.
[8] Khosravi-Darani K, Mokhtari ZB, Amai T, et al. Microbial production of poly(hydroxybutyrate) from C₁ carbon sources. Appl Microbiol Biotechnol, 2013, 97(4): 1407–1424.
[9] Khmelenina VN, Rozova ON, Trotsenko YA. Characterization of the recombinant pyrophosphate-dependent 6-phosphofructokinases from Methylomicrobium alcaliphilum 20Z and Methylococcus capsulatus Bath. Meth Enzymol, 2011, 495:1–14.
[10] Khmelenina VN, Trotsenko YA. Characterization ofthe ectoine biosynthesis genes of haloalkalotolerant obligate methanotroph "Methylomicrobium alcaliphilum 20Z". Arch Microbiol, 2006, 184(5): 286–297.
[11] Vuilleumier S, Khmelenina VN, Bringel F, et al. Genome sequence of the haloalkaliphilic methanotrophic bacterium Methylomicrobium alcaliphilum 20Z. J Bacteriol, 2012, 194(2): 551–552.
[12] Kao WC, Chen YR, Yi EC, et al. Quantitative proteomic analysis of metabolic regulation by copper ions in Methylococcus capsulatus (Bath). J Biol Chem, 2004, 279(49): 51554–51560.
[13] Anthony C. The Biochemistry of Methylotrophys. London: Academic Press, 1982.
[14] Yang S, Sadilek M, Synovec RE, et al. Liquid chromatography-tandem quadrupole mass spectrometry and comprehensive two-dimensional gas chromatography-time-of-flight mass spectrometry measurement of targeted metabolites of Methylobacterium extorquens AM1 grown on two different carbon sources. J Chromatogr A, 2009, 1216(15): 3280–3289.
[15] Yang S, Sadilek M, Lidstrom ME. Streamlined pentafluorophenylpropyl column liquid chromatography-tandem quadrupole mass spectrometry and global13C-labeled internal standards improve performance for quantitative metabolomics in bacteria. J Chromatogr A, 2010, 1217(47): 7401–7410.
[16] Yang S, Synovec RE, Kalyuzhnaya MG, et al. Development of a solid phase extraction protocol coupled with liquid chromatography mass spectrometry to analyze central carbon metabolites in lake sediment microcosms. J Sep Sci, 2013, 4(24): 3597–3605.
[17] Matsen JB, Yang S, Stein LY, et al. Global molecular analyses of methane metabolism in methanotrophic alphaproteobacterium, Methylosinus trichosporium OB3b. Part I: transcriptomics study. Front Microbiol, 2013, doi: 10.3389 /fmicb. 2013. 00040.
[18] Yang S, Matsen JB, Konopka M, et al. Global molecular analyses of methane metabolism in methanotrophic alphaproteobacterium, Methylosinus trichosporium OB3b, Part II: Metabolomics and13C-labeling study. Front Microbiol, 2013, 4: 70. doi: 10.3389/fmicb.2013.00070.
[19] Peyraud R, Kiefer P, Christen P, et al. Demonstration of the ethylmalonyl-CoA pathway by using 13C metabolomics. Proc Natl Acad Sci USA, 2009, 106(12): 4846–4851.
[20] Ward N, Larsen Ø, Sakwa J, et al. Genomic insights into methanotrophy: the complete genome sequence of Methylococcus capsulatus (Bath). PLoS Biol, 2004, 2(10): e303.
[21] Stein LY, Yoon S, Semrau JD, et al. Genome sequence of the obligate methanotroph Methylosinus trichosporium strain OB3b. J Bacteriol, 2010, 192(24): 6497–6498.
[22] Khadem AF, Wieczorek AS, Pol A, et al. Draft genome sequence of the volcano-inhabiting thermoacidophilic methanotroph Methylacidiphilum fumariolicum strain SolV. J Bacteriol, 2012, 194(14): 3729–3730.
[23] Leak DJ, Dalton H. Growth yields of methanotrophs 2. A theoretical analysis. Appl Microbiol Biotechnol, 1986, 23(6): 477–481.
(本文责编 陈宏宇)
Highly efficient methane assimilation through Embden-Meyerhof-Parnas pathway in Methylomicrobium alcaliphilum 20Z
Jinyu Cui1, Lu Yao1, Xiaole Sun2, Marina G. Kalyuzhnaya3, and Song Yang1,4
1 Shangdong Province Key Laboratory of Applied Mycology, School of Life Sciences, Qingdao Agricultural University, Qingdao 266109, Shandong, China
2 Institute of Quality Standards and Testing, Shandong Academy of Agricultural Sciences, Jinan 250100, Shandong, China
3 Department of Microbiology, University of Washington, Seattle, WA 98105, USA
4 Key Laboratory of Systems Bioengineering, Ministry of Education, Tianjin University, Tianjin 300072, China
In order to understand metabolic functions essential for methane assimilation, we investigate dribulose monophosphate pathway and adjacent pathways in gammaproteobacterial Methylomicrobium alcaliphilum 20Z by using combined approaches of RNA-seq, LC-MS, and 13C-labeled techniques. The absolute quantification of metabolome showed that the concentrations of intermediates, such as glucose-6-phosphate and 2-dehydro-3-deoxy-phosphogluconate, involved in Entner-Doudoroff (EDD) pathway were (150.95±28.75) µmol/L and below the limit of detection of mass spectrometry. In contrast, fructose-1, 6-bisphosphate, glyceraldehyde-3-phosphate/dihydroxyacetone and phosphoenolpyruvate in Embden-Meyerhof-Parnas (EMP) pathway had significantly higher concentrations with (1 142.02±302.88) µmol/L, (1 866.76±388.55) µmol/L and (3 067.57±898.13) µmol/L, respectively. 13C-labeling experiment further indicated that the enrichment of [3-13C1]-pyruvate involved in EMP pathway was 4–6 fold higher than [1,13C1]-pyruvate in EDD pathway in a dynamic course. Moreover, gene expression profile showed that the expression levels of genes in EMP pathway (e.g. fbaA, tpiA, gap and pykA) were 2 479.2, 2 493.9, 2 274.6 and 1 846.0, respectively, but gene expressionlevels in EDD pathway (e.g. pgi, eda and edd) were only 263.8, 341.2 and 225.4, respectively. Overall our current results demonstrated that EMP pathway was the main route for methane assimilation in M. alcaliphilum 20Z. This discovery challenged our understanding of methane assimilation pathway in gammaproteobacterial methanotrophic bacteria, and further provided an important insight for efficient methane biocatalysis in the future.
methanotrophs, methane assimilation, methane biocatalysis,13C-labeling metabolomics, transcriptomics
July 24, 2013; Accepted: October 8, 2013
Supported by: the USA Department of Energy (No. DE-SC0005154), Open Funding Project of the Key Laboratory of Systems Bioengineering, Ministry of Education of China, and Start-up Funding of High-level Talent at Qingdao Agricultural university (No. 1312). Corresponding author: Song Yang. Tel/Fax: +86-532-88030327; E-mail: yangsong15@hotmail.com
美国能源部基金 (No. DE-SC0005154) ,中国系统生物工程教育部重点实验室开放课题基金,青岛农业大学高层次人才启动基金(No. 1312)资助。
时间:2013-11-27 网络出版地址:http://www.cnki.net/kcms/detail/11.1998.Q.20131127.1120.005.html

