粗糙脉孢菌木质纤维素降解利用研究进展
林良才,李金根,王 邦,裴 雪,2,田朝光
(1.中国科学院 天津工业生物技术研究所,天津 300308;2.吉林大学 植物科学学院,长春 130062)
丝状真菌粗糙脉孢菌(Neurospora crassa)又称粗糙脉孢霉(以下简称脉孢菌),作为生物遗传学模式生物已有近百年的研究历史,其中“一种基因一种酶学说”获得了1958年诺贝尔奖。作为天然的纤维素快速降解菌,粗糙脉孢菌可以快速降解木质纤维素,并具有利用木糖、纤维二糖、寡糖等组分的能力,有关脉孢菌降解木质纤维素及其纤维素酶的研究早在1950年就有相关报导[1]。20世纪70年代第3次石油危机时,就掀起了一个研究脉胞菌纤维素酶和半纤维素酶的小高潮,不仅对脉孢菌产纤维素酶的条件、纤维素酶及半纤维素酶酶活性质进行了研究[2-3],而且深入探讨了利用脉孢菌直接发酵木质纤维素生产乙醇的可能性[4]。随着分子生物学技术的不断发展,20世纪90年代初期,脉孢菌纤维素的外切酶cbh-1首次被克隆、测序[5]。在我国,最早利用脉孢菌开展纤维素酶研究的是山东大学曲音波教授,其团队先后对脉孢菌产纤维素酶及其木糖发酵进行了系统研究[6-7]。此外,天津理工大学冯炘教授针对脉孢菌发酵纤维素酶过程优化进行了深入研究[8]。近年来,伴随着转录组、蛋白组等组学技术的兴起,脉孢菌纤维素降解机制和应用的研究再次成为热点,并取得了多个方面的显著进展,包括从组学水平系统研究纤维素降解过程、多糖单加氧酶(PMO)(GH61)家族在纤维素降解中功能、纤维素酶表达调控以及生物质糖转运蛋白功能的研究等。本文中笔者将就这些方面进行重点综述。
1 粗糙脉孢菌木质纤维素降解全基因组水平研究
组学技术的兴起将真菌木质纤维素降解的研究迅速提升到全基因水平。一批重要的木质纤维素降解真菌的基因组相继被测序。脉孢菌是第一个被测序的丝状真菌[9],随后公布了里氏木霉(Trichoderma reesei)的基因组数据[10]。比较基因组学分析显示,脉孢菌含有的纤维素酶数目是里氏木霉的两倍之多,同时还拥有诸多半纤维素酶和相关转录因子[10]。2009 年,Tian 等[11]以脉孢菌为研究对象,利用丰富的突变体库,综合运用转录组学和蛋白质组学技术,首次对纤维素降解真菌进行了全基因组学水平的系统研究。基因差异分析显示,脉孢菌在芒草秸秆培养基上有700多个基因转录本表达显著变化(相比于蔗糖培养基)。其中在23个预测的纤维素酶基因中有18个快速显著上调;在预测的19个半纤维素基因中有13个显著上调。同时,一些转录因子、糖转运蛋白以及未知功能蛋白也被显著诱导表达。通过对胞外分泌蛋白进行质谱分析,确证了10个纤维素酶和8个半纤维素酶。这些组学研究结果为后续挖掘新型纤维素降解酶系以及遗传改造脉孢菌提供了丰富的数据。随后,美国加州大学伯克利分校Louise Glass实验室继续以脉孢菌为对象,对半纤维素降解调控进行了转录组和分泌蛋白组水平的研究[12]。除鉴定了353个基因在木聚糖条件下显著上调表达以及34个胞外分泌蛋白以外,还证明了xlr-1(NCU06971)是脉孢菌主要的半纤维素酶基因表达正调控因子,指明xlr-1对半纤维素降解调控作用普遍存在于脉孢菌、里氏木霉、黑曲霉等纤维素降解真菌中。Schmoll等[13]发现光感受基因wc-1/2(white collar)及vvd参与了纤维素酶表达调控。研究指出,WC-1/2组成的复合物WCC(white collar complex)在脉孢菌生长早期负调控纤维素酶表达,但是随着vvd感受光刺激产生光适应,WCC活性被抑制,进而增强了细胞对纤维素的利用能力。值得注意的是,wc基因除了形成复合物间接对纤维素酶表达起调控作用之外,还能分别独立地间接负调控纤维素酶表达,但其具体的调控通路仍不明晰。此外,伯克利分校的 Phillips等[14]实验室运用蛋白质谱定量技术,首次对脉孢菌在微结晶纤维素上的分泌蛋白做了定量分析,测定了胞外分泌纤维素酶的成分比例。结果显示,CBH-1(NCU07340)、 GH6-2(NCU09680)、 GH5-1(NCU00762)和GH3-4(NCU04952)是脉孢菌最主要的纤维素酶系成分,占到胞外蛋白总质量的63%~65%。随后,对这4种酶进行了cocktail配比实验,发现比例趋势基本符合脉孢菌本源的分泌蛋白组。然而,这个最优配比的纤维素酶系的酶活只能达到本源分泌酶混合物的43%,由此推测纤维素酶水解促进蛋白(包括PMO、纤维二糖脱氢酶(CDH)等)也起着不可忽略的作用。充分利用脉孢菌全基因组突变体库和功能基因组学工具,开展纤维素降解途径新基因和新蛋白的挖掘,必将大大加深对木质纤维素降解和纤维素酶表达分泌的基因组水平的理解。
2 粗糙脉孢菌PMO家族纤维素酶研究
纤维素降解是通过内切葡聚糖酶、外切葡聚糖酶以及β-葡糖苷酶3种酶协同作用完成。基因组预测显示,脉孢菌含有10个内切葡聚糖酶、6个外切葡聚糖酶和7个β-葡萄糖苷基因。早在20世纪50年代,Reese等[15]就提出可以通过添加一种能破坏底物聚合结构的非水解组分来提高纤维素酶的降解效率,促进纤维素的水解。PMO家族的酶就具有这种功能,该家族蛋白是铜依赖的溶解多糖单加氧酶,具有较弱的内切葡聚糖酶酶活,其与CDH结合可增强纤维素酶的水解能力,如GH61家族蛋白。在商业化的纤维素酶中添加少量的该家族的蛋白即可显著提高对纤维素的降解作用,故在木质纤维素糖化产业化中有巨大的应用前景,也是近年来的研究热点和重点。
迄今为止,在很多纤维素降解真菌中都发现了pmo基因,脉胞菌基因组中有14个 pmo基因。Phillips等[14]对N.crassa在微晶纤维素上生长所分泌的胞外蛋白进行定量分析,检测到4个PMO蛋白,共占分泌蛋白总量的15%。由此可以推断,无论是从该家族基因数量还是其分泌量都可看出PMO家族蛋白在纤维素降解过程中发挥着重要的作用。
Phillips 等[14]、Beeson 等[16]通过对脉孢菌中的PMO家族蛋白的研究发现,该家族蛋白的N-端有保守的氨基酸结构域以及2个与金属离子相结合的保守的组氨酸位点。PMO家族的酶通过与CDH或者一些低相对分子质量的还原剂(如抗坏血酸)发生氧化还原反应,氧化纤维素、破坏糖苷键、裂解纤维素链上的不同部位C原子(C-1、C-4和C-6),进而达到裂解纤维素的目的[17]。Beeson 等[16]的研究表明 PMO-5 氧化裂解纤维素作用位点位于纤维素链上的C-1位置,而PMO-4则是作用在C-4和C-6位置。
Kittl等[17]在 Picha pastoris中表达了 N.crassa的4个PMO家族蛋白,分别是PMO3、PMO5、PMO6和PMO10,其中PMO-3、PMO-5和PMO-10具有碳水化合物结合模块(CBM)结构域。通过对N.crassa在芒草上的转录表达水平分析显示,PMO-5表达水平提高了107倍,PMO-3提高了85倍,PMO-6上调了26倍,而PMO-10表达水平几乎没有变化。在P.pastoris中重组表达后证实,这些酶为铜依赖的PMO,并且CDH可以有效提高其活性,可以促进纤维素的氧化裂解。然而,已公布的80%的PMO家族蛋白的序列在C端都不具有纤维素结合域。Harris等[18]指出是否存在该结构域与其增加纤维素水解的能力无关;同时也指出在加入Ca2+、Mg2+、Co2+、Mn2+、Ni2+和 Zn2+时,PMO 家族蛋白可显著提高纤维素酶对底物的水解能力;然而当不含有这些金属离子时,添加PMO家族蛋白对纤维素酶的水解能力无显著影响。同时,Quinlan等[19]通过研究Thermoascusaurantiacus中的 PMO家族蛋白(TaGH61A),发现当存在没食子酸(gallic acid)等电子供体的情况下,该蛋白可促进纤维素的氧化裂解。Kittl等[17]也在N.crassa中证实了这一观点。
由于PMO家族蛋白可以加速纤维素酶对生物质的降解,从而降低纤维素酶的用量,并缩短其反应时间,因此PMO家族蛋白的发现和应用将会对木质纤维素的利用、转化发挥重要的作用。在T.reesei中共表达一个高活性的PMO蛋白能够使其纤维素酶活提高2倍[19]。诺维信公司(Novozymes)生产的一个高效纤维素酶Cellic®CTec2中就添加了PMO蛋白,其酶活相对于之前的产品就有了显著的提高[20]。PMO家族基因的开发与利用可有效提高纤维素酶活,并且减少纤维素酶的用量,未来如果能够大规模应用,将有助于进一步降低生物糖化成本,从而促进生物质资源的有效利用。
3 纤维素酶表达调控研究进展
纤维素酶是在胞内表达合成后经分泌系统分泌至胞外的,而诱导纤维素酶合成的天然底物是不溶性的,无法进入胞内产生诱导表达响应,因此它如何产生诱导作用成为了关注的热点。基于对一些寡糖及其衍生物如纤维二糖、δ-纤维二糖内酯、乳糖和槐糖对纤维素酶表达合成具有强信号诱导作用的研究认识,通常认为木质纤维素降解产生的寡糖及其衍生物是作为事实上的诱导底物,产生信号分子,进一步诱导木质纤维素酶系基因的大量表达[21]。槐糖是由β-1,2糖苷酶连接的葡萄糖组成,能够诱导高水平的纤维素酶表达,通常被认为是极佳的天然可溶诱导物[22-23],尤其是在 T.reesei中,通常认为它是由纤维二糖在β-葡萄糖苷酶的转糖基作用形成的[24]。纤维二糖是纤维素降解中的重要可溶性中间产物,能够产生信号分子,诱导纤维素酶的表达,被认为是纤维素酶表达的自然诱导物,但纤维二糖诱导纤维素酶的分子水平机制尚不清楚。在通常条件下,纤维二糖被β-葡萄糖苷酶降解成为葡萄糖,并且引起阻遏效应,其对纤维素酶的诱导作用无法体现。粗糙脉孢菌通过敲除 3个主 要 的 β-葡 萄 糖 苷 酶(NCU00130、NCU04952和NCU08755)可以实现在纤维二糖条件下直接表达纤维素酶。突变体Δ3βG在纤维二糖的诱导条件下其蛋白水平和转录水平的响应与野生型菌株在纤维素诱导条件下的响应相类似。由此推测,其他纤维素降解丝状真菌的β-葡萄糖苷酶缺失菌株可以直接以可溶性的纤维二糖作为诱导剂诱导表达纤维素酶。
近年来,对纤维素酶基因表达调控研究主要集中在木霉、曲霉等工业微生物上[25](图1)。目前已知的调控基因有转录激活因子XInR[26-29]、转录抑制因子Ace1[30-31]、激 活 因 子 Ace2[32-34]、葡 萄 糖 阻 遏 蛋 白Cre1[35-36]、锌指结构转录因子 PacC[37-39]、CCAAT 结合复合体 Hap2/3/5[40-42]和 GATA 因子 AreA[43-44]。在曲霉和木霉中,转录因子XlnR/XYR1对纤维素酶和半纤维素酶具有重要的调控作用。然而,XlnR/XYR1在脉孢菌和镰刀霉中的同源基因并不是纤维素酶表达所必须的,它们是菌体利用半纤维素所必需的,并且调控半纤维素酶的表达。加州大学伯克利分校Glass实验室对200多个粗糙脉孢菌转录因子敲除菌株在纤维二糖上进行了生长和酶活力检测的筛选,获得了2个在子囊真菌中较保守的Zn(Ⅱ)2Cys6家族转录因子clr-1和clr-2,研究结果表明这2个转录因子是所有主要的纤维素酶基因和大部分半纤维酶基因诱导表达所必须的转录因子[45]。突变株Δclr-1和Δclr-2丧失了在纤维素条件下表达、分泌纤维素酶的能力。然而,这2个突变株在木聚糖条件下依旧可以正常地生长和分泌半纤维素酶。由此推测,在一些纤维素降解丝状真菌中,纤维素酶和半纤维素酶的表达调控相互独立,而clr-1和clr-2正是粗糙脉孢菌纤维素酶表达调控的重要组成部分。纤维二糖及其转糖基产物作为纤维素酶最初的诱导物。在纤维二糖或是其转糖基产物的诱导下,clr-1被激活,促进一系列可有效利用纤维二糖的基因表达,如clr-2等。clr-2可以进一步直接诱导纤维素酶和某些半纤维素酶的表达。在构巢曲霉中,诱导纤维素酶表达所需的是clrB(clr-2的同源基因),而不是clrA(clr-1的同源基因)。然而,clrA的缺失将导致构巢曲霉利用纤维二糖的能力的丧失。因此,clr-1/clrA在丝状真菌纤维二糖感受途径中具有重要的保守作用。
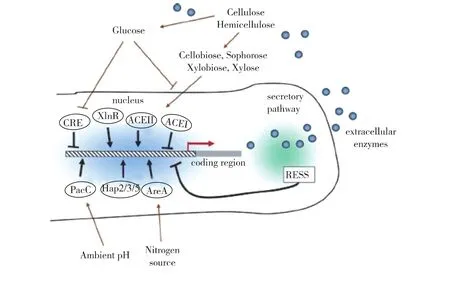
图1 丝状真菌中参与纤维素酶调控机制示意[25]Fig.1 Schematic representation of the different fungal trans-acting factors and regulatory responses affecting cellulase expression[25]
转录因子clr-1和clr-2都属于锌指蛋白转录因子超家族,锌指蛋白是一类具有手指结构域的转录因子,对基因表达调控、细胞分化、胚胎发育、增强植物抗逆性等方面具有重要的作用,该家族蛋白最初于1983年在非洲爪蟾卵母细胞的转录因子TFⅢA中被发现[46-47],是迄今在真核生物基因组中分布最广的一类蛋白,也是真核生物转录调控因子中最大的家族之一。锌指蛋白可以根据高度保守的氨基酸序列分为三大类:①经典锌指蛋白Cys2His2型,是真核生物中最为普遍存在的转录调控因子,通常以单体的形式与核酸相互作用,如人的转录因子Sp1、曲霉的碳代谢阻遏调控因子creA;②Cys4型锌指蛋白,与Cys2His2型不同,以二聚体的形式与DNA相作用,其同源二聚体识别目标基因的反向重复序列,异源二聚体可以与正向重复序列相结合;③C6型锌指又称锌簇或锌双核簇(Zn(Ⅱ)2Cys6或Zn2C6),包含一个由6个半胱氨酸围绕着2个锌离子而形成的DNA结合域。值得指出的是,该锌指家族蛋白是真菌所特有的转录因子,其可以与DNA以单体、同源二聚体或异源二聚体形式结合[48]。
笔者通过对粗糙脉孢菌锌指蛋白超家族转录因子敲除突变体库的进一步系统筛选,获得了多个与纤维素酶表达异常的突变体。研究表明,在以结晶纤维素作为唯一C源的条件下,有3株突变体的蛋白分泌量和纤维素酶活力显著上升,而显著下降的有2株(未发表)。目前,正在深入研究这些潜在的基因对纤维素酶表达、分泌的调控机制以及与其他转录调控因子之间的关系。这些重要的转录因子功能的揭示将有助于对丝状真菌木质素降解途径的认识,更有利于形成高产纤维素酶的工程菌株,从而提高木质纤维素生物质的利用效率。
4 生物质糖转运蛋白克隆鉴定及其在生物质乙醇发酵中的应用
作为木质纤维素主要成分,纤维素和半纤维素是多种单糖分子聚合而成的复杂高聚碳水化合物,通过复合酶系可以水解为各种单糖和寡糖(图2)。而对水解产物的利用,是纤维素转化为生物燃料和化学品的重要研究内容。特别是关于木质纤维素降解真菌,是如何将降解的单糖和寡糖运入细胞,转运的过程如何受到细胞内外因素的调控以及不同的糖分子如何被特异性地转运,这些方面的研究仍然非常薄弱。深入研究纤维素降解真菌(包括脉孢菌)对纤维素的利用,对于理清纤维素降解机制及糖到化学品的微生物转化都十分必要。
从脉孢菌在纤维素和半纤维素条件下的转录组数据出发,已经先后克隆了脉孢菌的3个非常重要的糖转运蛋白 CDT1(NCU00801),CDT2(NCU08114)和An25(NCU00821)。其中,CDT1和CDT2为纤维寡糖转运蛋白,An25为木糖特异的转运蛋白。CDT1自被克隆以来,在纤维二糖发酵以及与木糖、半乳糖等戊糖共发酵方面取得了显著进展,这方面工作主要来自美国伊利诺伊大学金永株(Yong-su Jin)实验室和赵惠民实验室。他们在能发酵木糖的酵母中引入脉孢菌的CDT1和胞内β-葡萄糖苷酶(NCU04952),构建出能够同时利用纤维二糖和木糖发酵产乙醇的工程酵母,而胞内水解纤维二糖的策略也大大减缓了胞外葡萄糖产生的阻遏效应[49-50]。基于相同的思路,金永株实验室还构建了纤维二糖/半乳糖共发酵的工程酵母,拓宽了酵母发酵的底物谱[51]。最近,赵慧民实验室采用纤维二糖代谢途径定向进化的策略,大大提高了酵母利用纤维二糖的能力,同时将乙醇产量提高到1 g/L/h[52]。此外,研究者也尝试用含纤维素酶cocktail和含 CDT1的酵母做 CBP(consolidated bioprocess)产乙醇[53],以及利用纤维二糖发酵产其他化学品[54-55]。总的说来,脉孢菌高亲和性纤维寡糖转运蛋白的鉴定与应用极大地促进了混合糖发酵的研究,为纤维素乙醇及其他纤维素化学品发酵提供了新的思路和工程改造靶点。
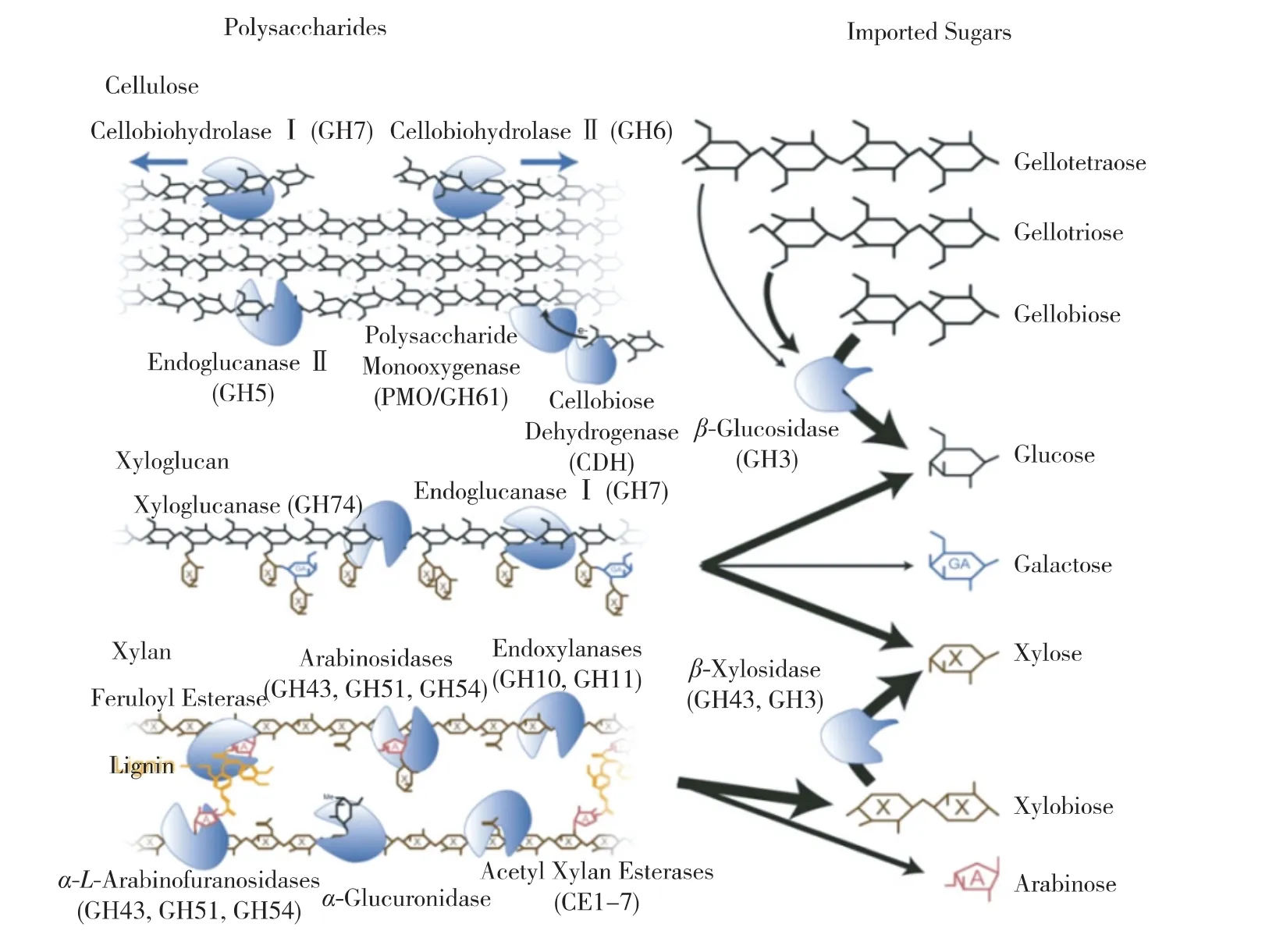
图2 粗糙脉孢菌中参与降解木质纤维素的各种酶系[45]Fig.2 Characterized enzyme classesinvolved in lignocelluloses breakdown identified in the N.crassa genome[45]
在五碳糖转运蛋白方面,过去的研究多集中在利用己糖转运蛋白等底物广谱性转运蛋白来转运五碳糖,而对于特异性五碳糖转运蛋白(pentose specific transporter)的研究才刚刚起步。美国伊利诺伊大学赵慧民教授实验室分别从粗糙脉孢菌和毕赤酵母中鉴定了2个木糖特异转运蛋白(An25和Xyp29)[56]。作为半纤维素的主要成分之一,阿拉伯糖的利用对木质纤维素彻底转化利用至关重要,国际上关于阿拉伯糖转运蛋白研究逐渐开始涉及。2011年,芬兰VTT研究中心Richard实验室的科学家从Ambrosiozyme monospora酵母中克隆2个阿拉伯糖特异性转运基因(LAT1,LAT2),但该阿拉伯糖转运蛋白亲和力不高[57],寻找亲和力更高的阿拉伯糖转运蛋白是研究阿拉伯糖转化利用的核心问题之一。
基于转录组学数据和序列结构域分析,目前脉孢菌全基因组被预测有39个糖转运蛋白(未发表数据),基本属于膜蛋白家族MFS(major facilitator superfamily)[58]。依据转运的机制,MFS 又分为简单转运蛋白(uniporter)(又称协助扩散蛋白(facilitateddiffusion protein)、同向转运蛋白(symporter)以及反向转运蛋白(antiporter)。Uniporter,依靠底物浓度梯度驱动转运,转运蛋白主要起着协助运输的作用。Symporter,向同一个方向同时转运2种及以上的底物,并以其中一种底物的电化学梯度作为推动力,常见的有 sugar/H+、glucose/Na+、phosphate/H+、nucleoside/H+以 及nitrate/H+等等。Antiporter,向反方向协同转运2种及以上的底物,驱动力来源和Symporter一样,这一类里很多都是drag/H+反向转运蛋白。真菌中的糖转运蛋白主要是uniporter和symporter/H+类型[58]。早在1974年已有报道指出,脉孢菌在主动转运糖进入细胞的同时会协同转运质子[59],但在分子水平上哪些糖转运蛋白的作用导致表观上电势的变化至今仍没有被阐释。此外,由于自然界中脉孢菌能生长在树枝、秸秆等各种干枯的木质纤维上,可以预见其糖转运蛋白家族具有很高的功能多样性。同时,在长期的进化适应过程中,细胞面对不同的生长环境,如酸性-碱性、碳充足-碳匮乏,会有很好的策略来协同这些转运蛋白之间的转运工作,以使得其能高效地吸收外界的营养。全基因组系统解析丝状真菌中糖转运蛋白动力类型和生化特征,对于研究丝状真菌环境适应性,理清纤维素降解菌利用木质纤维素的机制有重要意义。更进一步地,这种高效的糖转运吸收方式,有可能对改造工程菌(包括酵母、曲霉和木霉)以充分利用培养基中的残糖发酵,提高糖的转化利用率有一定的指导意义。
5 粗糙脉孢菌与生物质一步转化
生物质具有成本低、可再生以及产量大等特点,是生产生物燃料及化学品的重要原料。利用生物质进行发酵一般要经过预处理、酶解、发酵以及最后产品的分离纯化。其中,预处理和酶解过程是整个过程中成本最重的部分。尽管木霉、曲霉和白腐菌等木质纤维素降解真菌已被广泛地应用于生物质纤维素预处理和生产纤维素酶过程中。目前为止,直接利用它们将生物质转化为糖或是化学品却未见报道。因此,如何直接利用廉价的生物质作为底物生产清洁能源或是工业化学品成为了今后研究的发展方向。粗糙脉孢菌作为丝状真菌的模式菌株不仅具有降解木质纤维素所需的酶系,而且还能够利用降解产物进行发酵生产乙醇等化学品[7]。这种直接从初始的生物质原料一步发酵生产生物产品的工艺将极大地降低生产成本,并且大幅提高生物质中的碳转化效率。
脉孢菌能够利用生物质降解物中的各种糖类(葡萄糖、木糖及阿拉伯糖等)进行发酵生产乙醇等。张潇等[7]研究了不同条件对脉孢菌AS3.1602木糖发酵的影响,研究表明脉孢菌具有较强的发酵木糖产生乙醇及木糖醇的能力。在木糖发酵中,乙醇及木糖醇等产物产率很大程度上决定于通氧条件、培养基初始pH值及糖浓度等环境因素。由于辅因子的不同,真菌中木糖代谢具有氧化还原不平衡的特点。Zhao等[60]经过研究证明,在脉孢菌中存在2种木糖还原酶的同工酶(EI和EII),其中木糖还原酶EII能够同时利用NADH与NADPH作为辅因子催化氧化还原反应,从而有助于减缓细胞内氧化还原不平衡,促进的木糖的代谢,进而提高发酵效率。Zhang等[61]探讨了在限氧条件下脉孢菌的木糖代谢,在最佳通氧条件,乙醇产量达到6.7 g/L,转化率为66%。
脉孢菌发酵木质纤维素生产乙醇一般要经过两个阶段:第一阶段在有氧条件下,分泌降解纤维素的各种酶类;第二阶段,在厌氧或微好氧条件下,将水解产物发酵生产乙醇。Xiros等[62]将脉孢菌DSM 1129在高粱残渣固体中培养,而后在厌氧或微好氧条件下进行液体发酵,其乙醇产量为75 g/L,研究表明用碱液对生物质进行预处理有利于乙醇产量的提高,且产生的潜在抑制物对发酵无抑制作用。Okonko等[63]报道了脉孢菌在37℃下经过可以直接发酵甘蔗渣生产乙醇。
除了纤维素乙醇工艺的整合之外,Wu等[64]通过有性杂交得到β-葡萄糖苷酶的六突变体,其中菌株F5能够降解纤维素积累纤维二糖(7.7 g/L)。在过量表达纤维二糖脱氢酶的情况下,菌株F5能够利用纤维素发酵生产纤维二糖酸(0.4 g/L)及纤维二糖(6.5 g/L),从而将纤维素酶的酶的生产和酶解过程进行整合。
此外,脉孢菌体内还存在丰富的蛋白质、维生素B12及脂类等,同时还具有良好的蛋白表达、分泌能力,然而利用木质纤维素直接发酵生产这些高附加值产品还有待进一步的研究。
6 展望
粗糙脉孢菌作为纤维素降解模式真菌,在国际上有较大的研究群体,在技术方法和文献资源上具有其他真菌所没有的优势,使得脉孢菌纤维素降解机理研究能够在真菌中处于领先地位。可以预见,除了已经取得进展之外,未来几年,利用粗糙脉孢菌系统在木质纤维素降解利用方面的研究将会有更大进展。美国伯克利能源生命科学研究所(EBI)重点以粗糙脉孢菌系统为依托,部署了一系列的研究项目(表1)。在该方向正在开展的8个项目(Program)中有4个涉及粗糙脉孢菌体系,其中2个主要研究粗糙脉孢菌(蛋白分泌途径和木质纤维素转化生物燃料),另外2个利用粗糙脉孢菌研究果胶降解组学和纤维素酶系的表达筛选。此外,在该方向14个资助课题(Project)中有3个是粗糙脉孢菌研究(脂肪酸合成,代谢组学重构和生物质降解机制)。预计该系列研究将带动整个脉孢菌在木质纤维素降解机制,蛋白质合成分泌等方面取得新一轮进展,同时也表明粗糙脉孢菌木质纤维素降解利用研究竞争会更加激烈,包括机理研究和利用该菌进行纤维素酶生产、蛋白表达系统开发以及燃料化学品生产等方面。

表1 EBI木质纤维素降解领域资助的有关粗糙脉孢菌的项目和课题Table 1 Long-term programs and short-term projects in cellulose degradation supported by Energy Biosciences Institute(EBI)
[1] Perlman D.Observations on the production of ethanol by fungi and yeasts[J].Am J Bot,1950,37(3):237-241.
[2] Eberhart B M,Beck R S,Goolsby K M.Cellulase of Neurospora crassa[J].J Bacteriol,1977,130(1):181-186.
[3] Rao M,Deshpande V,Keskar S,et al.Cellulase and ethanol production from cellulose by Neurospora crassa[J].Enzyme Microb Technol,1983,5(2):133-136.
[4] Deshpande V,Keskar S,Mishra C,et al.Direct conversion of cellulose/hemicelluloses to ethanol by Neurospora crassa[J].Enzyme Microb Technol,1986,8(3):149-152.
[5] Taleb F,Radford A.The cellulase complex of Neurospora crassa:cbh-1 cloning,sequencing and homologies[J].Gene,1955,161(1):137-138.
[6] 张潇,朱冬青,王丹,等.粗糙脉孢菌(Neurospora crassa)木聚糖酶的合成及性质研究[J].维生素科学与技术,2002,10(4):20-26.
[7] 张潇,朱冬青,王丹,等.粗糙脉孢菌(Neurospora crassa)木糖发酵的研究[J].微生物学报,2003,43(4):466-472.
[8] 冯炘,王丹,辛丽霞,等.粗糙脉胞菌(Neurospora crassa)产纤维素酶发酵条件研究[J].食品科学,2005,26(1):67-70.
[9] Galagan J E,Calvo S E,Borkovich K A,et al.The genome sequence of the filamentous fungus Neurospora crassa[J].Nature,2003,422:859-868.
[10] Martinez D,Berka R M,Henrissat B,et al.Genome sequencing and analysis of the biomass-degrading fungus Trichoderma reesei(syn.Hypocrea jecorina)[J].Nat Biotechnol,2008,26(5):553-560.
[11] Tian C,Beeson W T,Iavarone A T,et al.Systems analysis of plant cell wall degradation by the model filamentous fungus Neurospora crassa[J].PNAS,2009,106(52):22157-22162.
[12] Sun J,Tian C,Diamind S,et al.Deciphering transcriptional regulatory mechanisms associated with hemicellulose degradation in Neurospora crassa[J].Eukaryotic Cell,2012,11(4):482-493.
[13] Schmoll M,Tian C,Sun J,et al.Unravelling the molecular basis for lightmodulated cellulase gene expression:the role of photoreceptors in Neurospora crassa[J].BMC Genomics,2012,doi:10.1186/1471-2164-13-127.
[14] Phillips C M,Iavarone A T,Marletta M A,et al.Quantitative proteomic approach for cellulose degradation by Neurospora crassa[J].J Proteome Res,2011,10(9):4177-4185.
[15] Reese E T,Siu R G H,Levinson H S,et al.The biological degradation of soluble cellulose derivatives and its relationship to the mechanism of cellulose hydrolysis[J].J Bacteriol,1950,59(4):485-497.
[16] Beeson W T,Phillips C M,Cate J H,et al.Oxidative cleavage of cellulose by fungal copper-dependent polysaccharide monooxygenases[J].J Am Chem Soc,2012,134(2):890-892.
[17] Kittl R,Kracher D,Burgstaller D,et al.Production of four Neurospora crassa lytic polysaccharide monooxygenases in Pichia pastorismonitored by a fluorimetric assay[J].Biotechnol Biofuels,2012,5(1):79.
[18] Harris P V,Welner D,McFarland K C,et al.Stimulation of lignocellulosic biomass hydrolysis by proteins ofglycoside hydrolase family 61:structure andfunction of a large,enigmatic family[J].Biochemistry,2010,49(15):3305-3316.
[19] Quinlan R J,Sweeney M D,Lo Leggio L,et al.Insights into the oxidative degradation of cellulose by a copper metalloenzyme that exploitsbiomasscomponents[J].PNAS,2011,108(37):15079-15084.
[20] Cannella D,Hsieh C W,Felby C,et al.Production and effect of aldonic acids during enzymatic hydrolysis of lignocellulose at high dry matter content[J].Biotechnol Biofuels,2012,doi:10.1186/1754-6834-5-26.
[21] Mach R L,Zeilinger S.Regulation of gene expression in industrial fungi:Trichoderma[J].Appl Microbiol Biotechnol,2003,60(5):515-522.
[22] Mandels M,Reese E T.Induction of cellulase in fungi by cellobiose[J].J Bacteriol,1960,79(6):816-826.
[23] Mandels M,Parrish F W,Reese E T.Sophorose as an inducer of cellulase in Trichoderma viride[J].J Bacteriol,1962,83(2):400-408.
[24] Vaheri M,Leisola M,Kauppinen V.Transglycosylation products of cellulase system of Trichoderma reesei[J].Biotechnol Lett,1979,1(1):41-46.
[25] Aro N,Pakula T,Penttilä M.Transcriptional regulation of plant cell wall degradation by filamentous fungi[J].FEMS Microbiol Rev,2005,29(4):719-739.
[26] Gielkens M M,Dekkers E,Visser J,et al.Two cellobiohydrolaseencoding genes from Aspergillus niger require D-xylose and the xylanolytic transcriptional activator XlnR for their expression[J].Appl Environ Microbiol,1999,65(10):4340-4345.
[27] Hasper A A,Visser J,de Graaff L H.The Aspergillus niger transcriptionalactivator XlnR,which is involved in the degradation of the polysaccharides xylan and cellulose,also regulates D-xylose reductase gene expression[J].Mol Microbiol,2000,36(1):193-200.
[28] Rauscher R,Würleitner E,Wacenovsky C,et al.Transcriptional regulation of xyn1,encoding xylanase I,in Hypocrea jecorina[J].Eukaryot Cell,2006,5(3):447-456.
[29] Stricker A R,Steiger M G,Mach R L.Xyr1 receives the lactose induction signal and regulates lactose metabolism in Hypocrea jecorina[J].FEBS Lett,2007,581(21):3915-3920.
[30] Saloheimo A,Aro N,Ilmén M,et al.Isolation of the ace1 gene encoding a Cys2-His2transcription factor involved in regulation of activity of the cellulase promoter cbh1 of Trichoderma reesei[J].J Biol Chem,2000,275(8):5817-5825.
[31] PortnoyT,MargeotA,Seidl-Seiboth V,etal.Differential regulation of the cellulase transcription factors XYR1,ACE2,and ACE1 in Trichoderma reesei strains producing high and low levels of cellulase[J].Eukaryot Cell,2011,10(2):262-271.
[32] Stricker A R,Mach R L,deGraaffL H.Regulation of transcription of cellulases-and hemicellulases-encoding genes in Aspergillus niger and Hypocrea jecorina(Trichoderma reesei)[J].Appl Microbiol Biotechnol,2008,78(2):211-220.
[33] Aro N,Saloheimo A,Ilmén M.ACEII,a novel transcriptional activator involved in regulation of cellulase and xylanase genes of Trichoderma reesei[J].J Biol Chem,2001,276(26):24309-24314.
[34] Kubicek C P,Mikus M,Schuster A,et al.Metabolic engineering strategies for the improvement of cellulase production by Hypocrea jecorina[J].Biotechnol Biofuels,2009,doi:10.1186/1754-6834-2-19.
[35] de Graaff L H,van den Broeck H C,van Ooijen A J,et al.Regulation of the xylanase-encoding xlnA gene of Aspergillus tubigensis[J].Mol Microbiol,1994,12(3):479-490.
[36] de Vries,R P,Visser J,de Graaff L H.CreA modulates the XlnR-induced expression on xylose of Aspergillus niger genes involved in xylan degradation[J].Res Microbiol,1999,150(4):281-285.
[37] Suárez T,Peñalva M A.Characterization ofa Penicillium chrysogenum gene encoding a PacC transcription factor and its binding sites in the divergent pcbAB-pcbC promoter of the penicillin biosynthetic cluster[J].Mol Microbiol,1996,20(3):529-540.
[38] Negrete-Urtasun S,Reiter W,Diez E,et al.Ambient pH signal transduction in Aspergillus:completion of gene characterization[J].Mol Microbiol,1999,33(5):994-1003.
[39] MacCabe A P,Orejas M,Pérez-González J A,et al.Opposite patterns of expression of two Aspergillus nidulans xylanase genes with respect to ambient pH[J].J Bacteriol,1998,180(5):1331-1333.
[40] Zeilinger S,Mach R L,Kubicek C P.Two adjacent protein binding motifs in the cbh2(cellobiohydrolase II-encoding)promoter of the fungus Hypocrea jecorina(Trichoderma reesei)cooperate in the induction by cellulose[J].J Biol Chem,1998,273(51):34463-34471.
[41] Zeilinger S,Ebner A,Marosits T,et al.The Hypocrea jecorina HAP 2/3/5 protein complex binds to the inverted CCAAT-box(ATTGG)within the cbh2(cellobiohydrolase II-gene)activating element[J].Mol Genet Genomics,2001,266(1):56-63.
[42] Würleitner E,Pera L,Wacenovsky C,et al.Transcriptional regulation of xyn2 in Hypocrea jecorina[J].Eukaryot Cell,2003,2(1):150-158.
[43] Muro-Pastor M I,Gonzalez R,Strauss J,et al.The GATA factor AreA is essential for chromatin remodelling in a eukaryotic bidirectional promoter[J].EMBO J,1999,18(6):1584-1597.
[44] Lockington R A,Rodbourn L,Barnett S,et al.Regulation by carbon and nitrogen sources of a family of cellulases in Aspergillus nidulans[J].Fungal Genet Biol,2002,37(2):190-196.
[45] Coradetti S T,Craig J P,Xiong Y,et al.Conserved and essential transcription factors for cellulase gene expression in ascomycete fungi[J].PNAS,2012,109(19):7397-7402.
[46] Miller J,McLachlanA D,KlugA.Repetitivezinc-binding domains in the protein transcription factor IIIA from Xenopus oocytes[J].EMBO J,1985,4(6):1609-1614.
[47] Lee M S,Cavanagh J,Wright P E.Complete assignment of the1H NMR spectrum of a synthetic zinc finger from Xfin:sequential resonance assignments and secondary structure[J].FEBS Lett,1989,254(1/2):159-164.
[48] Todd R B,Andrianopoulos A.Evolution of a fungal regulatory gene family:the Zn(II)2Cys6 binuclear cluster DNA binding motif[J].Fungal Genet Biol,1997,21(3):388-405.
[49] Li S,Du J,Sun J,Galazka J M,et al.Overcoming glucose repression in mixed sugar fermentation by co-expressing a cellobiose transporter and a β-glucosidase in Saccharomyces cerevisiae[J].Mol Biosyst,2010,6(11):2129-2132.
[50] Ha S J,Galazka J M,Kim S R,et al.Engineered Saccharomyces cerevisiae capable of simultaneous cellobiose and xylose fermentation[J].PNAS,2011,108(2):504-509.
[51] Ha S J,Wei Q,Kim S R,et al.Cofermentation of cellobiose and galactose by an engineered Saccharomyces cerevisiae strain[J].Appl Environ Microbiol,2011,77(16):5822-5825.
[52] Eriksen D T,Hsieh P C,Lynn P,et al.Directed evolution of a cellobiose utilization pathway in Saccharomyces cerevisiae by simultaneously engineering multiple proteins[J].Microb Cell Fact,2013,12(1):61.
[53] Chang J J,Ho F J,Ho C Y,et al.Assembling a cellulase cocktail and a cellodextrin transporter into a yeast host for CBP ethanol production[J].Biotechnol Biofuels,2013,6(1):19.
[54] Mizuno R,Ichinose H,Maehara T,et al.Properties of ethanol fermentation by Flammulina velutipes[J].BiosciBiotechnol Biochem,2009,73(10):2240-2245.
[55] Zha J,Li B Z,Shen M H,et al.Optimization of CDT-1 and XYL1 expression for balanced co-production of ethanol and xylitol from cellobiose and xylose by engineered Saccharomyces cerevisiae[J].PLoS One,2013,8(7):e68317.
[56] Du J,Li S,Zhao H.Discovery and characterization of novel dxylose-specific transporters from Neurospora crassa and Pichia stipitis[J].Mol Biosyst,2010,6(11):2150-2156.
[57] Verho R,Penttilä M,Richard P.Cloning of two genes(LAT1,2)encoding specific L-arabinose transporters of the L-arabinose fermenting yeast Ambrosiozyma monospora[J].Appl Biochem Biotechnol,2011,164(5):604-611.
[58] Reddy V S,Saier M H Jr.BioV suite:a collection of programs for the study of transport protein evolution[J].FEBS J,2012,279(11):2036-2046.
[59] Slayman C L,Slayman C W.Depolarization of the plasma membrane of Neurospora during active transport of glucose:evidence for a proton-dependent cotransport system[J].PNAS,1974,71(5):1935-1939.
[60] Zhao X,Gao P,Wang Z.The production and properties of a new xylose reductase from fungus Neurospora crassa[J].Appl Biochem Biotechnol,1998,70-72:405-414.
[61] Zhang Z,Qu Y,Zhang X,et al.Effects of oxygen limitation on xylose fermentation,intracellular metabolites,and key enzymes of Neurospora crassa AS3.1602[J].Appl Biochem Biotechnol,2008,145(1/2/3):39-51.
[62] XirosC,TopakasE,Katapodis P,etal.Hydrolysis and fermentation of brewer's spent grain by Neurospora crassa[J].Bioresour Technol,2008,99(1/2/3):5427-5435.
[63] Okonko I O,Adejoye O D,Ogun A A,et al.Hazards analysis critical control points(HACCP)and microbiology qualities of seafoods as affected by handler's hygience in Ibadan and Lagos,Nigeria[J].Afr J Food Sci,2009,3(2):35-50.
[64] Wu W,Hildebrand A,KasugaT,etal.Directcellobiose production from cellulose using sextuple beta-glucosidase gene deletion Neurospora crassa mutants[J].Enzyme Microb Technol,2013,52(3):184-189.

