壳聚糖与DNA相互作用的单分子方法研究
张旭,王艳伟,杨光参
(温州大学物理与电子信息工程学院,浙江 温州 325035)
DNA凝聚是将一根或几根自由伸长的DNA凝聚成紧凑、有规律形态的过程,是基因治疗的重要组成部分[1]。因聚合物的尺寸及分散性对基因治疗效果有很大的影响,所以凝聚剂的选择尤为重要[2-4]。壳聚糖是一种天然的高分子,具有优良的生物相容性、生物可降解性、低毒以及高正电荷的性质,所带的阳离子可以与DNA上的阴离子有效结合,并且保护其免受核酸酶的降解,以保障DNA所受的破坏最小,同时装载DNA的壳聚糖微粒性质比较稳定[1,5-13]。这些特点使得壳聚糖被广泛应用在生物医学的很多领域中,如制药、组织工程和基因传递等[11-15]。
目前对壳聚糖的研究很多,Stranad等[16]通过实验证明了壳聚糖凝聚DNA的能力与乙酰化单位含量及聚合度紧密相关,即环状与棒状聚合物的比,随着乙酰化单位含量的增加而降低,同时证明了壳聚糖的电荷密度是决定聚合物形态的重要因素。Liu等[17]排除乙酰基对壳聚糖与DNA相互作用的影响,用CD光谱、静电荧光和原子力显微镜(AFM)等方法研究在不同壳聚糖与DNA电荷比k下,壳聚糖-DNA聚合物的形态学特征,并验证了其稳定性与pH值紧密相关。Cao等[18]进一步验证了上述结论,即pH值为5时聚合物是稳定存在的,当pH值上升到9时,凝聚态的DNA被释放出来。Strand等[16]也通过研究证实了壳聚糖的电荷密度及每条链上的电荷数是聚合物形态的决定因素,同时也验证了当pH值大于7.4时聚合物的稳定度下降,这就为基因治疗中生理pH值附近的聚合物稳定度降低提供了理论依据。
单分子磁镊(magnetic tweezers,MT)、AFM等技术的迅速发展使我们能够深入地研究生物大分子的一些特性,实时观测单分子的运动轨迹,通过对这些分子机械性能的研究,使我们了解它们在细胞内的作用机制。因此,单分子技术对了解DNA凝聚及分子性质有很大的帮助。应用问题可以在单根DNA分子上施加一个恒定的力,同时测量它的长度随时间的变化,进而对其作用过程得以深入地了解[19-21]。本文我们采用AFM及MT技术,系统地研究了壳聚糖作用下的DNA凝聚。用AFM技术对聚合物形态及尺寸随电荷比k的变化规律进行统计,然后应用MT技术观察凝聚力及拉伸步距随k值的变化规律,并进一步研究其相互作用机制。
1 实验材料与方法
1.1 材料
实验选用的噬菌体λ-DNA及bpr322-DNA购自New England Biolabs公司,原始浓度是500 ng/μL,使用前无需提纯。纯水(电阻率为18.2 MΩ·cm)经密理博超纯化系统去离子,紫外灭菌处理。壳聚糖、MgCl2及配置壳聚糖溶液所使用的NH4AC及乙酸均购自美国Sigma公司。壳聚糖用分1%乙酸溶液稀释至1 mg/mL作为母液,后期用150mmol/L NH4AC(pH=7.4)稀释到不同浓度。
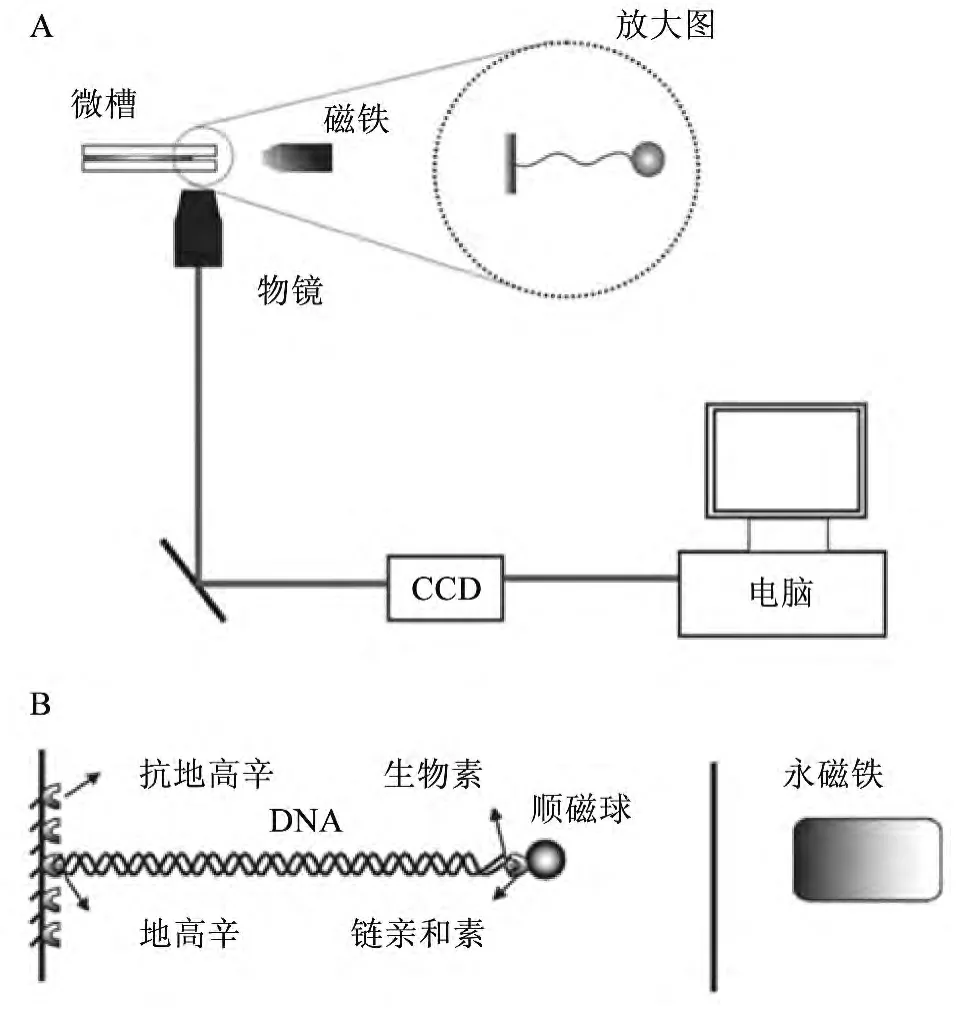
图1 横向单分子MT装置Fig.1 Horizontal single molecule MT device
1.2 MT实验装置及方法
本实验采用横向单分子MT装置(图1A),首先将0.17 mm厚的盖玻片一侧磨光,然后夹到两个载玻片中间,四周用玻璃胶密封,形成一个流动的密封槽,两边接两个玻璃微管,用于导入及导出缓冲液与药品等。样品槽放到倒置显微镜的40倍物镜上面观察,用三维操纵仪控制顺磁铁对焦平面上的DNA施加力。事先磨光的盖玻片的侧壁涂有抗地高辛与DNA一端的地高辛连接。当DNA与磁球的混合液冲入样品槽后,就会形成侧壁-DNA-顺磁球的结构(图1B)。导入试剂的样品槽在室温下垂直静止放置半小时,放置过程中应确保磨光的盖玻片表面向上,确保DNA分子连接到磨光的侧壁上。然后用缓冲液冲洗,去除多余的未连接在侧壁上的DNA分子,确保实验过程视野清晰。用显微镜找到连接好的DNA分子,观察并录像。通过测量磁球与盖玻片侧壁之间的距离得到DNA的长度,通过磁球在垂直于DNA伸长方向上的布朗运动得到DNA所受的拉力[20,22-23]。其磁力可表示为。

其中KB为玻尔兹曼常量,T为热力学温度,δx为磁球布朗运动x方向的均方差。
通过分析软件分析力与长度的关系,对比已知数据来确定是否为单根DNA。根据不同的k值计算出壳聚糖浓度,用1 mg/mL的壳聚糖作为母液,并用150mmol/L NH4AC(pH=7.4)稀释到所需要的浓度并注入到样品槽里面,培养15 min后,观察不同k值下DNA的长度随时间的变化关系。
1.3 AFM实验装置及方法
实验装置为日本京都岛津公司生产的AFM,型号为SPM-9600。AFM在空气中扫描图像采用轻敲模式获得。扫描速度为3 Hz,均方振幅是2 V,驱动频率约350 kHz,硅探针具有42 N/m的固定弹性系数,图片像素512×512。首先按不同的k值计算出100 μL溶液所需的DNA的量(实验中所使用的DNA的浓度均为原始浓度)。取出DNA后放入离心管中,按计算所得加入壳聚糖溶液后,用NH4AC溶液稀释,最终保证DNA的浓度恒定为4 ng/μL备用。实验时,用移液器取出15 μL的DNA-壳聚糖混合液滴到新鲜剥离的云母片表面上,静置2~3 min,然后用20 μL的Milli-Q超纯水冲洗云母片,大约冲洗10~15次,最后用氮气吹干样品,将样品在干燥器中保存2 h以上待用。
2 结果与讨论
应用MT技术研究不同k值下DNA在壳聚糖作用下的凝聚过程及凝聚力的变化,并得出其变化的规律。应用AFM技术观察不同种类的DNA在相同k值下聚合物的形态学上的区别,同时观察同种DNA在不同k值下的形态学的变化规律并与MT结果相对照,进而深入地了解DNA在壳聚糖作用下的凝聚过程。
2.1 用MT研究λ-DNA的力谱变化
壳聚糖是目前常用的基因治疗的载体,我们利用MT技术研究不同浓度的壳聚糖作用下的λ-DNA的力谱曲线变化。在恒定外力作用下,没有加入任何药品时λ-DNA的长度是不随时间变化的,即DNA的长度在恒力作用下保持不变,只有本身布朗运动引起的微小长度变化。而在溶液中加入k=2的相应浓度的壳聚糖后,我们会观测到λ-DNA的凝聚过程。因凝聚过程中控制外力难以恒定,而拉伸过程中可有效地克服此缺点,所以我们选取拉伸曲线为研究对象。控制外力为5.9 pN,观测λ-DNA的拉伸过程(图2),从侧面证明了在壳聚糖的作用下DNA发生了折叠及凝聚。另外,壳聚糖浓度较小且施加在DNA上的外力很大时,也会发现λ-DNA的长度有所收缩,证明壳聚糖对λ-DNA的作用比较强。
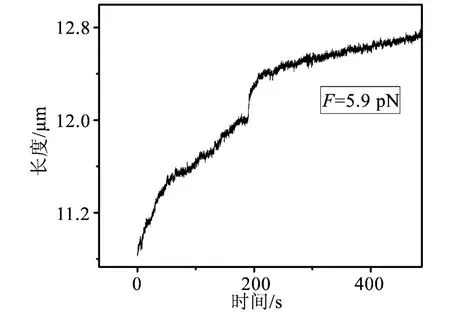
图2 k=2时,加入壳聚糖后DNA的长度与时间关系曲线Fig.2 Extension-time curve ofthe DNA corresponding to k=2 after chitosan adding
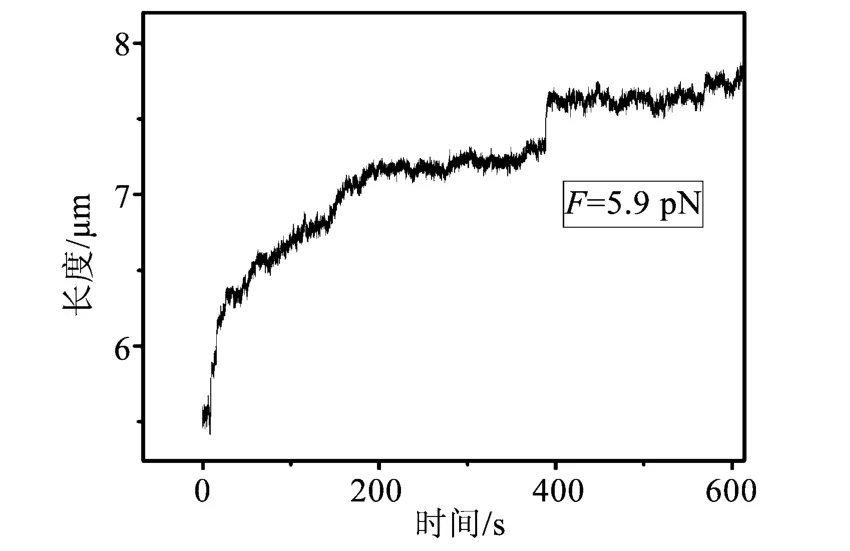
图3 k=4时,加入壳聚糖后DNA的长度与时间关系曲线Fig.3 Extension-time curve ofthe DNA corresponding to k=4 after chitosan adding
图3给出了k=4时壳聚糖-DNA聚合物在5.9 pN的恒力作用下的拉伸曲线,与图2比较可知,当k值增大时拉伸曲线中大跳跃步距明显增多,且初始阶段的长度变化趋势明显加快。这可能是因为随着k值的增加,DNA凝聚得更加紧凑且凝聚的节点增多,因而被拉伸的机会也相应地增加。而当k=2或k=4时均发现有500 nm左右的大步距存在,这可能是因为DNA在凝聚过程中形成的几个环状结构同时被打开造成的。
2.2 用AFM研究壳聚糖与不同类型DNA的作用
配制不同浓度的壳聚糖溶液,按不同的k值与环状pbr322-DNA混合,制成AFM样品进行观察,发现聚合物的形态随着k值的变化而明显变化,图4是不同k值下,壳聚糖与环状pbr322-DNA分子相互作用的AFM图像。图4A为没有加入任何药品的环状pbr322-DNA分子的AFM图像,当没有与药物分子相互作用时,DNA成圆环状结构。图4B~D分别对应k值为2、5、6时,加入壳聚糖后的DNA图像,从中可发现DNA发生凝聚,而且有环状、棒状及球状结构出现。当k值不断增加时,球状和棒状结构的比值不断减小,且凝聚物的尺寸也逐渐变小。由统计结果可知在k值由2增加到6时,球状与棒状聚合物的比由2减小到0.75。此部分结果与前人实验所得结果基本一致[24]。
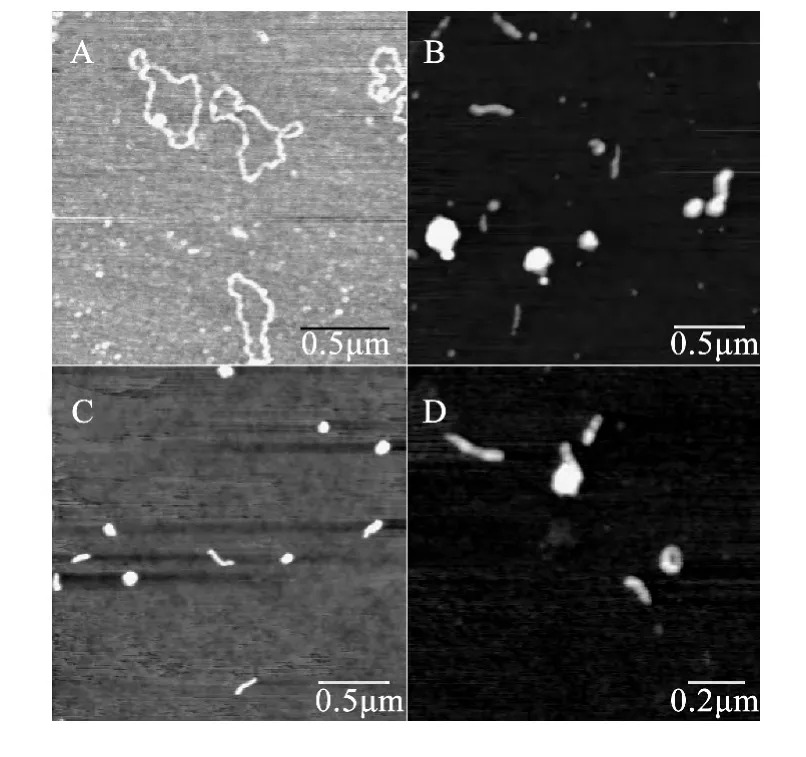
图4 环状pbr322-DNA分子的AFM图像及不同k值下的壳聚糖与环状pbr322-DNA分子相互作用的AFM图像。Fig.4 AFM imageofcirclepbr322-DNA molecules and AFM images of the interaction between circle pbr322-DNA and chitosan

图5 线状λ-DNA分子以及不同k值下的壳聚糖与线状λ-DNA分子相互作用的AFM图像Fig.5 AFM image of linear λ-DNA molecules and AFM images of the interaction between linear λ-DNA and chitosan
为了深入地研究DNA与壳聚糖的相互作用的过程,我们变换DNA的种类,研究壳聚糖与线状λ-DNA分子的相互作用。图5 A是裸λ-DNA的AFM图像,DNA处于自由伸展状态。向λ-DNA溶液里面加入壳聚糖,当k值=0.5时,线状λ-DNA分子明显聚集并出现凝聚的棒状及球状结构,当k值继续增大时,λ-DNA聚集得更加紧凑,旁边的松散结构全部消失并出现了环状聚合物,在k值增大的过程中聚合物的尺寸也在逐渐地减小(如图5 B ~D k值分别为0.5、2、4。).
通过比较图6与图7,我们发现对于不同DNA凝聚物的球状与棒状结构比随k值的变化规律基本是相同的,即都是随着k值的增加而降低。同时发现相同的k值下,使用pbr322-DNA比使用λ-phage DNA形成的粒子分散性好,且形成的粒子的尺寸基本相同。在MT实验中我们选用线性λ-phage DNA代替pbr322进行实验,以便于实时地观测壳聚糖与DNA的作用过程,对其相互作用有较为深入地了解。
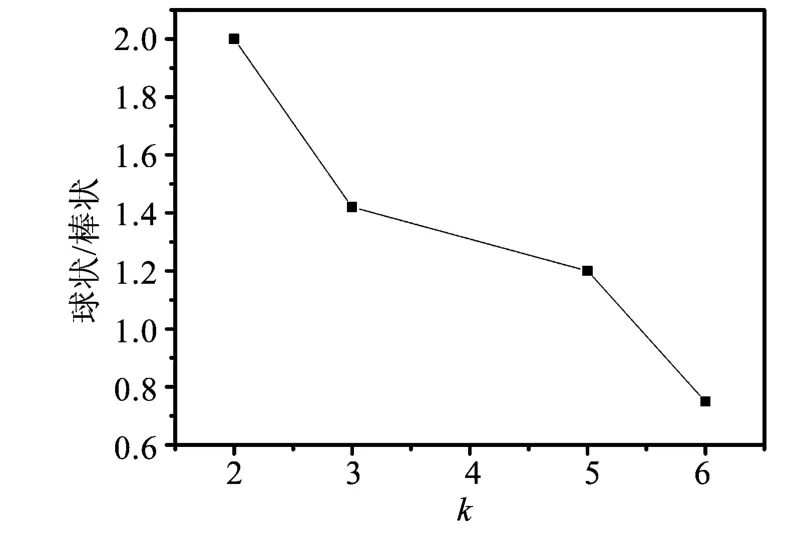
图6 pbr322-DNA-壳聚糖聚合物的球状/棒状比值随着k值变化的统计图Fig.6 Illustration of the variety of torod/rad ratio of pbr322-DNA-chitosan polymer with k
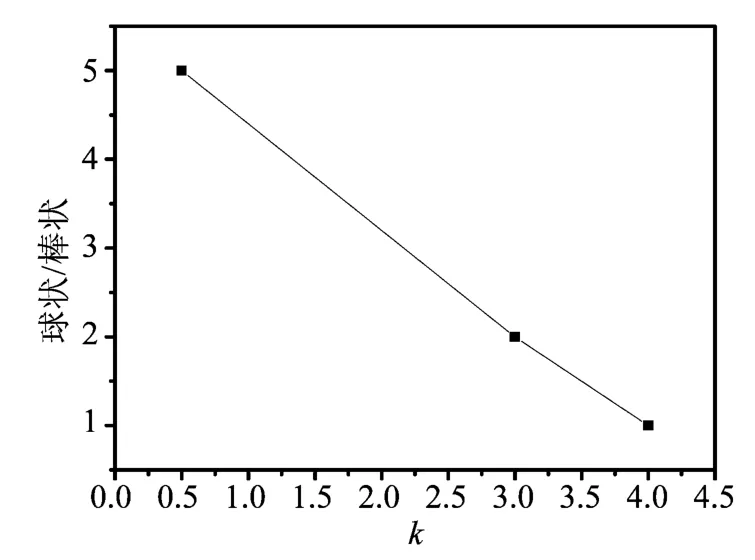
图7 λ-phage DNA-壳聚糖聚合物的球状/棒状比值随着k值变化的统计图Fig.7 Illustration of the variety of spherical/rad ratio of λphage DNA-chitosan polymer with k
3 壳聚糖与DNA作用的模型简图
图8将DNA在壳聚糖作用下所形成聚合物的形态以直观的方式表示出来,即DNA在壳聚糖的作用下所形成的聚合物的形态主要以环状、棒状和球状为主,同时也会有一些不规则形态的聚合物存在。而且随着k值的不同,各形态粒子之间的比例明显的不同。原因可能是壳聚糖通过与DNA的碱基对结合,使DNA折叠和凝聚。

图8 DNA与壳聚糖作用的模型图Fig.8 Model image of the interaction between λ-DNA and chitosan
4 结论
本文应用AFM及MT技术研究了壳聚糖与不同种类的DNA的相互作用过程,并对电荷比k取不同值时,DNA的凝聚过程及所形成聚合物的形态进行了统计。DNA与壳聚糖相互作用所形成聚合物的形态由初始时的不规则块状慢慢凝聚成具有规则形态的聚合物,其中包括球状、环状及棒状,且由统计结果可知,球状或环状与棒状聚合物的数量之比随着k值的增加而不断地降低。同时,聚合物的尺寸也随着k值的增加逐渐地减小。通过MT实验可知,随着k值的不断增加,DNA的凝聚过程所需的时间越来越短,即长度随时间的变化越来越明显。
[1]BLOOMFIELD V A.DNA condensation[J].Current opinion in structural biology,1996,6(3):334 -341.
[2]PELTA J Jr,DURAND D,DOUCET J,et al.DNA mesophases induced by spermidine:Structural properties and biological implications[J].Biophysical journal,1996,71(1):48 -63.
[3]RASPAUD E,DURAND D,LIVOLANT F.Interhelical spacing in liquid crystalline spermine and spermidine-DNA precipitates[J].Biophysical journal,2005,88(1):392 -403.
[4]HUD N V,VILFAN I D.Toroidal DNA condensates:Unraveling the fine structure and the role of nucleation in determining size[J].Annual Review of Biophysics and Biomolecular Structure,2005,34:295-318.
[5]LANKALAPALLI S,KOLAPALLI V.Polyelectrolyte complexes:A review of their applicability in drug delivery technology[J].Indian journal of pharmaceutical sciences,2009,71(5):481 -487.
[6]NARAMBUENA C F,LEIVA E P M,CHÁVEZ-PÁEZ M,et al.Effect of chain stiffness on the morphology of polyelectrolyte complexes.A Monte Carlo simulation study[J].Polymer,2010,51(14):3293 -3302.
[7]ALATORRE-MEDA M,TABOADA P,SABÍN J,et al.DNA-chitosan complexation:A dynamic light scattering study[J].Colloids and Surfaces A:Physicochemical and Engineering Aspects,2009,339(1/2/3):145-152.
[8]IVANOV V A,MARTEMYANOVA J A,MÜLLER M,et al.Conformational changes of a single semiflexible macromolecule near an adsorbing surface:A Monte Carlo simulation[J].The Journal of Physical Chemistry B,2009,113(12):3653 -3668.
[9]CUI Z,MUMPER R J.Chitosan-based nanoparticles for topical genetic immunization[J].Journal of controlled release,2001,75(3):409-419
[10]ILLUM L,JABBAL-GILL I,HINCHCLIFFE M,et al.Chitosan as a novel nasal delivery system for vaccines[J].Advanced drug delivery reviews,2001,51(1/2/3):81 -96
[11]SALAS T,MARTÍNEZ B P,GODÍNEZ R R T,et al.Evaluation of humoral and cellular immune response in amurine model using vesicles outer membrane of Brucella ovis in chitosan nanoparticles[J].Asian Chitin Journal,2008,4:59-66.
[12]JAYAKUMAR R,NWE N,TOKURA S,et al.Sulfated chitin and chitosan as novel biomaterials[J].International Journal of Biological Macromolecules,2007,40(3):175 -181.
[13]KAI E,OCHIYA T.A method for oral DNA delivery with N-acetylated chitosan[J].Pharmaceutical research,2004,21(5):838-843
[14]PURAS G,ZARATE J,ACEVES M,et al.Low molecular weight oligochitosans for non-viral retinal gene therapy[J].European Journal of Pharmaceutics and Biopharmaceutics,2013,83(2):131-140.
[15]BLOOMFIELD V A.DNA condensation by multivalent cations[J].Biopolymers,1997,44(3):269 -282.
[16]STRAND S P,DANIELSEN S,CHRISTENSEN B E,et al.Influence of chitosan structure on the formation and stability of DNA-chitosan polyelectrolyte complexes[J].Biomacromolecules,2005,6(6):3357 -3366.
[17]LIU W G,SUN S J,CAO Z Q,et al.An investigation on the physicochemical properties of chitosan/DNA polyelectrolyte complexes[J].Biomaterials,2005,26(15):2705 -2711
[18]CAO W D,EASLEY C J,FERRANCE J P,et al.Chitosan as a polymer for pH-induced DNA capture in a totally aqueous system[J].Analytical chemistry,2006,78(20):7222-7228.
[19]SUN B,WEI K J,ZHANG B,et al.Impediment of E.coli UvrD by DNA-destabilizing force reveals a strained-inchworm mechanism of DNA unwinding[J].The EMBO Journal,2008,27(24):3279 -3287.
[20] RAN SY,WANG Y W,YANG G C,etal. Morphologycharacterizationandsingle-moleculestudyofDNA-dodecyltrimethylammonium bromide complex[J].The Journal of Physical Chemistry B,2011,115(16):4568 -4575.
[21]RICHARDSON S W,KOLBE H J,DUNCAN R.Potential of low molecular mass chitosan as a DNA delivery system:Biocompatibility,body distribution and ability to complex and protect DNA[J].International journal of pharmaceutics,1999,178(2):231-243.
[22]WANG Y W,RAN S Y,MAN B Y,et al.Ethanol induces condensation of single DNA molecules[J].Soft Matter,2011,7(9):4425-4434.
[23]STRICK T R,ALLEM J-F,BENSIMON D,et al.Behavior of supercoiled DNA[J].Biophysical journal,1998,74(4):2016 -2028.
[24]DANIELSEN S,VÅRUM K M,STOKKE B T.Structural analysis of chitosan mediated DNA condensation by AFM:Influence of chitosan molecular parameters[J].Biomacromolecules,2004,5(3):928 -936.

