利用正交设计优化羊食道口线虫SRAP-PCR反应体系
李 芬,胡 涛,谢汝婷,刘 伟,程天印,李 娜
(1.湖南农业大学动物医学院 ,长沙 410128;2. 湖南农业大学园艺园林学院,长沙 410128)
·简报·
利用正交设计优化羊食道口线虫SRAP-PCR反应体系
李 芬1,胡 涛1,谢汝婷1,刘 伟1,程天印1,李 娜2
(1.湖南农业大学动物医学院 ,长沙 410128;2. 湖南农业大学园艺园林学院,长沙 410128)
本研究利用正交设计L16(4)对羊食道口线虫SRAP-PCR反应体系的4因素:Taq酶、Mg2+、dNTPs、引物进行摸索。结果表明:各因素的不同水平对SRAP-PCR反应结果都有显著的影响,正交设计组合4中扩增的条带最清晰;羊食道口线虫SRAP-PCR最佳反应条件:最佳引物给合为Me1/Em7,最佳反应体系为25 μL:2.5 μL 10×PCR buffer、20 ng模板DNA、Mg2+(25 mmol/L)3 μL、dNTPs (2.5 mmol/L/μL)3 μL、引物(10 pmol/μL)4μL、rTaq DNA 聚合酶(5U/μL)0.2μL、灭菌ddH20补足。本研究结果为进一步采用SRAP技术来研究食道口线虫遗传进化奠定了坚实基础。
羊;食道口线虫;SRAP;正交设计
食道口线虫病是由圆形目(Strongyloidea)食道口属(Oesophagostomum)的食道口线虫寄生于家畜的大肠中所引起的,因在大肠壁上形成淡黄色的结节又称结节虫病[1]。引起羊食道口线虫病的食道口线虫有哥伦比亚食道口线虫、微管食道口线虫、粗纹食道口线虫和甘肃食道口线虫。传统寄生虫分类依靠寄生虫的形态、生活史、寄生的宿主等特征进行描述,但是对形态相似的虫种、株的鉴定很困难,且寄生虫虫株的遗传进化分析依靠传统的方法很难解决。分子生物学技术的发展,为食道口线虫虫株之间或者与其他寄生虫之间的鉴定提供了新思路。有报道采用PCR-SSCP、PCR-RFLP方法扩增ITS序列对食道口线虫进行分子遗传标记[1,2]。但是,PCR-RFLP方法建立在已知物种序列的基础上,对未知物种序列的检测效果不佳;PCR-SSCP方法对150~300 bp的核酸序列检出率最高,而对序列是否有突变还需要经过测序检测。
SRAP (sequence-related amplified polymorphism,即相关序列扩增多态性)是由Li和Quiros[3]创建的一种基于PCR的新型DNA分子标记技术,通过独特的引物扩增基因组内的开放阅读框(open reading frames,ORFs),SRAP标记的正、反引物分别与基因外显子里GC 含量、启动子和内含子里AT 含量丰富的区域配对而使基因呈现多态性[4-9]。此技术具有操作简便迅速、成本低、重复性高、高稳定性等特点,适合进行基因定位、克隆、遗传图谱构建及遗传多样性分析等研究,目前SRAP技术已被用于荷花等多种植物[8,9]、草鱼[10]、异尖线虫[11]、肝片吸虫[12,13]、日本血吸虫[14]等遗传图谱的构建和遗传多样性分析。作为遗传标记SRAP在寄生虫研究中应用很少,主要见于吸虫的报道[12-14],是否适用于线虫的研究报道仅见会议摘要[11]。在SRAP中PCR的反应体系很重要,它受很多因素的影响,为此何正文等[15]提出了采用正交设计来进行PCR反应体系的优化。本文通过正交实验设计,针对rTaq酶、Mg2+、dNTPs、引物这4个PCR反应因素进行SRAP-PCR反应体系优化,来研究SRAP技术是否适合于食道口线虫遗传标记。
1 材料与方法
1.1 虫体样品的采集及处理食道口线虫采样于屠宰场屠宰的1头山羊结肠内。生理盐水洗净后,逐级过30%、50%、70%的酒精,最后于70%的酒精4℃保存。
1.2 主要试剂及仪器WizardTMDNA Clean-Up System为Promega公司产品;ExTaq酶、PCR试剂、DNA Marker DL2000为TaKaRa宝生物工程(大连)有限公司产品;蛋白酶K 为天根公司产品; Dolophin-Doe凝胶成像系统;Gene Amp PCR System 9700型PCR仪;DYY—Ⅲ型电泳仪。
1.3 虫体DNA的提取取出70%酒精保存的虫体,用纯净水反复吹打冲洗6次后,置于1.5 mL Eppendorf管中,用灭菌且经紫外灯照射过的眼科剪刀将虫体组织剪碎,加入250 μL GT buffer反复研磨,再加入20 μL蛋白酶K,混匀后,55℃培养箱中16~18 h,其间摇动数次,使虫体组织充分裂解。将消化好的虫体悬液按WizardTMDNA Clean-Up System使用说明进行虫体DNA提取,将提取出的DNA样品浓度稀释至20 ng/μL,-20℃保存备用。
1.4 SRAP反应体系优化
1.4.1 引物 根据Li 等[3]报道,SRAP-PCR体系中引物有以下组合(见表1),引物由金思瑞生物科技有限公司合成。
1.4.2 正交体系设计表格 按照表2采用L16(4 )正交试验设计,制备总体积25 μL的PCR反应体系,除表中变化因素外,每个组合中还含有2.5 μL 10×PCR buffer和20 ng模板DNA,3次重复实验,取2 μL扩增产物用6%变性聚丙烯酰胺凝胶电泳检测。SRAP-PCR的扩增程序:94℃预变性5 min;94℃变性30 s,35℃ 退火1 min,72℃延伸60 s,5个循环;94℃变性30 s,50℃退火1min,72℃延伸60 s,35个循环;循环结束后,72℃延伸7 min,4℃保存备用。
2 结果与讨论
2.1 引物筛选结果按照扩增要求(条数清晰,带数多,多态性好),通过筛选引物选用Me1/Em7组合作为本次试验的引物。
2.2 正交实验扩增结果按照正交试验设计PCR扩增结果见图1。从图1中可见:体系1、4扩增的条带清晰,背景清晰;体系2、3、6、13扩增的背景浅,且条带无或者是只有1~2条,不可以采用;体系8、9、10、14、15扩增的条带多,背景深,但是扩增的条带没有体系1、4的清晰;体系11、12扩增的条带多,背景浅,也存在着条带不清晰的现象;体系16扩增的背景深,条带不清晰。
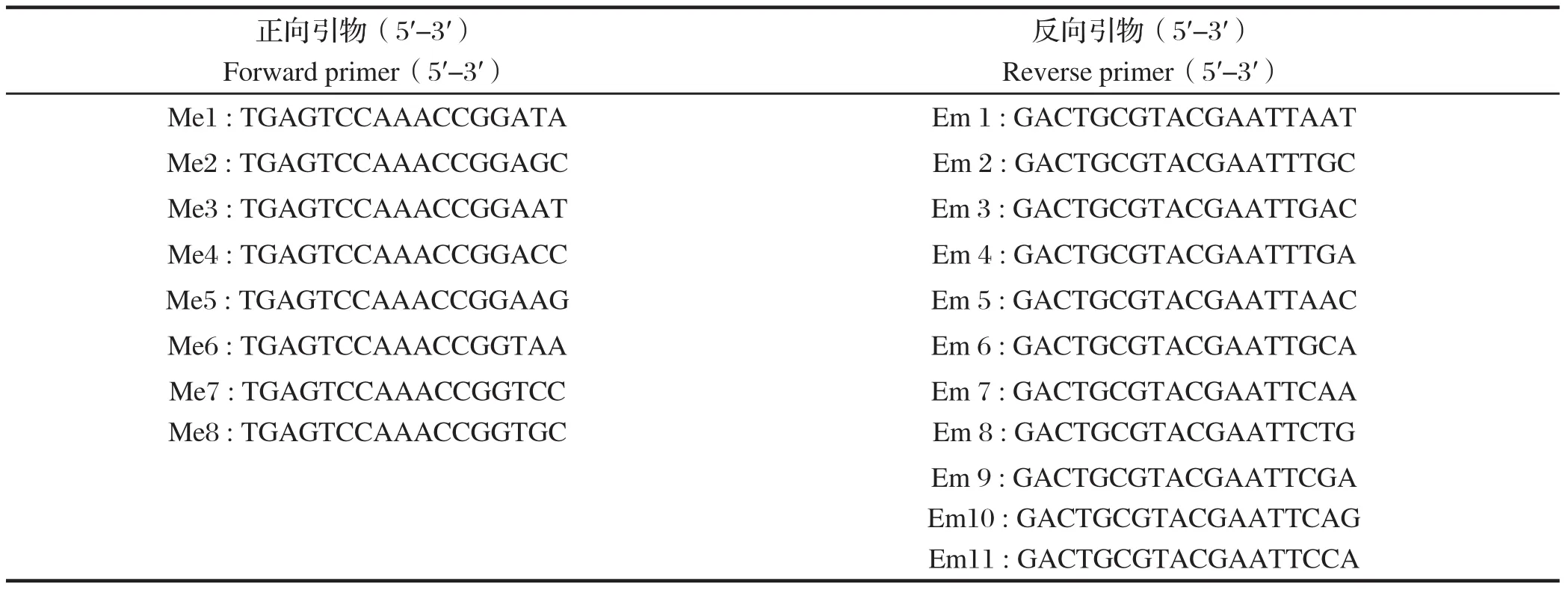
表1 SRAP引物Table 1 SRAP primers
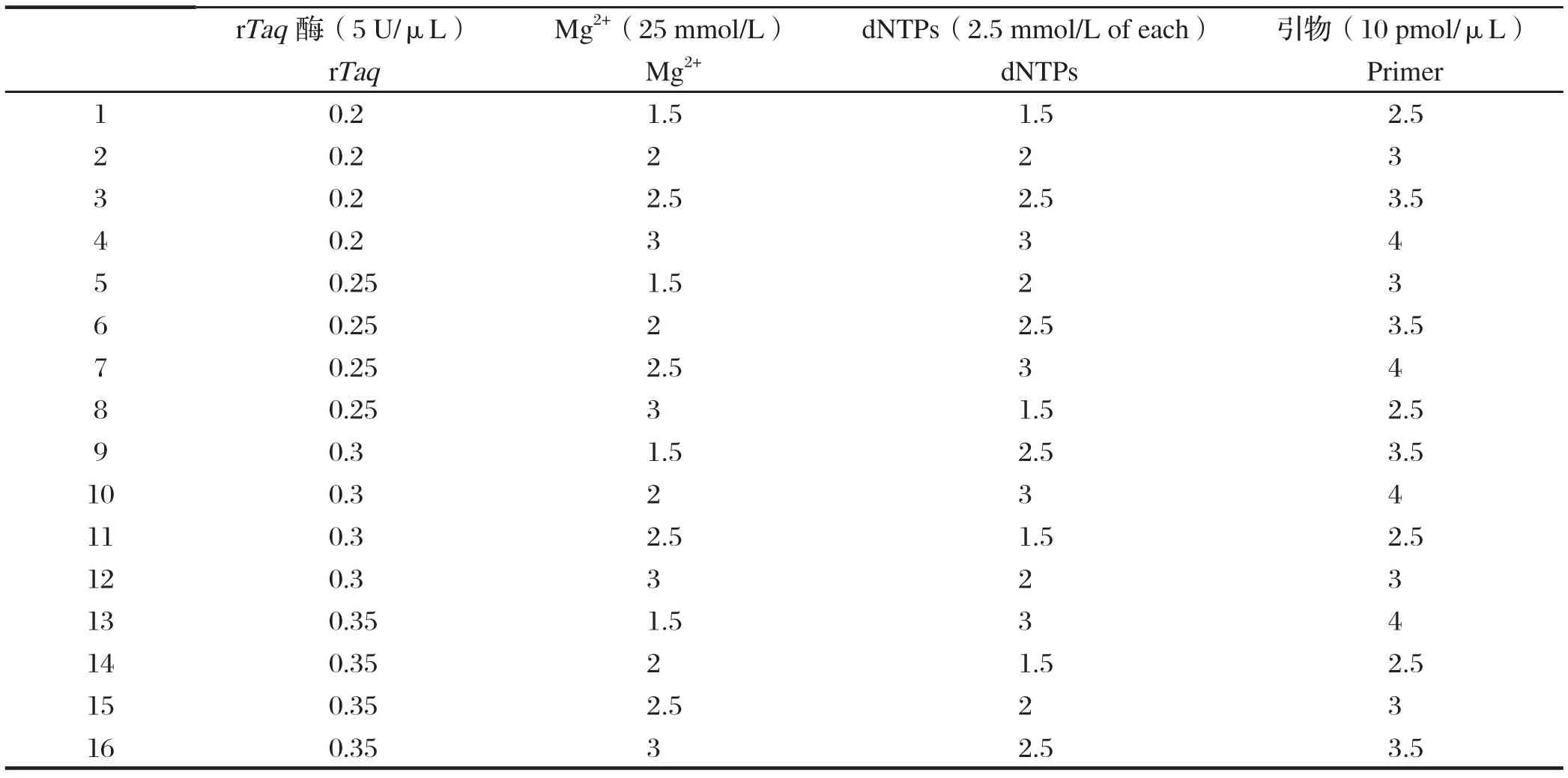
表2 SRAP-PCR正交实验设计反应体系L16(44)Table 2 SRAP-PCR orthogonal experimental design L16(44)
2.3 rTaq酶对PCR扩增的影响如图1所示,体系7与体系4比较,引物浓度与dNTPs浓度一样,rTaq酶浓度增加,Mg2+浓度减少,扩增的条带在减少且背景不清晰;体系8到体系11之间的变化也是一样的规律;体系5与1比较,两者Mg2+浓度一样,rTaq酶浓度与引物浓度增加,体系1的条带比体系5的清晰且明显;体系3与7的变化规律也是一样。说明酶的浓度影响了扩增基因条带的清晰度。
2.4 Mg2+对PCR扩增的影响图1显示,随着Mg2+浓度的变化,各体系扩增的结果不一,按照扩增条带清晰且背景清晰的选择条件,应当选择Mg2+浓度为3时的体系4与8组合,这两个体系组合能把250~1000 bp段之间的条带分开。Mg2+为3 μL时,体系4中的条带清晰且背景清晰。随着rTaq酶浓度与dNTPs浓度的增加,扩增的条带并没有增加而是有所减少,且背景变浅(体系8和12)。
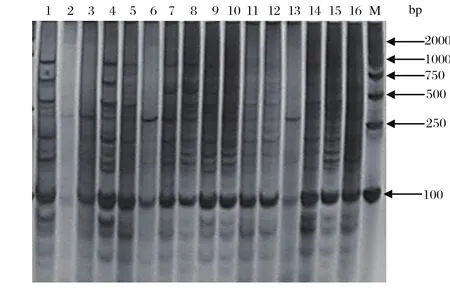
图1 正交组合电泳结果Fig.1 Electrophoresis results of orthogonal combinationM: DNA分子量标准(DL2000);1~16: 扩增体系组合1-16M: DNA Marker(DL2000);1-16: PCR amplifi cation system1-16
2.5 引物浓度对PCR扩增的影响如图1所示,扩增体系1~4 rTaq酶浓度一样,引物为4 μL(10 pmol/ μL)时扩增的条带数多且背景清晰,扩增体系5~8有相同的变化规律,说明引物浓度影响扩增结果。体系4与8比较,两者的Mg2+浓度一样,引物浓度减少,扩增的基因条带数存在1~2个差异,扩增的背景变得不是很清晰,且条带变的很小。图1中的体系10与14的变化与此相同,说明基因扩增的条带与引物浓度变化有关系。
通过正交试验筛选得到扩增食道口线虫的最终的SRAP-PCR反应条件:2.5 μL10×PCR buffer、20 ng模板DNA、Mg2+(25 mmol/L)3 μL、dNTPs(2.5 mmol/μL)3 μL、引物(10 pmol/μL)4 μL、rTaq DNA(5 U/μL)聚合酶0.2 μL,总体积25 μL。
通过正交设计SRAP-PCR反应体系,可以更全面的考虑各因素及其之间的交互作用,降低了试验成本,这也与其他报道相同[17]。SRAP技术在寄生线虫研究中的应用目前在国内还没有相关实验性文章报道。本研究将不同的因素和水平进行比较分析,确定了一个优化的反应体系,应用该体系在食道口线虫DNA研究中可获得清晰稳定的扩增结果。因此,SRAP标记技术有望在食道口线虫的遗传多头性的研究中得到广泛应用,并为其他寄生虫类的基因组分析奠定基础。
[1] 林瑞庆, 张媛, 朱兴全. 食道口线虫与食道口线虫病的研究进展[J]. 中国预防兽医学报, 2010, 32(9): 737-740.
[2] 胡冰. 粗纹食道口线虫和哥伦比亚食道口线虫遗传多样性和分子分类研究[D]. 杨凌: 西北农林科技大学, 2013.
[3] Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging iniBrassica[J]. Theor Appl Genet, 2001, 103: 455-103.
[4] Li G, Gao M, Yang B, et al. Gene for gene alignment between the Brassica and Arabidopsis genomes by direct transcriptome mapping[J]. Theor Appl Genet, 2003, 107: 168-107.
[5] 李巧燕, 林瑞庆, 朱兴全. SRAP分子标记及其应用概述.热带医学杂志[J]. 2006, 6(4): 467-469, 478.
[6] 任羽, 王得元, 张银东. 相关序列扩增多态性(SRAP):一种新的分子标记技术[J]. 中国农学通报, 2004, 20(6): 11-14.
[7] 柳李旺, 龚义勤, 黄浩, 等. 新型分子标记--SRAP与TRAP及其应用[J]. 遗传, 2004, (5): 777-781.
[8] Jehan T, Vashishtha A,Yadav S R, et al. Genetic diversity and genetic relationships in Hyacinthaceae in India usingRAPD and SRAP markers[J]. Physiol Mol Biol Plants, 2014, 20(1):103-114.
[9] Deng C L, Qin R Y, Gao J, et al. SRAP analysis of DNA base sequence changes in lotus mutants induced by Fe+ implantation[J]. Genet Mol Res, 2013, 12(1): 335-343.
[10] 张志伟, 韩曜平, 仲霞铭, 等. 草鱼野生群体和人工繁殖群体遗传结构的比较研究[J]. 中国水产科学, 2007, 9: 720-725.
[11] 李巧燕, 林瑞庆, 宋慧群, 等. SRAP技术应用于异尖线虫的初步研究[C] //中国畜牧兽医学会家畜寄生虫学分会第九次学术研讨会论文摘要集, 2006: 234.
[12] Alasaad S, Li Q Y, Lin R Q, et al. Genetic variability among Fasciola hepatica samples from different host species and geographical localities in Spain revealed by the novel SRAP marker[J]. Parasitol Res, 2008, 103(1): 181-186.
[13] Li Q Y, Dong S J, Zhang W Y, et al . Sequence-related amplified polymorphism, an effective molecular approach for studying genetic variation in Fasciola spp. of human and animal health significance[J]. Electrophoresis, 2009, 30(2): 403-409.
[14] Song H Q, Mo X H, Zhao G H, et al. Electrophoretic detection of genetic variability among Schistosoma japonicum isolates by sequence-related amplified polymorphism[J]. Electrophoresis, 2011, 32(11): 1364-1370.
[15] 何正文, 刘运生, 陈立华, 等. 正交设计直观分析法优化PCR条件[J]. 湖南医科大学学报, 1998, 23(4): 403-404.
[16] 张平湖, 刘冠明, 橄榄SRAP-PCR体系的建立和优化[J].中国农学通报, 2010, 26(15): 86-88.
OPTIMIZATION OF SRAP-PCR REACTION SYSTEM FOR OESOPHAGOSTOMUM OF GOAT USING ORTHOGONAL DESIGN
LI Fen1, HU Tao1, XIE Ru-ting1, LIU Wei1, CHENG Tian-yin1, LI Na2
( 1.College of Veterinary Medicine, Hunan Agricultural University, Changsha 410128, China; 2. College of Horticulture and Landscape, Hunan Agricultural University, Changsha 410128, China)
In order to know the genetic diversity of Oesophagostomum of goat using SRAP-PCR, four factors, including DNA polymerase, Mg2+, dNTPs and primers in SRAP-PCR system were optimized using orthogonal design. The result should 25 μL optimized SRAP-PCR system contains 2.5 μL 10×PCR buffer, 20 ng DNA template, 3 μL 25 mmol/L Mg2+, 3 μL 2.5 mmol/L dNTPs, 4 μL 10 pmol/μL primers, 0.2 μL rTaq DNA polymerase (5U/μL) and dd H2O. The results built a base for the study of genetic evolution of Oesophagostomum using SRAP technology.
Goat; Oesophagostomum; SRAP; orthogonal design
S852.731
B
1674-6422(2014)04-0067-05
2014-02-25
国家自然科学基金项目(31000901);湖南省教育厅科学研究优秀青年项目(141392)
李芬,女,博士研究生,讲师,主要从事分子寄生虫学研究
程天印,E-mail:hn5368@163.com;李娜,E-mail:1112lina@163.com

