基于16S rDNA和COⅠ基因序列探讨四种溞类的系统关系和分类地位
徐 敏 张海军 邓道贵 王文平 张晓莉 查岭生
(淮北师范大学生命科学学院, 资源植物生物学安徽省重点实验室, 淮北 235000)
基于16S rDNA和COⅠ基因序列探讨四种溞类的系统关系和分类地位
徐 敏 张海军 邓道贵 王文平 张晓莉 查岭生
(淮北师范大学生命科学学院, 资源植物生物学安徽省重点实验室, 淮北 235000)
为了检验已记录的4 个 溞属种类(隆 线 溞Daphnia carinata 、透明 溞D. hyalina 、蚤状 溞D. pulex和大型 溞D. magna)的系统分类, 用试剂盒法分别提取4 种 溞类的基因组DNA。利用特异性引物, 通过PCR扩增了4 种 溞属种类的16S rDNA和COⅠ基因部分序列, 并与来自GenBank中每个种类相似度较高的同种序列进行分析。结果表明, 基于16S rDNA和COⅠ基因, 4 个 溞属种类的平均种间相似度分别为85.56%和80.67%,碱基中A+T含量均明显高于G+C含量。基于COⅠ基因, 巢湖隆线 溞与来自GenBank 上的拟同形 溞(Daphnia similoides AB549199)相似度为99%, 分歧度为0.4%—0.9%。就16S rDNA和COⅠ基因而言, 巢湖透明 溞更接近于盔形 溞(Daphnia galeata)。基于16S rDNA和COⅠ基因构建的NJ树和贝叶斯树, 两者的主要分枝基本一致。结果暗示, 巢湖隆线 溞 与拟同形 溞为一个种, 透明 溞 应属于盔形 溞。由于缺乏核基因的研究, 有关巢湖 溞属的分类地位还需进行更深入的探讨。
溞属; 16S rRNA; COⅠ基因; 系统分类
溞属隶属节肢动物, 又称水蚤或 溞, 是枝角类中重要的类群。本属描述的种类超过 100种, 但仅有三十余种被准确地鉴定, 我国现存的 溞属种类有10余种[1,2]。 早期的 溞类分类研究主要依据其形态学特征。由于 溞属动物有十分丰富的变异类型, 属内近源种之间可以杂交, 因此 溞属种类可能存在种间杂种。例如, 盔型 溞(Daphnia galeata)、 僧帽 溞(D. cucullata)、 透明 溞(D. hyalina)、 玫瑰 溞(D. rosea)、长刺 溞(D. longispina)都 属于盔型 溞的种复合体, 可以形成种间杂交种[3,4]。而近年来我国在枝角类的分类方面研究尚不多见, 在 溞属种类的分类鉴定中存在界定模糊, 甚至出现鉴定错误的现象[2,5]。我国的《中国动物志·淡 水枝角类》中把同形 溞 归到隆线溞一类, 没有关于拟同形 溞的记载。此外, 只介绍了透明 溞的几种变异类型, 也没有对盔形 溞进行描述。据报道, 我国一些水体中的同形 溞可能一直被命名为隆线 溞或者鹦鹉 溞[5]。
随着分子生物学的发展, 分子标记被逐渐应用到物种的系统进化研究中。Schwenk等用线粒体16S rRNA基因对部分枝角类种类进行分子系统进化分析, 建立了比较直观的系统进化树[6]; Gelas和Meester用线粒体COⅠ 基因分析了欧洲大型 溞的不同的单倍型[7]; Briski等采用线粒体COⅠ基因和16S rRNA基因的分子标记方法, 利用船舶压载水所携带的休眠卵识别外来入侵的无脊椎动物[8]。近几年来, 我国学者在象鼻 溞属(Bosmina)[9]、盔形 溞(D. galeata)[10]、 秀体 溞属(Diaphanosoma)[11]、 薄皮 溞属(Leptodora)[12]、 圆形盘肠 溞(Chydorus sphaericus)[13]等枝角类的遗传多样性方面做了一些工作, 但对国内 溞类种间系统分化的研究尚未见报道。本研究通过对安徽已发现的大型 溞、隆线 溞、透明 溞、蚤状溞等四种 溞属种类的16S rDNA和COⅠ基因序列进行分析, 探讨这些 溞属种类的归属以及他们之间的分子系统关系, 初步解决一些传统分类上存在争议的分类问题, 以期为国内 溞属的分子系统学研究提供基础资料。
1 材料与方法
1.1 实验材料
研究用大型 溞于2007年3月用25号浮游生物网在淮河采集; 蚤状 溞 、隆线 溞 、透明 溞由2012年3月采集的巢湖沉积物中的休眠卵孵化而来, 并分离鉴定[2,14](表1)。 几种 溞类均在智能光照培养箱中用斜生栅藻培养, 2—3d更换一次培养液, 温度为20 ℃ ±1℃、光照时间L︰D=12h︰12h。实验时用吸管吸取单只个体饥饿一昼夜后用双蒸水冲洗, 离心管收集备用。
1.2 基因组DNA提取
本实验采用微量样品基因组 DNA试剂盒(TIANamp Micro DNA Kit)提 取 溞属DNA 。由于溞属种类属于小型甲壳动物, 外被甲壳, 在提取前用灭菌的牙签捣碎 溞体, 有利于蛋白酶K对组织进行消化。
1.3 PCR扩增及序列测定
用于扩增COⅠ基因引物:
COIF (5′-AYCAATCATAAGGACYATTGGRAC-3′)
COIR (5′-KGTGATWCCNACHGCTCAKAC-3′)
用于扩增16S rDNA基因引物[15]:
L2510(5′-CGCCTGTTTAACAAAAACAT-3′)
H3059(5′-CCGGTCTGAACTCAGATCATGT-3′)
PCR反应体系25 μL, 每个反应体系内含PCR Buffer 2.5 μL、dNTP 4.0 μL、Mg2+0.5 μL、模板DNA 1.0 μL、Taq酶0.25 μL、上下游引物各1.0 μL, 双蒸水补足到25 μL。
扩增条件为 95℃预变性 3min, 95℃变性 45s, 50℃退火 45s, 72℃延伸 45s, 共 35个循环, 72℃10min充分延伸, 4℃结束。
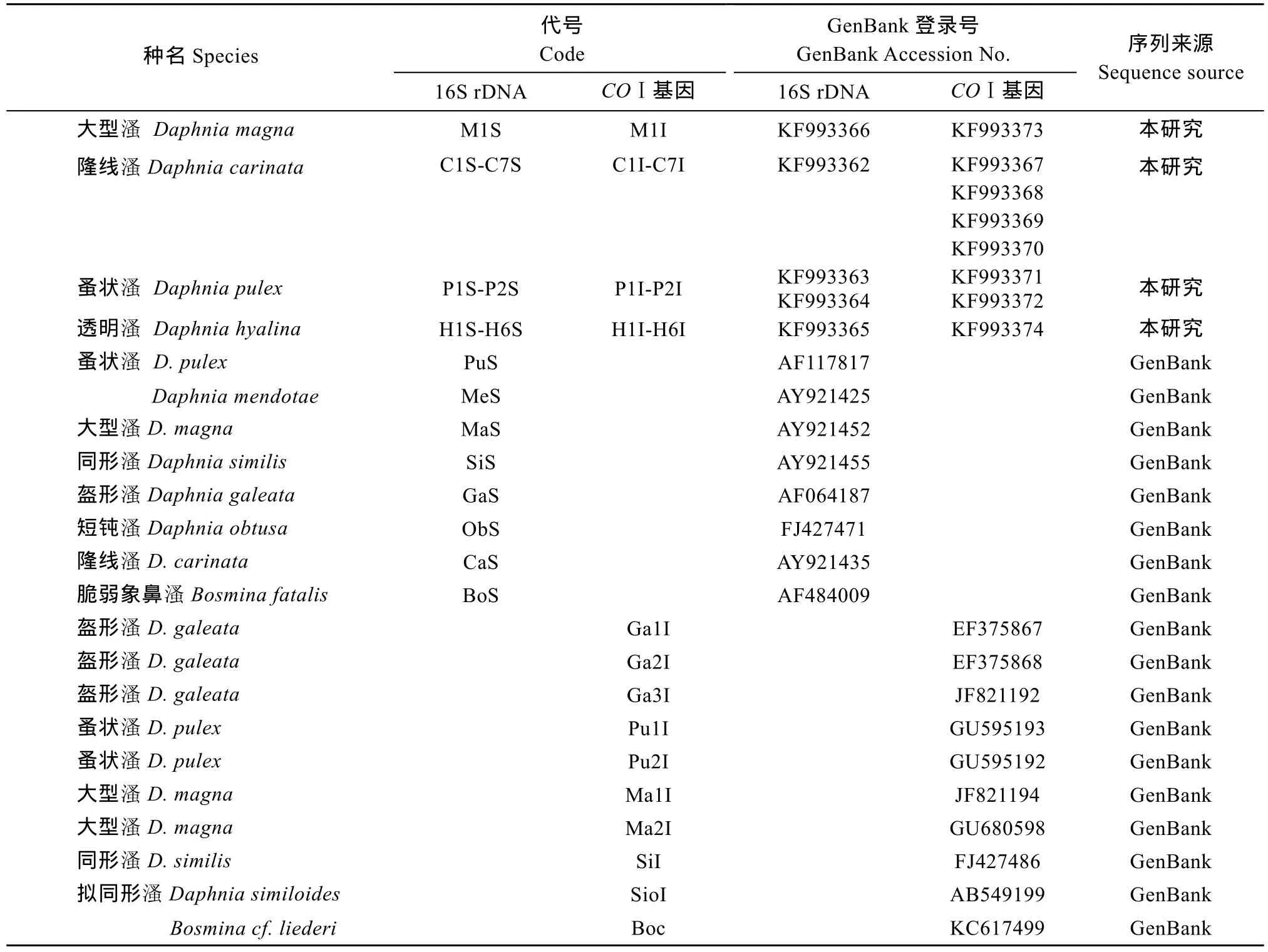
表1 本研究中样本的核苷酸序列的来源及编号Tab. 1 The origin and number of nucleotide sequence of samples in this study
扩增产物经电泳检测后, 选择目的带清晰且明亮的 PCR反应产物送交生工公司纯化, 并测序。其中16S rDNA采用双向测序, 正反向对比后确定分析用序列, 用DNASTAR中的seqnam比对后获得隆线溞539个碱基, 对比峰图修除两端不可靠的碱基, 经人工校正后5 溞个隆线 个体共获得有效碱基为510-19个(C1S510、C2S517、C3S517、C4S515、C5S519)。同样地, 溞蚤状 的有效碱基为 479-22个(P1S479、P2S522), 溞透明 为482-20个碱基(H1S513、H2S520、H3S520、H4S482、H5S520、H6S520), 大溞型 为519个有效碱基。来自5个休眠卵孵化的隆线溞的16S rDNA序列完全相同, 来自6个休眠卵孵化溞的透明 的16S rDNA序列也完全相同, 溞蚤状 的两个序列存在多态性, 分别用P1、P2构建进化树。在序列对比中, 溞除大型 只有1个个体外, 溞每种 取片段最长的个体进行比对, 分别用C5S、H2S、M1S、P1S、P2S进行分析(表1)。COⅠ基因采用单向测序,获得5个种类16个个体长度为499个碱基的COⅠ部分序列, 其中7 溞个隆线 个体中有4个单倍型, 透溞明 6个个体的COⅠ序列完全一致, 溞蚤状 2个个体有2个单倍型。由于物种内存在多态性, 溞隆线 、溞溞溞透明 、蚤状 、大型 分别取C1I、C2I、C5I、C7I、H1I、P1I、P2I和M1I参与比对(表 1)。通过NCBI中BLAST比对, 分别找出与本研究相似度最高的序列放在文中一起分析。参考已测出Daphnia pulex全序列[16](GenBank登录号AF117817)中16S rRNA基因定位剪出AF117817的16S rRNA基因进行比对, 其他用于分析的下载序列见表1。
1.4 系统发生分析
用DNASTAR对测序结果进行人工校正, 并去除两端冗余序列。使用BioEdit建立序列相似矩阵图,用Clustalx1.81软件多重序列比对, 用DNAspV5分析变异位点, 用Mega4.1计算不同序列间转换/颠换数、生成序列差异位点表、并计算遗传距离和构建NJ进化树。采用K2P双参模型计算两两遗传距离,系统树设置采用 P-distance模型, 两种分子标记分溞别以脆弱象鼻 和Bosmina cf. liederi为外群, Bootstrap Replications1000次; 用 Mrbayes3.2构建贝叶斯树, 替换模型设置参数nst=6 (GTR模型), 位点速率变异设置 invgamma, 其余设置为默认值; 采用mcmc法运算1000000代, 每100代取样一次, 在舍弃25%老化样本后, 用剩余样本构建一致树。
2 结果
2.1 基于16S rDNA的结果分析
C5S、H2S、M1S、P2S在DNASTAR中经过两两序列比对, 平均种间相似度为 85.56%, 碱基中A+T(66.2%)含量明显高于G+C(33.8%)含量。与来自GenBank上Daphnia的16S rDNA进行序列比对, 并计算遗传距离(表2); 用DNAspV5进行序列变异位点分析, 表2中12 溞个 个体可识别位点(包括缺失与丢失的位点)445个, 其中保守位点 326个, 变异位点119个, 单一位点18个, 简约信息点101个; 用MEGA软件分析平均转换/颠换(si/sv)数为 1.25, 转换数明显高于颠换数。就构建的NJ树(图1)和贝叶斯树来看, 两者主要分枝基本一致。

表2 12个溞类个体16S rDNA的遗传距离(对角线下)和序列相似度(对角线上)Tab. 2 The genetic distances (below diagonal) and sequence similarity (above diagonal) of 16S rDNA in twelve Daphnia individuals

图1 基于12 溞个 属个体16S rDNA序列构建的系统发生树(NJ树)Fig. 1 NJ tree based on the sequence of 16S rDNA in twelve Daphnia individuals
2.2 基于COⅠ基因序列的系统发育分析
本研究发现4个种类16个个体的COⅠ基因部分序列中, 隆线 溞7个个体中, 有4个单倍型, 平均相似率为 99%, 有三个多态性位点均发生转换。蚤状 溞2个个体序列相似度为98%(表3)。参与分析的4个种类8个个体中平均种间相似率为80.67%, 低于16S rDNA的种间相似度。A+T含量(69.25%)明显高于 G+C(30.75)含量, 符合无脊椎动物 A、T含量高的特点。用 DNAspV5分析可识别的位点数为569个, 其中保守性位点 368个, 变异位点 201个,远大于16S rDNA的变异。单一位点15个, 简约信息点186个(图2)。用Mega 4.1软件分析碱基的转换/颠换树之比为1.42。就构建的NJ树(图2)和贝叶斯树来看, 两者主要分枝也基本一致。
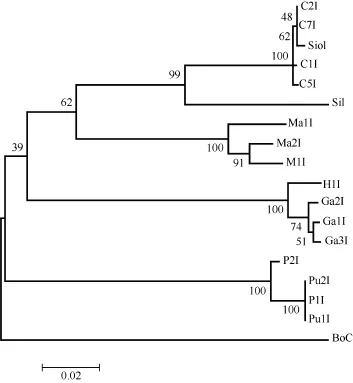
图2 基于17 溞个 属个体线粒体COI基因序列构建的系统发生树(NJ树)Fig. 2 NJ tree based on the sequence of mitochondrial COI gene in seventeen Daphnia individuals

表3 17个溞类个体基于线粒体COI基因的遗传距离(对角线下)及序列相似度(对角线上)Tab. 3 The genetic distances (below diagonal) and sequence similarity (above diagonal) based on mitochondrial COI gene in seventeen Daphnia individuals
3 讨论
3.1 有关几种争议溞类的命名和分类地位的探讨
鹦鹉 溞和隆线 溞由于在形态上十分接近, 国内学者在参考中国动物志进行鉴定时有可能把两者混为一谈, 统称为隆线 溞[5,6]。近几年来, 关于同形溞的报道逐渐增多, 以前以隆线 溞命名的种名逐渐更正为同形 溞[5]。 隆线 溞最早由King在1853年进行绘图描述, 我国的中国动物志中也参考了King的形态描述, 记载了我国隆线 溞的分布。通过形态比对,国内学者早年描述的隆线 溞在形态上与 King的记载存在很大的区别[17]。在本研究中, 选用的 7个个体16S rRNA基因均相同而COⅠ基因有所不同, 这验证了16S rRNA基因相对保守、COⅠ基因变异较大的特点。基于16S rDNA序列可以看出, 与C5S相似度最大的为同形 溞(Daphnia similis), 相似度为94%。基于K2P模型分歧度为4.9%(表2), 接近5%的枝角类线粒体的种内分歧度[18]。同时, 基于COⅠ基因序列, 与C1I-C7I 相似度最大均为拟同形溞(Daphnia similoides)。基于 K2P模型分歧度为0.4%—0.9% (表3), 明显小于枝角类种内差异5%的数值。因此, 仅仅从线粒体序列差异来看, 巢湖隆线溞应为拟同形 溞, 两种不同标记的进化树均支持以上结论(图1、图2)。这与顾杨亮等[17]结合形态分类的研究结果一致。
关于透明 溞和盔形 溞的归属也存在问题, 国内学者分类一般参考《中国动物志·淡水枝角类》[2]均以透明 溞命名。近年来, 国内学者将透明 溞的命名逐渐更正为盔形 溞[19]。在本研究中, 巢湖透明 溞与盔形 溞基于16S rDNA的相似度为98%, 基于K2P模型的遗传距离为0.7% (表2); 同时基于COⅠ基因相似度为97%—98%, 基于K2P模型的遗传距离为1.8%—2.3% (表3); 两种分子标记均支持透明 溞为盔形 溞的结论。本实验所构建的两种进化树也均显示巢湖透明 溞与盔形 溞的亲缘关系最近(图1、图2)。据报道, 透明 溞 、盔形 溞 、僧帽 溞 、长刺 溞等近源物种之间存在种间杂交的现象, 其杂交种具有外形相近、核基因相同线粒体基因相近或线粒体基因相同核基因相近的特点[6,20]。仅根据线粒体基因来划分, 巢湖的透明 溞 应与盔形 溞为同一个物种。在本研究中, 来自GenBank的D. mendotae在国内尚未见记载, 且与巢湖透明 溞和来自GenBank 的盔形溞遗传距离在种内范围内, 该种也可能为盔形 溞的种复合体。在今后的研究中将增加分子标记的数量,以及增加核基因与线粒体基因进行对比, 以确定巢湖 溞属种类间是否存在种复合体。
3.2 四种溞类的系统进化关系分析
据报道, 16S rDNA进化速度较慢且相对保守[21],而COⅠ基因变异相对较大, 约是16S rDNA的1.5倍[22]。在本研究中, 12 个 溞个体16S rDNA变异率(总变异数/可识别位点数)为 26.7%, COⅠ基因的变异率为35.3%, 是16S rDNA变异率的1.32倍。两种测序结果的A+T含量均明显高于G+C的含量, 这一结果与其他学者在无脊椎动物中研究结果相一致[23—25]。基于 COⅠ基因的 K2P双参模型遗传距离, 属内种间距离最小为 8.7%, 最大为 29.9%, 与端足类钩虾属的种间距离一致(min 5.58—max 31.39)[26]。通过序列比对可以看出, 四种 溞类的 16S rRNA基因变异区较集中, 而COⅠ基因变异区较分散。两种分子标记构建的进化树均显示, 大型 溞 与拟同形 溞亲缘关系最近, 这也符合形态分类标准, 他们同为一个亚属。大型 溞与拟同型 溞先聚为一支, 然后与盔形溞聚为一支, 最后与蚤状 溞聚在一起; 根据进化树物种分枝时间不同, 四种 溞的进化趋势由低到高为蚤状 溞— 盔形 溞— 大型 溞— 拟同形 溞。在本研究中,基于16S rDNA, 与P1S、P2S相似度最大和遗传距离最小的均为短钝 溞(D. obtusa), 其次才为蚤状 溞;而在COⅠ基因的比对中与P1S、P2S相似度较高的均为蚤状 溞; 在构建的系统树中, 基于16S rDNA的进化树出现了巢湖蚤状 溞最先与短钝 溞并为一支,而在基于COⅠ的进化树中却没有出现这种现象。另外, 基于16S rDNA, 巢湖蚤状 溞与来自GenBank的蚤状 溞种内遗传距离(0.81—0.86)大 于其他 溞种内遗传距离, 巢湖蚤状 溞与来自GenBank 上的短钝溞间的分类关系有待于进一步研究。线粒体为非编码蛋白质基因, 进化过程中没有受到密码子选择压力影响, 其大部分区域发生的变异为中性突变[27], Schubart等提出的16S rDNA分子钟理论, 即每百万年序列变异为0.4%—0.9%[28]。P1S、P2S均来自巢湖休眠卵的孵化, 其基因序列差异为1%, 可以推测2个蚤状 溞个体的分化时间约为1.11百万—2.5百万年前, 这在其他几个个体中均没有出现过。由此可见, 巢湖 溞属种类的遗传多样性和种类的系统进化关系还需进一步探讨。
参考文献:
[1] Zhao W. Hydrobiology [M]. Beijing: China Agricultural University Press. 2005, 181 [赵文. 水生生物学. 北京: 中国农业大学出版社. 2005, 181]
[2] Jiang X Z, Du N S. Fauna Sinica: Crustacean, Freshwater Cladocera [M]. Beijing: Science Press. 1979, 102—118 [蒋燮治, 堵南山. 中国动物志·淡水枝角类. 北京: 科学出版社. 1979, 102—118]
[3] Wolf H G, Mort M A. Inter-specific hybridization underlies phenotypic variability in Daphnia populations [J]. Oecologia, 1986, 68: 507—511
[4] Hebert P D N, Schwartz S S, Hrbacek J. Pattern of genotypic diversity in Czechoslovakian Daphnia [J]. Heredity, 1989, 62: 207—216
[5] Benzie J A H. The Genus Daphnia (Including Daphniopsis) [M]. In: Dumont H J F (Eds.), Guides to the Identification of the Microinvertebrates of the Continental Waters of the World. Leiden: Backhuys Publishers. 2005, 305—310
[6] Schwenk K, Sand A, Boersma M, et al. Genetic markers, genealogies and biogeographic patterns in cladocera [J]. Aquatic Ecology, 1998, 32: 37—51
[7] Gelas K D E, Meester L D E. Phylogeograph of Daphnia magna in Europe [J]. Molecular Ecology, 2005, 14: 753—764
[8] Briski E, Cristescu M E, Bailey S A, et al. Use of DNA barcoding to detect invertebrate invasive species from dispausing eggs [J]. Biology Invasions, 2011, 13: 1325—1340
[9] Fan F J. Morphology and molecular phylogeny of Bosmina in China [D]. Thesis for Master of Science, Jinan University, Guangzhou. 2010 [范风娟. 溞中国象鼻 属的形态与分子系统发育学研究. 硕士学位论文, 暨南大学, 广州. 2010]
[10] Ren J J. Analysis of random amplified polymorphic DNA of genetic diversity of four geographic populations of Daphnia galeata [D]. Thesis for Master of Science, Jinan University, Guangzhou. 2010 [任晶晶. 4 溞个盔型 地理种群遗传多样性的RAPD分析. 硕士学位论文, 暨南大学,广州. 2010]
[11] Chen H. The morphology and molecular phylogeny of Diaphanosoma in China [D]. Thesis for Master of Science, Jinan University, Guangzhou. 2011 [陈花. 溞中国秀体 属的形态与分子系统发育学研究. 硕士学位论文, 暨南大学,广州. 2011]
[12] Xu L, Han B P, Damme K V, et al. Biogeograph and evolution of the Holarctic zooplankton genus Leptodora (Crustacea: Branchiopoda: Haplopoda) [J]. Journal of Biogeography, 2011, 38: 359—370
[13] Hu H J, Yan Q Y, Ni J J, et al. Genetic structures of Chydorus sphaericus populations from lake Donghu, Wuhan [J]. Acta Hydrobiologica Sinica, 2013, 37(6): 1007—1012 [胡红娟, 颜庆云, 倪加加, 等. 溞武汉东湖圆形盘肠 种群遗传结构分析. 水生生物学报, 2013, 37(6): 1007—1012]
[14] Deng D G, Xie P, Zhou Q, et al. Field and experimental studies on the combined impacts of cyanobacterial blooms and small algae on crustacean zooplankton in a large, eutrophic subtropical Chinese lake [J]. Limnology, 2008, 9: 1—11
[15] Bouchon D, Souty-Grosset C, Raimond R. Mitochondrial DNA variation and markers of species identity in two penaeid shrimp species: Penaeus monodon Fabricius and P. japonicus Bate [J]. Aquaculture, 1994, 127(2): 131—144
[16] Crease T J. The complete sequence of the mitochondrial genome of Daphnia pulex (Cladocera: Crustacea) [J]. Gene, 1999, 233(1—2): 89—99
[17] Gu Y L, Xu L, Lin Q Q, et al. A new subspecies of Daphnia: Daphnia similoides sinensis [J]. Ecological Science, 2013, 32(3): 308—312 [顾杨亮, 徐磊, 林秋奇, 等. 新亚种—中
溞华拟同形 (Daphnia similoides sinensis). 生态科学, 2013, 32(3): 308—312]
[18] Adamowicz S J, Petrusek A, Colbourne J K, et al. The scale of divergence: a phylogenetic appraisal of intercontinental allopatric speciation in a passively dispersed freshwater zooplankton genus [J]. Molecular Phylogenetics and Evolution, 2009, 50(3): 423—436
[19] Deng C. Effects of nutrient enrichment and fish stocking on zooplankton communities [D]. Thesis for Master of Science, Jinan University, Guangzhou. 2011 [邓琛. 营养盐加富和鱼类补充对浮游动物群落的影响. 硕士学位论文, 暨南大学,广州. 2011]
[20] Dlouhá S, Thielsch A, Kraus R, et al. Identify hybridizing taxa within the Daphnia longispina species complex:a comparison of genetic methods and phenotypic approaches [J]. Hydrobiologia, 2010, 634: 107—122
[21] Dai Y J, Liu P, Gao B Q, et al. Sequence analysis of mitochondrial 16S rRNA and COiv gene fragments of four wild populations of Portunus trituberculatus [J]. Periodical of Ocean University of China, 2010, 40(3): 54—60 [戴艳菊,刘萍, 高保全, 等. 三疣梭子蟹 4个野生群体线粒体16SrRNA和COiv基因片段的比较分析. 中国海洋大学学报, 2010, 40(3): 54—60]
[22] Taylor D J, Finston T L, Hebert P D N. Biogeography of a widespread freshwater crustacean: Pseudocongruence and cryptic endemism in the North American Daphnia leavis complex [J]. Evolution, 1998, 52: 1648—1670
[23] Anderson F E. Phylogeny and historical biogeography of the loliginid squids (Mollusca: Cephalopoda) based on mitochondrial DNA sequence data [J]. Molecular Phylogenetics and Evolution, 2000, 15(2): 191—214
[24] Gao T X, Zhang X M, Yoshizaki G, et al. Study on mitochondrial DNA sequences of Japanese mitten crab, Eriocheir Japonica I. 12S rRNA [J]. Journal of Ocean University of Qingdao, 2000, 30(1): 43—47 [高天翔, 张秀梅, 吉崎悟朗, 等. 日本绒螯蟹线粒 DNA序列研究 I.12SrRNA. 青岛海洋大学学报, 2000, 30(1): 43—47]
[25] Liu J, Li Q, Kong L F, et al. COⅠ-based DNA barcoding in mytilidae species (Mollusca: Bivalvia) [J]. Acta Hydrobiologica Sinica, 2011, 35(5): 874—881 [刘君, 李琪, 孔令锋,等. 基于线粒体COⅠ的DNA条形码技术在贻贝科种类鉴定中的应用. 水生生物学报, 2011, 35(5): 874—881]
[26] Costa F O, deWaard J, Boutillier J, et al. Biological identifications through DNA barcodes: the case of the Crustacea [J]. Canadian Journal of Fisheries and Aquatic Sciences, 2007, 64: 272—295
[27] Zheng L, He M L, Jin J R, et al. Primary and secondary structure of 3’-end of the large subunit ribosomal RNA of silkorm Attacus ricini and evolution implications as Inferred from the gene sequence [J]. Acta Genetica Sinica, 1997, 24(4): 296—304 [郑雷, 何明亮, 靳嘉瑞, 等. 蓖麻蚕核糖体大亚基RNA基因3′端序列分析及进化研究. 遗传学报, 1997, 24(4): 296—304]
[28] Schubart C D, Diesel R, Hedges S B. Rapid evolution to terrestrial life in Jamaican crabs [J]. Nature, 1998, 393: 363—365
PHYLOGENETIC RELATIONSHIP AND TAXONOMIC STATUS OF FOUR DAPHNIA SPECIES BASED ON 16S rDNA AND COⅠSEQUENCE
XU Min, ZHANG Hai-Jun, DENG Dao-Gui, WANG Wen-Ping, ZHANG Xiao-Li and ZHA Ling-Sheng
(Anhui Key Laboratory of Resource and Plant Biology, School of Life Science, Huaibei Normal University, Huaibei 235000, China)
To examine the systematic classification of four previously-recorded Daphnia species, D. carinata, D. hyalina, D. pulex, and D. magna, genomic DNAs of these species were extracted and PCR was used to amplify fragments of 16S rDNA and COⅠ gene by employing specific primers. The sequences of amplified fragments were compared with Daphnia species from the GeneBank. Based on 16S rDNA and COⅠ gene, the average interspecific similarity rate of four Daphnia species was 85.25% and 80.67% respectively, and the content of A+T was obviously higher than that of G+C. The similarity of D. carinata from Lake chaohu and D. similoides (AB549199) from the GeneBank was 99% according to COⅠ gene and the divergence rate was 0.4%—0.9%. D. hyalina from Lake Chaohu was more close to D. galeata from the GeneBank based on 16S rDNA or COⅠgene. Main branches of both NJ tree and bayes tree were similar. The results indicated that D. carinata and D. similoides were the same species, and D. hyalina belongs to D. galeata. Due to the lack of nuclear gene analysis, the classification status of Daphnia species in Lake Chaohu needs further investigation.
Daphnia; 16S rDNA; COⅠgene; Systematic classification
Q951+.3
A
1000-3207(2014)06-1040-07
10.7541/2014.153
2013-10-09;
2014-03-11
国家自然科学基金 (31370470)资助
徐敏(1983—), 女, 安徽蒙城人; 硕士研究生; 研究方向为浮游生物系统进化。E-mail: minxuzheng@126.com
邓道贵, E-mail: dengdg@263.net

