串联亲和纯化技术在研究细胞内相互作用蛋白质中的应用
刘 侠(综述),韦 薇(审校)
(1.广西医科大学研究生院,广西 530021; 2.广西医科大学附属肿瘤医院乳腺外科,广西 530021)
随着基因组计划的进行,仅了解基因DNA序列及表达已不能解决基因的表达时间、表达量以及蛋白质翻译后的情况。蛋白质行使的功能多样化,包括信息传递、生物化学反应等,所有的这些功能是通过蛋白质之间的相互作用来实现的。因此,大规模、全细胞分析蛋白质之间的相互作用,建立细胞内蛋白质复合体之间的相互作用网络,为准确理解蛋白质功能,揭示生命活动的机制提供重要信息。目前用于蛋白质组间相互作用的方法主要有酵母双杂交、谷胱甘肽巯基转移酶亲和层析、噬菌体展示技术、免疫共沉淀、串联亲和纯化(tandem affinity purification,TAP)技术和蛋白芯片等及其基于生物信息学的分析方法,各种技术都有其优劣,在不同研究中功能互补。以下主要介绍TAP技术及其在细胞系内使用时需要注意的问题。
1 TAP技术的特点
1.1TAP技术原理 TAP技术最早由德国Rigaut等[1]发明,并在鉴定酵母菌蛋白复合物中获得成功,随后扩展到其他细胞系及组织[2]。TAP技术基本原理包括:重组构建带有两种亲和纯化标签的融合靶蛋白,然后转染到宿主细胞或组织,带标签的靶蛋白表达并结合上相关蛋白质后,利用标签与相应磁珠作用,两步纯化得到相关蛋白质。
1.2TAP技术优点 该方法的主要优点概括有四方面:①可以在细胞生理状态下或者接近生理状态表达融合靶蛋白,研究靶蛋白及其未知的相关蛋白复合体,更真实地反映蛋白质在生物体内的功能;②经过两次洗脱,降低了非特异蛋白量,相比较于酵母双杂交更敏感,更少假阳性;③可以反映复杂的蛋白相关性,除了鉴定到直接结合蛋白,亦能检测到非直接结合蛋白,甚至捕捉到除蛋白质以外的小分子物质。④TAP标签种类多样,能根据研究需要选择和设计标签,技术适用广泛且实用性强。
1.3TAP技术的限制 每种技术在研究方面会有其局限性,TAP技术也不例外。除TAP标签本身对蛋白质结构的影响外,在构建融合基因导入细胞系中,哺乳动物细胞比酵母的同源重组效率差,而通过瞬时转染和外源性启动子稳定转染获得的融合蛋白呈现高表达,往往会引起细胞生理活动的变异,虽然可以利用这点研究特殊情况下的蛋白相互作用,但过量表达蛋白结合大量的分子伴侣和热激蛋白而绝缘,出现蛋白非正常折叠,细胞定位变化和细胞应激反应产生大量假阳性相互作用,可能导致实验结果偏离[3]。
2 TAP技术的应用
2.1TAP技术流程 利用TAP技术鉴定细胞内相互作用蛋白质的技术,应用于真核细胞系流程大致如下:首先选取感兴趣的靶蛋白质,从细胞或组织中通过聚合酶链反应获得该蛋白质DNA全长,然后选择含有标签序列的亲本质粒,将靶蛋白的与TAP标签蛋白的DNA进行重组。如果没有带标签的亲本质粒,也可以使用全基因合成将靶蛋白和标签蛋白的DNA融合入表达载体,使用质粒或者慢病毒转染至细胞系,选定稳定转染表达带标签靶蛋白的细胞株,提取蛋白后纯化蛋白复合体,纯化的样本进行质谱分析等后续研究。
2.2TAP技术的成功应用 Ogawa等[4]首次使用FLAG/HA(influenza hemagglutinin epitope)串联亲和标签,在人海拉细胞系内使用带标签的转录因子E2F-6蛋白鉴定到E2F-6与Mga和Max组成的蛋白复合体,进而阐释该复合体在生物体内的功能。Gloeckner等[5]使用StrepⅡ/FLAG标签在人胚肾细胞HEK293鉴定到Raf(rapidly accelerated fibrosarcoma)和其相互作用蛋白促分裂原活化蛋白激酶1(mitogen-activated protein kinase kinase,MEK1)和14-3-3蛋白;使用该标签能快速有效地纯化目标靶蛋白,并由此建立了该纯化标签的纯化标准流程[6]。TAP标签除了鉴定到相互作用蛋白质,更可以鉴定到蛋白质以外的小分子物质。Nonne等[7]在2010年利用TAP技术上捕获到mRNA相关的miRNA,使TAP标签的应用更为广泛。以下按年份整理了部分TAP标签在哺乳类,尤其是人类细胞系及其鉴定到的蛋白复合物(表1)。
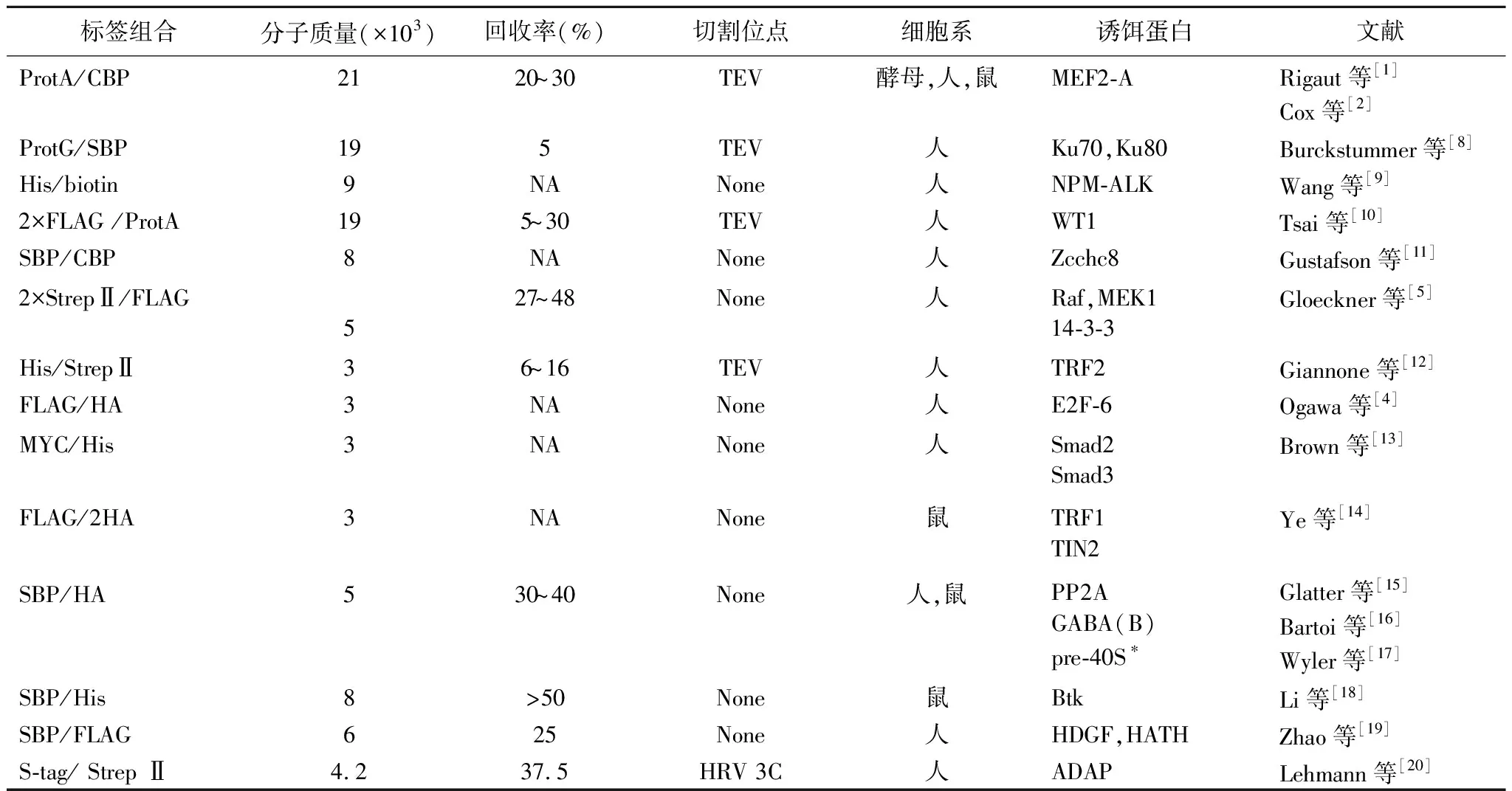
表1 串联亲和纯化标签的应用
*:为RNA; NA:没有数据; ProtA:蛋白A免疫球蛋白伽马结合位点;CBP:钙调蛋白结合位点;ProtG:蛋白G免疫球蛋白伽马结合位点;SBP:链霉亲和素结合位点;His:多组氨酸结合位点;biotin:生物素;StrepⅡ:两个链霉素亲和位点;MYC:MYC结合位点;HA:influenza hemagglutinin epitope;S-tag:S肽标签;MEF2-A蛋白:肌细胞增强因子2抗原;Ku70,Ku80:DNA依赖蛋白激酶调节亚单位;NPM-ALK:间变性淋巴瘤激酶核磷酸蛋白融合蛋白;WT1:Wilms肿瘤基因;Zcchc8:锌指8;Raf:加速纤维肉瘤蛋白,rapidly accelerated fibrosarcoma;MEK1:促分裂原活化蛋白激酶;14-3-3:14-3-3蛋白;TRF2:端粒重复序列结合因子2;E2F-6:一种真核细胞转录因子;Smad2:丝氨酸/苏氨酸激酶受体2;Smad3:丝氨酸/苏氨酸激酶受体3;TRF1:端粒重复序列结合因子1;TIN2:TRF1作用核蛋白2;PP2A:蛋白磷酸酶2;GABA(B):γ-氨基丁酸;pre-40S:预成熟核糖体亚基40;Btk:酪氨酸蛋白激酶;HDGF:肝癌衍生生长因子;HATH:肝癌衍生生长因子同源氨基末端;ADAP:黏附脱颗粒促进适配蛋白
3 应用TAP技术中需注意的问题
3.1常用的TAP标签及其选择 随着TAP技术的发展,越来越多的标签供不同串联组合[3],常见的TAP标签有钙调蛋白结合肽、蛋白A免疫球蛋白伽马结合位点(IgG binding domains of protein A,ProtA)、His、FLAG、StrepⅡ和链霉抗生物素蛋白结合肽等,在选择上需要考虑的问题包括:纯化回收率、纯度、对融合蛋白的结构和生物学的影响,以及成本。因此,标签的大小、TAP标签之间有无酶切位点,TAP标签的亲和率和洗脱条件等都是需要注意的要点。
在最开始的实验中,由于TAP标签体积对靶蛋白影响明显,融合蛋白出现表达变异,蛋白质空间结构改变使其性状无法表达,蛋白质功能的改变。通过小TAP标签可以改善由于亲和标签分子质量太大对靶蛋白的影响,目前最小的TAP标签组合的分子质量仅有3×103。此外,把TAP标签加在不同的末端也可能改善对靶蛋白的影响。Cipak等[21]报道,在TAP标签和靶蛋白之间加入柔性片段可以有效降低TAP标签带来的负面影响。
TAP标签及对纯化柱的亲和指数越高,结合到纯化柱上的概率越大,但同时洗脱回收率可能下降,对于难以培养的细胞系需要选择高回收率的TAP标签,如FLAG。对于那些容易降解的靶蛋白,则应选择洗脱条件温和的TAP标签,如链霉亲和素结合位点(streptavidin-binding peptide,SBP)。需多者兼顾的可以考虑使用StrepⅡ标签。多组氨酸结合位点(Polyhistidine,His)作为商业纯化蛋白是使用最多的标签之一[22],因其苛刻的洗脱条件,可以用于检测细胞蛋白经交联处理后的瞬时作用或者较弱的相互作用蛋白质[23]。
酶切位点作为TAP标签之间的连接,烟草蚀纹病毒酶切位点(tobacco etch virus,TEV)最为常见,另外还有人鼻病毒酶切位点HRV 3C蛋白酶[20]。TEV蛋白酶高效特异,基本不含其他细胞蛋白识别点,使用TEV蛋白酶切掉相关蛋白质的概率极低,可以在第一步纯化洗脱条件苛刻情况下使用,不同的标签在第一步可以使用同样的洗脱方法。标签的改良和纯化条件的改善使标签对靶蛋白影响减少,可不用酶切位点,这样的优点是不担心TEV酶的污染,但要求两步纯化的标签在洗脱时都比较温和。
3.2重组蛋白标签融合的位置 融合标签加在靶蛋白的N端还是C端,需要根据靶蛋白的特点,例如蛋白功能区域结合位点、信号肽、加工剪切位置和蛋白的三维结构等选择TAP标签加入的位置。多数实验初始设计两条融合蛋白,来应对可能无法顺利表达的问题。如果有融合基因构建后的mRNA水平与实际蛋白表达量不相符的情况出现,可能导致无法用TAP标签抗体检测并纯化,则需更换TAP标签位置。
3.3重组蛋白过表达和内源性蛋白竞争的问题 实现融合靶蛋白内源水平的表达和去除内源性蛋白竞争一直是TAP技术运用在真核细胞,尤其是哺乳动物体系的难题。目前最多使用反转录病毒稳定转染使蛋白质表达量接近生理表达量,或者用RNA干扰获得稳定表达生理量水平的带标签蛋白质。另有研究利用基于蜕皮素及其受体的可控诱导表达系统,根据需要调整蜕皮素类似物的添加量调整融合蛋白的表达水平[24]。对于内源性蛋白竞争,Forler等[25]使用RNA干扰和TAP结合建立的干扰TAP,选择果蝇相应蛋白同源的人类蛋白做诱饵,用双链RNA沉默果蝇的内源基因,然后再稳定转染表达外源基因。还有利用转基因或使用TAP重组基因小鼠[26],但因为成本高、时间长,无法实现高通量化。
4 展 望
TAP技术属于蛋白质组学中“Bottom-up”策略之一,对研究蛋白质功能有很强的实用性。TAP技术联合质谱对纯化后的蛋白质进行分析,通过生物信息学获得的蛋白相关性可信度高,为后续的验证工作节省大量时间。TAP和酵母双杂交得到的蛋白相互作用图谱中重合性不高,各技术策略侧重点不同,TAP技术倾向于蛋白在细胞自然生理条件下的功能和相互作用,除了检测直接相互作用蛋白,更可捕获间接相互作用蛋白,从而发现复杂的蛋白复合体。TAP技术同其他方法获得的数据进行补充印证,不断完善蛋白质相互作用图谱。TAP技术作为研究相互作用蛋白有不可替代的作用,该技术联合质谱技术将是蛋白质组学研究中发现蛋白功能,揭示复杂蛋白作用网络的重要手段。
[1] Rigaut G,Shevchenko A,Rutz B,etal.A generic protein purification method for protein complex characterization and proteome exploration[J].Nat Biotechnol,1999,17(10):1030-1032.
[2] Cox DM,Du M,Guo X,etal.Tandem affinity purification of protein complexes from mammalian cells[J].Biotechniques,2002,33(2):267-268.
[3] Li Y.The tandem affinity purification technology:an overview[J].Biotechnol Lett,2011,33(8):1487-1499.
[4] Ogawa H,Ishiguro K,Gaubatz S,etal.A complex with chromatin modifiers that occupies E2F-and Myc-responsive genes in G0 cells[J].Science,2002,296(5570):1132-1136.
[5] Gloeckner CJ,Boldt K,Schumacher A,etal.A novel tandem affinity purification strategy for the efficient isolation and characterisation of native protein complexes[J].Proteomics,2007,7(23):4228-4234.
[6] Gloeckner CJ,Boldt K,Schumacher A,etal.Tandem affinity purification of protein complexes from mammalian cells by the Strep/FLAG (SF)-TAP tag[J].Methods Mol Biol,2009,564:359-372.
[7] Nonne N,Ameyar-Zazoua M,Souidi M,etal.Tandem affinity purification of miRNA target mRNAs (TAP-Tar)[J].Nucleic Acids Res,2010,38(4):e20.
[8] Burckstummer T,Bennett KL,Preradovic A,etal.An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells[J].Nat Methods,2006,3(12):1013-1019.
[9] Wang P,Wu F,Ma Y,etal.Functional characterization of the kinase activation loop in nucleophosmin (NPM)-anaplastic lymphoma kinase (ALK) using tandem affinity purification and liquid chromatography-mass spectrometry[J].J Biol Chem,2010,285(1):95-103.
[10] Tsai A,Carstens RP.An optimized protocol for protein purification in cultured mammalian cells using a tandem affinity purification approach[J].Nat Protoc,2006,1(6):2820-2827.
[11] Gustafson MP,Welcker M,Hwang HC,etal.Zcchc8 is a glycogen synthase kinase-3 substrate that interacts with RNA-binding proteins[J].Biochem Biophys Res Commun,2005,338(3):1359-1367.
[12] Giannone R,McDonald WH,Hurst G,etal.Dual-tagging system for the affinity purification of mammalian protein complexes[J].Bio Techniques,2007,43(3):296-302.
[13] Brown KA,Ham AJ,Clark CN,etal.Identification of novel Smad2 and Smad3 associated proteins in response to TGF-beta1[J].J Cell Biochem,2008,105(2):596-611.
[14] Ye JZ,Hockemeyer D,Krutchinsky AN,etal.POT1-interacting protein PIP1:a telomere length regulator that recruits POT1 to the TIN2/TRF1 complex[J].Genes Dev,2004,18(14):1649-1654.
[15] Glatter T,Wepf A,Aebersold R,etal.An integrated workflow for charting the human interaction proteome:insights into the PP2A system[J].Mol Syst Biol,2009,5:237.
[16] Bartoi T,Rigbolt KT,Du D,etal.GABAB receptor constituents revealed by tandem affinity purification from transgenic mice[J].J Biol Chem,2010,285(27):20625-20633.
[17] Wyler E,Zimmermann M,Widmann B,etal.Tandem affinity purification combined with inducible shRNA expression as a tool to study the maturation of macromolecular assemblies[J].RNA,2011,17(1):189-200.
[18] Li Y,Franklin S,Zhang MJ,etal.Highly efficient purification of protein complexes from mammalian cells using a novel streptavidin-binding peptide and hexahistidine tandem tag system:application to Bruton′s tyrosine kinase[J].Protein Sci,2011,20(1):140-149.
[19] Zhao J,Yu H,Lin L,etal.Interactome study suggests multiple cellular functions of hepatoma-derived growth factor (HDGF)[J].J Proteomics,2011,75(2):588-602.
[20] Lehmann R,Meyer J,Schuemann M,etal.A novel S3S-TAP-tag for the isolation of T-cell interaction partners of adhesion and degranulation promoting adaptor protein[J].Proteomics,2009,9(23):5288-5295.
[21] Cipak L,Spirek M,Novatchkova M,etal.An improved strategy for tandem affinity purification-tagging of Schizosaccharomyces pombe genes[J].Proteomics,2009,9(20):4825-4828.
[22] Hellman LM,Zhao C,Melikishvili M,etal.Histidine-tag-directed chromophores for tracer analyses in the analytical ultracentrifuge[J].Methods,2011,54(1):31-38.
[23] Woodcock SA,Jones RC,Edmondson RD,etal.A modified tandem affinity purification technique identifies that 14-3-3 proteins interact with Tiam1,an interaction which controls Tiam1 stability[J].J Proteome Res,2009,8(12):5629-5641.
[24] Holowaty MN,Zeghouf M,Wu H,etal.Protein profiling with Epstein-Barr nuclear antigen-1 reveals an interaction with the herpesvirus-associated ubiquitin-specific protease HAUSP/USP7[J].J Biol Chem,2003,278(32):29987-29994.
[25] Forler D,Kocher T,Rode M,etal.An efficient protein complex purification method for functional proteomics in higher eukaryotes[J].Nat Biotechnol,2003,21(1):89-92.
[26] Zhou D,Ren JX,Ryan TM,etal.Rapid tagging of endogenous mouse genes by recombineering and ES cell complementation of tetraploid blastocysts[J].Nucleic Acids Res,2004,32(16):e128.

