AcMNPV lef-10在Sf9细胞中的表达与定位研究
刘田田,蒋 向,许晓东
(西北农林科技大学 生命科学学院,陕西 杨凌 712100)
杆状病毒是一类主要感染节肢动物的病毒,其宿主通常为鳞翅目昆虫,此外还有膜翅目和双翅目昆虫[1]。苜蓿银纹夜蛾核型多角体病毒(Autographacalifornicamultiple nucleopolyhedro virus,AcMNPV)是杆状病毒科的模式种,其基因组长134 kb,共有154个开放阅读框。AcMNPV的基因表达为级联式调控,按基因表达时序可分为4个阶段:立即早期基因表达、早期基因表达、晚期基因表达和极晚期基因表达。
早期基因的上游序列是昆虫细胞的调节序列,其启动子可以被宿主细胞的RNA聚合酶Ⅱ及相关转录因子所识别,表达产物可满足完成病毒复制所需的条件。病毒DNA复制是晚期基因转录的先决条件,晚期基因的表达依赖于早期基因及其产物,并通过病毒自身编码的RNA聚合酶催化晚期基因的转录;此外,晚期基因的表达还需要一类蛋白因子调控,即晚期表达因子(Late expression factor,lef)。
瞬时表达试验证明,AcMNPV中与病毒DNA复制相关的基因有:DNA引物酶lef-1[2]、DNA引物酶辅助因子lef-2(也是病毒蛋白组分)[3]、单链DNA结合蛋白lef-3[4-5]、DNA聚合酶dnapol[6]、反式激活因子ie1[7]、DNA复制辅助因子lef-7[8]、转录激活因子ie2(ien)[9]、DNA解旋酶p143[5]和抗细胞凋亡基因p35[10]。在已知功能的10个晚期表达因子中,有4个编码RNA聚合酶的亚基:lef-4、lef-8、lef-9和p47[11];而另外6个晚期表达因子为:lef-5(为转录起始因子而非延伸因子)[12]、lef-6(可以加速侵染循环)[13]、lef-10、lef-11(是DNA复制所必需的)[14]、lef-12(可以刺激晚期因子的表达)[15]和pp31(可以增强大多数病毒的转录水平)[16]。
体外晚期基因瞬时表达试验表明,有19个AcMNPV基因被确定为晚期表达因子[17-18]。而在这19个晚期表达因子中,目前关于lef-10的功能知之甚少。lef-10基因全长234 bp,编码78个氨基酸,相对分子质量为8.7 ku,是已知的相对分子质量最小的晚期表达因子。
本研究以AcMNPV全基因组为模板,分别构建包含lef-10、innateP-lef10和GFP融合基因的表达载体,感染草地贪夜蛾(Spodopterafrugiperda)Sf9细胞,对其编码产物进行亚细胞定位及时空表达分析,以期为进一步研究lef-10基因的功能奠定基础。
1 材料与方法
1.1 菌株、质粒和细胞
大肠杆菌Top10细胞、BL21(DE3)细胞、载体pTriEx-SP-GFP、Sf9细胞和pAc-GFP,皆由西北农林科技大学生命科学学院分子病毒学实验室保存;AcMNPV Bacmid来源于University of Reading,Prof. Ian Jones实验室。
1.2 主要试剂
PCR产物纯化试剂盒、凝胶回收试剂盒和质粒DNA小量抽提试剂盒,均购自上海生工生物工程有限公司;限制性内切酶BspHⅠ、NcoⅠ、BamHⅠ和XbaⅠ及T4 DNA连接酶、Unstained Protein MW Marker,均购自Fermentas公司;酵母浸粉和蛋白胨均购自北京奥博星生物技术有限公司;DNA Marker、抗GFP标签兔单克隆抗体、FITC标记的羊抗兔抗体,均购自康为世纪生物公司;Fugene HD转染试剂、碘化丙啶(Propidium Iodide,PI),均购自Roche公司;胎牛血清,购自Thermo Scientific HyClone公司。
1.3 引物设计与合成
参照AcMNPV基因组全序列[19],构建lef-10及innateP-lef10基因重组质粒,用Primer Premier 5软件设计特异性扩增引物(表1)。引物均由上海生工生物工程有限公司合成。
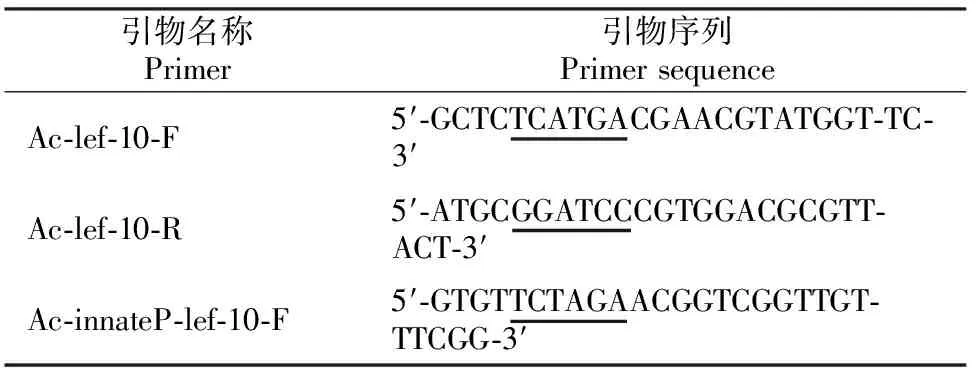
表1 lef-10及innateP-lef10基因重组质粒构建所用引物
1.4 lef-10及innateP-lef10基因重组质粒的构建
1.4.1lef-10及innateP-lef10基因的PCR扩增 以AcMNPV基因组DNA为模板,用引物Ac-lef-10-F/Ac-lef-10-R、Ac-innateP-lef-10-F/Ac-lef-10-R进行PCR扩增,扩增体系为:AcMNPV基因组100 ng,25 mmol/L MgCl22 μL,10×TaqBuffer 2 μL,25 mmol/L dNTP 0.4 μL,10 μmol/L上游引物Ac-lef-10-F/Ac-innateP-lef-10-F 1 μL,10 μmol/L下游引物Ac-lef-10-R 1 μL,Taq酶 0.2 μL,ddH2O补足20 μL;反应条件为:95 ℃预变性3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s(lef-10)/2 min(innateP-lef10),30个循环;72 ℃ 10 min,最后4 ℃保存。PCR产物用10 g/L琼脂糖凝胶电泳检测,并用PCR产物纯化试剂盒纯化后,-20 ℃保存。
1.4.2 pTriEx-lef10-GFP质粒的构建 用限制性内切酶BspHⅠ和BamHⅠ对上述PCR回收产物进行双酶切,然后用PCR产物纯化试剂盒回收,与以NcoⅠ和BamHⅠ双酶切pTriEx-SP-GFP质粒切胶回收的载体用T4 DNA连接酶16 ℃连接过夜,然后按常规方法转化大肠杆菌Top10感受态细胞,涂布于氨苄青霉素(50 μg/mL)抗性平板,培养过夜后挑单菌落进行PCR鉴定,将阳性菌落接种至含50 μg/mL氨苄青霉素的LB液体培养基中,37 ℃、250 r/min过夜摇菌,用质粒DNA小量抽提试剂盒提取质粒,并对其进行XbaⅠ、BamHⅠ双酶切鉴定,将鉴定为阳性克隆的重组质粒送往上海生工生物工程有限公司测序,构建正确的重组质粒命名为pTriEx-lef10-GFP。
1.4.3AcMNPVlef-10天然启动子的确定 根据杆状病毒早期基因核心启动子元件的保守基序,即TATA元件、起始子基元序列(INR)ATCA(G/T)T(C/T)、下游活化区元件(DAR)(A/T)CACNG及远侧上游活化区元件(UAR)A(A/T)CGT(G/T),找到了位于lef-10上游近1 676 bp处的启动子元件(图1)。
另外,在AcMNPV全基因组序列中,lef-10与下游基因vp1054有141 bp重叠,且位于vp1054上游523 bp处有3个TAAG基序[20],而TAAG基序为晚期与极晚期基因启动子的重要元件。

图1 lef-10和vp1054顺式作用启动子元件
引物Ac-innateP-lef-10-F就是根据杆状病毒早期基因核心启动子元件的保守基序设计的,用引物Ac-innateP-lef-10-F和Ac-lef-10-R进行PCR扩增所得的片段包含了lef-10启动子和编码区的全部序列。
1.4.4 pTriEx-innateP-lef10-GFP重组质粒的构建 将innateP-lef10 PCR产物及构建好的pTriEx-lef10-GFP质粒均进行XbaⅠ、BamHⅠ双酶切,产物回收,将酶切好的PCR产物片段与切胶回收的载体大片段进行连接,16 ℃过夜连接,其余操作方法同1.4.2。将构建正确的重组质粒命名为pTriEx-innateP-lef10-GFP。
1.5 lef-10在Sf9细胞中的时空表达及定位
1.5.1lef-10在Sf9细胞中的表达定位分析 将构建好的pTriEx-lef10-GFP与Bacmid共转染Sf9细胞,获得重组病毒。转染步骤为:取0.5 mL离心管,向其中加入100 μL无菌水、5 μL构建好的质粒和3 μL线性化Bacmid,混匀后加入5 μL转染试剂,充分混匀,静置10~30 min后均匀加入到铺好的Sf9细胞中。转染4 d后收集上清病毒,然后取100 μL病毒重新感染Sf9细胞,3 d后,置于荧光显微镜下观察。
1.5.2 天然启动子控制下的lef-10在Sf9细胞中的时空定位分析 将构建好的pTriEx-innateP-lef10-GFP与Bacmid共转染Sf9细胞。转染步骤同1.5.1。转染4 d后,收集上清病毒用于后续试验。将Sf9细胞铺于6孔板平皿中,每孔感染100 μL重组病毒,感染4,8,16,24和36 h后收集细胞,经固定、抗体孵育及碘化丙啶(Propidium Iodide,PI)染色。操作步骤如下:收集细胞于1.5 mL离心管中,200g离心15 min,弃上清;PBS洗3次,每次5 min,200g离心5 min,弃上清;质量分数4%多聚甲醛固定细胞15 min,200g离心5 min,弃上清;PBS洗2次,200g离心5 min,弃上清;用含有质量分数5%脱脂奶粉的PBS封闭30 min,200g离心5 min,弃上清;一抗37 ℃孵育2 h,200g离心5 min,弃上清;PBS洗3次,每次5 min,200g离心5 min,弃上清;二抗37 ℃黑暗孵育1 h,200g离心5 min,弃上清;PBS洗3次,每次5 min,200g离心5 min,弃上清;向细胞中加入PI(20 μg/mL)。取一干净载玻片,在其上滴1滴处理好的细胞悬浮液,盖上盖玻片,倒置,置于激光共聚焦显微镜下观察。
2 结果与分析
2.1 lef-10 PCR扩增及重组质粒pTriEx-lef10-GFP的双酶切鉴定
以AcMNPV基因组为模板,通过PCR扩增lef-10基因,得到234 bp的特异性片段,大小与预期结果吻合(图2-A)。将构建的重组质粒pTriEx-lef10-GFP经XbaⅠ和BamHⅠ双酶切后,得到约533 bp的片段(图2-B),与预期片段大小相同,表明载体构建正确。
2.2 innateP-lef10 PCR扩增及重组质粒pTriEx-innateP-lef10-GFP的双酶切鉴定
以AcMNPV基因组为模板,通过PCR扩增innateP-lef-10基因,得到2 182 bp的特异性片段,大小与预期结果吻合(图3-A)。将构建的重组质粒pTriEx-innateP-lef10-GFP经XbaⅠ和BamHⅠ双酶切后,得到约2 204 bp的片段(图3-B),与预期片段大小相同,表明载体构建正确。

图2 lef-10基因的PCR扩增结果(A)及重组质粒pTriEx-lef10-GFP的双酶切鉴定(B)
2.3 AcMNPV lef-10在Sf9细胞中的表达定位
由图4可见,在荧光显微镜下可以观察到绿色荧光蛋白,由于lef-10和GFP形成的融合蛋白在强晚期启动子p10下共表达,所以观察到的绿色部分为lef-10和GFP形成的融合蛋白。从图4可以看到,在重组病毒感染Sf9细胞的晚期,表达的lef-10蛋白主要定位于细胞核,初步确定了其在宿主细胞内的定位情况。

图4 AcMNPV lef-10在Sf9细胞中的表达定位
2.4 天然启动子控制下的lef-10在Sf9细胞中的时空表达定位
为了进一步确定lef-10在Sf9细胞中的时空表达情况,本试验对天然启动子启动表达的lef-10进行了时空表达定位分析。在激光共聚焦显微镜下观察的结果(图5)显示,在重组病毒感染Sf9细胞的早期,即感染4 h,与GFP融合的lef-10已开始表达,但表达量较低;在向晚期时相过渡时期(即感染8 h后),lef-10表达量增加并逐渐向细胞核转移,在晚期和极晚期时相,蛋白成簇聚集在细胞核中。据此推测,在病毒感染宿主细胞晚期,lef-10作为晚期表达因子在细胞核中发挥一定的功能,用于调控其他基因的转录;同时,也进一步证明试验早期核心启动子元件能够启动lef-10的早期表达。

图5 天然启动子控制下的lef-10在Sf9细胞中的时空表达及定位
3 讨 论
先前研究表明,无论在原核表达系统还是昆虫细胞表达系统中,lef-10都会形成一个巨大的复合物[21]。但由于得不到lef-10单体蛋白而无法获得抗lef-10的抗体,而GFP可以作为融合标签用于蛋白的亚细胞定位[22],所以本试验将融合了GFP的lef-10蛋白用于亚细胞定位分析。
由于目前没有关于AcMNPVlef-10及其启动子的报道,为了尽量保留其启动子及有关旁侧序列,本试验在找到的核心启动子元件上游延伸了近180 bp,从而使lef-10能够得到完整表达。
在AcMNPV基因组中,polh基因表达的蛋白是形成病毒包涵体的主要蛋白,它对感染性病毒的产生及复制是非必需的[23],所以在杆状病毒表达载体系统中,polh基因可以作为外源基因的插入位点。本试验构建的pTriEx-lef10-GFP和pTriEx-innateP-lef10-GFP载体可以与Bacmid发生重组,形成可以表达外源基因的重组病毒。
载体pTriEx-SP-GFP包含了可以在原核细胞(如大肠杆菌)、昆虫细胞及动物细胞中启动基因表达的3类启动子。本研究结果显示,pTriEx-lef10-GFP与Bacmid共感染Sf9细胞后,lef-10可以在载体自带的晚期强启动子p10的启动下进行表达;感染晚期,lef-10表达量较大且主要集中于细胞核中。
本试验分析了天然状态下的lef-10在宿主细胞中的表达定位情况,结果显示,在病毒感染细胞早期,lef-10就已开始表达,随着感染时间的推移,lef-10逐渐向细胞核移动,在晚期和极晚期时相,蛋白成簇聚集在细胞核中。这些结果也进一步说明,在受染细胞核中,lef-10是作为晚期表达因子发挥功能的,并且对其他基因的转录起一定的调控作用。
Lin等[24]和Durantel等[25]在研究AcMNPVlef-11和lef-4基因的表达及亚细胞定位时,利用anti-LEF-11和anti-LEF-4多克隆抗体,分析了在受感染细胞Sf9中lef-11和lef-4的表达水平及其在细胞中的定位,结果显示:lef-11和lef-4在病毒感染Sf9细胞的早期时相开始表达,且在晚期时相均定位在细胞核中,这与它们作为晚期表达因子行使功能是一致的;同时,2位学者还通过5′RACE、3′RACE及RT-PCR对它们的转录本进行了进一步分析。而在本试验中,由于抗体的限制,AcMNPV在感染Sf9细胞后,无法通过Western-blot方法对lef-10蛋白表达量进行分析,后续试验将在转录水平上通过对lef-10转录本进行分析,从而对lef-10的表达水平进行进一步验证。
4 结 论
本研究采用构建GFP融合标签的方法分析了lef-10在天然启动子控制下的表达及亚细胞定位情况,结果显示:lef-10在感染宿主细胞早期已开始表达,在晚期和极晚期时相,主要集中于细胞核中,推测lef-10对晚期其他基因的转录具有一定的调控作用。这为进一步研究lef-10的功能提供了一定的理论基础。
[参考文献]
[1] Marc H V,Claude M.Virus taxonomy:Classification and nomenclature of viruses [R]//Seventh Report of the International Committee on Taxonomy of Viruses.San Diego:Academic Press,2000.
[2] Mikhailov V S,Rohrmann G F.Baculovirus replication factor LEF-1 is a DNA primase [J].J Virol,2002,76:2287-2297.
[3] Wu C P,Huang Y J,Wang J Y,et al.Autographacalifornicamultiple nucleopolyhedrovirus LEF-2 is a capsid protein required for amplification but not initiation of viral DNA replication [J].J Virol,2010,84:5015-5024.
[4] Ito E,Sahri D,Knippers R,et al.Baculovirus proteins IE-1,LEF-3,and P143 interact with DNAinvivo:A formal dehyde cross-linking study [J].Virol,2004,329:337-347.
[5] Yu M,Carstens E B.Identification of a domain of the baculovirusAutographacalifornicamultiple nucleopolyhedrovirus single strand DNA-binding protein LEF-3 essential for viral DNA replication [J].J Virol,2010,84:6153-6162.
[6] McDougal V V,Guarino A.Autographacalifornicanuclear po-lyhedrosis virus DNA polymerase:Measurement of processing and strand displacement [J].J Virol,1999,73:4908-4918.
[7] Olson V A,Wetter J A,AFriesen P D.Oligomerization mediated by a helix-loop-helix-like domain of baculovirus IE1 is required for early promoter transactivation [J].J Virol,2001,75:6042-6051.
[8] Chen C J,Thiem S M.Differential infectivity of twoAutographacalifornicanucleopolyhedrovirus mutants on three permissive cell lines is the result of lef-7 deletion [J].Virol,1997,227:88-95.
[9] Yoo S,Guarino L A.TheAurographacalifornicanuclear pol-yhedrosis virus ie2 gene encodes a transcriptional regulator [J].Virol,1994,202:746-753.
[10] Kool M,Ahrens C H,Goldbach R W,et al,Identification of genes in DNA replication of theAutographacalifornicabaculovirus [J].Proc Natl Acad Sci USA,1994,91:11212-11216.
[11] Acharya A,Gopinathan K P.Characterization of late gene expression factors lef-9 and lef-8 fromBombyxmorinucleopolyhedrovirus [J].J Gen Virol,2002,83:2015-2023.
[12] Guarino L A,Dong W,Jin J.Invitroactivity of the baculovirus late expression factor LEF-5 [J].J Virol,2002,76:12663-12675.
[13] Lin G,Blissard G W.Analysis of anAutographacalifornicamulticapsid nucleopolyhedrovirus lef-6-null virus:LEF-6 is not essential for viral replication but appears to accelerate late gene transcription [J].J Virol,2002,76:5503-5514.
[14] Lin G,Blissard G W.Analysis of anAutographacalifornicanucleopolyhedrovirus lef-11 knockout: LEF-11 is essential for viral DNA replication [J].J Virol,2002,76:2770-2779.
[15] Guarino L A,Mistretta T A,Dong W.Baculoviruslef-12 is not required for viral replication [J].J Virol,2002,76:12032-12043.
[16] Yamagishi J,Burnett E D,Harwood S H,et al.The AcMNPV pp31 gene is not essential for productive AcMNPV replication or late gene transcription but appears to increase levels of most viral transcripts [J].Virol,2007,365:34-47.
[17] Lu A,Miller L K.The roles of eighteen baculovirus late expression factor genes in transcription and DNA replication [J].J Virol,1995,69:975-982.
[18] Rapp J C,Wilson J A,Miller L K.Nineteen baculovirus open reading frames,including LEF-12,support late gene expression [J].J Virol,1998,72:10197-10206.
[19] Ayres M D,Howard S C,Kuzio J,et al.The complete DNA sequence ofAutographacalifornicanuclear polyhedrosis virus [J].Virol,1994,202:586-605.
[20] Olszewski J,Miller L K.Identification and characterization of a baculovirus structural protein,VP1054,required for nucleocapsid formation [J].J Virol,1997,71:5040-5050.
[21] Xu X,Jones I M.Rapid parallel expression inE.coliand insect cells:Analysis of five lef gene products of theAutographacalifornicamultiple nuclear polyhedrosis virus (AcMNPV) [J].Virus Gen,2004,29:191-197.
[22] Chalfie M,Tu Y,Euskirchen G,et al.Green fluorescent protein as a marker for gene expression [J].Science,1994,263:802-805.
[23] Smith G E,Fraser M J,Summers M D.Molecular engineering of theAutographacalifornicanuclear polyhedrosis virus genome:Deletion mutations within the polyhedrin gene [J].J Virol,1983,46:584-593.
[24] Lin G,Slack J M,Blissard G W.Expression and localization of LEF-11 inAutographacalifornicanucleopolyhedrovirus-infectedSf9 cells [J].J Gen Virol,2001,82:2289-2294.
[25] Durantel D,Croizier G,Ravallec M,et al.Temporal expression of the AcMNPV lef-4 gene and subcellular localization of the protein [J].Virol,1998,241:276-284.

