昆明葡萄酒相关酵母菌的分离与鉴定
刘小珍,张汉尧
(1 农业部农业微生物资源收集与保藏重点实验室,北京 100081;2 西南林业大学 西南地区生物多样性保育国家林业局重点实验室,云南 昆明 650224)
酵母作为主要的发酵微生物不仅对葡萄酒质量和发酵过程影响很大,而且对葡萄酒色泽、香气、感官质量及风格的形成有重要贡献[1]。用不同菌株发酵生产的葡萄酒,其酒质和风味差别很大。所以寻找能够适应当地葡萄原料特性、有利于形成地域葡萄酒风格的新土著酵母菌株及新酵母种已成为研究的焦点[2-6]。目前,ITS序列的差异分析已被广泛用于酵母种属水平的鉴定[7-8]。Zhang等[9]在新西兰8个酒厂中分离了1 279个菌株,这些菌株分属9个属27个种,这些菌种的ITS与已发表的大部分酵母菌ITS序列存在或多或少的差异。Li等[10]对中国葡萄产区的酿酒酵母进行了ITS分析,发现共有8个属的17个酵母种在中国有分布。朱丽霞等[11]则对新疆慕萨莱思地区的葡萄酒相关酵母进行了多样性分析,发现共有7个属13个种的酵母。王泽举等[12]通过对酵母菌5.8S核糖体RNA ITS区域的PCR扩增,以及限制性酶切片段多态性(RFLP)图谱分析和26S rDNA D1/D2区、5.8S-ITS区的序列分析,将供试菌株鉴定为4个属4个种。此外,分子指纹图谱已被广泛用于群体中个体差异的研究[13]。Schuller等[14]对葡萄园中发酵相关的酿酒酵母进行了微卫星指纹分析。Howell等[15]则对发酵期间酿酒酵母的遗传多样性进行了微卫星分析。云南昆明具有典型的气候特征和独特的地理条件,是我国早熟葡萄的重要产地之一,蕴含着丰富的酵母菌资源。但目前尚未见有关云南昆明葡萄产区酿酒酵母资源的相关研究报道。
本研究拟分离云南昆明葡萄产区的酿酒酵母资源,并运用ITS分析鉴定其种属,采用微卫星分析鉴定不同酿酒酵母菌株,旨在为这些高原酵母资源的进一步利用奠定基础。
1 材料与方法
1.1 材 料
葡萄浆果于2012-08采自昆明官渡区三瓦村(在葡萄园内多点采样,并注意采集植株各个部位的浆果),取果皮 1.0 g,加入50 mL灭菌的液体SelMed富集培养基中,28 ℃摇床培养2 d。梯度稀释后涂布于分离纯化平板上,28 ℃培养3 d后,结合菌落颜色和形态选取多个菌落进行保藏。
1.2 试 剂
所用引物由上海生物工程公司合成;TaqDNA聚合酶、dNTPs、RNase购自上海生物工程公司;pMD18-T Vector、DNA Marker购自大连宝生物工程公司;酵母基因组DNA提取试剂盒购自天根生化科技(北京)有限公司;2×PowerTaqPCR MasterMix为北京百泰克生物技术有限公司产品。其他常规试剂为国产分析纯产品。
1.3 DNA提取
按照试剂盒说明书进行提取。将菌种采用划线法接种到新的培养基上,获得单菌落。接种单菌落酵母到10 mL酵母试管培养基(YPD培养基)中,28 ℃培养18 h;取1.5 mL酵母培养液,12 000 r/min离心1 min收集菌体;加入600 μL山梨醇buffer,5 μL 10 U/μL溶菌酶(Lyticase),30 ℃处理30 min;4 000 r/min离心10 min,向沉淀中加入200 μL缓冲液GA重悬;加入20 μL蛋白酶K混匀,加入4 μL RNase(100 mg/mL),室温放置5 min;加入220 μL缓冲液GB,颠倒混匀,70 ℃放置10 min;加220 μL 无水乙醇充分颠倒混匀,所得溶液和沉淀都加入吸附柱中,12 000 r/min离心30 s,向吸附管中加入500 μL缓冲液GD,12 000 r/min离心30 s,向吸附柱中加入500 μL漂洗液PW洗2次,最后加入30 μL ddH2O洗脱柱子上的酵母基因组DNA。
1.4 ITS序列的扩增及RFLP分析
使用通用引物进行分析,其中上游引物ITS1:5′-TCCGTAGGTGAACCTGCGG-3′;下游引物ITS4:5′-TCCTCCGCTTATTGATATGC-3′。PCR反应体系为15 μL:双链总DNA模板1.0 μL,上下游引物各1.0 μL,2×PowerTaqPCR MasterMix 7.5 μL,用双蒸水补足至15 μL。PCR反应程序:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸60 s,共35 个循环;最后72 ℃终延伸7 min,产物于-20 ℃ 保存。PCR产物检测:取PCR产物5 μL用EB染色,用质量浓度为30 g/L的琼脂糖凝胶进行电泳检测,用凝胶成像仪照相。
采用2种限制性内切酶HaeⅢ 和HinfⅠ对大于700 bp的PCR 扩增产物进行酶切分析,反应体系及反应条件见文献[9]。
1.5 ITS序列分析
将PCR产物委托中美泰和生物技术(北京)有限公司测序。运用NCBI 网站上的BLAST 比对测序结果,从GenBank 中查找与目的基因序列同源性最高且已知分类地位的菌种进行分离菌株鉴定。另外,从GenBank中提取所有与目的基因序列相关且已知的标准菌株序列。
1.6 微卫星分析
酿酒酵母的微卫星指纹图谱构建参照Zhang等[9]的方法进行,所用引物见表1。

表1 酿酒酵母微卫星分析所用的引物及序列
2 结果与分析
2.1 昆明地区葡萄表皮酵母菌株的菌落特征
本研究从采集的葡萄桨果表皮中共分离到了207个单克隆菌落。培养观察显示,其中以菌落形态深红色的平滑菌落居多,为37个;外观形态为奶油色且粗糙凸起的最少,仅有5个;外观形态为乳白色且平滑凸起的有35个;外观形态为奶油色平滑凸起、黄色平滑凸起、灰色平滑凸起的菌落数分别为31,26和23个;还有较少部分菌落外观形态呈黄色干燥凸起、乳白色粗糙凸起和灰白色粗糙凸起,分别为19,17和14个。
2.2 酿酒相关酵母的ITS分析与鉴定
ITS扩增产物在30 g/L的琼脂糖凝胶中电泳检测得到350~800 bp条带,符合理论预期的ITS区片段大小,部分扩增结果见图1。再进一步对大于700 bp的PCR扩增产物进行RFLP分析,最终筛选得到了23个酿酒酵母单菌落。随机选取ITS扩增片段大小相同且外观形态有一定差异的菌落各3个送中美泰和生物技术(北京)有限公司测序。ITS序列分析结果表明:实测的ITS长度为318~771 bp,而G+C的含量则为39.9%~48.9%(表2),所有菌落分属于3个属5个种,其中梅奇酵母属(Metschnikowia)有3个种,分别为金佩梅奇酵母(M.chrysoperlae)、美极梅奇酵母(M.pulcherrima)和核果梅奇酵母(M.fructicola);红酵母属(Rhodotorula)有1个种,为胶红酵母(R.mucilaginosa);酿酒酵母属(Saccharomyces)有1个种,为酿酒酵母(S.cerevisiae)。在鉴定出的3个属中,梅奇酵母属(Metschnikowia)在昆明地区葡萄果粒中出现频率最高,为昆明地区的优势酵母属。
2.3 酿酒酵母的SSR分析
SSR分析结果表明,所分离出来的23个酿酒酵母分属4个不同的菌株(具有不同的SSR指纹图谱),其中菌株KA17的SSR图谱见图2。将这些菌株的指纹图谱与国际酿酒酵母指纹图谱库比对后,发现其中有1个与欧洲商用菌株X5的指纹图谱完全一致,另外3个菌株的指纹图谱并未匹配到亲缘关系明显较近的国际菌株,表明在昆明的葡萄植株上栖息着具有自己遗传特征的酿酒酵母居群。
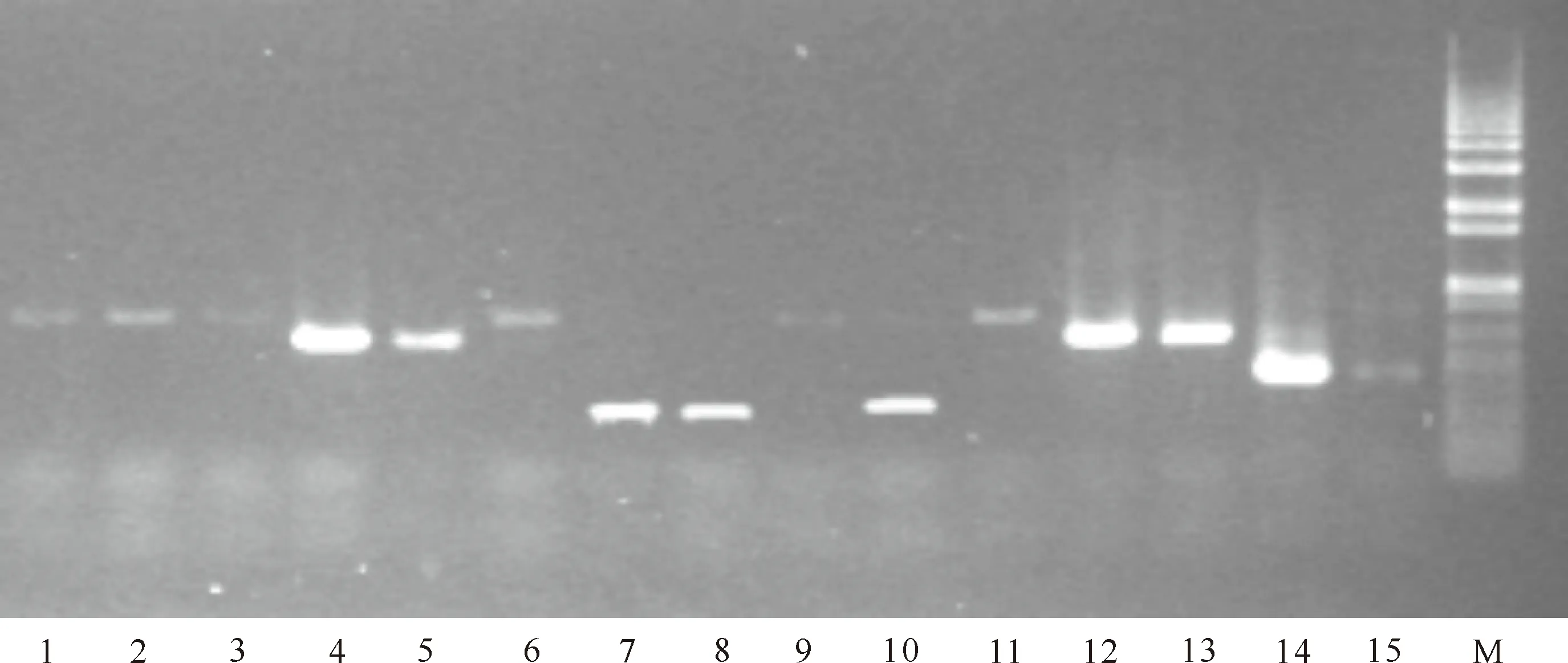
图1 云南昆明部分酵母菌落ITS区PCR扩增电泳图谱

表2 5种酿酒相关酵母的ITS序列长度和G+C含量
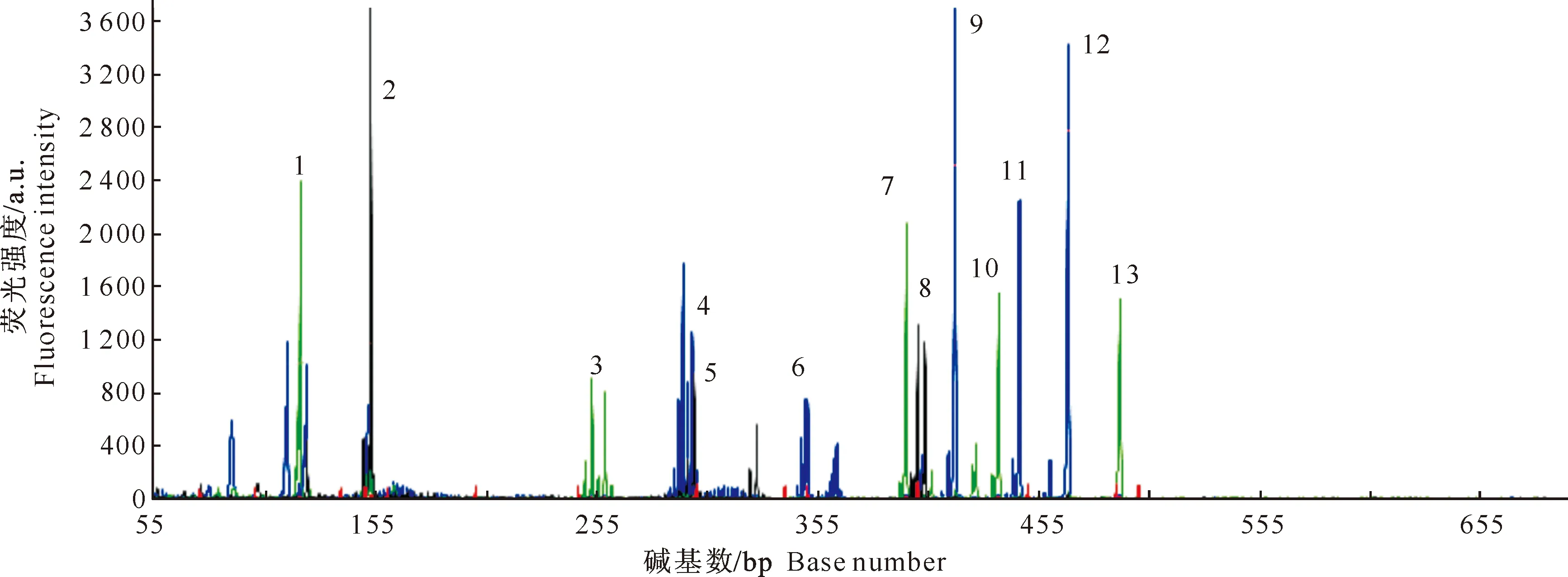
图2 酿酒酵母菌株KA17的微卫星指纹图谱
3 讨 论
目前,传统多是通过形态学、生理生化性状等对酿酒酵母进行分析,然后将试验结果与标准系统进行对比得出鉴定结论。但在菌落形态研究中发现,外形相似的菌落,却可能分属于不同的属,如外形虽皆为乳白色、奶酪状中间突起,但有些是酿酒酵母(如KA04),有些却是梅奇酵母(如KA13);有些外形有一定差异的菌落却是同一种菌种,如表面光滑、光亮的KA17与边缘光滑、暗淡的KB38均属于酿酒酵母。酵母属菌种显微形态主要以单细胞形式存在,显微观察结果表明,各属间的细胞性状很接近,属内种间的细胞差异几乎观察不到(数据未列出)。因此,形态学观察无法提供有效的分类学信息。酵母属各种间生理生化差异有限,而且这些菌种的表型性状不稳定。有研究表明,以生理生化指标为依据,有时可将同一菌株的冷冻干燥保藏株和多代斜面保藏株鉴定为不同的种[16]。因此,仅将生理生化特征差异作为酵母属各种间的分类依据并不可靠,而且该方法也可能将种水平之下菌株之间的差异误认为是种间差异。
本研究收集了昆明官渡区三瓦村的葡萄酒相关酵母,对其形态特征、培养特征等进行了研究,并运用5.8S-ITS扩增片段的RFLP分析及序列分析确定了菌株的种类归属。本研究结果表明,自昆明葡萄桨果果皮中分离鉴定出的所有酿酒相关酵母菌株分属于梅奇酵母属、红酵母属、酿酒酵母属等,其中梅奇酵母属出现频率最高,为昆明地区葡萄表皮酵母的优势属。需要指出的是,本研究是用SelMed富集培养基培养后再分离鉴定的,而富集培养后的分离鉴定不易反映真实的菌落结构。上述酵母在以往的研究[7-9]中均出现过,且以往关于葡萄园中葡萄酒相关酵母的研究结果以梅奇酵母属为优势或常见属的报道也较多[17-20]。另外,本研究还进一步用微卫星指纹图谱对分离筛选的23个酿酒酵母进行了鉴定,并发现了1株与欧洲商用菌株X5指纹图谱完全一致的菌株,但该菌株在昆明地区并未用于酿酒,而在欧洲得到了广泛应用,因此推测该菌株很可能是从欧洲的酒厂逃逸到葡萄植株上,而后随葡萄植株(或接穗)被引进到昆明。值得注意的是,不同研究报道中关于微卫星的菌株区分能力有一定差异[1,13],这可能与其所用的微卫星引物不同有关。传统的酵母菌分类及鉴定准确率较低,本研究在分子水平上以DNA序列为分类的依据,大大提高了菌种分类的准确率。
4 结 论
1)本研究在分子水平上以DNA序列及微卫星指纹图谱为鉴定的依据,大大提高了菌种鉴定的准确率,并可将鉴定水平提高至种下水平。
2)在昆明的葡萄园中至少栖息了5种酵母,包括酿酒酵母(S.cerevisiae)、胶红酵母(R.mucilaginosa),以及梅奇酵母属的金佩梅奇酵母(M.chrysoperlae)、美极梅奇酵母(M.pulcherrima)和核果梅奇酵母(M.fructicola)等。
3)分离自昆明官渡区三瓦村的葡萄酒相关酵母部分可能源于欧洲,是随葡萄植株(或接穗)被引进到昆明的。
[参考文献]
[1] Romano P,Capece A,Serafino V,et al.Biodiversity of wild strains ofSaccharomycescerevisiaeas tool to complement and optimize wine quality [J].World Journal of Microbiology and Biotechnology,2008,24(9):1797-1802.
[2] Pretorius I S.Tailoring wine yeasts for the new millenium:Novel approaches to the ancient art of winemaking [J].Yeast,2000,16(8):675-729.
[3] Caruso M,Fiore C,Contursi M,et al.Formation of biogenic amines as criteria for the selection of wine yeasts [J].World Journal of Microbiology & Biotechnology,2002,18(2):159-163.
[4] Cocolin L,Manzano M,Rebecca S,et al.Monitoring of yeast population changes during a continuous wine fermentation by molecular methods [J].American Journal of Enology and Viticulture,2002,53(1):24-27.
[5] Espinosa J C,Fernandez-Gonzalez M,Ubeda J,et al.Identification of wine yeasts by PCR-RFLP without previous isolation on plate [J].Food Technology and Biotechnology,2002,40(2):157-160.
[6] Martinez C,Cosgaya P,Vásquez C,et al.High degree of correlation between molecular polymorphism and geographic origin of wine yeast strains [J].Journal of Applied Microbiology,2007,103(6):2185-2195.
[7] Esteve-Zarzoso B,Belloch C,Uruburu F,et al.Identification of yeasts by RFLP analysis of the 5.8S rRNA gene and the two ribosomal internal transcribed spacers [J].International Journal of Systematic Bacteriology,1999,49(1):329-337.
[8] Dlauchy D,Tornai-Lehoczki J,Peter G.Restriction enzyme analysis of PCR amplifed rDNA as a taxonomic tool in yeast identification [J].Systematic and Applied Microbiology,1999,22(3):445-453.
[9] Zhang H Y,Lee S A,Bradbury J,et al.Yeasts isolated from New Zealand vineyards and wineries [J].Australian Journal of Grape and Wine Research,2010,16(3):491-496.
[10] Li S S,Cheng C,Li Z,et al.Yeast species associated with wine grapes in China [J].International Journal of Food Microbiology,2010,138(1/2):85-90.
[11] 朱丽霞,李红梅,郭东起,等.新疆慕萨莱思自然发酵过程中酵母菌表型多样性及优势菌分析 [J].食品科学,2012,33(7):142-147.
Zhu L X,Li H M,Guo D Q,et al.Phenotype diversity and dominant species of yeasts during spontaneous fermentation process of Musalais from Xinjiang [J].Food Science,2012,33(7):142-147.(in Chinese)
[12] 王泽举,王汝瑱,孙 悦,等.几株酵母菌的分子系统学鉴定 [J].食品科学,2012,33(15):195-200.
Wang Z J,Wang R Z,Sun Y,et al.Molecular phylogenic identification of wine-related yeasts [J].Food Science,2012,33(15):195-200.(in Chinese)
[13] Bradbury J E,Richards K D,Niederer H A,et al.A homozygous diploid subset of commercial wine yeast strains [J].Antonie van Leeuwenhoek,2005,89(1):27-37.
[14] Schuller D,Casal M.The genetic structure of fermentative vi-neyard-associatedSaccharomycescerevisiaepopulations revealed by microsatellite analysis [J].Antonie van Leeuwenhoek,2007,91(2):137-150.
[15] Howell K S,Bartowsky E J,Fleet G H,et al.Microsatellite PCR profiling ofSaccharomycescerevisiaestrains during wine fermentation [J].Letters in Applied Microbiology,2004,38(4):315-320.
[16] 白逢彦,贾建华,梁慧燕.假丝酵母属疑难菌株大亚基rDNA D1/D2区域序列分析及其分类学意义 [J].菌物系统,2002,21(1):27-32.
Bai F Y,Jia J H,Liang H Y.Molecular taxonomic study on the problematicCandidastrains based on 26S rDNA D1/D2 domain sequence comparison [J].Mycosystema,2002,21(1):27-32.(in Chinese)
[17] Nurgel C,Erten H,Canbas A,et al.Yeast flora during the fermentation of wines made fromVitisviniferaL.cv.Emir andKalecikkarasigrown in Anatolia [J].World Journal of Microbiology & Biotechnology,2005,21(6/7):1187-1194.
[18] Sipiczki M.Metschnikowiastrains isolated from botrytized gr-apes antagonize fungal and bacterial growth by iron depletion [J].Applied and Environmental Microbiology,2006,72(10):6716-6724.
[19] Lopandic K,Tiefenbrunner W,Gangl H,et al.Molecular profiling of yeasts isolated during spontaneous fermentations of Austrian wines [J].FEMS Yeast Res,2008,8(7):1063-1075.
[20] Comitini F,Ciani M.Influence of fungicide treatments on the occurrence of yeast flora associated with wine grapes [J].Annals of Microbiology,2008,58(3):489-493.

