不同引物扩增对东海陆架沉积物真核微型生物多样性的影响*
俞凯成 王健鑫① 陶 诗 刘明华 蒋 然 刘雪珠 黄 备
(1.浙江海洋学院海洋微生物分子生态与应用实验室 舟山 316022;2.浙江省舟山海洋生态环境监测站 舟山 316021)
海洋沉积物覆盖了超过地球表面2/3的面积,是海洋生态系统的重要组成部分,更是地球上主要的微生物栖息地(Whitmanet al,1998)。由于海底沉积物的特殊地理环境,其中的微生物往往具有一些适应其环境的形态学、生理学、遗传学上的特异性(戴欣等,2001),同时沉积物微生物还在整个海洋生态系统的生物地球化学循环过程中发挥着重要的作用(Kösteret al,2008),因此了解海洋沉积物微生物群落结构和多样性有着重要的意义。
东海是西太平洋构造活动带中一个大型边缘海,处于 21°54′—33°17′N,117°05′—131°03′E 之间,是西太平洋沟-弧-盆体系的典型发育地区(李家彪,2008)。东海不仅有着多样的水文条件、显著的物理化学梯度、显著的季节差异,同时有很高的初级生产力,是碳和其它物质在全球生物地球化学循环的重要场所,也是研究海底各种沉积作用和微生物群落多样性的有利场所(王健鑫等,2012)。东海陆架沉积环境微生物多样性的研究多集中在细菌(宋志刚等,2006;Zenget al,2007;王健鑫等,2012)和古菌(张林宝等,2010)等原核生物,对于真核微型生物多样性的研究较少(Parket al,2008),同时分析方法也比较单一,导致对于东海沉积环境真核微型生物缺乏系统科学的了解。
在真核微型生物分子多样性研究中,18S rDNA是最常使用的靶标基因之一,既可针对保守区进行引物设计,又可根据可变区来区分不同种真核生物的差异(汪岷等,2009);另外核糖体基因间隔区(ITS区)也是常用的靶标基因,由于其进化速率较快,被认为比较适合在较小的分类阶元层次进行鉴定和多样性分析(Rodríguezet al,2005)。在对真核微型生物分子多样性进行研究时,应根据各个靶标基因的特点来综合分析,以增加结果准确性,虽然针对陆生环境已有部分不同引物扩增效果的比较研究(Andersonet al,2003),但在海洋沉积环境中不同引物扩增微型真核生物的相关比较研究尚未涉及。
本文以东海陆架DH-13站点沉积物为研究对象,选择三对真核微型生物的通用引物(NS1/NS4、EF3/EF4、ITS1/ITS4),通过 18S rDNA、ITS区的基因克隆和文库构建,进行分类学、多样性和系统发育分析,以期为海洋沉积物真核微型生物多样性研究的引物选择提供科学参考。
1 材料和方法
1.1 样品采集
本文分析所用的沉积物样品是2007年4—5月海洋调查项目所采集的DH-13号沉积物柱样表层样品,采样点坐标为(31°30.103′N,123°29.844′E),水深 40m,位于东海陆架长江古河口区。样品采集后立即将0—10cm的沉积物柱芯以3cm为单位分为4层,分装于无菌样品袋中,–20°C保存,后放置于实验室–80°C超低温冰箱中长期保存,沉积物以中细砂为主。
1.2 样品总DNA的提取与纯化
利用 FastPrep®-24 快速核酸提取仪(MP Biomedicals公司)和FastDNA spin kit for soil试剂盒进行DNA提取,核酸蛋白检测仪(Bio-Rad公司)测定DNA浓度和纯度,纯化后用于PCR扩增。
1.3 PCR扩增和产物回收
将上述总DNA在梯度PCR仪(Biometra公司)中进行扩增,扩增采用通用引物ITS1/ITS4、NS1/NS4和EF3/EF4,见表1所示。50μL PCR扩增体系:DNA模板 2μL,顺反引物(10μmol/ L)各 1μL,dNTP(2.5mmol/L)4μL,10×PCR buffer(含 MgCl2,1.5mmol/L)5μL,rTaq 聚合酶(5U/μL)1μL,双蒸水补足至 50μL。PCR扩增条件:95°C预变性4min;94°C变性30s,50°C退火 30s,72°C 延伸 60s,循环 35次;最后 72°C 延伸10min。1%琼脂糖凝胶电泳检测PCR扩增结果,并用DNA胶回收试剂盒(QIAGEN公司)回收产物。
1.4 克隆和测序
PCR回收产物与pMD-18T vector(TaKaRa公司)在 16°C连接过夜,将连接产物转化到大肠杆菌(E.coli)DH5α感受态细胞,蓝白斑筛选转化子,挑选阳性克隆,重新扩增插入片断,将含有合适大小插入片段的克隆送上海美季生物技术有限公司进行测序。

表1 三对通用引物的详细信息Tab.1 Particular information of three universal primers
1.5 序列分析
将序列提交到RDP(ribosomal database project)Ⅱ数据库,利用在线工具CHECK- CHIMERA检测嵌合体;应用 BLASTN程序(www.ncbi.nlm.nih.gov/BLAST/)搜索相似性序列,进行系统发育分析。采用ClustalX(Version 1.8)对序列进行比对分析,通过MEGA 5软件构建系统发育树(Kumaret al,2004),采用 Neighbor-joining建树方法,建树结果进行bootstrap1000次系统检验;利用 Phylip软件包中DNASIS程序计算距离矩阵,利用 DOTUR计算OTU(Schlosset al,2005)。引物 ITS1/ITS4、EF3/EF4、NS1/NS4扩增的部分序列在 GenBank核苷酸数据库中的接受号分别为 KM067396—KM067409、KM067410—KM067425、KM067426—KM067449。
2 结果与分析
2.1 三对引物的真核微型生物文库数据分析与对比
将测序返回的序列经拼接去载体后,三对引物ITS1/ITS4、EF3/EF4和NS1/NS4分别获得94、100和129个有效克隆子。通过 DOTUR软件将相似性大于97%的克隆子归类为同一个OTU,ITS1/ITS4序列属于35个OTU;EF3/EF4序列属于61个OTU;NS1/NS4序列属于80个OTU。三对引物克隆文库的多样性指数见表2 ,通过DOTUR软件制作稀释性曲线见图1。
文库分析结果表明:ITS1/ITS4、EF3/EF4、NS1/NS4三对引物克隆文库的Chao 1指数、ACE丰富度指数和 Shannon-Weiner指数依次递增,而Simpson优势度指数则依次递减,4个指数都说明三个文库的多样性以 NS1/NS4最高,EF3/EF4其次,ITS1/ITS4最后。从稀释性曲线上可以看出,多样性由高到低排列依次为NS1/NS4、EF3/EF4、ITS1/ITS4,与多样性指数分析结果相一致;随着克隆抽样次数的增加,稀释性曲线的斜率都有一定程度的变小,三对引物克隆文库的 OTU类型都有一定程度的饱和,但ITS1/ITS4曲线相较于NS1/NS4和EF3/EF,明显趋于平坦,说明用于ITS1/ITS4多样性分析的取样数量要明显少于其它两对引物,相对较少的克隆子可以反映多数微型真核生物的物种信息。

表2 东海陆架表层沉积物真核微型生物多样性指数Tab.2 The microeukaryotic diversity from the surface layer sediments of the East China Sea

图1 三个文库的稀释性曲线Fig.1 Rarefaction curves of three clone librares
2.2 三对引物的克隆文库OTUs物种类群总述
应用 BLASTN搜索相似性序列,显示东海陆架DH13站点176个OTU可分为4界24个类群,如图2所示。其中原生生物界(Protist,125个OTU)具有显著的优势种群地位(占71%),包含丝足虫门(Cercozoa,64个OTU)、横裂甲藻纲(Dinophyceae,25个OTU)、不等鞭毛门(Stramenopiles,13个 OTU)、纤毛虫门(Ciliophora,11个 OTU)、无根虫门(Apusozoa,5个OTU)、顶复门(Apicomplexa,3个OTU)、囊泡虫总门(Alveolata,2个OTU)、隐藻门(Cryptophyta,1个OTU)和眼虫门(Euglenozoa,1个OTU)等9个类群。真菌界(Fungi)共有 35个 OTU(占 19.9%),包含子囊菌门(Ascomycota,13个 OTU)、壶菌门(Chytridiomycota,11个OTU)、真菌类(Fungi environmental samples,8个OTU)、毛霉亚门(Mucoromycotina,2个OTU)和担子菌门(Basidiomycota,1个OTU)等5个类群。动物界(Metazoa,13个 OTU,占 7.4%)包含了环节动物门(Annelida,4个OTU)、腕足动物门(Brachiopoda,3个OTU)、扁形动物门(Platyhelminthes,1个OTU)、线虫动物门(Nematoda,1个OTU)、软体动物门(Mollusca,1个OTU)、轮形动物门(Rotifera,1个OTU)和刺胞动物门(Cnidaria,1个OTU)等8个类群。丰度最少的植物界(Plantae,3个 OTU,占 1.7%)则是包含绿藻门(Chlorophyta,1个OTU)和维管植物门(Tracheophyta,1个OTU)2个类群。
2.3 三对引物的克隆文库OTUs物种类群比较
将三对引物ITS1/ITS4、EF3/EF4和NS1/NS4所获得的克隆文库(OTU分型)结果,以“门”为基准,比较三个文库 OTU的物种多样性,分类标准以 NCBI Taxonomy上的分类为基础,结果见图3、图4、图5。
综合分析图3、图4、图5结果表明:三对引物的克隆文库OTUs都至少包含了原生生物界(Protist)、真菌界(Fungi)、植物界(Plantae)三大界,且原生生物界(Protist)占据了很大的比重,均超过了 50%,而特异的是 NS1/NS4同时还有相当多类群的动物界(Metazoa)OTUs。
ITS1/ITS4克隆文库分为4界11个类群(见图3),其中主要为原生生物界(57.1%)和真菌界(34.3%)。原生生物界涵盖了:横裂甲藻纲(Dinophyceae,9个OTU),纤毛虫门(Ciliophora,7个 OTU),无根虫门(Apusozoa,1个OTU),丝足虫门(Cercozoa,1个OTU),不等鞭毛门(Stramenopiles,1个 OTU)以及囊泡虫总门(1个 OTU)。真菌界分为 3类群,子囊菌门(Ascomycota,10个OTU)含量最高,另有少量担子菌门(Basidiomycota,1个 OTU)和真菌类(Fungi,2个OTU)。除此之外,动物界的刺胞动物门(Cnidaria,1个 OTU)和植物界的绿藻门(Chlorophyta,1个 OTU)也有被检测到类似序列的存在。
EF3/EF4克隆文库分为3界6个类群(见图4),同样以原生生物界(70.5%)和真菌界(27.9%)为主。原生生物界均为丝足虫门(Cercozoa,43个OTU)。真菌界分为4个类群,以壶菌门(Chytridiomycota,7个OTU)和真菌类(Fungi,5个 OTU)为主,另有子囊菌门(Ascomycota,3个OTU)和毛霉亚门(Mucoromycotina,2个 OTU)。植物界则是唯一的维管植物门(Tracheophyta,1个OTU)。

图2 DH13站点中176个OTU的物种分类示意图Fig.2 Species classification for 176 OTUs from station DH-13

图3 ITS1/ITS4引物克隆文库的OTU类群比例Fig.3 Ratio of OTU groups from clone library of primer pairs ITS1/ITS4
NS1/NS4克隆文库展现了最高的多样性,共含4界 19个类群(见图5),原生生物界(占 77.5%)仍是最主要的优势种群,其中丝足虫门(Cercozoa,20个OTU)、横裂甲藻纲(Dinophyceae,16个OTU)和不等鞭毛门(Stramenopiles,12个 OTU)为主要的三个门,此外还有眼虫门(Euglenozoa,1个 OTU)、隐藻门(Cryptophyta,1个 OTU)、无根虫门(Apusozoa,4个OTU)、纤毛虫门(Ciliophora,4个 OTU)、顶复门(Apicomplexa,3个 OTU)以及囊泡虫总门(Alveolata environmental samples,1个OTU)。真菌界(占6.25%)以壶菌门(Chytridiomycota,4个OTU)为主。与其它两对引物克隆文库不同的是,NS1/NS4中动物界(15%)占到了第二位,且多样性较高,具有 7个类群:环节动物门(Annelida,4个 OTU)、扁形动物门(Platyhelminthes,1个OTU)、线虫动物门(Nematoda,1个OTU)、腕足动物门(Brachiopoda,3个OTU)、软体动物门(Mollusca,1个OTU)、轮形动物门(Rotifera,1个OTU)和刺胞动物门(Cnidaria,1个OTU)。植物界则以绿藻门(Chlorophyta,1个OTU)为代表。

图4 EF3/EF4引物克隆文库的OTU类群比例Fig.4 Ratio of OTU groups from clone library of primer pairs EF3/EF4

图5 NS1/NS4引物克隆文库OTU类群比例图Fig.5 Ratio of OTU groups from clone library of primer pairs NS1/NS4
2.4 三对引物的克隆文库系统发育学分析
2.4.1 三对引物的真菌克隆文库系统发育分析分别从三对引物克隆文库中选取代表性OTUs构建真菌系统发育树,结果见图6、图7、图8所示。
ITS1/ITS4克隆文库构建的真菌系统发育树主要含子囊菌门(Ascomycota)和担子菌门(Basidiomycota)2个类群(图6)。子囊菌门(Ascomycota)中的代表性基因型 DH132F36-A与一株来自于云南水稻根性寄生的未培养克隆子 R3-6有高同源性(99%),基因型DH132F27、DH132F14与地毯草(Axonopus compressus)根圈中分离的未培养土壤真菌克隆子 FITS_HRP1_W16具有高同源性(99%),基因型DH132F17-c与用于分解芒草和甘蔗细胞壁的Phaeosphaeriopsissp.TMS-2011有高同源性(99%),基因型 DH134F09与白黄笋顶孢霉(Acrostalagmus luteoalbusisolate L03)有高同源性(99%),此类霉菌同样在深海环境中被发现,并用于细胞毒素的研究(Wanget al,2012)。担子菌门(Basidiomycota)中的代表性基因型 DH132F30-B与夏威夷海域的银耳目(Tremellales)LM477有较高同源性(95%)。
EF3/EF4克隆文库构建的真菌系统发育树中主要类群为壶菌门(Chytridiomycota)和子囊菌门(Ascomycota)2个类群(图7)。壶菌门(Chytridiomycota)中的基因型DH133F27与法国湖泊Pavin和Aydat中的未培养壶菌 PA2009B6有较高同源性(95%),基因型DH131F01-a与红根囊壶菌(Rhizophlyctis roseaJEL 318)有 93%的同源性,基因型 DH134F60、DH134F06、DH134F11与喜马拉雅山脉、落基山脉的未培养壶菌克隆子有 90%的同源性,先前的研究显示高海拔土壤真菌中壶菌占主导地位(Freemanet al,2009)。子囊菌门(Ascomycota)中的基因型DHE134-1与海洋中的二尖梅奇酵母(Metschnikowia bicuspidate,DQ520881)有高同源性(99%),该酵母被认为是三疣梭子蟹乳化病的致病菌(Wanget al,2007)。此外基因型DHE132-10与一株未培养Fungus clone nco32b11c1有高同源性(99%)。
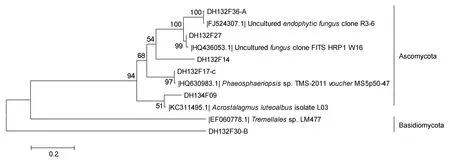
图6 根据ITS1/ITS4克隆文库构建的真菌系统发育树Fig.6 Phylogenetic tree of Fungi according to clone library of primer pairs ITS1/ITS4
NS1/NS4克隆文库构建的真菌系统发育树中基因型都为壶菌门(Chytridiomycota),如图8所示。基因型 DH1331F43与海洋藻类病原体菌株-壶菌polysiphoniae有 93%的同源性,基因型 DH1321F31与 EF3/EF4中的部分基因型(DH134F60,DH134F06,DH134F11)相同,与喜马拉雅山脉、落基山脉的未培养壶菌克隆有较高同源性(94%)。
2.4.2 三对引物的原生生物克隆文库系统发育分析分别从三对引物克隆文库中选取代表性OTUs构建原生生物系统发育树,结果见图9、图10、图11所示。
ITS1/ITS4克隆文库构建的原生生物系统发育树主要分为囊泡虫总门(Alveolata)、不等鞭毛门(Stramenopiles)、无根虫门(Apusozoa)和丝足虫门(Cercozoa)四大类。而囊泡虫总门(Alveolata)中的纤毛虫门(Ciliophora)和双鞭毛虫门(Dinoflagellata)下的横裂甲藻纲(Dinophyceae),两者占到了ITS1/ITS4克隆文库原生生物界的绝大多数(图9)。纤毛虫门(Ciliophora)基因型 DH133-5和海洋拟急游虫(Strombidinopsis Sopsis99-1)有着高相似度(98%),基因型DH131-1和急游虫属(StrombidiumLLK-2005)有着较高相似度(94%),而基因型DH131-2则与中国大陆海洋鱼类中分离到的刺激隐核虫(Cryptocaryon irritans)相似,但相似度不高(85%)。横裂甲藻纲(Dinophyceae)基因型 DH134-12与异帽藻(Heterocapsa)相似(89%),基因型DH133-31与裸甲藻(Gymnodinium beii)高度相似(99%)。基因型DH131-23与 MAST-4类群的未培养不等鞭毛门克隆子 M_IND70.7有着较高相似度(94%),MAST-4被认为是一个 低 进 化 多 样 性 的 类 群(Rodríguez-Martínezet al,2012)。无根虫门(Apusozoa)基因型DH131-7与海洋中分离的Apusomonas proboscidea相似,但相似度不高(87%)。丝足虫门(Cercozoa)基因型DH132F19与海洋丝足虫类Thaumatomastix克隆子有着较高相似度(93%)。

图7 根据EF3/EF4克隆文库构建的真菌系统发育树Fig.7 Phylogenetic tree of Fungi according to clone library amplificated with primer pairs EF3/EF4

图8 根据NS1/NS4克隆文库构建的真菌系统发育树Fig.8 Phylogenetic tree of Fungi according to clone library amplificated with primer pairs NS1/NS4
EF3/EF4克隆文库构建的原生生物系统发育树显示均为丝足虫门(Cercozoa),这与 ITS1/ITS4和NS1/NS4显示出的原生生物多样性显著不同,如图10所示。基因型DH131F02-a与未培养丝足虫类4-E2相似(95%),DH131F03与未培养丝足虫类3b-F5相似(95%),两个相似序列均来自于波罗的海的海雪(sea snow)中。基因型 DH132F33-a和 DH133F22分别与来自加拿大不列颠哥伦比亚省海洋底栖生物栖息地中的丝足虫类CC-2009c和CC-2009e相似,相似度为96%和94%。基因型DH131F04-a与广泛分布于国内外沿海浮游生物群落中的吞噬鞭毛虫(Ebria tripartiteisolate 2)有94%的相似度。基因型DH133F46和基因型 DH132F15-a分别与海洋丝足虫类的CryothecomonasAPCC MC5-1Cryo和ThaumatomastixCC002有较高相似度,相似度为97%和96%。
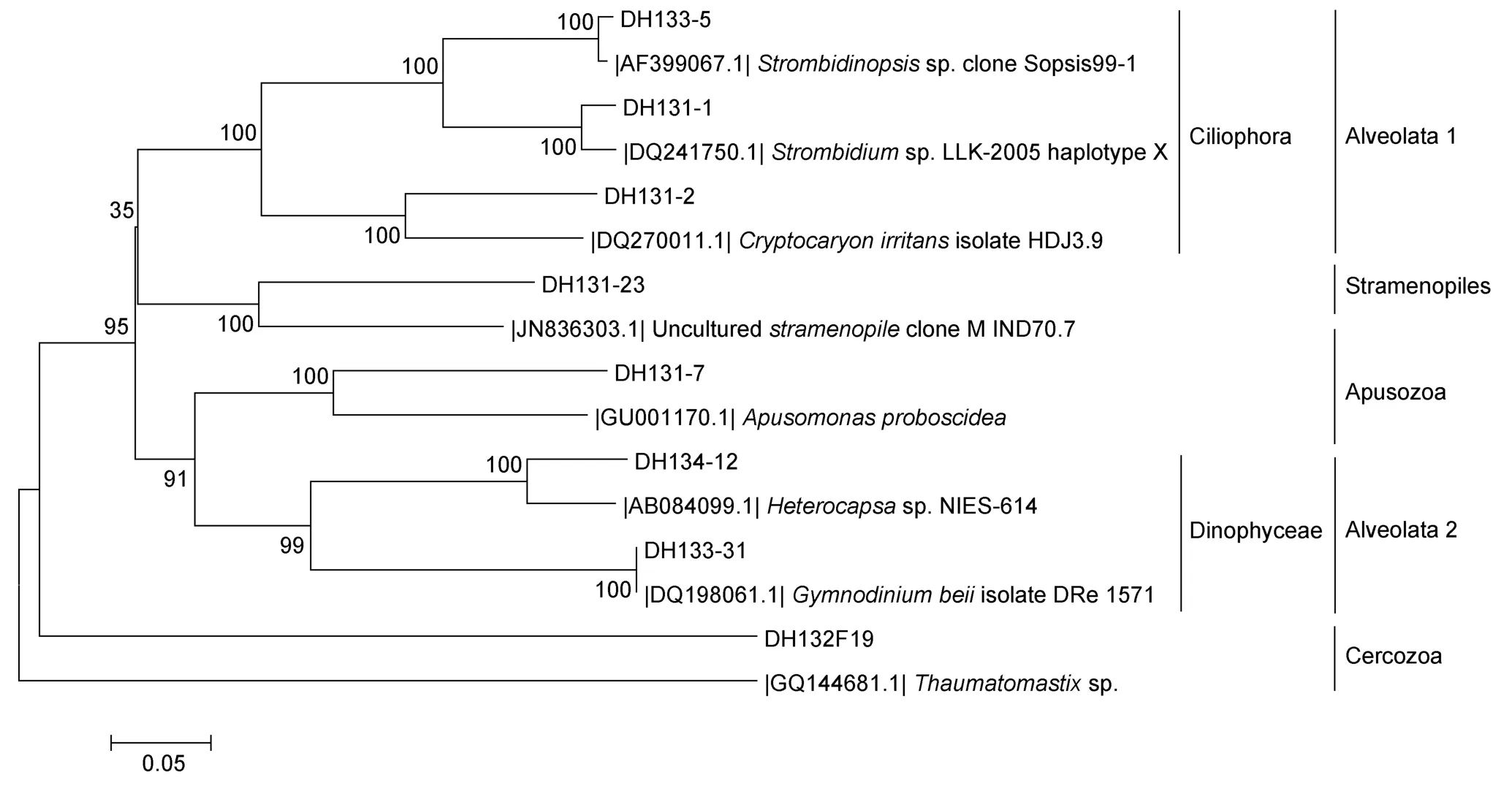
图9 根据ITS1/ITS4克隆文库构建的原生生物系统发育树Fig.9 Phylogenetic tree of Protist according to clone library amplificated with primer pairs ITS1/ITS4

图10 根据EF3/EF4克隆文库构建的原生生物系统发育树Fig.10 Phylogenetic tree of Protist according to clone library amplificated with primer pairs EF3/EF4
NS1/NS4克隆文库表现出了丰富的原生生物多样性,选取其中4个主要类群囊泡虫总门(Alveolata)、不等鞭毛门(Stramenopiles)、丝足虫总门(Cercozoa)和无根虫门(Apusozoa)的代表性OTUs构建系统发育树(图11)。

图11 根据NS1/NS4克隆文库构建的原生生物系统发育树Fig.11 Phylogenetic tree of Protist according to clone library amplificated with primer pairs NS1/NS4
囊泡虫总门(Alveolata)分为纤毛虫门(Ciliophora)和双鞭毛虫门(Dinoflagellata)下的横裂甲藻纲(Dinophyceae)两个类群,与 ITS1/ITS4相同,纤毛虫门(Ciliophora)基因型 DH1311F40与韩国西部海岸的Strombidinopsis jeokjoSBNS0310HJ有高度同源性(99%),基因型 DH132F32与黄海的Paradiscoce-phalus elongatus克隆子有高相似度(98%)。横裂甲藻纲(Dinophyceae)基因型 DH1331F18与海洋环境中的Euduboscquella克隆子有较高相似度(95%),基因型DH1311F04与Gonyaulax spinifera克隆子有较高相似度(95%),基因型DHY1与Alexandrium satoanum克隆子有高同源性(99%),基因型 DH1331F38与贫氧海洋环境中的未培养双鞭毛虫 CCW105有 90%的相似度。基因型DH132F81与韩国西部海岸的Woloszynskia cincta克隆子有高同源性(99%),基因型DH1331F29与Lepidodinium chlorophorum克隆子有较高相似度(96%),基因型 DH1341F07与异帽藻(HeterocapsaHE04)有高相似度(99%),基因型 DHR3与Gyrodinium fusiforme克隆子有较高相似度(94%)。
不等鞭毛门(Stramenopiles)有代表性基因型OTU6个,其中基因型 DH1324B08与一株为未识别的类似Haliphthoros克隆子NJM 0034相似度为97%;基因型 DH1311F11与北太平洋海域的未培养不等鞭毛类 897st19-43有高相似度(99%);基因型DH1331F06与来自于红海东北海岸一个贫营养站点的未培养不等鞭毛类 RS.12f.10m.367有较高相似度(95%),该站点的海洋不等鞭毛门(Marine Stramenopiles)类群多样性丰富,是第二大类群(OTUs 16.8%)(Acostaet al,2013);基因型1331F46与南极洲海域的Thalassiosira CCMP982有高相似度(99%);基因型1311F14与美国纽约长岛海域沉积物的一个未培养LabyrinthulidPJS101305_35有98%的相似度;基因型1311F38与AplanochytriumTs1有高相似度(99%)。
丝足虫门(Cercozoa)与 EF3/EF4非常相似,基因型DH1341F20和基因型DH132F92分别与海洋丝足虫类的CryothecomonasAPCC MC5-1Cryo和ThaumatomastixCC002相似,相似度为99%和94%,基因型DH1341F23的相似序列来自于加拿大不列颠哥伦比亚省海洋底栖生物栖息地中的丝足虫类克隆子,相似度为98%,基因型DH1321F11与波罗的海海雪中的未培养丝足虫类 CC-2009b,相似度为98%。
无根虫门(Apusozoa)基因型 DH1331F03与AmastigomonasJJP-2003有较高同源性(96%),基因型DH132F05与Apusomonas proboscideaHFCC47有91%的相似度。
3 讨论
3.1 真核微型生物多样性研究中的引物选择
随着分子生物学的不断发展,越来越多的引物被设计应用于真核微型生物分子生态学研究,除了本文使用的三对引物外,Euk1A/Euk516r(鲍磊等,2008)、nu-SSU-0817/nu-SSU-1196 or 1536(Bornemanet al,2000)、18S-42F or 82F/18S-1498R or 1520R(Takishitaet al,2006)、Euk-A/Euk-B(Koidet al,2012)等也同样被应用于真核微型生物的研究。
本文使用的三对引物最初被设计用于子囊菌、小麦根系等陆地真核微型生物多样性的研究(Whiteet al,1990;Bernieret al,1994;Smitet al,1999),后来也被用于海洋环境真核微型生物研究,如 Lai等(2007)应用ITS1/ITS4引物对南海深海甲烷水合沉积物中真菌群落结构进行了初步研究,扩增得到的大多数真菌可归类为Ascomycota和Basidiomycota;Gao等(2008)应用ITS1/ITS4、EF3/EF4等8对引物扩增夏威夷海绵共附生真菌;Nagano等(2010)应用 ITS1/ITS4引物扩增到了深海沉积物中新的真菌类群;高远等(2012)应用 ITS1/ITS4对鄂霍次克海冷泉沉积物真核微型生物多样性进行研究;Singh等(2012)分别使用NS1/NS4和ITS1/ITS4对深海沉积物中可培养和不可培养两种形式的真菌进行了扩增。
但上述多数研究仅使用单一引物对环境中真核微型生物进行分析,研究者们也逐渐发现仅用单一引物来研究复杂环境样本中的真核微型生物多样性存在着偏倚和局限性,如Smit等(1999)在分析了小麦根系样本后认为引物 EF3/EF4略微偏向于扩增担子菌和接合菌。因此采用多种引物进行比较分析的相关研究在近些年也逐渐被研究人员关注(Andersonet al,2003),但是对于海洋沉积环境的相关比较分析还未发现,本文研究结果对更深入地开展海洋沉积环境真核微型生物的多样性研究有着借鉴作用。
3.2 不同引物的扩增特异性及其多样性
本文三对引物扩增得到的克隆文库均包含了沉积物中真核微型生物的主要类群:原生生物(Protist)和真菌(Fungi),NS1/NS4克隆文库还独特的扩增到了一些动物界(Metazoa)类群。
原生生物类群在三对引物中比例都达到了 50%以上,ITS1/ITS4克隆文库与NS1/NS4克隆文库均包含了横裂甲藻纲(Dinophyceae)、纤毛虫门(Ciliophora)、丝足虫门(Cercozoa)、无根虫门(Apusozoa)和不等鞭毛门(Stramenopiles)5个类群,且横裂甲藻纲(Dinophyceae)在两个文库中均为优势类群(45%和 25.8%),此外NS1/NS4扩增到了独有的 3个类群(Apicomplexa,Cryptophyta,Euglenozoa),在三对引物对原生生物的扩增中展现了最高的多样性。但 EF3/EF4克隆文库中所有原生生物克隆子均为丝足虫门(Cercozoa),这可能是由于 EF3/EF4引物对海洋沉积物中原生生物类群扩增具有局限性。
真菌方面,三对引物扩增结果各异。ITS1/ITS4克隆文库主要是子囊菌门(Ascomycota)和担子菌门(Basidiomycota);EF3/EF4克隆文库是子囊菌门(Ascomycota)、壶菌门(Chytridiomycota)和毛霉亚门(Mucoromycotina);NS1/NS4克隆文库则基本都是壶菌门(Chytridiomycota)。这些类群与国际上沉积物真菌多样性研究中的主要类群基本一致(Laiet al,2007;Naganoet al,2010;Singhet al,2012)。值得注意的是,Smit等(1999)认为EF3/EF4引物在培养生物和环境样本的扩增中轻微偏向于扩增担子菌门(Basidiomycota)和接合菌门(Zygomycota),但这两个类群在东海陆架表层沉积物中都未被扩增到,这可能是由于小麦根系与海洋沉积物的环境差异性所导致的,而 Burgaud等(2013)应用EF3/EF4引物对美国特拉华河河口沉积物的扩增所得主要类群为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和壶菌门(Chytridiomycota),与本文结果类似。
3.3 引物选择对海洋原生生物多样性研究的影响
虽然三对引物在国际上的研究中多被用于真菌多样性的研究,但根据本文的数据结果显示,三对引物都扩增到了较多的原生生物类群,尤其是NS1/NS4引物,原生生物类群的多样性在三对引物中是最高的。Park等(2008)应用引物18S-82F/18S- 1520R扩增东海表层沉积物的研究中显示,90%的克隆序列为原生生物,主要类群为纤毛虫门(Ciliophora,18%)、双鞭毛虫门(Dinoflagellata,19%)、不等鞭毛门(Stramenopiles,11%)和丝足虫门(Cercozoa,20%),与本文三对引物扩增结果基本一致,且除领鞭毛虫门(Choanozoa)外,Park等(2008)文中所扩增得到的原生生物类群本文同样扩增得到。因此本文推断,ITS1/ITS4、NS1/NS4引物同样可应用于海洋沉积物原生生物多样性的研究中,作为其进一步的补充和延伸,而 EF3/EF4则由于其在丝足虫门(Cercozoa)扩增上的相对专一性,可能更适用于丝足虫门(Cercozoa)下不同种属多样性的研究。
4 结论
本文选择三对真核微型生物的通用引物(NS1/NS4,EF3/EF4,ITS1/ITS4),通过18S rDNA、ITS区的基因克隆和文库构建,对东海陆架DH-13站点沉积物真核微型生物类群进行分类学、多样性和系统发育分析,研究发现三对引物的选择不同对真核微型生物多样性的影响较显著,尤其是在原生生物的扩增上。本文的研究结果再次说明了对特异性PCR引物进行严格检测以适用于目的类群扩增的重要性和迫切性,同时也说明了单一引物扩增的局限性,通过多种引物综合使用来评估复杂环境中真核微型生物多样性是必要的,未来的研究和分析应更注重于综合性的处理方法。
王健鑫,许贤恩,周链链等,2012.东海陆架表层沉积物微生物多样性初步研究.海洋与湖沼,43(4):806—813
李家彪,2008.东海区域地质.北京:海洋出版社,1—105
汪 岷,江雪娇,2009.海洋微微型真核生物分子多样性的研究方法.中国海洋大学学报:自然科学版,39(4):627—632
宋志刚,许强芝,鲁心安等,2006.中国东海海洋微生物种群多样性初步研究.微生物学通报,33(1):63—67
张林宝,李铁刚,党宏月等,2010.东海内陆架泥质区沉积物古菌群落垂向分布特征.地球科学——中国地质大学学报,35(2):255—260
高 远,李富超,姜 鹏等,2012.鄂霍次克海冷泉沉积物真核生物多样性的初步研究.海洋科学,36(5):29
鲍 磊,陈纪新,黄邦钦等,2008.应用变性梯度凝胶电泳研究厦门西海域超微型真核浮游生物多样性.海洋环境科学,26(6):504—509
戴 欣,蔡创华,2001.海洋沉积物中特有细菌类群的初步探讨.中山大学学报:自然科学版,40(6):51—54
Acosta F,Ngugi D K,Stingl U,2013.Diversity of picoeukaryotes at an oligotrophic site off the Northeastern Red Sea Coast.Aquatic Biosystems,9(1):16
Anderson I C,Campbell C D,Prosser J I,2003.Potential bias of fungal 18S rDNA and internal transcribed spacer polymerase chain reaction primers for estimating fungal biodiversity in soil.Environmental Microbiology,5(1):36—47
Bernier L,Hamelin R C,Ouellette G B,1994.Comparison of ribosomal DNA length and restriction site polymorphisms in Gremmeniella and Ascocalyx isolates.Applied and Environmental Microbiology,60(4):1279—1286
Borneman J,Hartin R J,2000.PCR primers that amplify fungal rRNA genes from environmental samples.Applied and Environmental Microbiology,66(10):4356—4360
Burgaud G,Woehlke S,Rédou Vet al,2013.Deciphering the presence and activity of fungal communities in marine sediments using a model estuarine system.Aquatic Microbial Ecology,70:45—62
Freeman K R,Martin A P,Karki Det al,2009.Evidence that chytrids dominate fungal communities in high-elevation soils.Proceedings of the National Academy of Sciences,106(43):18315—18320
Gao Z,Li B,Zheng Cet al,2008.Molecular detection of fungal communities in the Hawaiian marine spongesSuberites zetekiandMycale armata.Applied and Environmental Microbiology,74(19):6091—6101
Köster M,Wardenga R,Blume M,2008.Microscale investigations of microbial communities in coastal surficial sediments.Marine Ecology,29(1):89—105
Koid A,Nelson W C,Mraz Aet al,2012.Comparative analysis of eukaryotic marine microbial assemblages from 18S rRNA gene and gene transcript clone libraries by using different methods of extraction.Applied and Environmental Microbiology,78(11):3958—3965
Kumar S,Tamura K,Nei M,2004.MEGA3:Integrated software for molecular evolutionary genetics analysis and sequence alignment.Briefings in Bioinformatics,5:150—163
Lai X,Cao L,Tan Het al,2007.Fungal communities from methane hydrate-bearing deep-sea marine sediments in South China Sea.The ISME Journal,1(8):756—762
Nagano Y,Nagahama T,Hatada Yet al,2010.Fungal diversity in deep-sea sediments——the presence of novel fungal groups.Fungal Ecology,3(4):316—325
Park S J,Park B J,Pham V Het al,2008.Microeukaryotic diversity in marine environments,an analysis of surface layer sediments from the East Sea.The Journal of Microbiology,46(3):244—249
Rodríguez F,Derelle E,Guillou Let al,2005.Ecotype diversity in the marine picoeukaryote Ostreococcus(Chlorophyta,Prasinophyceae).Environmental Microbiology,7:853—859
Rodríguez-Martínez R,Rocap G,Logares Ret al,2012.Low evolutionary diversification in a widespread and abundant uncultured protist(MAST-4).Molecular Biology and Evolution,29(5):1393—1406
Schloss P D,Handelsman J,2005.Introducing DOTUR,a computer program for defining operational taxonomic units and estimating species richness.Applied and Environmental Microbiology,71(3):1501—1506
Singh P,Raghukumar C,Meena R Met al,2012.Fungal diversity in deep-sea sediments revealed by culture-dependent and cultureindependent approaches.Fungal Ecology,5(5):543—553
Smit E,Leeflang P,Glandorf Bet al,1999.Analysis of fungal diversity in the wheat rhizosphere by sequencing of cloned PCR-amplified genes encoding 18S rRNA and temperature gradient gel electrophoresis.Applied and Environmental Microbiology,65(6):2614—2621
Takishita K,Tsuchiya M,Reimer J Det al,2006.Molecular evidence demonstrating the basidiomycetous fungusCryptococcus curvatusis the dominant microbial eukaryote in sediment at the Kuroshima Knoll methane seep.Extremophiles,10(2):165—169
Wang F Z,Huang Z,Shi X Fet al,2012.Cytotoxic indole diketopiperazines from the deep sea-derived fungusAcrostalagmus luteoalbusSCSIO F457.Bioorganic &Medicinal Chemistry Letters,22(23):7265—7267
Wang X,Chi Z,Yue Let al,2007.A marine killer yeast against the pathogenic yeast strain in crab(Portunus trituberculatus)and an optimization of the toxin production.Microbiological Research,162(1):77—85
White T J,Bruns T,Lee Set al,1990.Amplification and Direct Sequencing of Fungal Ribosomal RNA Genes for Phylogenetics.PCR Protocols:a Guide to Methods and Applications.Academic Press,New York,18:315—322
Whitman W B,Coleman D C,Wiebe W J,1998.Prokaryotes:The unseen majority.Proceedings of the National Academy of Sciences,USA,95:6578—6583
Zeng Y H,Li H Y,Jiao N Z,2007.Phylogenetic diversity of planktonic archaea in the estuarine region of East China Sea.Microbiological Research,162:26—36

