牛支原体分离株全基因组序列测定及初步分析
王艳杰,李平安,吴太平,关平原*
(1.内蒙古农业大学兽医学院农业部动物临床诊疗技术重点实验室,内蒙古呼和浩特010018;2.内蒙古农业大学食品科学与工程学院,内蒙古呼和浩特010018)
牛支原体于1961年首次由Hale等从美国的患乳腺炎的牛乳中分离到,1976年被确定为引起牛呼吸系统疾病的病原之一,其引起的临床症状主要表现为乳腺炎、关节炎、肺炎、角膜炎、不孕症甚至引起中耳炎[1]。牛支原体对牛的危害主要在于引起牛支原体相关性疾病(Mycoplasma bovis association disease,MbAD),即牛支原体感染后继发感染巴氏杆菌等自然界常在菌往往会造成巨大的经济损失。在欧洲,由牛呼吸道疾病引起的经济损失每年都高达57 600万欧元,而其中大约有1/4的经济损失与牛感染支原体有关。在美国,每年由于牛支原体感染而引起的经济损失高达3 200万美元,并且牛支原体感染已经成为欧洲和北美洲地区牛发病和死亡的重要原因之一[2]。2008年我国首次报道从患肺炎的犊牛病例中分离得到牛支原体,随后不断有牛支原体被分离鉴定的报道,因而牛支原体引起的疾病未来可能会成为我国养牛业的重大隐患。
牛支原体是柔膜体纲支原体目支原体科支原体属的一员,形成典型菌落形态为煎蛋样,无细胞壁,G+C%含量低,仅由3层膜构成,形态多样,对外界环境抵抗力低,但其却广泛存在于自然界。有学者认为,这可能与其可变表面脂蛋白家族有关。可变表面脂蛋白家族是指一类大小和抗原表位具有高频变异性的蛋白,它造成牛支原体表型的多样性,从而逃避宿主的免疫应答[3]。牛支原体引起疾病的临床症状与丝状支原体丝状亚种引起的疾病即牛肺疫的症状非常相似。抗生素的大量使用导致疾病的非典型症状出现,使得牛支原体与丝状支原体丝状亚种的鉴别诊断越来越困难,牛支原体与无乳支原体16 S rRNA序列的同源性高,利用扩增16SrRNA目的片段也很难鉴别两者,这阻碍了牛支原体临床病例的诊断[4]。我国2011年和2012年分别完成了Hubei和HB0801两株牛支原体的测序,对Hubei分离株与牛支原体PG45株比较发现,两者可能具有不同的执行基因水平转移的能力,HB0801株分离自有肺脏损伤的牛,并且牛体内试验证实其与牛支原体PG45具有相同的毒力,其具有惊人的基因组可塑性[5-6]。
牛支原体NM2012菌株分离自消瘦,胸部皮下水肿,四肢腕关节、膝关节和跗关节明显肿大的犊牛,处死后采集关节液病料分离菌株,经形态学观察、生化鉴定以及特异性PCR鉴定为牛支原体,命名为NM2012菌株。
1 材料与方法
1.1 材料
1.1.1 菌株来源 分离自患疑似支原体病犊牛的关节液病料,由内蒙古农业大学兽医学院动物传染病学实验室分离、鉴定、保存并命名为牛支原体NM2012菌株。
1.1.2 培养基 PPLO (21g/L)121 ℃高压灭菌20min,由马血清(200mL/L)、250g/L 的酵母粉(100mL/L)、10 单 位 青 霉 素 (10g/L)组 成,pH 7.6,若配制固体培养基,则加12g/L的琼脂。
1.1.3 试剂及仪器设备 PPLO为BD公司产品;酵母粉为OXOID公司产品;马血清为Gibco公司产品;琼脂为BIOSHARP公司产品;苯酚、氯仿、异戊醇、3mol/L乙酸钠、无水乙醇、RNaseA、蛋白酶 K、TE缓冲液(pH8.0)、ddH2O、琼脂糖、6×Loading buffer、DNA标准DL 15 000、青霉素为宝生物工程(大连)有限公司产品。
酶标仪(SynergyH4)、紫外凝胶成像系统(G∶BOX)、水浴振荡器(HZS-H)、低温高速离心机(Multifuge X3R)等,由内蒙古农业大学动物传染病实验室提供。
1.2 方法
1.2.1 菌株基因组提取 将支原体NM2012菌株在液体培养基中大量培养,做3个平行分别命名为1号、2号和3号样品,参照郝瑞霞[7]的方法进行全基因组DNA提取。
1.2.2 提取全基因组DNA质量检测
1.2.2.1 琼脂糖凝胶电泳检测提取的DNA的大小 配制6g/L的琼脂糖凝胶,将提取的基因组DNA 120V,电泳30min,上样量为6μL的DNA样品与2μL的6×Loading的混合液,DNA标准DL 15 000上样量为5μL。
1.2.2.2 酶标仪检测DNA浓度 按酶标仪操作说明书进行样品DNA浓度的检测,用2μL的ddH2O校准,上样量为2μL。
1.2.3 基因公司对样品合格性检测方法
1.2.3.1 利用 Qubit Fluorometer检测样品浓度采用DNA BR检测试剂盒检测样品浓度,上样量为1μL。
1.2.3.2 利用琼脂糖凝胶电泳检测样品完整性配制6g/L的琼脂糖凝胶,120V,40min进行电泳,上样量分别为 M1(DNA标准 DL 23 130)3μL,1号样品 0.21μL,2 号 样 品 0.44μL,3 号 样 品1.44μL,M2(DNA 标准 DL 2 000)6μL。
1.2.4 测序及注释方法 采用Illumina Hiseq 2 000平台进行全基因组测序及注释,具体步骤及方法为:①采用超声法Covaris将大片段DNA随机打断并产生主带小于等于800bp的一系列DNA片段;②用 T4DNA Polymerase、Klenow DNA Polymerase和T4PNK将打断形成的粘性末端修复成平末端,再通过3′端加碱基“A”,使DNA片段能与3′端带有“T”碱基的特殊接头连接,用电泳法选择需回收的目的片段连接产物,再使用PCR技术扩增两端带有接头的DNA片段;③用合格的文库进行cluster制备和测序,测序后数据首先进行数据过滤获得Clean Data,采用SOAPdenovo 2.04软件对Clean Data进行组装,依据K-mer分析以及GC-depth分析对组装结果进行评价后进行基因组组分分析:①串联重复分析;串联重复序列及小卫星序列分析、微卫星序列分析;②非编码RNA分析;rRNA、tRNA及sRNA分析;③基因预测及基因组功能分析,主要进行常规基因注释,使用GO、KEGG和Swiss-Prot,以及NR和COG数据库对基因预测获得的ORF进行功能注释。
2 结果
2.1 提取全基因组DNA质量检测
2.1.1 琼脂糖凝胶电泳检测提取的基因组DNA大小 如图1所示,3份样品的主条带都大于15 000bp,不存在拖尾现象。
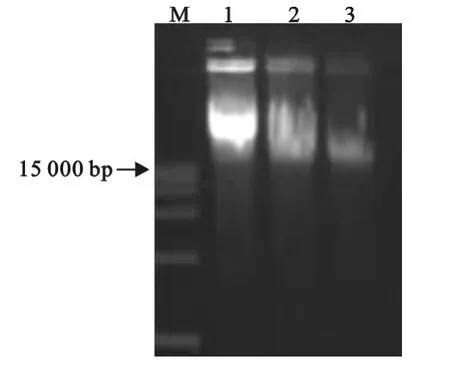
图1 DNA长度检测Fig.1 The DNA length detection
2.1.2 DNA 浓度检测 1号样品浓度为3 689.34ng/μL,OD 260/OD 280=2.059;2号样品浓度为2 977.25ng/μL,OD 260/OD 280=2.006;3号样品浓度为987.78ng/μL,OD 260/OD 280=2.012。
2.2 基因公司对样品合格性检测结果
2.2.1 利用Qubit Fluorometer检测样品浓度 1号样品检测浓度为476ng/μL,2号样品检测浓度为229ng/μL,3号样品检测浓度为69.4ng/μL。
2.2.2 利用琼脂糖凝胶电泳检测样品完整性 如图2所示,3个样品主条带都在23 130bp,无拖尾现象,样品符合全基因组测序要求。
2.3 测序及注释结果
牛支原体NM2012菌株基因组测序选择2号样品,其K-mer频率分布中显示其预测基因组大小为1.02Mb,错误率为0.000 7,错误率非常低(图3),实际其基因组大小为993 483bp。GC-depth分析呈泊松分布,G+C%不存在偏移,G+C%实际含量为29.26%(图4)。牛支原体NM2012菌株组装共产生3个scaffolds,24个contigs。基因组组分分析后发现,牛支原体NM2012菌株的基因组含有885个基因,总长度为834 042bp,平均长度942bp,占基因组全长的83.95%。串联重复序列共79个,总长为9 203bp,占基因组全长的0.926 3%,小卫星序列53个,微卫星序列5个。tRNA 34个,rRNA 4个。COG数据库分析显示与数据库基因匹配最高的基因是J类(翻译、核糖体结果及生物合成)和L类(复制、重组和修复),匹配最低的基因是Q类(脂质转运及代谢)和Q类(二级代谢产物的生物合成、转运及分解代谢)(表1)。
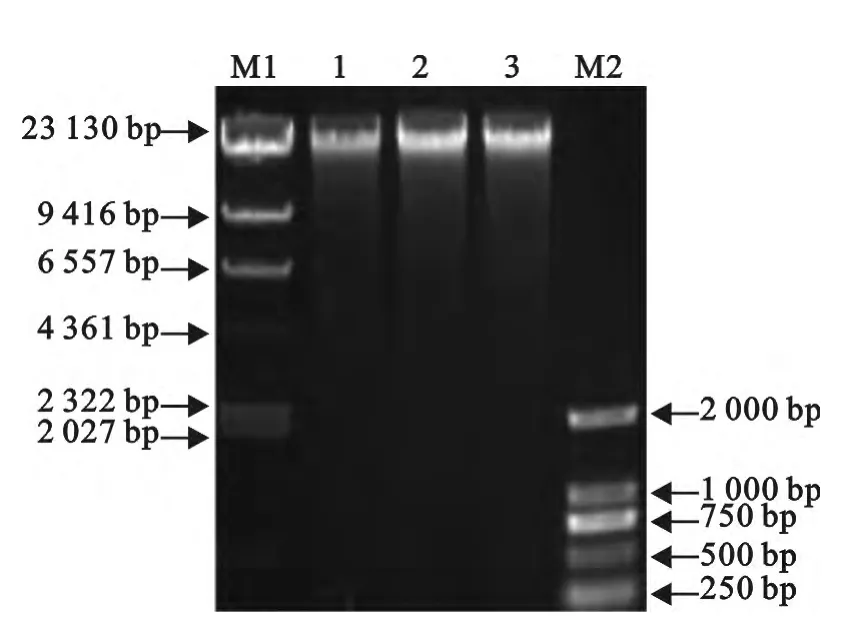
图2 样品完整性检测结果Fig.2 Sample integrity detection

图3 K-mer频率分布结果Fig.3 The results of K-mer frequency distribution

图4 GC-depth分析Fig.4 Analysis of GC-depth

表1 COG数据库分析结果Table 1 Analysis results of COG data
2.4 牛支原体NM2012株16SrRNA序列及与致牛羊病支原体属主要菌株的进化树分析
牛支原体NM2012株16SrRNA序列与致牛羊病支原体属主要菌株16SrRNA序列进行进化树分析见图5。牛支原体NM2012菌株16SrRNA序列如下:


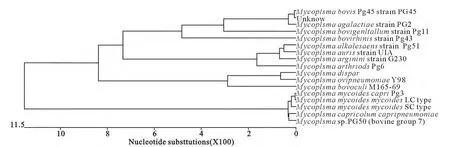
图5 牛支原体NM2012株16SrRNA序列及与致牛羊病支原体属主要菌株的进化树分析Fig.5 Phylogenetic tree analysis with 16SrRNA sequence from Mycoplasma bovis NM2012 strains and Mycoplasma mainly infecting cattle and sheep
从图5中可见牛鼻支原体PG43、牛生殖道支原体PG11、无乳支原体PG2、牛支原体PG45及牛支原体NM2012菌株牛支原体位于同一支,相比于其他2种菌株而言,牛支原体NM2012菌株与牛支原体PG45和无乳支原体PG2关系更近,但是牛支原体NM2012菌株与牛支原体PG45更相近,在同一分支,所以我们进一步确定牛支原体NM2012菌株为牛支原体。
3 讨论
本试验所获3个样品中,选择2号样品进行后续试验,因本试验提取基因组DNA采用的是最原始的酚-氯仿提取法,DNA样品中会存在一定的蛋白污染,从图1可见1泳道和2泳道相比,1泳道在点样孔中存在的杂质要比2泳道多,在1、2号样品浓度都符合条件的情况下(构建两个文库要求样品浓度为133ng/μL),我们选择2号样品以减少杂质对后续试验的影响;酶标仪与Qubit Fluorometer检测3份样品浓度差异大的原因是Qubit Fluorometer用于检测双链DNA,其特异性相对高于酶标仪;从测序的K-mer频率分布图可见其预测的基因组大小为1.02Mb与最终组装结果差异不大,说明组装结果非常好。牛支原体是柔膜体纲支原体目支原体属的成员,支原体属以无细胞壁,G+C%含量低及油煎蛋样菌落形态为特征[8],牛支原体NM2012菌株G+C%含量仅为29.26%,符合支原体属的特征之一。通过基因组COG数据库注释可见其复制、转录和翻译及修复相关的基因占据绝大部分,这可能与支原体属基因组小有关,例如生殖道支原体就被作为生物所需最小核心基因被研究[9]。牛支原体基因组小,其绝大部分基因用于最基本的复制、转录及翻译过程,而用于合成其所需营养物质的基因就少,从而其对宿主的依附性非常强,它的体外分离培养除基础营养物质以外,还必须添加额外的必须的营养物质才能生长,例如常添加200mL/L马血清来供给其所需的胆固醇,因此其培养非常困难[10]。牛支原体NM2012菌株的测序结果与牛支原体标准株PG45全基因序列相比,都含有34个tRNA,G+C%含量非常相似分别为29.25%和29.3%,这可能因两者都为牛支原体,其序列的保守性所致,但是牛支原体NM2012菌株与标准株PG 45相比少了2个sRNA,其仅含有4个s RNA,分别为1个16sRNA、1个23sRNA和2个5sRNA,这可能因牛支原体非保守序列所致,也可能是在原始数据处理过程中将小的测序片段过滤掉的原因导致的。
通过对牛支原体NM2012菌株16SrRNA序列与致牛羊病支原体属主要菌株16SrRNA序列的比较分析发现,牛支原体NM2012菌株与牛鼻支原体PG43、牛生殖道支原体PG11、无乳支原体PG2、牛支原体PG45位于同一支,而与丝状支原体的成员距离较远,牛支原体NM2012菌株虽然与牛鼻支原体PG43、牛生殖道支原体PG11位于同一支,但相对于他们而言,牛支原体NM2012菌株与无乳支原体和牛支原体关系更近,并且与牛支原体在同一支,进一步确定牛支原体NM2012菌株为牛支原体,该进化树的构建进一步证实牛支原体和无乳支原体16SrRNA序列的同源性高,所以oppD/F基因和uvrC基因被用于PCR扩增以鉴定牛支原体和无乳支原体,可变表面脂蛋白家族被研究其可通过序列的插入缺失甚至倒置而产生不同的后代,因每个可变表面脂蛋白的序列上游都含有2个保守的序列片段,序列1位于起始密码子上游,大约71bp,序列2位于序列1的上游,并被认为是可变表面脂蛋白家族基因的启动子,通过其可调控部分可变表面脂蛋白基因沉默[11]。可变表面脂蛋白家族的存在被认为是牛支原体逃避宿主免疫系统的机制,另有学者认为烯醇化酶可能也与牛支原体的致病性相关,本研究将为后期进一步研究牛支原体提供依据。
[1]张 瑞,崔 朋,巴晓亮,等.牛支原体临床分离株牛体毒力和免疫原性比较[J].动物医学进展,2013,34(6):126-132.
[2]郭 磊,何生虎,邵 倩,等.奶牛支原体病的诊断[J].动物医学进展,2012,33(12):200-204.
[3]Wise K S,Calcutt M J,Foecking M F,et al.Complete genome sequence of Mycoplasma bovis type strain PG45(ATCC 25523)[J].Infection and Immunity,2011,79(2):982-983.
[4]伍晓红,储岳峰,张 轩,等.牛支原体的分离鉴定及16S rRNA基因序列分析[J].动物医学进展,2012,33(12):35-37.
[5]Li Y,Zheng H J,Liu Y,et al.The complete genome sequence of Mycoplasma bovis strain Hubei-1[J].PLoS One,2011,6(6):e20999.
[6]Qi J J,Guo A Z,Cui P,et al.Comparative geno-plasticity analysis of Mycoplasma bovis HB0801 (Chinese isolate)[J].PLoS ONE,2012,7(5):e38239.
[7]郝瑞霞.绵羊肺炎支原体的分离鉴定及全基因组序列的测序与分析[D].内蒙古呼和浩特:内蒙古农业大学,2012.
[8]Soehnlen M K.Molecular characterization and epidemiology of Mycoplasma bovis[D].Department of Veterinary and Biomedical Sciences,Pennsylvania State University,2011,ProQuest LLC.
[9]Huang C H,Hsiang T,Trevors J T.Comparative bacterial genomics:defining the minimal core genome[J].Antonie van Leeuwenhoek,2013,103:385-398.
[10]石 刚,王静梅,剡根强,等.牛支原体改良培养基培养效果观察[J].动物医学进展,2013,34(3):129-132.
[11]Song Z Q,Li Y,Liu Y,et al.A-enolase,an adhesion-related factor of Mycoplasma bovis[J].Plos One,2012,7(6):e38836.

