基于可视化抗体宏阵列技术的出血性大肠杆菌O157∶H7定量检测
李浩林,刘 箐,刘 芳,董庆利,邱 实,杨玉萍,郭慧琴,韩舜愈
(1.甘肃农业大学食品科学与工程学院,甘肃 兰州 730070;2.上海理工大学医疗器械与食品学院,上海 200093;3.甘肃出入境检验检疫局,甘肃 兰州 730000;4.上海慧耘生物科技有限公司,上海 200437)
基于可视化抗体宏阵列技术的出血性大肠杆菌O157∶H7定量检测
李浩林1,2,刘 箐2,刘 芳3,董庆利2,邱 实2,杨玉萍2,郭慧琴4,韩舜愈1,*
(1.甘肃农业大学食品科学与工程学院,甘肃 兰州 730070;2.上海理工大学医疗器械与食品学院,上海 200093;3.甘肃出入境检验检疫局,甘肃 兰州 730000;4.上海慧耘生物科技有限公司,上海 200437)
目的:探索建立一种新的出血性大肠杆菌O157∶H7(Escherichia coli O157∶H7)的快速定量检测技术。方法:以硝酸纤维素膜为基片,用生物芯片点样仪制作抗体宏阵列,采用“双抗夹心”原理,对出血性大肠杆菌O157∶H7进行定量检测研究。结果:本方法对出血性大肠杆菌O157∶H7的检出限为3.4×105CFU/mL,在105~107CFU/mL细菌浓度范围内,宏阵列的灰度值与细菌浓度之间有较好的线性关系。该方法可在2.5 h内同步检测多个样品中出血性大肠杆菌O157∶H7的浓度。结论:通过用标准菌株、模拟带菌等研究显示,用宏阵列技术快速定量检测出血性大肠杆菌O157∶H7,结果肉眼可见,稳定准确,同时操作简便、成本低廉、无需大型设备,制作好的抗体宏阵列可用于快速评估食品中出血性大肠杆菌O157∶H7的污染状况,尤其适合于基层实验室进行快速高通量样品筛查。
出血性大肠杆菌O157∶H7;抗体宏阵列;可视化;定量检测
出血性大肠杆菌O157∶H7(Escherichia coli O157∶H7)是一种人畜共患病病菌,该菌对酸、低温等有较强的抗逆性,且致病性较强,可通过食品、人畜粪便等造成大规模爆发流行;该菌感染后引起呕吐、腹泻、发烧等症状,严重时伴有并发症如溶血性尿毒综合征、出血性肠炎等[1]。大肠杆菌O157∶H7型是引起出血性肠炎最常见的致病菌之一,1982年,美国首次发现大肠杆菌O157∶H7感染的疫情,是由牛肉汉堡传播,调查结果发现是牛肉污染引起;2005年,瑞典首次暴发大肠杆菌O157∶H7疫情,由污染的莴苣引发[2]。
常见的致病菌检测方法有传统的微生物培养方法、酶联免疫分析、聚合酶链式反应[3]等,也有近几年出现的生物传感器、免疫层析、电化学免疫法、微流控芯片[4-8]等新型检测技术,但除去传统平板计数方法以外,以上新型方法只能进行定性检测,且均有操作复杂、必须配合较高级设备、检测成本过高等问题。GB 29921—2013《食品中致病菌限量》已于2013年12月26日颁布,并于2014年7月1日实施,此会对微生物定量检测技术提出新要求。传统的定量检测方法如平板菌落计数法,耗时费力,无法适应大规模筛查检测的需要。近年来诸多学者对细菌的定量检测进行了研究探索,如结合实时荧光聚合酶链式反应(polymerase chain reaction,PCR)技术可以实现对细菌的快速定量检测[9-11],但该方法在对实际样品的检测中,由于受到PCR检测模板体系较少等限制,不仅会造成较高的漏检率,且操作相对复杂,成本较高,同时需要昂贵仪器,难以普及到基层实验室。本实验室在多年可视化蛋白芯片研究的基础上,采用了可视化抗体宏阵列、酶标抗体化学显色等技术,研制适合新标准要求的定量检测技术,以期为未来的致病菌定量检测研发实用技术。
1 材料与方法
1.1 材料与试剂
图1 可视化抗体宏阵列点样设计Fig.1 Visual antibody macroarray spot design
表1 实验所用标准菌株Table1 Standard strains used in this study
出血性大肠杆菌O157∶H7多克隆抗体、辣根过氧化物酶(horseradish peroxidase,HRP)标记大肠杆菌O157∶H7单克隆抗体以及各标准菌株(表1) 上海慧耘生物科技有限公司;硝酸纤维素膜(nitrocellulose membrane(NC膜),孔径0.22 μm) 美国Pall公司;增强型HRP-DAB底物显色液 北京天根生化科技有限公司;大肠杆菌O157∶H7显色培养基 北京陆桥技术有限公司。
1.2 仪器与设备
AD6010生物芯片点样仪 美国Bio-Dot公司;D90数码相机、AF-S VR Micro-Nikkor 105mm f/2.8G IF-ED微距镜头 日本Nikon公司。
1.3 方法
1.3.1 宏阵列制作
用含有20%甘油的碳酸盐缓冲液(pH 9.6)作为点样液稀释大肠杆菌多克隆抗体(一抗)至工作质量浓度200 μg/mL,作为捕获抗体,用生物芯片点样仪喷点在1 cm2的NC膜表面,每点80 nL,样点之间相距2 mm。点样设计如图1所示,第1行为捕获抗体。第2行为纯点样液,作为阴性控制,以确保点样液未受污染,避免假阳性的出现;第3行为羊抗猪IgG-HRP,作为阳性控制,以确保显色液的有效性[12]。点样完成后将基片置于4 ℃过夜备用。
1.3.2 宏阵列检测
封闭:用含有3%脱脂奶粉的磷酸缓冲液洗液(phosphate buffered saline tween 20,PBST)封闭阵列基片,同时轻微振荡;洗涤:吸除封闭液,PBST轻微振荡洗涤3 次;免疫反应:用菌液与抗体阵列反应,同时轻微振荡,之后吸除菌液,洗涤;加入二抗:将稀释600 倍的HRP标记大肠杆菌O157∶H7单克隆抗体与宏阵列基片反应,同时轻微振荡,之后吸除二抗,洗涤;显色:加入二氨基联苯胺(diamino aniline blue,DAB)显色液,避光显色,同时轻微振荡,之后吸除显色液,并加入双蒸水终止反应;最后用镊子从反应槽中取出反应完成的宏阵列基片,平铺于滤纸上避光阴干。
1.3.3 阵列灵敏度实验
用灭菌生理盐水依次1 0 倍梯度稀释单菌落培养的标准菌株纯菌液,并倒平板计数,浓度为3.4×100~3.4×108CFU/mL,生理盐水作为空白对照,用制作好的宏阵列基片进行检测。
1.3.4 标准曲线的拟合
平板计数后,用生理盐水10 倍稀释纯菌液至3.4×107CFU/mL,再将该浓度菌液用生理盐水依次连续对半稀释成不同浓度的菌液作为标准液,选择3.4×107、1.7×107、8.5×106、4.25×106、2.125×106、1.062 5×106、5.312 5×105CFU/mL作为标准曲线细菌浓度,用制作好的宏阵列基片进行实验。之后用数码相机拍照,此过程是在暗室中完成的,其间相机的各参数设置均保持一致,并且将相机固定在特定高度的支架之上,然后由上向下进行拍照,以确保不同的宏阵列结果均取自于相同的光源、光强以及成像距离。成像过程中的相机参数设置为:感光度3 200,快门速度1/30,闪光指数17,成像距离固定的58.5 cm。最后,照片不做任何处理,用Photoshop图像软件在灰度模式下对照片中的检测样点进行灰度分析,用Excel软件制作标准曲线。
1.3.5 模拟增菌实验
按照GB/T 4789.36—2008《食品卫生微生物学检验:大肠埃希氏菌O157∶H7/NM检验》中的规定,对超市购买的食品面包、果冻、牛奶进行前处理,在处理好的250 mL样品中加入事先培养并且稀释106倍的标准菌液1 mL,经计数为380 个/mL,然后于37 ℃进行增菌培养,分别于2、4、6、8、10、12、14、16 h后进行宏阵列检测,并同步用大肠杆菌O157∶H7显色培养基涂布平板计数,之后分析平板计数结果与标准曲线推算结果的符合程度。
1.3.6 特异性检测
用生理盐水分别将金黄色葡萄球菌、单核细胞增生李斯特菌、猪霍乱沙门氏菌、鼠伤寒沙门氏菌、肠炎沙门氏菌、福氏志贺氏菌、宋内氏志贺氏菌、痢疾志贺氏菌、鲍氏志贺氏菌、副溶血性弧菌、阪崎肠杆菌、小肠结肠炎耶尔森氏菌等常见食源性致病菌制备成107CFU/mL菌悬液,用制作好的宏阵列进行检测,验证其特异性。
2 结果与分析
2.1 灵敏度实验
本研究中所有用到的信号值,都是用软件分析各个检测样点的单点灰度峰值,并换算成信号值,最终的信号值为4 个平行信号值的平均值(灰度值范围为0~255,颜色越深,灰度值越低,本研究将所有检测样点信号值定义为:信号值=255-灰度值)。如图2a所示,3 次重复实验结果,随着菌液浓度依次降低,信号值也依次减弱,菌液浓度在3.4×104CFU/mL以下时,均不能检出,可以看到,本抗体宏阵列的检测灵敏度为3.4×105CFU/mL。通过软件分析各检测样点灰度值,制作如图2b所示直方图,可以直观地看到,3.4×108~3.4×105CFU/mL菌液浓度之下,其平均信号值依次为120.5、129.75、114.75、60,其中3.4×107CFU/mL菌液浓度时检测信号值最强,而在3.4×108CFU/mL菌液浓度时,检测信号值为120.5,弱于3.4×107CFU/mL菌液浓度时的信号值129.75,并且从107CFU/mL菌液浓度开始,浓度越低,检测信号值越弱,此平均数值来自相同的3 次重复实验。表2为4 个菌液浓度条件下检测样点信号值的变异系数。

图2 灵敏度实验Fig.2 Sensitivity of the method

表2 各菌液浓度条件下信号值的变异系数Table2 Coefficient of variations of signal values for different bacterial concentrations
2.2 标准曲线拟合
标准曲线阵列的实验结果如图3a所示,A、B、C为相同的3 次重复实验。以检测样点平均信号值为纵坐标,将各梯度标准菌液的浓度值以10为底取其对数作为横坐标,分别对应拟合b、c、d 3 条标准曲线,如图3b、c、d所示。
由图3可见,各检测样点的信号随着标准菌液浓度的增加而逐渐增强,拟合的3 条标准曲线也有着相似的增长趋势,经过对各检测样点的灰度值进行分析,各菌液浓度条件下3 组平均信号值的变异系数如表3所示,其中,4.25×106CFU/mL菌液浓度条件下的变异系数最小,为0.37%,2.125×106CFU/mL菌液浓度条件下的变异系数最大,为1.03%,但后者仍然远小于一般酶联免疫分析实验所要求的5%[13],因此,标准曲线的稳定性和重复性较强,由此得到较为理想的标准曲线。

图3 标准曲线拟合实验Fig.3 Standard curves
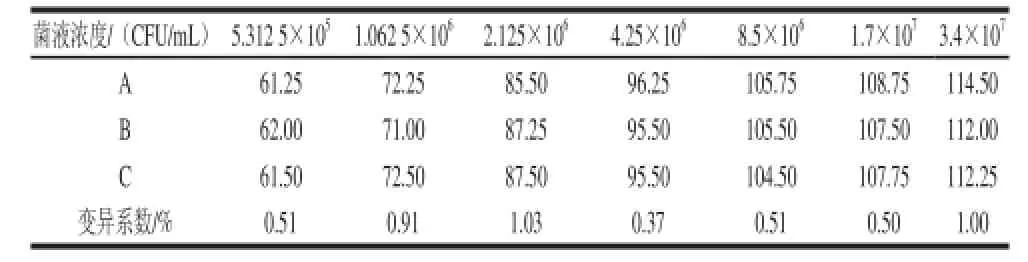
表3 各菌液浓度条件下3 组平均信号值的变异系数Table3 Coefficients of variations for three average signal values
2.3 模拟增菌定量检测
如图4a、c、e所示,抗体阵列对果冻、面包、牛奶样品在增菌8 h后即可检出,但10 h之后的检测样点信号值反而随抗原浓度的增大而降低,3个样品在不同增菌时间的检测平均信号强度如图4b、d、f所示。

图4 模拟增菌实验Fig.4 Detection of simulated enrichment culture
由图4可见,果冻样品在增菌8 h时检测信号值最强,达到了90.25,随后增菌10 h的检测信号有所减弱,为85.5,而之后再增菌培养12、14、16 h后的检测信号强度相比于8 h和10 h进一步减弱,但三者之间无明显差异。如图4d所示,面包样品增菌8 h的检测信号强度为79.75,弱于增菌10 h的107.75,随后的3 个增菌时间段的检测结果依次减弱,分别为97.5、94.75和94。如图4f所示,牛奶样品增菌8 h的检测强度为91,增菌10 h的检测强度为97.75,增菌12、14、16 h的检测强度依次为90、92.5、89.75,三者之间无明显差异。
对3 个样品增菌8 h和10 h的检测样点进行灰度值分析,并将信号值分别代入图3中的3 条标准曲线中,推算出相应浓度,然后与同步平板计数的结果进行比较,如表4及图5所示。增菌8 h和10 h的细菌数目在3 条标准曲线的线性范围之内,分析其各个检测样点的灰度值,换算成信号值并取平均值后代入图3b、c、d 3 条标准曲线中,得到相应的推算数目,如表4所示,在同一增菌时间范围内,3 条标准曲线推算所得到的3 个结果之间差异很小,可以忽略不计,因此可以确定标准曲线的稳定性。
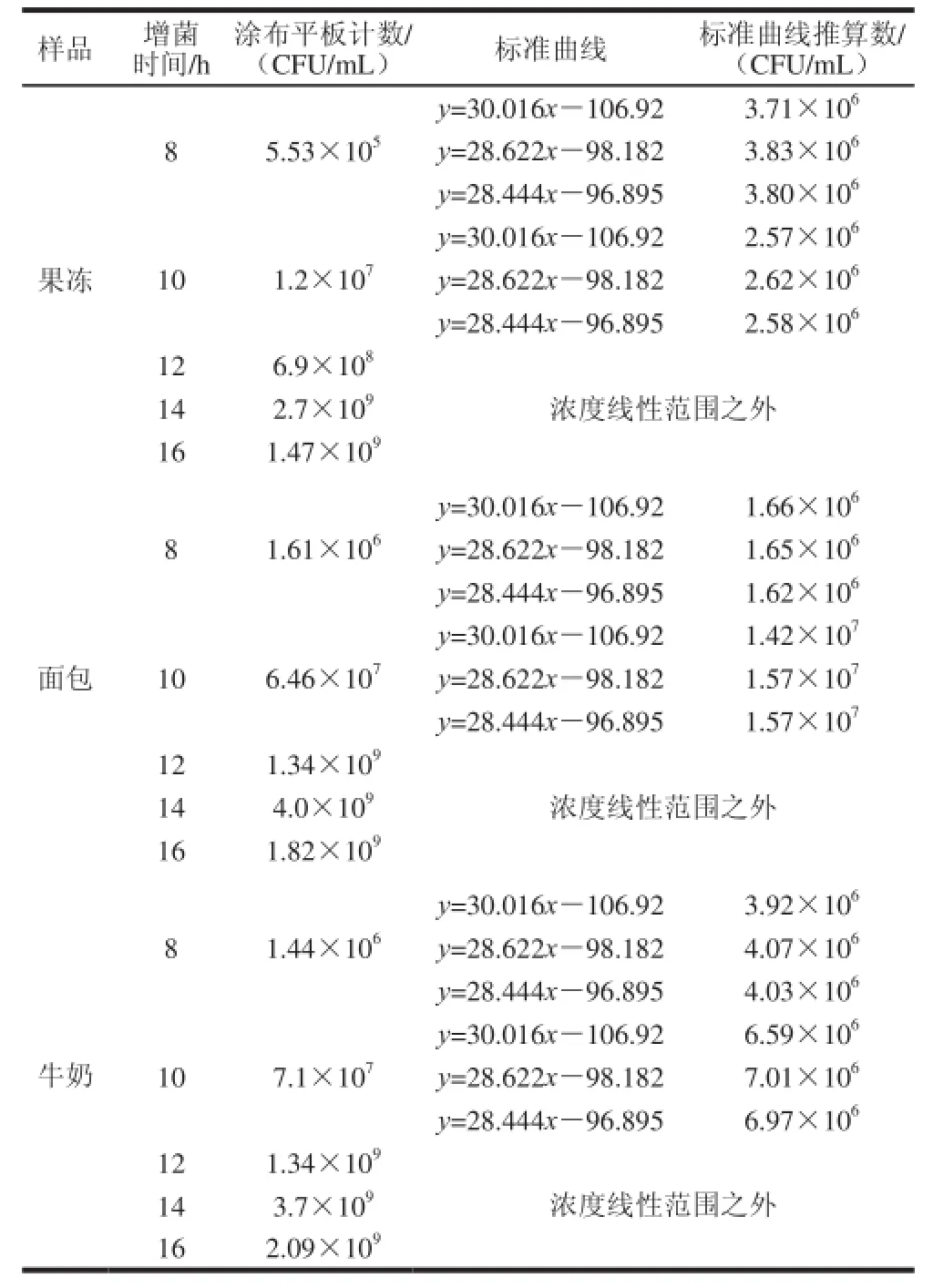
表4 实际样品检测中涂布平板计数与标准曲线推算结果的比较Table4 Comparison of spread plate counts and results calculated from the standard curves for real samples
另外,如表4所示,对涂布平板计数结果与标准曲线推算数结果进行比较,面包样品增菌8、10 h以及牛奶样品增菌8 h的推算结果与平板计数结果在同一数量级,而果冻样品增菌8、10 h以及牛奶样品增菌10 h的推算结果与平板计数结果相差一个数量级;分析有两部分原因,一方面,用作标准曲线的抗体宏阵列在实验过程中,各溶液、缓冲液组分单一、稳定,对抗原抗体特异性结合影响较小,而实际样品中成分复杂,对抗原抗体的特异性结合有较大影响,即“基质效应”[14],因而在显色完成后,标准曲线与实际样品的信号值会有差异,从而影响推算结果;另一方面,在增菌同步涂布平板的操作过程中,不可避免的会有少量细菌残留在涂布棒之上,从而造成平板计数的误差;结合这两方面原因,检测结果会产生一定程度的误差。同时,相比于10 h增菌的检测信号值,3 个样品在增菌10 h之后的信号值均弱于前者,但平板计数结果却远大于前者,这可能是因为,抗原抗体的结合力,主要有疏水作用、爱德华力、氢键、离子键等,过量的抗体或抗原,对于二者最佳的结合比列,都有消极的影响(即抗原之间相互竞争导致排斥,抗原抗体结合比率下降),而随着增菌时间的延长,细菌数目过多,即抗原的量远远大于抗体的量,从而对抗原抗体的特异性结合产生一定程度的负面影响。最后,从表4可以看出,平板计数的结果均在增菌14 h时达到最大值,增菌16 h后细菌数目反而有所下降,说明在增菌过程中,随着时间的延长,有限的营养物质逐渐被消耗殆尽,大肠菌群生长进入衰亡期,有大量细菌死亡。

图5 增菌过程中显色培养基同步涂布平板计数结果Fig.5 Spread plate counts from Escherichia coli O157∶H7 chromogenic medium
2.4 抗体宏阵列特异性实验
如图6a所示,除去两株出血性大肠杆菌O157∶H7以外,抗体宏阵列与其他细菌均无交叉反应。


图6 大肠杆菌O157∶H7抗体阵列特异性实验Fig.6 Specificity of the antibody macroarray for Escherichia coli O157∶H7
3 讨 论
本实验利用可视化抗体宏阵列技术,探讨将高通量的抗体宏阵列,运用于单样品多指标食源性致病菌快速定量检测,结果显示:抗体宏阵列对出血性大肠杆菌O157∶H7具有很好的特异性,与其他常见食源性致病菌均无交叉反应,模拟增菌实验结果良好,其检出限为3.4×105CFU/mL,灵敏度与酶联免疫吸附测定技术(enzyme linked immunosorbent assay,ELISA)相同[15],但耗时仅为2.5 h,并且节省抗体,操作简便快捷,无需特殊设备,对实验人员要求低,适合基层实验室进行快速高通量样品筛查。
在实验前期,原期望采用3.4×108、3.4×107、3.4×106、3.4×105、3.4×104CFU/mL 5 个菌液浓度作为标准曲线的坐标浓度,但结果显示,相比于3.4×107CFU/mL,108菌液的检测信号值反而弱于前者,这与模拟增菌实验中出现的10 h之后信号值减弱的现象是相同原因,因此该方法对于目标菌浓度过高的情况下,定量检测可能与实际细菌数有较大差异;另外本实验检测灵敏度为3.4×105CFU/mL,对104浓度菌液无法检出,因此不能用于拟合标准曲线。
常见的蛋白质芯片技术大都采用荧光标记物释放检测信号,需要相应荧光信号检测系统,成本较高且结果不稳定[16-21],亦或是对检测基片进行各种修饰加工,旨在提高检测灵敏度及特异性[22-23],虽然有所进步,但无形之中又增加了综合成本,均不适合基层实验室进行前期样品的快速高通量筛查。本实验研制的可视化抗体宏阵列无需信号采集系统,可直接用肉眼识别检测结果,若借助常用计算机软件,则可实现定量检测。
食品安全事故的频繁发生,推动着检测技术的不断进步,简洁、快速、高通量已成为目前检测技术的发展趋势,基于胶体金免疫层析技术的试纸条近年来在快速检测领域应用逐渐增多[24-25],但要实现大量样品的快速筛查,则其成本较高,并且为定性检测;抗体宏阵列检测虽然耗时长于试纸条方法,但仍可在2.5 h之内完成检测,同时更适合高通量检测。本研究随后将尝试在同一芯片阵列之上固定多种捕获抗体,从而达到同步检测多种目标细菌的目的,同时为研制蛋白质芯片快速检测试剂盒做前期准备。
[1] ZHANG Yi, WANG Gaochan, WU Yupeng, et al. PCR-DGGE analysis of earthworm gut bacteria diversity in stress of Escherichia coli O157:H7[J]. Advances in Bioscience and Biotechnology, 2013, 4(3): 437-441.
[2] 朱蓓. 肠出血性大肠杆菌感染的流行病学及临床医学资料概述[J].解放军预防医学杂志, 2011, 29(4): 309-311.
[3] 席美丽, 只帅, 王小璞, 等. 食源性大肠杆菌的PCR检测[J]. 西北农业学报, 2011, 20(12): 188-191.
[4] 斯城燕, 叶尊忠, 王一娴, 等. SPR生物传感器快速检测大肠杆菌O157:H7的研究[J]. 光谱学与光谱分析, 2011, 31(10): 2598-2601.
[5] LI Yan, FANG Lichao, CHENG Ping, et al. An electrochemical immunosensor for sensitive detection of Escherichia coli O157:H7 using C60based biocompatible platform and enzyme functionalized Pt nanochains tracing tag[J]. Biosensors and Bioelectronics, 2013, 49(15): 485-491.
[6] 刘慧杰, 滕瑛巧, 张新爱, 等. 基于Fe3O4@Au复合纳米粒子标记抗体的电化学免疫方法用于水体中大肠杆菌的检测[J]. 高等学校化学学报, 2010, 31(6): 1131-1136.
[7] 彭金兰, 徐溢, 吴永杰, 等. 微流控芯片上大肠杆菌的电化学阻抗检测方法研究[J]. 分析化学, 2011, 39(9): 1307-1312.
[8] C H O I H, I RU DAYA R A J J. L a t e r a l-f l ow e n z y m e immunoconcentration for rapid detection of Listeria monocytogenes[J]. Analytical and Bioanalytical Chemistry, 2013, 405(10): 3313-3319.
[9] 邵美丽, 董鑫, 赵燕丽, 等. 单增李斯特菌和金黄色葡萄球菌双重荧光定量PCR检测方法建立与初步应[J]. 食品科学, 2013, 34(16): 167-170.
[10] 程苏云, 张俊彦, 王赞信, 等. 海水产品副溶血性弧菌污染定量检测分析[J]. 中国卫生检验杂志, 2007, 17(2): 336-338.
[11] 李祖明, 王磊, 吴聪明, 等. 生乳阪崎肠杆菌、大肠杆菌和细菌总数的实时定量PCR检测[J]. 工业微生物, 2013, 43(2): 17-21.
[12] XIONG Liangbin, GAO Liping, LIU Qing, et al. A simple, rapid and visual antibody array for the simultaneous detection of multiple plant pathogens[J]. Analytical Methods, 2013, 5(9): 2413-2418.
[13] 刘雅莉, 刘芳, 刘箐, 等. 两种酶标抗体制备方法的比较及李斯特菌TAS ELISA试剂盒性能的优化[J]. 食品工业科技, 2012, 33(15): 301-310.
[14] LIAN Wei, WU Donghai, LIM D V, et al. Sensitive detection of multiplex toxins using antibody microarray[J]. Analytical Biochemistry, 2010, 401(2): 271-279.
[15] 葛萃萃, 钟青萍, 张旺, 等. 双抗夹心ELISA检测食品中大肠杆菌O157:H7方法研究[J]. 食品科学, 2007, 28(1): 171-175.
[16] ZHU Lingxiang, JIANG Guanglu, WANG Shengfen, et al. Biochip system for rapid and accurate identification of Mycobacterial species from isolates and sputum[J]. Journal of Clinical Microbiology, 2010, 48(10): 3654-3660.
[17] LEE B, TAJIMA A, KIM J, et al. Fabrication of protein microarrays using the electrospray deposition (ESD) method: application of microfluidic chips in immunoassay[J]. Biotechnology and Bioprocess Engineering, 2010, 15(1): 145-151.
[18] SARDESAI N P, KADIMISETTY K, FARIA R, et al. A microfluidic electrochemiluminescent device for detecting cancer biomarker proteins[J]. Analytical and Bioanalytical Chemistry, 2013, 405(11): 3831-3838.
[19] MARÍA G G, RICARDO J A, SERGIO M, et al. Nanotechniques in proteomics: protein microarrays and novel detection platforms[J]. European Journal of Pharmaceutical Sciences, 2012, 45(4): 499-506.
[20] CROMPTON P D, KAYALA M A, TRAORE B, et al. A prospective analysis of the Ab response to Plasmodium falciparum before and after a malaria season by protein microarray[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(15): 6958-6963.
[21] BRIAN B H. Applications of antibody array platforms[J]. Current Opinion in Biotechnology, 2006, 17(4): 415-421.
[22] TANG Jingfeng, XU Zhenghui, ZHOU Li, et al. Rapid and simultaneous detection of Ureaplasma parvum and Chlamydia trachomatis antibodies based on visual protein microarray using gold nanoparticles and silver enhancement[J]. Diagnostic Microbiology and Infectious Disease, 2010, 67(2): 122-128.
[23] SANG W H, SEULAH L, JUREE H, et al. Mutiscale substrates based on hydrogel-incorporated silicon nanowires for protein patterning and microarray-based immunoassays[J]. Biosensors and Bioelectronics, 2013, 45(15): 129-135.
[24] 夏敏, 杜美红, 贾瑜, 等. 胶体金免疫层析技术测定食品中黄原胶[J].食品科学, 2012, 33(20): 270-273.
[25] 张洋, 贾睿, 何培民. 免疫胶体金试纸条快速检测贝类体内的腹泻性贝毒[J]. 生物技术通报, 2013(6): 221-225.
Quantitative Detection of Escherichia coli O157:H7 Based on Visual Antibody Macroarray Technology
LI Hao-lin1,2, LIU Qing2, LIU Fang3, DONG Qing-li2, QIU Shi2, YANG Yu-ping2, GUO Hui-qing4, HAN Shun-yu1,*
(1. College of Food Science and Engineering, Gansu Agricultural University, Lanzhou 730070, China; 2. School of Medical Instrument and Food Engineering, University of Shanghai for Science and Technology, Shanghai 200093, China; 3. Gansu Entry-Exit Inspection and Quarantine Bureau, Lanzhou 730000, China; 4. Shanghai Prajan Bioiogy Technology Company, Shanghai 200437, China)
Purpose: To establish a new rapid quantitative assay for detecting Escherichia coli O157:H7. Methods: Using nitrocellulose membrane as substrate, an antibody macroarray was fabricated with a biochip spotting robot, and then a double antibody sandwich method was developed for quantitative detection of E. coli O157:H7. Results: The limit of detection (LOD) for E. coli O157:H7 was 3.4 × 105CFU/mL, and a good linear relationship was observed between grey value for the macroarray and bacterial concentration in the range of 105-107CFU/mL. This method could allow simultaneous determination of different concentrations of E. coli O157:H7 in multiple samples. Conclusions: As evaluated by applying it to standard strains and simulated enrichment culture, the antibody macroarray-based method could provide stable and accurate results visible to naked eyes with simple operation and low costs without the use of large equipment. Moreover, the antibody macroarray developed in this study could be used to rapidly assess E. coli O157:H7 pollution in food, especially suitable for rapid screening of samples in grassroots-level laboratories.
Escherichia coli O157:H7; antibody macroarray; visualization; quantitative determination
Q939.91
A
1002-6630(2014)20-0185-07
10.7506/spkx1002-6630-201420037
2013-10-21
国家质检总局科技项目(GSCIQ_2010IK220);上海市科委重点支撑项目(13430502400);甘肃省科技支撑计划项目(1304FKCA056)
李浩林(1987—),男,硕士研究生,研究方向为食品安全检测。E-mail:lihaolin36@sina.com
*通信作者:韩舜愈(1963—),男,教授,博士,研究方向为食品科学与工程。E-mail:lzhansy@126.com

