原位杂交检测microRNA-205在乳腺癌中的表达
范瑞琦 王 晟 揭克敏 熊小亮 肖影群 贺晓菊 刘卓琦 罗达亚*
(1南昌大学基础医学院生物化学与分子生物学教研室,南昌330006;2杭州电子科技大学研究生院,浙江310018;3南昌大学附属感染医院病理科,江西330002;4江西省妇幼保健院,江西330005)
microRNA(miRNA,miR)是一类内源性非蛋白编码的小RNA分子。作为动植物体内的重要调控分子,miRNA主要通过翻译抑制、mRNA降解以及由其引导快速脱腺苷化作用起始的mRNA衰变等方式调节基因的表达[1]。由于miRNA较为稳定,相对于mRNA不容易被降解等特性,使得其成为具有良好临床应用潜能的分子标志物。
临床疾病标本的应用,在验证miRNA与肿瘤发生、发展的关系及其调控机制中具有非常重要的价值。然而,常规收集的、不论是新鲜标本或福尔马林固定石蜡包埋的临床组织标本,都存在因组织标本的异质性和缺乏miRNA定位信息而使得研究并不容易获得满意的结果。为此,建立并应用具备定性、半定量以及定位信息的miRNA原位杂交(miRNA insitu hybridization,MISH)方法对临床组织标本进行分析将为miRNA的研究带来更多的便利。此项研究中,课题组选择前期利用miRNA表达芯片筛选的miR-205作为研究靶点,建立并应用原位杂交的方法对乳腺组织芯片标本进行miR-205的表达分析,以验证miR-205与乳腺癌发生、发展的关系。
材料和方法
1.组织芯片
人乳腺组织芯片购自西安艾丽娜生物技术有限公司(编号BR723),共71例/72点。71例病人均为女性,年龄18-83岁,中位年龄45岁。其中正常与乳腺良性病变36点,包括正常乳腺组织2点,癌旁正常乳腺组织3点,腺病9点,纤维腺瘤4点,乳腺增生病变18点;乳腺癌36点,包括导管原位癌3点,浸润性导管癌18点,髓样癌3点,粘液癌3点,小叶癌3点,远处转移癌6点。芯片上的组织均经常规4%中性甲醛固定,石蜡包埋,5μm厚度切片,直径1.5mm。
2.原位杂交
miR-205原位杂交检测试剂盒购自丹麦Exiqon公司,原位杂交封闭液与洗涤液购自美国Roche公司。基本过程如下:组织芯片脱蜡,于37℃蛋白酶K消化15min,漂洗后梯度乙醇脱水。55℃预杂交30min后,地高辛标记的 miR-205探针(100nmol/L)恒温杂交1h,碱性磷酸酶标记的羊抗地高辛抗体室温孵育1h,NBT/BCIP黑暗处30℃显色,核固红复染。杂交过程选择U6探针(1nmol/L)作为阳性对照,去除探针作为阴性对照。结果判定:miR-205呈蓝色或蓝紫色颗粒的阳性信号定位于乳腺腺上皮或癌细胞胞质和胞核中;U6呈蓝色或蓝紫色颗粒的阳性信号定位于乳腺导管上皮或间质细胞的胞核。根据阳性细胞着色的范围分为:阳性细胞<10%为0分;阳性细胞≥10%为1分;按着色细胞染色强度分为:无表达或弱表达为0分,中等强度或强表达为1分。以上两项积分相乘,积分为0分表示阴性(-),积分为1分表示阳性(+)。组织芯片的染色结果由两位病理医师独立镜检和评分。
3.细胞培养
9株人乳腺细胞均由美国佐治亚医科大学肿瘤研究中心石慧东副教授馈赠,包括高度恶性人乳腺癌细 胞 株 BT549、 HS578T、 MDA-MB-231 和SUM159PT;低度恶性人乳腺癌细胞株 MDA-MB-468、T-47D、ZR-75-1和SKBR3以及永生化正常乳腺上皮细胞株MCF10A。8株乳腺癌细胞均选择含10%小牛血清的DMEM培养液,置于37℃、5%CO2培养箱中培养。MCF10A则用含5%马血清、表皮生长因子(20ng/mL)、胰岛素(10μg/mL)、霍乱毒素(100ng/mL)和皮质醇(500ng/mL)的DMEM/F12培养液,置于37℃、5%CO2培养箱中培养。
4.miRNA提取、逆转录与实时定量PCR
miRNA 提 取 按 照 mirVanaTMmiRNA Isolation Kit(Invitrogen,美国)说明书进行。按照 RT2miRNA First Strand Kit(SA Biosciences,美国)说明书,取1μg miRNA进行逆转录操作。按照RT2SYBR Green Master Mixes(SABiosciences,美国)的操作说明,以U6为 内 参 照,使 用 Light Cycler 480II instrument(Roche,瑞士)进行PCR扩增反应,每个样品3个重复孔。U6、miR-205引物均购自美国SABiosciences公司。采用公式2-ΔCt(ΔCt= Ct(miR-205)– Ct(U6))计算 miR-205的相对表达量。
5.统计学处理
采用SPSS 20.0软件进行统计学处理,计数资料率的比较采用卡方检验,相关性分析采用Pearson相关分析,检验水准α=0.05。
结 果
1.miR-205的原位杂交分析
乳腺组织原位杂交后,阴性对照结果未见蓝色颗粒;U6探针杂交结果显示呈蓝紫色颗粒的阳性信号定位于乳腺导管上皮或间质细胞的胞核;miR-205探针杂交结果显示呈蓝紫色颗粒的阳性信号定位于乳腺导管上皮或癌细胞胞质和胞核。结果见图1、2。36例正常与良性乳腺病变中,33例(91.67%)积分为1分,为表达阳性(+);36例乳腺癌中,23例(63.89%)积分为1分,为表达阳性(+)。miR-205的表达在乳腺良恶性病变中的表达有统计学差异(P=0.011),结果见表1。miR-205的表达与乳腺癌TNM分期、临床分期无关(P>0.05)。
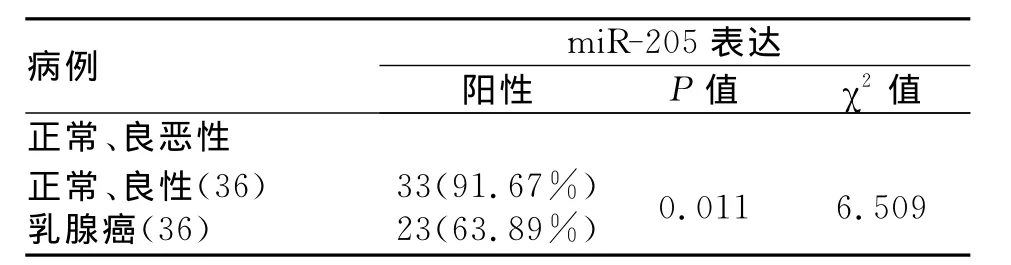
表1 miR-205在正常、良性乳腺病变和乳腺癌中的表达Table 1 miR-205expression in normal tissues,benign tumors and malignant breast cancer
2.miR-205在乳腺细胞株中的表达
采用 mirVanaTMmiRNA Isolation Kit试剂盒分别提取的总RNA和小分子量RNA,经1%琼脂糖电泳显示,总RNA泳道中大分子量的28SrRNA和18SrRNA条带清晰,5SrRNA等小分子量RNA的条带较弱;小分子量RNA泳道中只存在5S rRNA等小分子量RNA,条带较总RNA泳道中的小分子量RNA条带清晰。结果见图3。实时定量RT-PCR结果显示,四个高度恶性乳腺癌细胞株(MDA-MB-231、HS578T、BT549 和 SUM159PT)中miR-205的表达较四个低度恶性细胞株(MDA-MB-468、T-47D、ZR-75-1和SKBR3)和永生化正常乳腺上皮细胞株 MCF10A中为低(P<0.05)(图4)。

图1 乳腺组织中阴性对照、U6、miR-205探针的原位杂交(SP×100,NBT/BCIP显色)图2 组织芯片miR-205原位杂交(NBT/BCIP显色)图3 细胞总RNA(aRNA)与小分子量RNA(sRNA)的琼脂糖凝胶电泳分析图4 实时定量 RT-PCR检测正常乳腺上皮 MCF10A和低度恶性(MDA-MB-468、T-47D、ZR-75-1和SKBR3)、高度恶性(MDA-MB-231、HS578T、BT549和SUM159PT)乳腺癌细胞株中 miR-205的表达Fig.1In situ hybridization analysis for negative,U6,miR-205in breast tissue(SP×100,NBT/BCIP solution)Fig.2In situ hybridization for miR-205in tissue array(NBT/BCIP solution)Fig.3Agarose gel electrophoresis analysis for total RNA (aRNA)and small molecular RNA (sRNA)Fig.4qRT-PCR detection for miR-205expression in normal breast epithelial cell MCF10A,less aggressive (MDA-MB-468,T-47D,ZR-75-1and SKBR3)and aggressive(MDA-MB-231,HS578T,BT549and SUM159PT)breast cancer cell lines
讨 论
课题组前期利用miRNA表达芯片,在高度恶性、正常与低度恶性的12个乳腺细胞株中,共筛选到9个差异表达的miRNAs(P<0.01,倍数值≥20或≤-20)。与正常、低度恶性细胞株相比,miR-205的表达在高度恶性乳腺癌细胞株中明显下调[2]。为此,课题组选择9个乳腺细胞株检测miR-205的表达以验证芯片分析结果。实时定量RT-PCR结果显示,四个高度恶性乳腺癌细胞株中miR-205的表达较四个低度恶性细胞株和永生化正常乳腺上皮细胞株MCF10A中为低(P<0.05),这与多数研究分析结果一致[3-5]。为进一步验证 miR-205与乳腺癌发生、发展的关系,选择乳腺临床标本进行miR-205的表达分析甚为必要。
在组织标本中常用的低通量miRNA检测方法主要是Northern blotting和实时定量RT-PCR。作为经典的miRNA表达分析方法,Northern blotting是miRNA检测的金标准。该方法主要通过荧光或放射性寡核苷酸探针与靶miRNA杂交以检测目的miRNA的表达情况[6],因其检测中未经过PCR扩增而能准确反映组织样品的实际miRNA含量,在miRNA研究的初期被广泛应用。然而,尽管经过多次改良,该方法仍无法完全避免样本需求量大、耗时长、操作繁琐等缺点。实时定量RT-PCR法是现阶段使用最为普遍的方法。以较为先进的stemloop RT-PCR为例,该方法不仅特异性高,可精确区分同一家族中序列高度同源性的miRNA,并能检测只有一个碱基差别的不同miRNA的表达水平,而且该方法灵敏度也很高,样品消耗量少,能检测出低丰度靶miRNA[7]。然而,由于该方法可能因为PCR扩增后而失真miRNA表达结果以及组织标本异质性的特点,并不容易获得满意的结果。
因定性并半定量反映miRNA的表达,原位杂交在组织标本的miRNA研究中可以带来更多的便利。Qi等[8]应用miRNA原位杂交的方法,针对乳腺正常导管上皮组织、异型扁平上皮组织、原位导管癌及浸润型导管癌组织分析发现,组织恶性程度越高,miR-21的表达越高。Li等[9]通过建立荧光原位杂交方法对miR-375进行的研究发现,该方法不仅在食管癌细胞株miR-375的表达分析中与实时定量RT-PCR得出一致的结果,而且利用该方法还能有效地对食管癌组织芯片标本进行分析。为此,课题组成功建立并应用原位杂交的方法在乳腺标本上进行了miR-205的表达分析。杂交结果显示miR-205主要表达在乳腺腺上皮的胞质与胞核,且miR-205的表达与乳腺病变良恶性相关(P=0.011),在恶性病变中的表达低于正常与良性乳腺病变。该分析结果不仅验证了在不同恶性程度乳腺细胞株上的miRNA芯片与实时定量RT-PCR的检测结果,也与大多数应用其他方法在乳腺癌标本上的分析结果一致。Savad[10]与 Wu[3]课题组通过实时定量 RTPCR方法分别对17例和19例临床标本分析发现,乳腺癌标本中miR-205的表达低于配对癌旁组织。Iorio等[11]利用 Northern blotting方法发现乳腺癌组织中miR-205表达明显低于对应的癌旁正常组织。对组织芯片中36例乳腺癌标本的原位杂交结果分析显示,miR-205的表达与病例年龄、乳腺癌TNM分期、临床分期均无相关性(P>0.05)。该结果与Liu等[12]对20例乳腺癌病人的血清miR-205表达与病例资料分析结果一致。然而,Baffa等[13]采用miRNA芯片对13例原位乳腺癌及其对应转移淋巴结组织进行分析后发现,miR-205的表达在转移灶中明显下调。产生这种差异的原因,可能与miR-205表达检测的方法、病例来源以及病例样本偏小等诸多因素有关。
在临床标本上验证miRNA与其调控基因的关系是miRNA功能分析的重要环节。Laura Gramantieri等[14]在细胞水平上成功验证 miR-122a和周期蛋白G1的调控关系后,通过Northern blotting和免疫印迹的方法对肝细胞癌临床标本中的miR-122a和周期蛋白G1分别进行了表达分析。结果发现,两者表达在同一标本中呈现明显反变的关系。然而,因操作繁琐和组织异质性等原因,选择Northern blotting或实时定量RT-PCR在高通量临床样本中验证miRNA及其靶点的调控关系较在细胞水平上的验证更为困难。原位杂交结合连续切片免疫组化分析在组织样本中验证miRNA的靶点调控显现出较其他方法更大的优势。Yamamichi等[15]采用原位杂交与免疫组化相结合的方法,发现miR-21在结肠癌组织中表达量高于正常结肠组织,而PDCD4的表达情况相反,两者的表达定位存在明显的反相关系。结合细胞水平的实验,在临床标本上进一步验证了PDCD4是miR-21靶点的可能。这为miRNA原位杂交方法在临床标本上的广泛应用提出了更迫切的需求。
综上所述,本实验结果认为miR-205可能参与乳腺癌病变的发生、发展并在乳腺良性病变、癌变及其演进过程中呈明显下调倾向;原位杂交方法的应用为阐明miRNA的作用机制提供了更为便利的条件。尽管本课题对miR-205在乳腺细胞株与乳腺疾病组织标本进行了表达分析,但组织标本的数量仍然较少,将实验结论在更多乳腺组织标本的验证仍然非常必要;同时,如何将细胞水平筛选的miR-205靶点应用原位杂交结合免疫组化方法验证两者之间的关系,也为后续工作提供了方向。
[1]Zhang,B,Pan X,Cobb GP,et al.microRNAs as oncogenes and tumor suppressors.Dev Biol,2007,302(1):1-12
[2]王晟,肖影群,刘卓琦等.乳腺癌转移相关microRNA-200c调控的基因网络分析.肿瘤,2013,33(2):111-118
[3]Wu,H,Zhu S and Mo YY.Suppression of cell growth and invasion by miR-205in breast cancer.Cell Res,2009,19(4):439-448
[4]Wu H and Mo YY.Targeting miR-205in breast cancer.Expert Opin Ther Targets,2009,13(12):1439-1448
[5]Piovan C,Palmieri D,Di Leva G,et al.Oncosuppressive role of p53-induced miR-205in triple negative breast cancer.Mol Oncol,2012,6(4):458-472
[6]Pall,GS,Codony-Servat C,Byrne J,et al.Carbodiimide-mediated cross-linking of RNA to nylon membranes improves the detection of siRNA,miRNA and piRNA by northern blot.Nucleic Acids Res,2007,35(8):e60
[7]Chen,C,Ridzon DA,Broomer AJ,et al.Real-time quantification of microRNAs by stem-loop RT-PCR.Nucleic Acids Res,2005,33(20):e179
[8]Qi L,Bart J,Tan LP,et al.Expression of miR-21and its targets(PTEN,PDCD4,TM1)in flat epithelial atypia of the breast in relation to ductal carcinoma in situ and invasive carcinoma.BMC Cancer,2009,9:163
[9]Li J,Li X,Li Y,et al.Cell-specific detection of miR-375downregulation for predicting the prognosis of esophageal squamous cell carcinoma by miRNA in situ hybridization.PLoS One,2013,8(1):e53582
[10]Savad S,Mehdipour P,Miryounesi M,et al.Expression analysis of MiR-21,MiR-205,and MiR-342in breast cancer in Iran.Asian Pac J Cancer Prev,2012,13(3):873-877
[11]Iorio MV,Casalini P,Piovan C,et al.microRNA-205 regulates HER3in human breast cancer.Cancer Res,2009,69(6):2195-2200
[12]Liu J,Mao Q,Liu Y,et al.Analysis of miR-205and miR-155expression in the blood of breast cancer patients.Chin J Cancer Res,2013,25(1):46-54
[13]Baffa R,Fassan M,Volinia S,et al.MicroRNA expression profiling of human metastatic cancers identifies cancer gene targets.J Pathol,2009,219(2):214-221
[14]Gramantieri L,Ferracin M,Fornari F,et al.Cyclin G1is a target of miR-122a,a microRNA frequently down-regulated in human hepatocellular carcinoma.Cancer Res,2007,67(13):6092-6099
[15]Yamamichi N,Shimomura R,Inada K,et al.Locked nucleic acid in situ hybridization analysis of miR-21expression during colorectal cancer development.Clin Cancer Res,2009,15(12):4009-4016

