采用正交设计法优化梨SSR-PCR体系
毕红园 王长彪 段永红 郭黄萍 孙毅,5
(1.山西大学生物工程学院,太原 030006;2.山西省农业科学院生物技术研究中心,太原 030031;3.山西农业大学农学院,太谷 030801;4.山西农科院果树研究所,太谷 030815;5.农业部黄土高原作物基因资源与种质创制重点实验室,太原 030031)
梨属于蔷薇科(Rosaceae),梨属(Pyrus L.)植物[1]。全世界梨属植物约有60种,我国是梨属植物的起源中心,蕴藏着丰富的种质资源,原产我国的梨属植物现有18个种,7个亚种,栽培品种约有3 000多个,栽培面积和总产量在国内主要水果中均排第3位[2]。目前,随着我省农业产业结构的调整,出现了一批自有知识产权的梨树新品种和新材料,但由于市场混乱,现有的新品种得不到有效的保护和鉴定,极大地损害了相关单位和个人的利益,同时也阻碍了我省农业经济的大力发展。为此,进行梨树品种间鉴定,保证品种的真实性和纯度,维护生产者与育种者的利益就显得尤为重要。近年来,迅速发展的DNA分子标记技术为优良品种的引进,品种快速准确的鉴定和伪劣苗木的早期识别提供了有效的手段。
已有学者选用不同的分子标记技术对梨开展了研究。其中,SSR标记(Simple Sequence Repeat)是一种新兴的分子标记技术,可应用于遗传图谱的构建、基因定位、亲缘关系分析和指纹图谱构建等领域[3]。由于该标记分布于整个基因组,具有数量丰富,等位变异高,共显性,检测简单,结果稳定可靠等优点[4],在品种鉴定等方面已到了广泛应用[5-10]。虽然已有对梨品种进行分子标记研究的报道,但尚未见报道关于梨SSR-PCR体系优化的研究,并且由于不同的梨品种所要求的体系并不完全相同,因而无法建立一个完整统一的模式。本次试验采用正交设计L16(45)方法对梨SSR-PCR反应体系的5因素(Taq DNA 聚合酶、Mg2+、模板DNA、dNTPs、引物的浓度)在4水平上进行优化,并对结果用DPS数据处理软件进行较为详细地分析,又用梨的24个品种对此体系进行检测,以期建立一个较为完整可靠的梨属SSR-PCR反应体系,为梨品种鉴定和新品种保护提供依分子生物学依据。
1 材料与方法
1.1 材料
“硕丰梨”品质优良、抗寒性较强,在我省冷凉地区有较大的种植面积,因而本试验采用“硕丰梨”作为试材。该品种叶片取自山西省农科院果树研究所,采回后置于-70℃冰箱备用。引物参照NCBI网站中梨属的EST序列,经MISA软件分析,并用Primer3软件设计获得梨属SSR标记引物,该引物由上海生物工程有限公司合成。
1.2 方法
1.2.1 DNA提取及浓度纯度检测 梨树总DNA提取采用改良的CTAB法[11],在核酸蛋白检测仪(SXABRC,eppendorf-Bio-photometer)上 测 定DNA样本的浓度及纯度。用琼脂糖凝胶电泳检测DNA的质量。将DNA样品置于-20℃下保存备用。

表1 PCR反应的因素和水平
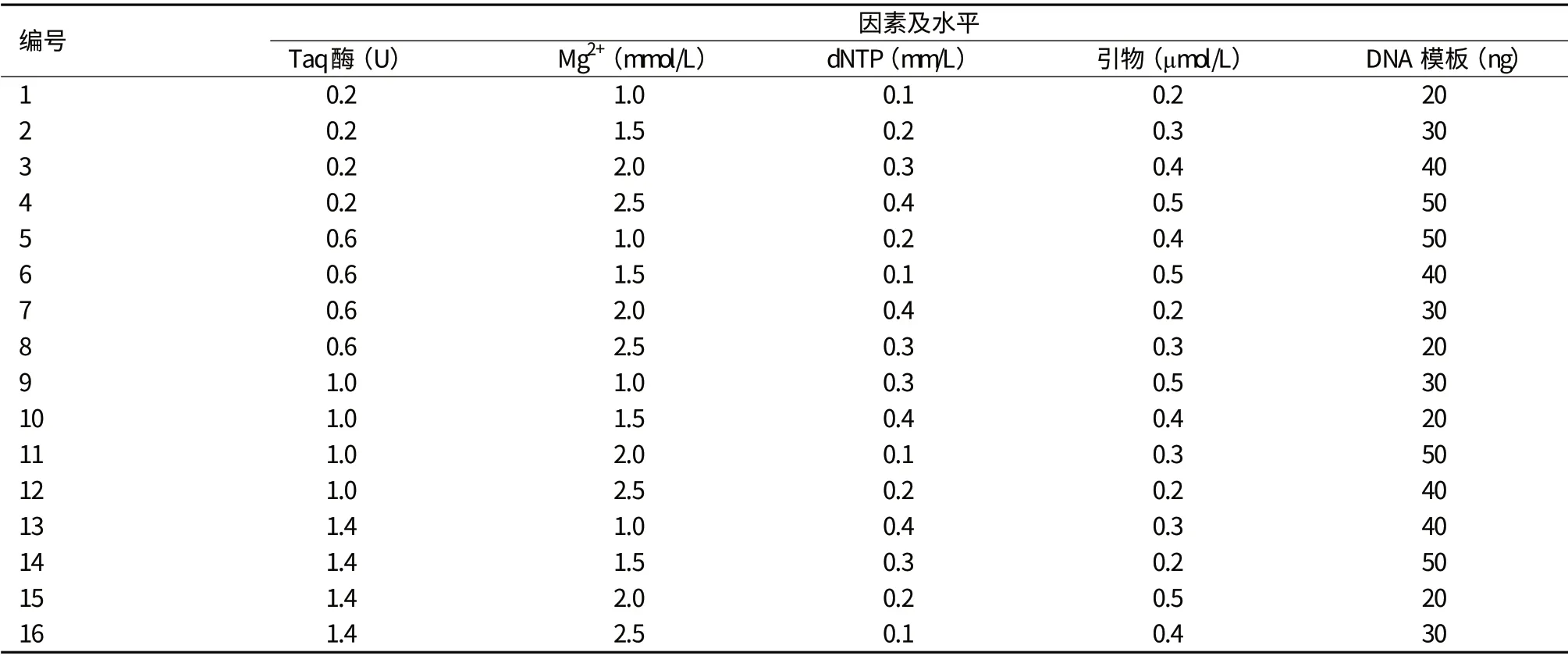
表2 PCR反应因素水平L16 (45 )正交试验设计
1.2.2 PCR反应因素水平的确定与正交表的设计 在PCR反应中,Taq 聚合酶、Mg2+、dNTP、引物、DNA模板是影响反应的5个主要因素。为确定它们在PCR反应中的最佳水平,针对这5个因素,每个因素设4个水平,见表1,采用正交设计L16(45)进行试验[12,13]。设计方案见表2,设3次重复,共48个PCR管,按表中的数据加样;另外每管还加入1 μL的10×PCR Buffer(终浓度为1×PCR Buffer),其余用ddH2O补足,反应体系总体积为10 μL。
1.2.3 PCR扩增及其产物的检测 PCR扩增反应在东胜创新生物科技有限公司的EDC-810型基因扩增仪上进行。反应程序为:94℃预变性3 min ;94℃变性1 min,57℃退火45 s,72℃延伸1 min,35个循环;72℃延伸10 min,4℃保存。以上海生物工程有限公司的Marker为对照,用6%的非变性聚丙烯酰胺凝胶电泳检测,在北京六一仪器厂DYCZ-30C型电泳仪上于300 V恒压下电泳40 min,拆板后观测并拍照分析。参照刘明珍、何正文等[14]的方法,依扩增条带的敏感性和特异性进行计分,分数越高,表示扩增带的敏感性、特异性越好。
1.2.4 最佳反应体系的验证 从梨属的SSR引物中,随机选择1对引物,对梨属24个品种的DNA进行扩增,以验证优化的梨属SSR-PCR反应的稳定性。
2 结果
2.1 DNA质量检测
SSR标记虽然对DNA的质量要求不高,但如果DNA样品不纯则会影响PCR反应,使试验结果不稳定。本试验采用改良的CTAB法提取梨属DNA,所提DNA无色透明,可溶性好。用核酸检测仪检测DNA溶液纯度和浓度,经琼脂糖凝胶电泳(0.8%)检测DNA的质量,观察到条带清晰且无降解,说明提取的DNA质量高,完整性好,可用于SSR分析(图1)。
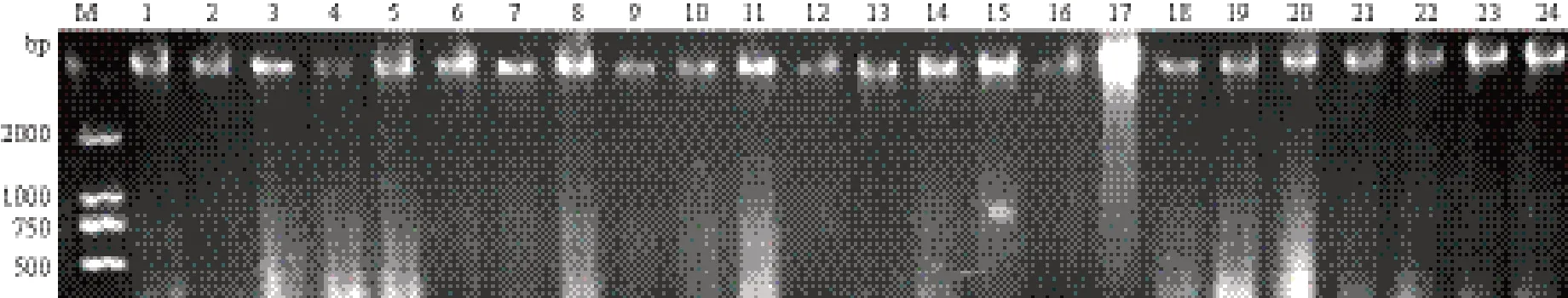
图1 24个梨品种的DNA电泳图
2.2 PCR扩增结果直观分析
2.2.1 梨SSR反应体系的优化设计及扩增结果 按照表2正交设计的16个处理组合,进行PCR反应和电泳检测,结果见图2。结果表明,5种反应因素浓度组合不同,其扩增的效果也不一。唯一相同的是16个处理组合都没有扩增出分子量小于100 bp的引物二聚体。其中2、8、14号处理组合扩增效果较差,条带不清晰且不易观察;6、15、16号处理组合扩增的条带目的条带多且容易观察。根据图片,结合遗传多样性分析的要求,将16个处理组合分别进行打分,即将条带数量丰富、清晰度高、背景低的扩增产物记为16分,而最差的记为1分。由图中1-16处理组合可知,条带数量最丰富且清晰的是6号处理组合,赋值为16,几乎无条带的13号处理赋值为1。按此方法对16个处理组合分别计分为4、3、12、13、10、16、5、2、6、7、8、9、1、11、15、14,并对不同因素与水平的电泳评分平均值和极差进行计算分析,结果如表3所示。
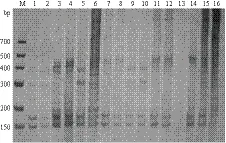
图2 正交体系凝胶电泳图
极差R值的大小表明各因素影响程度的高低,R越大,说明该因素对结果影响越大。因此,该体系中对结果影响的大小顺序是引物、Mg2+、dNTP、DNA模板、Taq聚合酶。其中,每一因素各水平均值大小K,反映了各因素不同水平对反应体系的影响情况,均值越大说明反应水平越好。
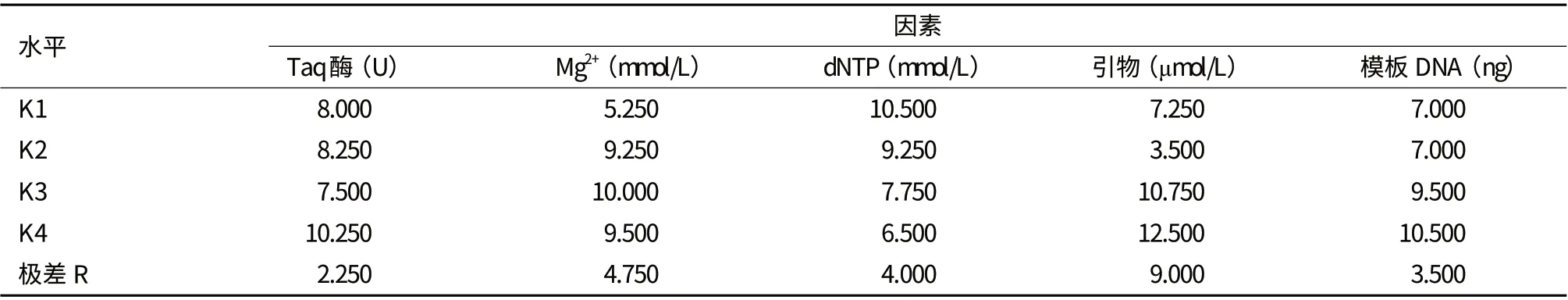
表3 正交设计直观分析
2.2.2 各因素浓度对梨PCR反应结果的影响 由表3可看出,Taq DNA聚合酶浓度处理组合中平均分数最低的是水平3,即Taq DNA聚合酶浓度为1.0 U时平均分值仅为7.500分;而平均分数最高的是水平4,即Taq DNA聚合酶浓度为1.4 U时平均分值最高为10.250分,说明1.4 U Taq DNA聚合酶浓度为梨PCR反应的适宜浓度。Mg2+处理组合中平均分数最低的是水平1,即Mg2+浓度为1.0 mmol/L 时平均分值为5.250分;而分数最高的是水平3,即Mg2+浓度为2.0 mmol/L 时平均分值最高为10.00分,说明2.0 mmol/L Mg2+浓度为梨PCR反应的适宜浓度。同理可得出dNTP最优浓度为0.1 mmol/L、引物最优浓度为0.5 μmol/L、DNA模板最优浓度为50 ng。
2.3 最佳反应体系稳定性检测
应用上述正交试验所获得的最佳反应体系,用1对引物对梨属24个品种的DNA进行扩增,结果如图3显示,每份DNA样品均能扩增出清晰的目的条带。说明该体系比较稳定,适用于梨属植物遗传多样性的SSR标记研究。
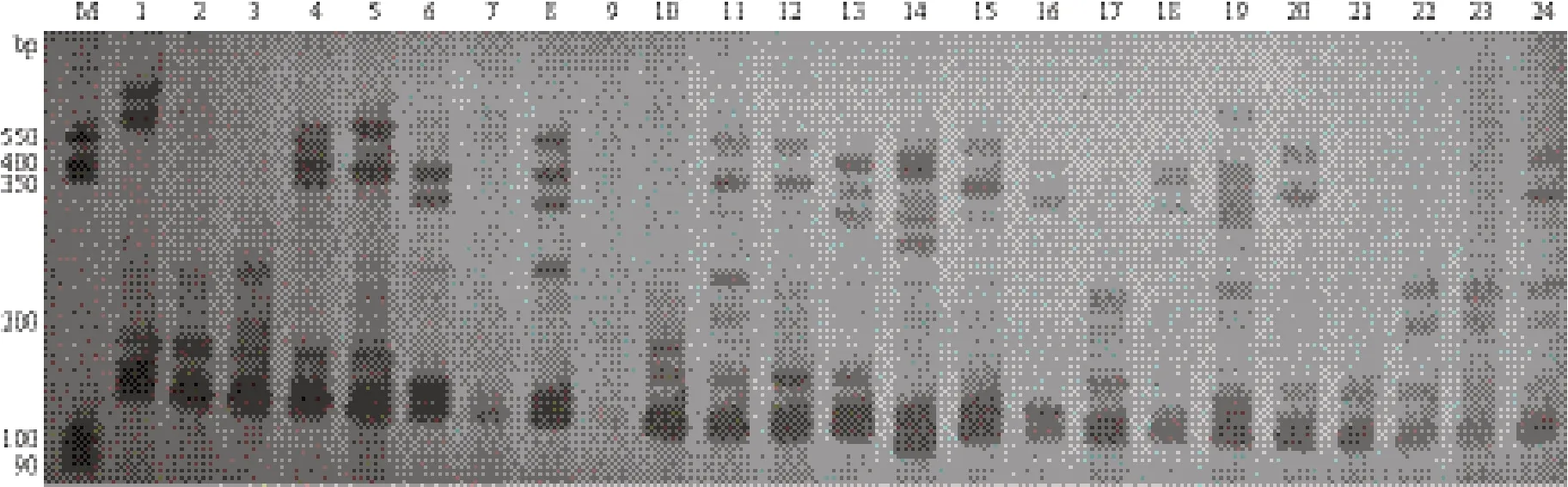
图3 最佳体系的24个梨品种电泳图
3 讨论
为了对梨进行遗传多样性研究,需要获得高质量的DNA。而梨的叶片中也存在大量的糖、酚类等物质,因而用传统的CTAB法所提取的梨DNA质量效果不太好,改良后的CTAB法可将细胞中的多糖,多酚去除,效果较好。
SSR分子标记技术以其便捷可操作性高,重复性高等优点,近年来在植物的种质鉴定、遗传多样性检测、亲缘关系分析和遗传图谱构建等方面得到广泛应用。但SSR-PCR 反应体系同样受到dNTPs、Taq DNA聚合酶、引物、DNA模板用量等因素的影响,例如,模板浓度过低会导致结果不稳定及条带模糊;浓度过高时引物与dNTPs过早耗尽,结果也不稳定。引物浓度偏高会引起碱基错配和非特异性产物的产生,形成引物二聚体。而Taq酶的浓度过高不仅增加试验成本,而且极易产生非特异性扩增产物;浓度过低则会导致产物的合成效率下降[15]。同时,Taq DNA聚合酶是Mg2+依赖性酶,对Mg2+浓度非常敏感。并且dNTP分子中的磷酸基团能定量地与Mg2+结合,使游离的Mg2+浓度降低[16]。因而确定其最优体系,对PCR反应是否成功、所得的结果是否准确,以及PCR的扩增效率都有重要的影响[17]。
传统的单因素试验筛选反应体系,需要进行多次的梯度试验,试验繁琐[18],但是正交试验因在减少试验规模的同时又不损失信息,能更加快速找到最优组合[19],同时也可借助于数学上的统计分析方法对试验结果进行分析与处理,获得可靠的结论,使分析结果更科学、完善和简便。不过不能忽视的是该方法也有不足,对照图片进行打分法,存在一定的主观性,因而也需要有进一步的改进。
4 结论
最终确立梨SSR-PCR最佳的反应体系(10 μL)为:1.0 μL 10×PCR Buffer、Taq DNA聚合酶1.4 U、Mg2+2.0 mmol/L、DNA模板50 ng、dNTPs 0.1 mmol/L、引物0.5 μmol/L。扩增程序:94℃预变性3 min ;94℃变性1 min,57℃退火45 s,72℃延伸1 min,35个循环;72℃延伸10 min,4℃保存。用此反应体系和扩增程序在24份梨品种材料中获得的结果清晰稳定,重复性好。因而初步确定此反应体系可用于梨属植物的PCR扩增分析。
[1] 胡芳名, 谭晓风, 刘惠民.中国主要经济树种栽培与利用[M].北京:中国林业出版社, 2006:402-407.
[2] 甘玲, 汤浩茹, 董晓莉. 梨种类和品种鉴定研究进展[J].中国农学通报, 2006, 22(5):302-306.
[3] 邹喻萍, 葛颂, 王晓东.系统与进化植物学中的分子标记[M].北京:科学出版社, 2000:103-107.
[4] McCouch SR, Temnykh S, Lukashova A, et al. Microsatellite markers in rice:abundance, diversity, and applications[M]. Rice GeneticsⅣ, 2001:117-135.
[5] 韩国辉, 向素琼, 汪卫星, 等. 沙田柚杂交后代群体的SSR鉴定与遗传多样性分析[J].中国农业科学, 2010, 43(22):4678-4686.
[6] 高华, 樊红科, 万怡震, 等. 苹果栽培品种的SSR鉴定及遗传多样性分析[J]. 西北农业学报, 2011, 20(2):153-158.
[7] 陈琛, 张兴桃, 程斐, 等. 秋甘蓝品种的SSR指纹图谱的构建[J]. 园艺学报, 2011, 38(1):159-164.
[8] 王省芬, 马峙英, 潘玉欣, 等. 转基因抗虫棉SSR和AFLP遗传变异研究[J]. 植物遗传资源学报, 2006, 7(2):153-158.
[9] 吴渝生, 杨文鹏, 郑用琏. 3个玉米杂交种和亲本SSR指纹图谱的构建[J]. 作物学报, 2003, 29(4):496-500.
[10] 韩宏伟, 杨敏生, 徐兴兴, 等. 利用SSR标记鉴定主要梨栽培品种[J]. 中国农学通报, 2006, 22(12) 496-500.
[11] 曹丽, 曲柏宏, 王成. 一种新的梨基因组DNA提取方法及其应用[J]. 湖北农业科学, 2006, 45(3):23-25.
[12] 袁志发, 周静芋. 试验设计与分析[M]. 北京:高等教育出版社, 2000:467.
[13] 唐启义, 冯明光.实用统计分析及其DPS 数据处理系统[M].北京:科学出版社, 2001.
[14] 何正文, 刘运生, 陈立华, 等.正交设计直观分析优化PCR条件[J].湖南医科大学学报, 1998, 23(4):403-404.
[15] 沈鹏, 董丽. 大苞萱草ISSR-PCR反应体系的建立与优化[J]. 生物技术通报, 2010(9):129-133.
[16] 张瑞丽, 许玉兰, 王大玮, 等.云南松SSR-PCR反应体系的建立与优化[J].生物技术通报, 2012(4):93-97.
[17] 刘天亮, 潘东明, 许长同, 等. 橄榄ISSR-PCR反应体系的优化[J].生物技术通报, 2010(7):137-141.
[18] 王士磊, 李玉鹏, 高树仁. 正交设计优化玉米SSR-PCR反应体系的研究[J]. 基因组学与应用生物学, 2009, 28(1):119-122.
[19] 续九如, 黄智慧. 林业试验设计[M]. 北京:中国林业出版社, 1998:65-79.

