油桐β-酮脂酰-ACP合酶Ⅲ基因克隆及序列分析
严方方,谭晓风* ,龙洪旭
(1.中南林业科技大学 经济林培育与保护教育部重点实验室,湖南 长沙 410004;2.中南林业科技大学 经济林育种与栽培国家林业局重点实验室,湖南 长沙 410004)
油桐(Vernicia fordii(Hemsl.)Airy-Shaw)是大戟科(Euphorbiaceae)油桐属(Vernicia Lour.),落叶乔木,原产我国,在南方15省(区)均有分布,被列为我国四大木本油料树种之一[1-2]。油桐种子榨出的桐油是优质干性油,化学性质极为活泼,是重要的工业油料,传统上广泛应用于涂料等化学工业[3],还可作为燃料[4-5],是一种优势明显的生物柴油原料[6-7]。近年研究还发现,桐油具有抗肿瘤作用[8]。桐油籽含油率高(50% ~70%),且脂肪酸含有较长的碳直链[9],抗性强,能在贫瘠土地上生长,不与粮食争地,这些优势都使其成为合成生物柴油的优势原料[10-13]。
油桐中的油脂是通过脂肪酸合成途径获得,植物脂肪酸合成途径属于已知两类脂肪酸合成途径中的第2类(FASⅡ)[14]。这一途径中β-酮脂酰-ACP合酶Ⅲ(beta-ketoacyl-ACP synthaseⅢ,KASⅢ)扮演着催化酰基ACP与丙二酸单酰ACP进行脂肪酸合成起始缩合反应(Claisen condensation)的角色[15]。目前,一些植物如蓖麻(Ricinus communis)、麻疯树(Jatropha curcas)和向日葵(Helianthus annuus)的KASⅢ基因已克隆完成[16-18]。植物KASⅢ除了主要以乙酰ACP为底物外,还能以另外一些酰基ACP为底物合成具有不同碳链结构的脂肪酸终产物,这一研究结果跟细菌KASⅢ的研究结果类似,表明在植物中KASⅢ也是脂肪酸碳链结构的主要决定因子之一[19-20]。有研究发现植物中脂肪酸合成的速率受到KASⅢ活性的影响,这一结果表明KASⅢ可能是脂肪酸合成途径的限速酶之一[21]。
1 材料与方法
1.1 实验材料
1.1.1 植物材料 采用的油桐近成熟种子来自湖南省长沙市天际岭林场。2011年8月底,采集对年桐果实,-80℃超低温冰箱保存。
1.1.2 试剂 PureLinkTM RNA Mini Kit,5'RACE System For Rapid Amplification of cDNA Ends,3'RACE System For Rapid Amplification of cDNA Ends试剂盒购自Invitrogen公司;RevertAid First Strand cDNA Synthesis Kit,2 ×Taq PCR MasterMix购自 Fermentas公司;pMD18-T Vector,PrimeSTAR HS DNA Polymerase购自Takara;Gel Extraction Kit购自安比奥公司;pEASY-Blunt Simple Cloning Kit购自全式金公司;引物合成及测序分别由华大基因和博尚公司完成。
1.2 实验方法
1.2.1 总RNA的提取 用Invitrogen公司的PureLinkTMRNA Mini Kit试剂盒提取油桐近成熟种子总RNA。利用质量分数为10 g/L琼脂糖凝胶电泳检测总RNA的完整性。
1.2.2 KASⅢ基因的克隆 根据已克隆的蓖麻(GenBank:XM_002529743)、大豆(GenBank:NM_001250806)、花生(GenBank:EU823328.1)、陆地棉(GenBank:HM236495.1)、麻疯树(GenBank:DQ987701.1)、拟南芥 (GenBank:L31891.1)、向日葵 (GenBank:EF514400.1)、油棕 (GenBank:DQ459441.1)、棕榈(GenBank:DQ459442.1)、紫苏(GenBank:AF026150.1)氨基酸保守区对应的碱基保守区域,利用Primer Premier 5.0软件设计引简并引物(表1)。以油桐近成熟种子总RNA,逆转录后得到第一条链cDNA为模板进行PCR扩增,得到片段序列。在片段序列基础上利用Primer Premier 5.0软件设计3'RACE和5'RACE引物通过巢式扩增得到全长序列(表1)。
1.2.3 PCR程序 以油桐种子的cDNA为模板进行PCR扩增,反应体系25 μL,简并PCR反应条件:94 ℃ 5 min,94 ℃ 40 s,62 ℃ 30 s,72 ℃ 2 min,3个循环;94 ℃ 40 s,60 ℃ /58 ℃ 30 s,72 ℃ 2 min,各15个循环;94℃ 40 s,56℃ 30 s,72℃ 2 min,3个循环,72℃ 8 min;4℃保存。5'末端扩增:94℃5 min,94℃ 40 s,58℃ 30 s,72℃ 2 min,36个循环,72℃延伸8 min;4℃保存。3'末端扩增:94℃5 min,94℃ 40 s,56℃ 30 s,72℃ 2 min,30个循环,72℃ 8 min;4℃保存。全长cDNA扩增:94℃5 min,94 ℃ 40 s,68 ℃ 30 s,72 ℃ 2 min,2 个循环,94 ℃ 40 s,66 ℃ 30 s,72 ℃ 2 min,4 个循环;94 ℃ 40 s,64 ℃ /62 ℃ /60 ℃ 30 s,72 ℃ 2 min,各7个循环;94 ℃ 40 s,58 ℃ 30 s,72 ℃ 2 min,4个循环,94℃ 40 s,56℃ 30 s,72℃ 2 min,4个循环,72℃延伸8 min;4℃保存。
1.2.4 序列分析和结构预测 利用gendoc将序列翻译成氨基酸,在线分析蛋白质和DNA同源性(http://www.ncbi.nlm.nih.gov/BLAST/)。然后利用 Vector NTI 10.3.0 进行 KASⅢ基因序列比对,用MEGA4.0进行多序列比对并计算构建聚类分析图。运用在线分析软件(http://us.expasy.org/tools/protparam.html)分析、预测蛋白质的理化性质,采用在线分析软件 http://www.expasy.org/cgi-bin/protscale.pl中Hphob./Kyte&Doolittle模式对蛋白的疏水性进行分析预测,并运用蛋白质二级结构预测软件(http://www.predictprotein.org/)以及跨膜区拓扑结构预测软件(http://bioweb.pasteur.fr/seqanal/interfaces/toppred.html)对油桐KASⅢ蛋白质二级结构进行分析和预测。
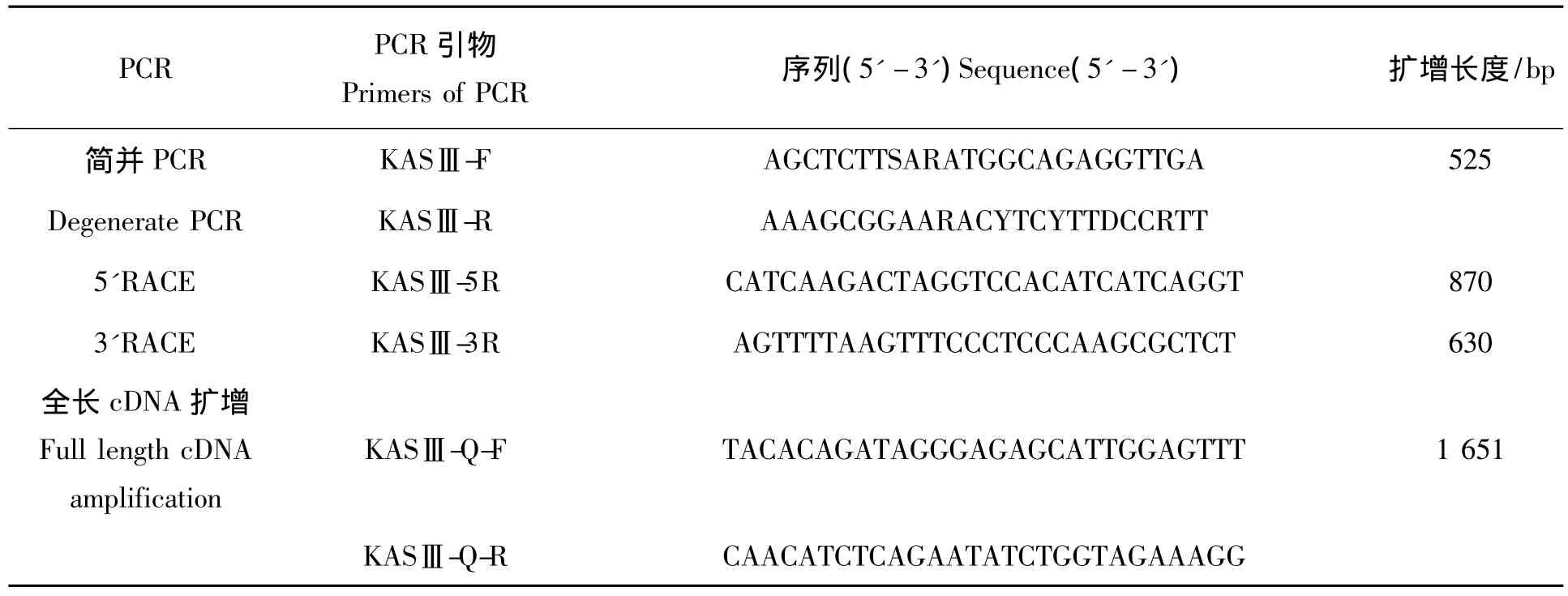
表1 PCR引物及扩增程序Tab.1 Primers used in this study and the processes of PCR
2 结果与分析
2.1 基因克隆与核苷酸序列分析
简并引物扩增获得526 bp的简并片段(图1-Ⅰ)。5'RACE和3'RACE分别得到870,630 bp的片段(图1-Ⅱ、图1-Ⅲ),将3个片段拼接,得到1条完整的cDNA序列,长度为1 901 bp。在5'端和3'端分别设计引物扩增cDNA完整编码区。测序结果表明,该片段长度为1 651 bp(图1-Ⅳ),其序列与拼接序列编码区的一致性为100%。cDNA序列及推导的氨基酸序列见图2,该cDNA含有1个1 209 bp的编码框,编码403个氨基酸。5'非编码区长445 bp,3'非编码区的长度为202 bp,在1 841 bp处发现了加尾信号AATAA,信号后19 bp处出现带有17个多聚A的尾巴,这表明扩增得到了KASⅢ基因的全长序列,在NCBI上进行BLAST比对,其结果也证实实验结果的正确性。

图1 油桐KASⅢPCR扩增结果Fig.1 PCR Products of KASⅢ gene from Vernicia fordii
2.2 蛋白质序列分析

图2 KASⅢ基因cDNA全长序列分析Fig.2 The cDNA completed sequence analysis of KASⅢ

图3 油桐KASⅢ与其他植物KASⅢ 基因编码的氨基酸序列比对Fig.3 Alignment of deduced amino acids of KASⅢ and other KASⅢ-like genes
将克隆得到的KASⅢcDNA序列推导出的氨基酸序列与其他物种的KASⅢ基因所编码的氨基酸通过软件Vector NTI 10.3.0和GENDOC进行同源性分析,发现油桐KASⅢ与其他植物的KASⅢ基因所编码的氨基酸同源性大多在75%以上,其中与麻疯树和蓖麻相似度达90%和87%(图3)。从图4中可以看出,KASⅢ基因在进化上较为保守,与其他植物KASⅢ的相似性较高,不同植物中的KASⅢ蛋白质序列N端保守性低,C端区域保守性较高。
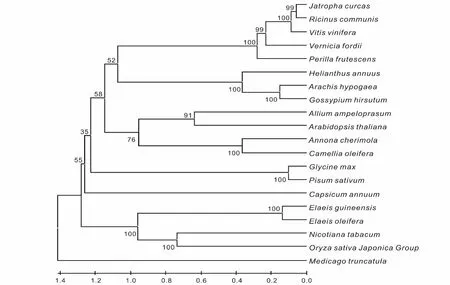
图4 油桐KASⅢ蛋白和其他植物KASⅢ蛋白的聚类分析Fig.4 Phylogenetic tree of the predicted KASⅢ homologous proteins from different species
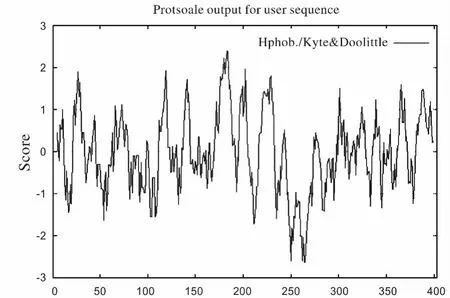
图5 蛋白质疏水性分析Fig.5 Hydrophobicity profile seed
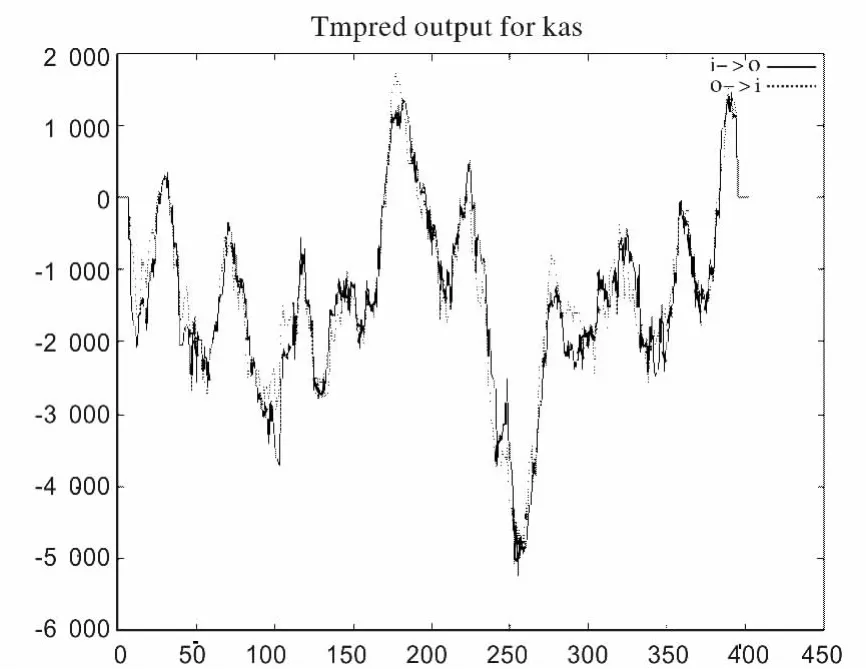
图6 Tmpred对跨膜区预测结果Fig.6 Result of Tmpred prediction
用软件MEGA4.0对20种植物KASⅢ氨基酸序列进行比对,结果显示,油桐KASⅢ基因编码蛋白与麻疯树、蓖麻、葡萄的KASⅢ基因在亲缘进化关系上最近(图4)。KASⅢ蛋白的理化性质预测分析结果表明,该蛋白分子式为C1858H2979N531O585S17,蛋白分子量为42 661.3 kv;它由20种氨基酸组成,等电点为5.78。疏水性分析结果如图5所示,由图可知KASⅢ整个蛋白的疏水指数从-2.644到2.389。大约在183位氨基酸表现出疏水性,整个蛋白质基本上是表现出亲水性的。一般蛋白质的疏水性与跨膜区是相互统一的,图6直观地给出了KASⅢ蛋白的跨膜区域(图中的实线代表跨膜方向由内向外,虚线表示跨膜方向由外向内),分析表明该蛋白有3个比较明显的跨膜区(评分超过500):一个是169~187位氨基酸的跨膜区,跨膜方向由外向内,一个是216~233位氨基酸的跨膜区,跨膜方向由内向外,另一个是380~400位氨基酸的跨膜区,跨膜方向由外向内。Predictprotein网站在线分析软件预测油桐KASⅢ蛋白中α螺旋结构为31.0%,β折叠为18.1%,无规则卷曲及其他结构达到了50.9%。
3 结论与讨论
根据GenBank中已经登录的β-酮脂酰-ACP合酶基因KASⅢ的序列,设计特异性引物,扩增出简并片段,通过5'RACE和3'RACE技术获得了油桐KASⅢ全长cDNA,含1 901 bp,开放阅读框为1 209 bp,编码403个氨基酸,5'UTR和3'UTR分别为445 bp和202 bp,表明该cDNA是一个完整度较好的全长cDNA。
油桐KASⅢ基因是脂肪酸合成的关键基因,因此克隆KASⅢ基因以及今后对其表达调控机理与功能的研究,将为木本植物脂肪酸合成网络的解析提供切入点并有助于更深入的阐述植物脂肪酸合成的分子机理,从而最终将研究结果应用到对油桐的遗传改良中去。另外,KASⅢ对植物油脂的组成成分和特性具有重要影响,因此对油桐KASⅢ基因的研究是油桐分子育种的基础,通过调控KASⅢ基因的表达可以提高种子的含油率,改善桐油的品质,从而加快油桐遗传改良的进程。
目前对油桐的研究主要是高产栽培和良种选育,相关的分子生物学研究还不够深入。利用基因工程手段定向改造油桐,增加桐油的产量,改善桐油的品质,将会极大提高油桐的综合利用能力,然而鉴定和分离脂肪酸合成代谢通路基因是进行基因工程操作的先决条件。
本文对油桐KASⅢ基因的cDNA序列进行了较系统的生物信息学分析,包括序列特征分析、多序列比对、相似性与同源性分析、蛋白质理化特性以及结构预测等,为油桐后续分子生物学研究提供基础数据,为进一步揭示油桐油脂合成规律及良种选育提供参考。
[1]方嘉兴.中国油桐[M].北京:中国林业出版社,1998:22-45.
[2]谭晓风,蒋桂雄,谭方友,等.我国油桐产业化发展战略调查研究报告[J].经济林研究,2011,29(3):1-7.
[3]谭晓风.油桐的生产现状及其发展建议[J].经济林研究,2006,24(03):62-64.
[4]龚榜初,蔡金标.油桐育种研究的进展[J].经济林研究,1996,14(1):5l-53.
[5]何方,何柏,王承南,等.油桐产品质量等级标准制订说明[J].经济林研究,2005,23(4):118-122.
[6]Chang C C,Wan S W.China’s motor fuels from tung oil[J].Ind Eng Chem,1947(39):1543 -1548.
[7]Grossley T D,Heyes T D,Hudson B J F.The effect of heat on pure trilycerides[J].Jaocs,1962(39):9 -14.
[8]Miki I,Temo M.Newly recognized cytotoxic effect of conjugated trienoic fatty acids on cultured human tumor cells[J].Cancer Letters,2000,148:173 -179.
[9]孙颖,卢彰显,李建安.中国油桐栽培利用与应用基础研究进展[J].经济林研究,2007,25(02):84-87.
[10]赵伟,刘拉平,杨健,等.桐油转化生物柴油的研究[J].西北农林科技大学学报,2007,35(11):770-971.
[11]周慧,黄帅,郭召智,等.桐油制备生物柴油的工艺研究[J].应用化工,2007,36(7):633 -635.
[12]龙洪旭,谭晓风,陈洪,等.油桐油体蛋白基因的克隆及序列分析[J].中南林业科技大学学报,2010,30(04):31-38.
[13]李元,汪阳东,李鹏,等.油桐种子FADX基因的克隆和序列分析[J].安徽农业科学,2008,36(11):4753-4755.
[14]Rock C O,Cronan J E.Escherichia coli as a model for the regulation of dissociable(type II)fatty acid biosynthesis[J].Biochim Biophys Acta,1996,1302(1):1 -16.
[15]White S W,Zheng J,Zhang Y M.The structural biology of type II fatty acid biosynthesis[J].Annu Rev Biochem,2005,74:791-831.
[16]Jones A L,Gane A M,Herbert D,et al.Beta-ketoacyl-acyl carrier protein synthase III from pea(Pisum sativum L.):properties,inhibition by a novel thiolactomycin analogue and isolation of a cDNA clone encoding theenzyme[J].Planta,2003,216(5):752-761.
[17]Li J,Li M R,Wu P Z,et al.Molecular cloning and expression analysis of a gene encoding a putative beta-ketoacyl-acyl carrier protein(ACP)synthase III(KAS III)from Jatropha curcas[J].Tree Physiol,2008,28(6):921 - 927.
[18]González-Mellado D,von Wettstein-Knowles P,Garcés R,et al.The role of beta-ketoacyl-acyl carrier protein synthase III in the condensation steps of fatty acid biosynthesis in sunflower[J].Planta,2010,231(6):1277 -1289.
[19]Choi K H,Heath R J,Rock C O.β-Ketoacyl-acyl carrier protein synthase III(FabH)is a determining factor in branchedchain fatty acid biosynthesis[J].J Bacteriol,2000,182(2):365 -370.
[20]Abbadi A,Brummel M,Schütt B S,et al.Reaction mechanism of recombinant 3-oxoacyl-(acyl-carrier-protein)synthase III from Cuphea wrightii embryo,a fatty acid synthase type II condensing enzyme[J].Biochem J,2000,345(1):153 -160.
[21]Dehesh K,Tai H,Edwards P,et al.Overexpression of 3-ketoacyl-acyl-carrier protein synthase IIIs in plants reduces the rate of lipid synthesis[J].Plant Physiol,2001,125(2):1103-1114.

