匍匐茎草本蛇莓RAPD反应体系优化
廖信军,李雅宜,胡雪华
匍匐茎草本蛇莓RAPD反应体系优化
*廖信军,李雅宜,胡雪华
(井冈山大学生命科学学院,江西,吉安 343009)
为了建立蛇莓RAPD反应的最佳反应体系,我们对每个反应因子进行优化研究。在本实验室常用的RAPD反应体系的基础上,根据RAPD扩增效果确定蛇莓基因组DNA的最佳RAPD反应体系。经优化后,我们建立了适合蛇莓的最佳RAPD反应体系:25 μL反应体系中含10×Buffer 2.5 μL,2 mmol/L Mg2+,1.5 UTaqDNA聚合酶,50 ng模板,0.22 mmol/L dNTPs,0.35 μmol/L随机引物S30。本研究结果为下一步野生蛇莓遗传多样评估奠定基础。
蛇莓;RAPD;扩增程序
蛇莓()为蔷薇科多年生草本植物,其性味甘、苦、寒,具有清热凉血、消肿解毒之功效,临床上应用于治疗热病、惊痫、咽喉肿痛、咳嗽吐血、蛇虫咬伤等[1-3]。目前,蛇莓中化学成分分析研究较多[4-6],而有关蛇莓分子遗传标记研究鲜有报道,仅姚旭丽等人2009年进行了蛇莓ISSR-PCR体系的建立和优化的研究[7]。RAPD由Williams和We1sh两个研究小组儿乎同时于1990年建立起来的一项分子标记技术,是目前中药研究者最常使用的DNA标记技术[8-9]。但RAPD易受各种因素的干扰,其最佳扩增条件在不同物种各有不同,因此,在对不同物种进行RAPD分析时,完全有必要对各因子的反应条件进行优化,建立稳定的、最佳的反应体系。本研究将对蛇莓RAPD体系进行优化,为进一步评估野生蛇莓群体遗传多样性奠定基础。
1 材料与方法
1.1 材料
实验材料为野生蛇莓群体,采自吉安市井冈山大学校园内。每个植株上摘取茎尖端的少量幼叶,编号然后置于保鲜袋中,密封保存,放到冰壶内,带回实验室后洗净晾干,并于当日提取基因组DNA。
1.2 基因组DNA提取
参考改良CTAB法[10]提取基因组DNA。
1.3 DNA浓度及纯度检测
提取的DNA先进行1%琼脂糖凝胶电泳,然后经紫外分光光度计(Eppendorf)检测其浓度和纯度,最后用ddH2O稀释至25 ng/μL。
1.4 试剂
试验所用MgCl2、dNTPs、Taq DNA聚合酶均购自上海生工,随机引物由上海生工合成。
1.5 RAPD反应
以蛇莓基因组DNA为模板,对已筛选的S30(5’-3’依次为GTGATCGCAG)随机引物进行扩增(德国Biometra-T3000ThermocyclerPCR仪)。本实验对影响扩增结果的模板浓度、镁离子浓度、Taq酶、引物浓度、dNTPs浓度、退火温度和循环次数等主要因子进行依次研究。在保持其他因子不变的情况下,按一定梯度改变单一因子进行扩增,筛选最优条件,以获得最优化、最稳定的反应体系(各因子梯度见表1)。
1.6 RAPD扩增产物检测
RAPD产物经1%琼脂糖凝胶(含Goldview)电泳(10 V/cm,35 min)分离,DNA Ladder范围为100~3000 bp,YLN-2000凝胶成像仪观察照相。
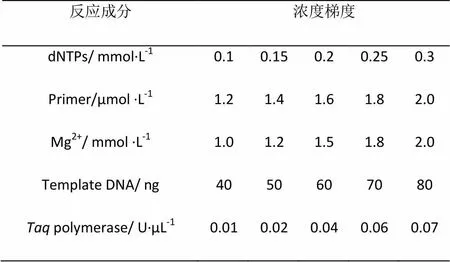
表1 RAPD反应体系各成分优化试验设计
注:反应总体积25μL(含10×buffer 2.5μL)
2 结果与分析
2.1 模板DNA
基本反应体系:2.5 μL 10*Buffer,2 μL 1.8 mmol/L MgCl2,0.5 μL 2.0 U TaqDNA聚合酶,2.5 μL dNTP,0.4 μL primer,dd H2O 补充至25 μL。 DNA优化分5个浓度梯度进行:0.5,1.0,1.5,2.0,2.5 ng/μL。结果见图1:模板DNA用量在0.5~2.5 ng/μL的范围内均能扩增出条带,当浓度为0.5 ng/μL时条带稍淡,浓度为2 ng/μL时条带最清晰。因此,我们最后选取了浓度为2.0 ng/μL (总含量50 ng)为最佳模板浓度。
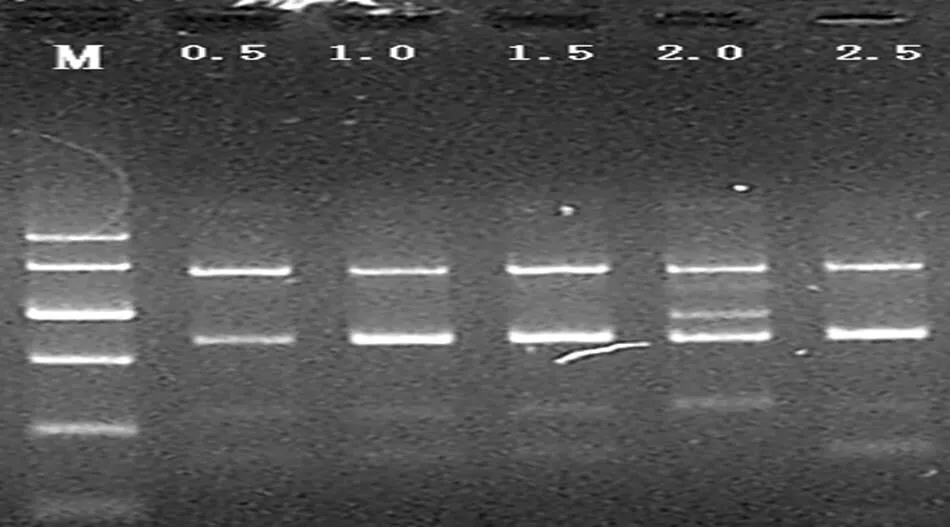
M: SM0321 Marker
2.2 引物
引物浓度低时,无法与所有的模板DNA结合位点结合,导致扩增结果不理想;引物浓度过高时,会增加碱基错配的机率,产生非特异性条带。本研究,基本反应体系:2.5 μL 10*Buffer,2 μL1.8 mmol/L MgCl2,0.5 μL 2.0 U TaqDNA聚合酶,2.5 μL dNTP,50 ng DNA,dd H2O 补充至25 μL,引物设计了5个梯度(依次为0.1,0.2,0.3,0.4,0.5 μmol/L)。结果见图3:引物浓度小于0.2 μmol/L时条带淡且数目明显更少,引物浓度在0.4~0.5 μmol/L较其它浓度更清晰,出于节约成本最终确定的引物采用量为0.4 μmol/L。
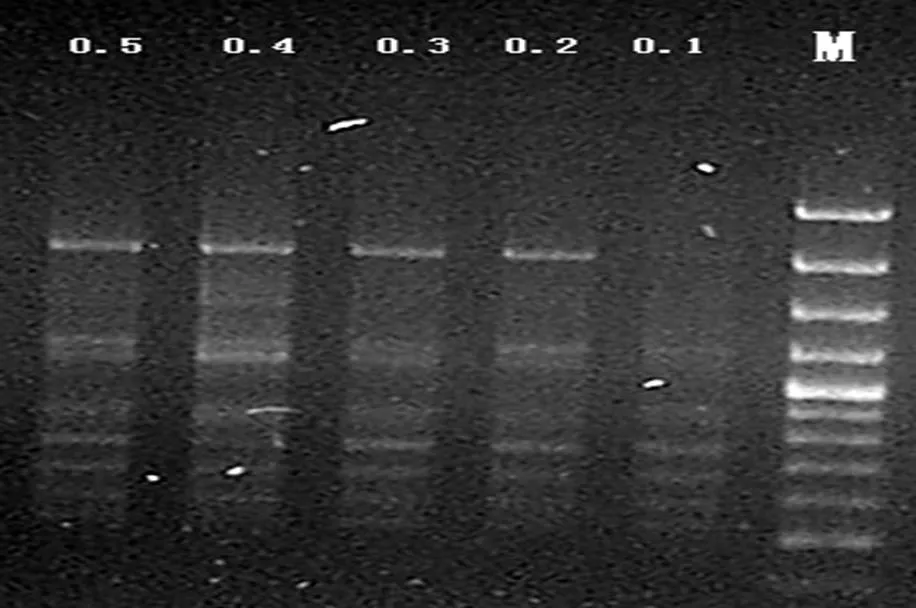
M: SM0321 Marker
2.3 dNTP
dNTPs是反应中磷酸根的主要来源,其浓度大小直接影响产物的质量,特异性及合成的可靠性[11]。本研究基本反应体系:2.5 μL 10*Buffer, 2 μL 1.8 mmol/LMgCl2,0.5 μL 2.0 U TaqDNA聚合酶,50 ng DNA,0.4 μL primer,dd H2O 补充至25μL,dNTP在120~240 μmol/L之间,设置了5个浓度梯度,分别为120,160,200,220,240 μmol/L。结果见图2。
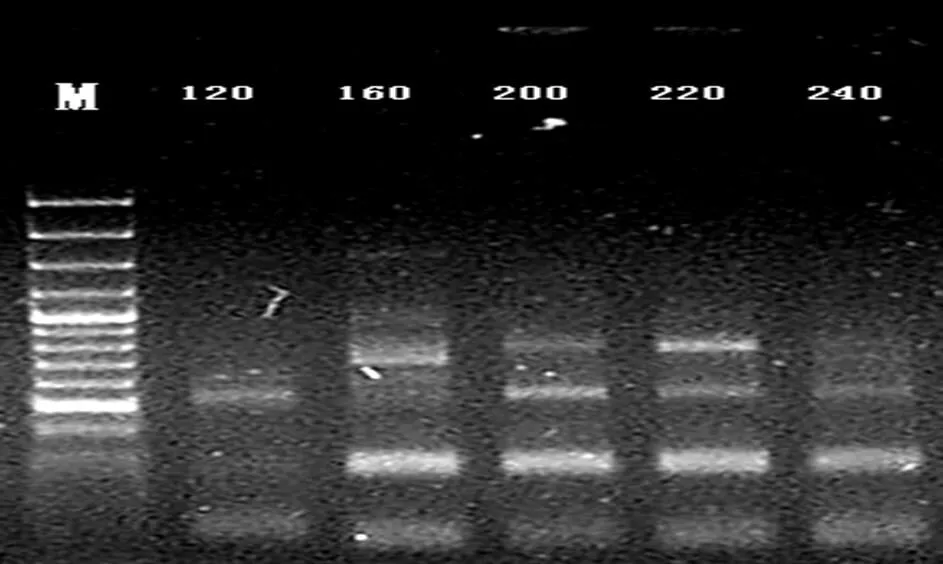
M: SM0321 Marker
注:条带从强到弱的浓度依次为,220,200,160,240,120μmol/L。故,选择dNTP终浓度为220 μmol/L进行RAPD扩增。
2.4 Mg2+浓度
Mg2+对PCR扩增的特异性和产量有显著的影响,浓度过高会使反应特异性降低,浓度过低又会降低Taq酶的活性,使反应产物减少[12]。本研究基本反应体系:2.5 μL 10*Buffer,2.2 μL dNTP,0.5 μL 2.0U TaqDNA聚合酶,50ng DNA,0.4 μL primer,dd H2O 补充至25 μL,Mg2+浓度设置了5个浓度梯度(1.0,1.5,2.0,2.5,3.0 mmol/L)。结果见图4:当Mg2+浓度当Mg2+浓度为1.0 mmol/L时,未见条带;1.5~3.0 mmo1/L之间均能扩增出较亮、较好的条带,在2.5 mmo1/L浓度时条带最清晰。综合考虑,本试验Mg2+最适宜浓度为2.5 mmol/L。
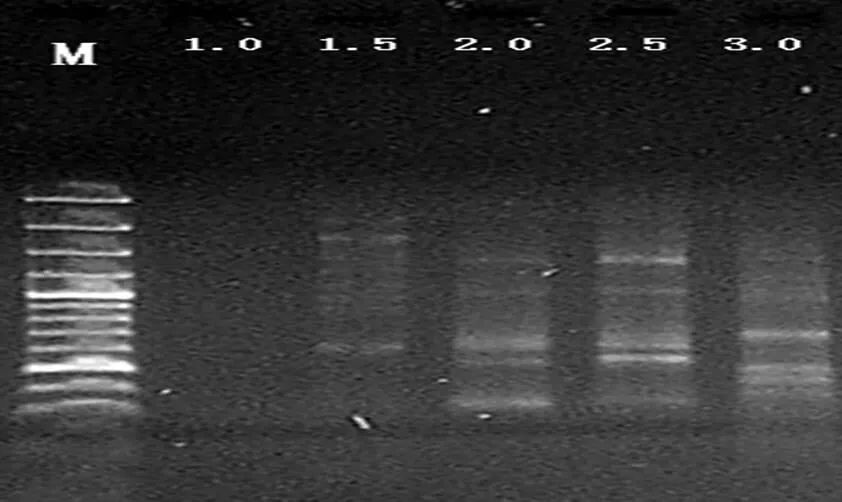
M: SM0321 Marker
2.5 Taq DNA聚合酶
Taq DNA聚合酶是扩增反应的关键因素,基本反应体系:2.5 μL 10*Buffer, 2.2 μL dNTP, 2.5 μL 1.8 mmol/LMgCl2, 50 ng DNA, 0.4 μL primer, dd H2O 补充至25 μL,T aq DNA聚合酶浓度从高到低共设置5个梯度:0.010,0.020,0.040,0.060,0.070 U/μL即总酶量为5个梯度:0.25,0.50,1.00,1.50,1.75 U。实验结果表明(图5):Taq DNA聚合酶具有很高的活性,即使在低浓度下(0.25 U)也有扩增结果,只是条带稍少且不稳定;在高浓度(1.75 U)下带型丰富,出现模糊现象,而且非特异性产物增多;1.50 U扩增出的条带多且清晰,故1.5 UTaqDNA聚合酶量最合适。

M: SM0321 Marker
2.6 反应程序对RAPD扩增的影响
图6显示,第1~9泳道都出现了条带,但条带不多且亮度比较弱,背景比较模糊,第10~12和第16~18泳道背景适宜,但部分条带比较弱且亮度不够,相比之下13~15泳道的条带多且清晰,这3个泳道中15泳道的条带最多且背景最清晰、亮度适宜。综合考虑,第15泳道的反应程序为最佳的反应程序。即预扩增时退火温度为36 ℃;扩增时退火温度为40 ℃,延伸时间为60 s。

图6 反应程序对扩增的影响
注: 1~9延伸时间75 s,10~18延伸时间60 s,1~3和10~12预扩增时退火温度为34 ℃,4~6和13~15预扩增时退火温度为36 ℃,7~9和16~18预扩增时退火温度为38 ℃,1、4、7、10、13、16扩增时退火温度为36 ℃,2、5、8、11、14、17扩增时退火温度为38 ℃,3、6、9、12、15、18扩增时退火温度为40 ℃。M: SM0321 Marker
2.7 优化的PCR反应体系扩增结果的稳定性
采用优化后的RAPD扩增体系对不同株个体DNA进行进行扩增,图7的扩增结果表明,该体系在这11株个体材料中均能扩增出清晰的带型,扩增出的条带较亮,条带数量适宜,且主带清晰,其中8号为用于RAPD条件优化的实验样品。

M: SM0321 Marker
3 讨论
RAPD标记的稳定性和可重复性,一直是受人们关注,这种随机扩增容易受到其反应体系中各成分含量的影响,所以在进行RAPD分析之前,完全有必要对各因子的反应条件进行优化[13]。模板DNA浓度的轻微变化不影响扩增产物的带型,但扩增的强度略有不同,总体来说DNA浓度可允许在一定范围内变动[14]。本文发现蛇莓的模板DNA用量在12.5~62.5 ng的范围内均能获得扩增条带,用量在50 ng时为最佳;随机引物与模板DNA的量之间有一种协同作用,当随机引物达到一定的浓度时,出现随机引物争夺模板DNA的情况,因此必须选择合适的引物浓度。本试验设定的引物浓度的范围相对于范惠玲等人[15]所设定的筛选范围更小,确定最佳引物浓度为0.4 μmol/L;Mg2+与dNTPs的比例应适当,否则会使扩增产物单链化或扩增产物减少导致扩增失败。张秀英等[16]得出Mg2+较小变动可导致扩增产物较大差异,因此确定Mg2+的浓度也是很关键的;Taq聚合酶在实验期间不可随意更换,本研究结果发现不同的酶得出的条带数量和大小均有差异,严奉坤等人(2007)也得出类似结论[17]。
[1] 叔慎.益寿野果蛇莓[J].中国土特产,1999(1):10-15.
[2] 薛红卫.蛇莓药理与临床研究进展[J]. 中国中医药信息杂志,2002,9(9):79-80.
[3] 吴英俊,王超男,刘洁婷,等. 蛇莓中齐墩果酸对肝癌细胞SMMC-7721 的抑制作用[J]. 中国生化药物杂志,2011,32(4):306-308.
[4] 段轲伟. 蛇莓中化学成分的分离与结构鉴定[D].郑州:郑州大学,2012.
[5] 赵萍. 蔷薇科植物蛇莓活性成分的化学研究[D].延边:延边大学,2011.
[6] 董国超. 蛇莓活性成分的分离及研究[D].哈尔滨:哈尔滨工业大学,2011.
[7] 姚旭丽,李钧敏,谢亚琴,等. 蛇莓ISSR-PCR反应体系的建立与优化[J]. 江苏农业科学,2009(2):55-56.
[8] Williams J G, Kubelik A R, Livak K J, et a.l DNA polymorphisms amplified by arbitrary primers are usefμLasgeneticmakers [J].Nucleic AcidsRes, 1990, 25(18): 6531.
[9] Welsh J, Mcclell. Fingerprinting genomes using PCR with arbitrary primers[J].Nucleic-AcidsRes, 1990, 25(18): 7213.
[10] 陈昆松,李方,徐昌杰,等. 改良CTAB法用于多年生植物组织基因组DNA的大量提取[J].遗传,2004,26(4): 529-531.
[11] 李瑞环,李新岗,黄建,等.枣树基因组DNA提取及其RAPD体系优化[J].安徽农业科学, 2007,35(17):5102-5104.5148.
[12] 陈永胜,郝卫国,朱国立,等.蓖麻RAPD- PCR反应体系的正交优化[J].农艺科学,2008,24(3):172-180.
[13] 王关林,方宏筠.植物基因工程原理与技术[M].北京:科学出版社,1998:100-106.
[14] 王丽,乔爱民,孙一铭,等. 菜心基因组DNA提取及RAPD反应体系的优化[J]. 2006, 31(2):124-132.
[15] 范惠玲,孙万仓,孟业雄.芸芥(Eruca. sativa Mill)总DNA提取方法及RAPD扩增体系研究[J].西北农业学报,2007,16(2):18-25.
[16] 张秀英,范亚文.蹄盖蕨科DNA的提取和几种因素对RAPD分析的影响[J].植物研究,2004,24(3):333-340.
[17] 严奉坤,许兴,殷敏,等. 枸杞RAPD反应体系的建立与优化[J].时珍国医国药, 2007, 18(8):1819-1821.
OPTIMIZATION OF RAPD REACTION SYSTEM IN THE STOLONIFEROUS HERB,
*LIAO Xin-jun, LI Ya-yi, HU Xue-hua
(School of Life Sciences, Jinggangshan University, Ji’an, Jiangxi 343009, China)
In order to establish the optimal RAPD reaction system in, every factor in the system was optimized. Based on the RAPD reaction system commonly used in our lab, the optimal RAPD reaction system of genome DNA ofwas determined according to the RAPD amplification effects. The optimal PCR system was as follows: 2.5 μL 10×buffer, 2.0 mmol/L Mg2+, 1.5 U Taq polymerase, 50 ng DNA template, 0.2 mmol/L dNTPs, 0.35 μmol/L random primer S30 in 25μl reaction system. The results of this paper will lay the basis for assessing the genetic diversity of the wild.
; RAPD; amplification procedure
Q785/Q948.1
A
10.3969/j.issn.1674-8085.2013.04.008
1674-8085(2013)04-0037-04
2013-03-01;
2013-06-17
井冈山大学科研项目(JZ0821)
*廖信军(1978-),男,江西吉安人,实验师,博士生,从事分子遗传学研究和教学(E-mail:liaoxinjun7811@126.com);
李雅宜(1987-),男,福建龙岩人,井冈山大学生命科学学院本科生(E-mail:liyayi56@126.com);
胡雪华(1977-),女,江西吉安人,实验师,硕士,主要从事植物学研究(E-mail:huxuehua2008@jgsu.edu.cn ).

