基于机器学习方法的丙型肝炎病毒聚合酶NS5B 非核苷抑制剂的定量构效关系研究
丛 湧 薛 英,2,*
(1四川大学化学学院,教育部绿色化学与技术重点实验室,成都610064;2西华大学四川省先进科学计算重点实验室,成都610039)
1 引言
丙型肝炎病毒(HCV)是通过血液传染的非甲、非乙型肝炎(non-A,non-B viral hepatitis)的主要致病因子.全世界约有1.7亿慢性丙肝病毒感染者,每年有35万余人死于与丙肝相关的肝脏疾病.1,2由于缺少特异有效的药物和疫苗,目前临床上采用α-2-聚乙二醇化干扰素(pegylated interferon-α-2a)与利巴韦林(ribavirin)联合用药治疗丙型肝炎,其治疗有效率仅为50%,治疗周期较长,不良反应发生频率高且比较严重.3HCV的高度变异性使治疗和预防面临巨大困难,急需研发有效的抗HCV药物,以补充完善目前现有的治疗方法.近年来,寻找HCV特定靶向抗病毒治疗药物(STAT-C)是抗HCV研究的重要方向,这些特定靶向的靶点包括NS3/NsS4A蛋白酶/解旋酶和非结构蛋白5B(NS5B)RNA依赖的聚合酶(NS5B RNA-dependent RNA polymerase)等,其中以丙型肝炎病毒NS5B RNA聚合酶为靶标的抗HCV药物研究颇受关注.4
NS5B是一种病毒编码的RNA依赖性RNA聚合酶,可以调控丙型肝炎病毒RNA模板(-)链的合成及(+)链基因组RNA的再生,5在丙型肝炎病毒复制进程中必不可少.研究发现通过对NS5B的抑制可以有效阻断丙肝病毒复制,以NS5B为靶点的抑制剂设计、合成及生物测试越来越成为该领域的研究热点,一系列具有不同骨架结构的核苷型(NIs)、非核苷型(NNIs)NS5B抑制剂被不断的合成及报道,6其中包括苯并咪唑,7吲哚,8噻吩,9吖啶酮衍生物,10苯并噻二嗪11等类型抑制剂.然而,这些研究主要集中在合成新的抑制剂化合物及测试其生物抑制活性,实验周期较长且资金消耗巨大.因此,在早期药物研发过程中,为了缩短药物开发周期和节约成本,我们急需大力发展计算机模拟技术对小分子抑制剂生物活性做出初步定性和定量的预测,并从建立的数据统计模型中挖掘大分子蛋白靶点与小分子配体的可能作用机理,进一步指导和辅助新型抗HCV药物分子的设计和发现.
结构-活性关系研究(SAR)已经成功应用于抗HCV抑制剂的发现.在这些方法当中,定量结构-活性关系(QSAR)研究分子结构与其所表达生物活性之间的相关性,在广泛实践中已被证明是一种非常有效的计算机辅助药物设计工具.与其他方法相比,QSAR方法的优点在于其建立的数据统计模型可以直观指示在生物抑制活性确定中发挥重要作用的小分子抑制剂结构性因素;构建模型所需的分子描述符独立于任何实验条件,可以通过小分子抑制剂三维结构直接计算得出;通过QSAR方法得到的构效关系能够提供非常有用的信息,这些信息可以进一步指导及辅助基于配体(ligand-based)和基于受体(receptor-based)的药物设计.然而,遗憾的是,基于机器学习的二维定量构效关系研究并不能明确揭示生物大分子蛋白受体与小分子抑制剂配体结合的三维构象;在这样的二维定量构效关系研究中,研究人员经常不会考虑样本分子集三维构象的叠合和取向.基于大分子靶蛋白和小分子抑制剂三维构象的3D-QSAR仍然是定量构效关系研究的发展趋势,也是我们工作组未来的研究重点.传统的化学信息学方法多采用多元线性回归(MLR)、启发式方法(HM)、主成分回归(PCR)和偏最小二乘方法(PLS)等线性定量构效关系方法建模.近年来,支持向量机(SVM)和径向基函数网络(RBF)等非线性回归方法在多样性分子结构样本集中对药效学、药代动力学和毒理学性质不断显示其优异的预测性能,12,13正越来越多的受到关注.Melagraki等14对98个苯并噻二嗪衍生物抗HCV抑制剂进行了定量构效研究,通过采用线性逐步回归特征消除选择方法(ES-SWR)从ChemSar和Topix软件计算的分子描述符集中筛选出5个重要的分子描述符建立QSAR模型.他们的QSAR模型对训练集的平方相关系数(R2)仅为0.74,对验证集的R2为0.81.Li研究小组15运用最佳多元线性回归方法(BMLR)选择建模最优描述符子集,并采用多元线性回归、径向基函数神经网络(RBFNN)和支持向量机方法建立线性和非线性QSAR模型对118个苯并噻二嗪衍生物抗HCV抑制剂进行了定量构效关系研究,15其中非线性RBFNN和SVM模型给出更为准确和理想的预测结果,两种非线性模型对于训练集的R2分别为0.850和0.875,对于测试集的R2分别为0.893和0.854.虽然苯并噻二嗪衍生物抗HCV抑制剂的定量构效关系研究已经取得了一些有成效的结果,然而如何选择合适的特征选择和建模机器学习方法仍然是影响QSAR模型预测能力的两个关键因素.本工作研究了89个最新报道的苯并异噻唑(benzoisothiazole)和苯并噻嗪(benzothiazine)类抗HCV抑制剂的定量构效关系.我们尝试使用遗传算法组合偏最小二乘(GA-PLS)和线性逐步回归分析(LSRA)特征选择方法选择最优描述符子集.对每种特征选择方法选择的描述符子集分别采用多元线性回归、偏最小二乘、遗传算法组合支持向量机(GA-SVM)三种方法用训练集建立QSAR模型,并用这些模型预测了测试集中化合物的NS5B抑制活性.
2 材料与方法
2.1 丙型肝炎病毒聚合酶NS5B抑制剂数据集的选取
本工作从最近发表的文献中总共收集了89个苯并异噻唑16,17和苯并噻嗪18-20类丙型肝炎病毒聚合酶NS5B抑制剂.这些化合物的IC50值(1-868000 nmol·L-1)均用相同的实验测量方法在相同的实验条件下测定,被测抑制剂中大部分是高效NS5B抑制剂.首先使用ChemDraw软件绘制每个小分子抑制剂的二维结构,随后通过CORINA软件将小分子二维平面结构转换成三维结构,再采用量子化学AM1计算方法优化每一个抑制剂分子的三维构型,然后对计算结果进行手动检查以确保每个优化分子都生成正确的手性结构且没有重复.为了便于研究,我们将抑制剂的IC50值转换成pIC50值(lg(109/IC50))作为QSAR模型的因变量.根据化合物的结构与化学性质在化学空间中的相似性和分布,21将所有的化合物分为训练集和测试集两大类.训练集(含45个小分子抑制剂)训练和优化回归模型,测试集(含44个小分子抑制剂)评价回归模型的预测能力.
2.2 分子描述符的计算
分子描述符在定量构效关系研究中经常被用于定量描述分子的结构和物理化学特性.在我们的研究中,通过手动方法从相关文献中找出一千多个分子描述符,并根据化合物拓扑结构、电子结构和几何结构等性质,剔除明显冗余和与预测药物性质不相关的描述符,22最终筛选出189个与化合物性质密切相关的分子描述符(见表1).其中包括18个简单分子性质描述符(如分子量、可旋转的键数),27个分子连接性和形状描述符(如分子连接性指数和分子卡伯形状指数),97个电拓扑态分子描述符(如电子拓扑态指数),22个量子化学性质分子描述符(如原子电荷和分子的偶极矩)和25个分子几何特性描述符(如溶剂可及表面积和疏水区域).我们应用实验室自编的分子描述符计算程序,根据AM1方法优化过后的化合物三维结构计算所有的分子描述符.为了减少建模中描述符之间高度自相关带来的多重共线性干扰,在特征选择之前对这189个分子描述符集做了预处理,步骤如下:(1)移除在90%的样本分子中具有相同数值的描述符;(2)移除相对标准偏差小于0.05的描述符;(3)对于Pearson相关系数超过0.95的一对描述符,留下与生物活性相关性较高的描述符,剔除另外一个.23经过上述预处理,我们最终保留了85个分子描述符进行下一步的特征选择.
2.3 特征选择方法
2.3.1 逐步回归分析法
我们借助SPSS软件自带的逐步回归分析程序选择最优描述符子集,逐步回归分析法中每步有两个过程即引进变量和剔除变量,且引进变量和剔除变量均需作F检验后方可继续进行,故又称为双重检验回归分析法.其具体步骤如下:(1)引入变量,引入变量的原则是未引进变量中偏回归平方和最大者并经过F显著性检验,若显著则引进,否则终止.(2)剔除变量,剔除原则是在引进的自变量中偏回归平方和最小者,并经过F检验不显著,则剔除.(3)终止条件即最优条件,再无显著自变量引进,也没有不显著自变量可以剔除.
2.3.2 遗传算法组合偏最小二乘方法
我们借助MATLAB偏最小二乘-遗传算法工具箱24实现最优描述符子集的选取.GA-PLS是一种基于遗传算法的优化工具,25,26其算法过程描述如下:(1)定义和编码染色体;(2)种群的初始化;(3)评价每个染色体的适应度;(4)保护染色体;(5)保留最好的染色体;(6)对种群进行交叉和变异遗传操作;(7)若满足终止条件停止程序,否则转入步骤3.本实验采用了GA-PLS工具箱里的三个函数,它们分别是GAPLSOPT(dataset,1),GAPLSOPT(dataset,2)和GAPLS(dataset,the number of evaluation,precision).
GAPLSOPT(dataset,1)函数测试样本数据集是否适用GA-PLS方法选取特征,根据GA-PLS软件设计者介绍,如果GAPLSOPT函数对于样本数据集的测试输出结果在0到5之间,使用GA-PLS方法对数据集进行特征选择是安全稳定的.图1中显示了本工作89个小分子抑制剂对应的85个分子描述符样本数据集的GAPLSOPT(dataset,1)输出结果,样本数据集的随机测试结果在0到4.9468之间,这说明采用GA-PLS方法对该数据集进行特征选择是可靠的.为了避免GA-PLS方法在训练过程中产生过拟合,我们使用GAPLSOPT(dataset,2)函数估计GAPLS函数所需的最优评价次数(the number of evaluation)参数,如图2所示,GAPLSOPT(dataset,2)的差异曲线在评价次数为115处有全局最大值,因此,评价次数被置为115作为GAPLS函数的最优控制参数.经过上述准备工作,我们运行GAPLS函数对数据集进行特征选择.为了减少随机误差,我们重复了10次GAPLS实验得到平均结果.图3显示了交叉验证响应和每个描述符的被选择频率,在85个分子描述符中,GAPLS函数最终选出7个分子描述符用于下一步的QSAR建模.
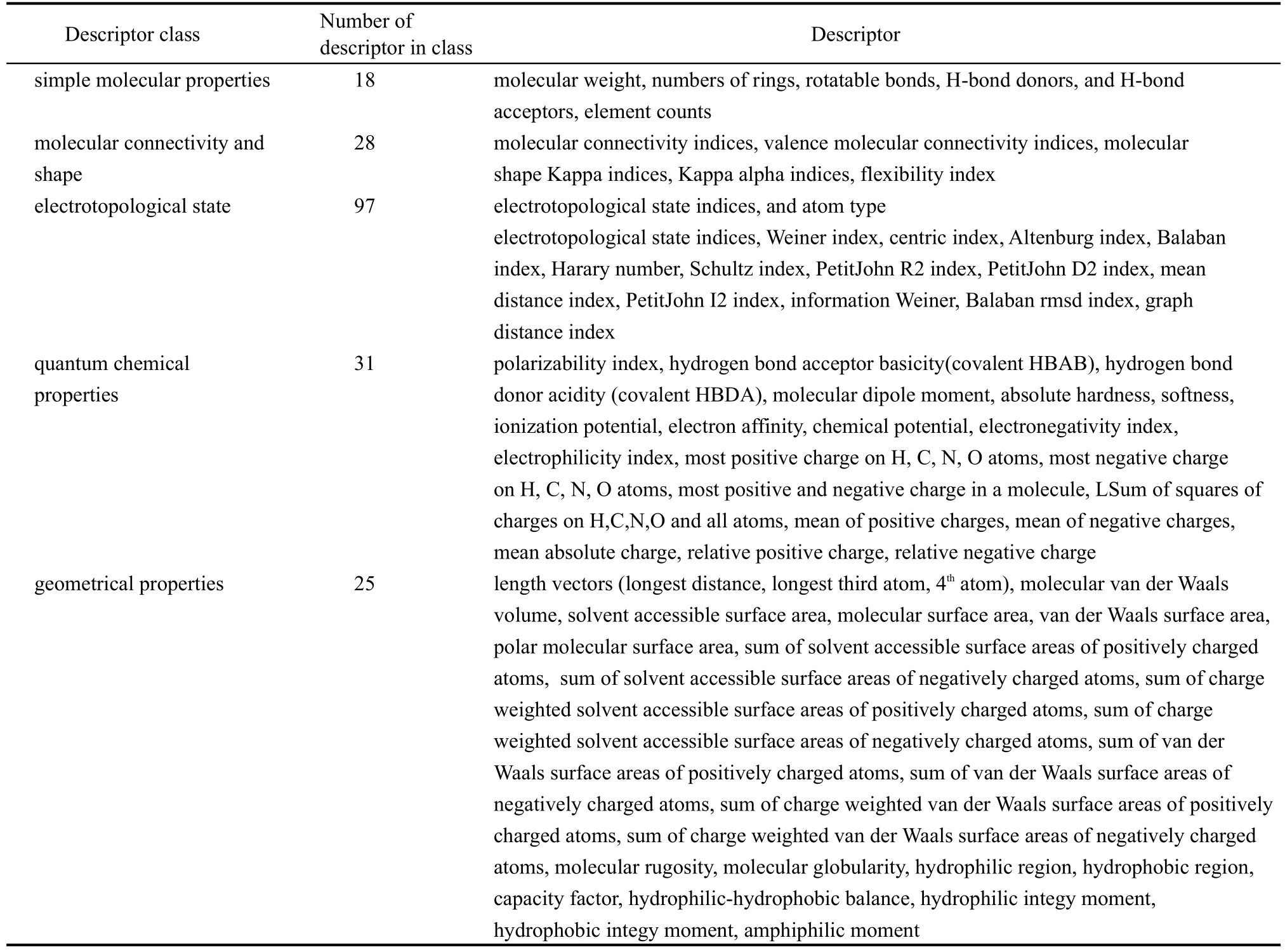
表1 所用的分子描述符Table 1 Molecular descriptors used in this work
2.4 遗传算法组合支持向量机方法

图2 GAPLSOPT(2)差异曲线Fig.2 GAPLSOPT(2)difference curve
支持向量机是基于结构风险最小化(SRM)理论的机器学习方法,其两分类理论经过多年发展已日趋成熟.通过引入ε-不敏感损失函数,支持向量机也可以扩展用来解决小样本数据集的回归问题.支持向量机经由核函数K(xi,x)将输入空间的X向量映射到高维希尔伯特空间H,其常用的核函数包括线性、多项式、径向基和S形等函数形式,其中径向基函数由于良好的非线性映像能力已在诸多领域得到了广泛应用.27本研究使用高斯径向基函数(RBF)构建支持向量机回归模型.高斯核函数具体表示如下:

我们在K(xi,x)特征空间构造决策函数(方程2)作为ε-支持向量机的最优解:

其中b为方程的偏置项.

图3 GAPLS函数描述符选择频率估计Fig.3 Selected frequency figure by GAPLS function
支持向量回归机的泛化性能取决于最优正则化参数C,不敏感参数ε和RBF核函数宽度σ的选取.28正则化参数C对回归函数的复杂性和泛化能力进行折衷.在确定的数据子空间中,参数C取得太小,则对样本数据中超出ε不敏感带的样本惩罚就越小,使训练误差变大,系统的泛化能力变差,会出现“欠学习”现象;C取得太大,相应的权重就小,系统的泛化能力变差,会出现“过学习”现象.不敏感参数ε控制着ε不敏感带的宽度,影响着支持向量的数目.ε值选得太小,回归估计精度高,但支持向量数目增多,ε选的太大,回归估计精度降低,支持向量数目减少,支持向量机的稀疏性大.RBF核函数宽度σ反映了训练样本数据的分布或范围特性,它确定了局部领域的宽度,较大的σ意味着较低的方差.
遗传算法是模拟达尔文生物进化论自然选择和遗传学机理的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优化解的方法.29本工作中我们采用遗传算法同时对支持向量回归机的三个参数(正则化参数C,不敏感参数ε和RBF核函数宽度参数σ)进行优化.新型进化计算框体—Python环境下分布式进化算法(DEAP)30被用来实现SVM参数优化的遗传算法框架,Libsvm程序31被用于构建ε-SVM回归模型,通过引入Python多路处理模块(The multi-processing techniques in Python),我们实现了GA-SVM算法的并发,大大提高了算法的计算速度.在遗传算法中,染色体使用二进制编码,每个染色体由C:(Ci,i=1-nc),ε:(εj,j=1-nε)和σ:(σk,k=1-nσ)三部分组成,Ci表示染色体中表征正则化参数C二进制位串中第i位的数值,σk表示染色体中表征RBF核函数宽度参数σ二进制位串中第j位的数值,εk表示染色体中表征不敏感参数ε二进制位串中第k位的数值,nc、nσ和nε分别表示染色体中表征C、σ和ε的二进制位串的长度(在本工作中nc=10,nσ=10,nε=10).根据解码公式(5),这三个二进制编码位串被分别转换成正则化参数C、不敏感参数ε和RBF核函数宽度参数σ的实数值.

其中R表示二进制位串所表征相应参数的真实值,maxR和minR为相应参数最大和最小指数幂取值范围(参数取值在2minR和2maxR
之间),d为二进制位串转十进制数值,l为二进制位串的长度.我们设置C的取值范围为2-10-215;ε的取值范围为:2-10
-210;σ的取值范围为2-10-28;n重交叉验证均方误差(MSECV)作为遗传算法的适应度函数,定义如下:

其中,yi是实验真实值,ŷi是模型的计算值,ntest是n重交叉验证集的样本化合物数目.遗传算法的选择、交叉和变异操作,我们通过调用DEAP内置函数——selTournament(individuals,k,tournsize)、cxTwo Points(ind1,ind2)和mutFlipBit(individual,indpb)来实现,设置种群规模为500,总共迭代100代,交叉率和变异率分别设置为0.6和0.2.当某代种群中95%以上的个体得到相同的均方误差适应度数值停止算法,作为GA-SVM的停机准则.
3 结果和讨论
3.1 两种特征选择方法选出的最优描述符子集及讨论
逐步回归分析法共选出6个分子描述符,其中包括2个简单分子性质描述符、1个分子连接性和形状描述符、2个量子化学性质描述符和1个分子几何特性描述符;遗传算法组合偏最小二乘方法共选出7个分子描述符,其中包括3个简单分子性质描述符、1个分子连接性和形状描述符、1个电拓扑态分子描述符、2个量子化学性质描述符和1个分子几何特性描述符.如表2所示,两种特征选择方法同时选出4个相同的分子描述符,分别为分子中杂原子数目(nhet)、氧原子数目(noxy)、分子最大负电荷(AQ,min)和亲水性指数(Hiwpl).
起初苯并噻嗪类抑制剂的构性关系研究集中在苯并噻嗪环的C-7位,18通过在该位置引入取代基来改善抑制活性.研究发现在苯并噻嗪环的C-7位引入极性基团如羟基或甲基磺酰胺基会提高抑制剂对酶NS5B的抑制效力.RNA聚合酶NS5B和其抑制剂的复合物晶体结构进一步证实在1,3-二羰基功能基团上的负电荷易在环上发生离域与聚合酶NS5B骨架上Tyr448残基的N-H基团及两个保守水分子反生静电相互作用;与此同时,苯并噻嗪环中的苯环正好与Phe193残基面对面接触,苯环之间发生π-π堆积作用.在C-7位引入甲基磺酰胺基的苯并噻嗪类抑制剂中,砜氧基团与桥接Ser556残基和甲基磺酰胺基团的结构水分子形成氢键静电相互作用;磺酰胺基团也会与NS5B的Asn291残基形成静电相互作用;氨磺酰基团中酸性的N-H与NS5B Asp318残基产生更强有力的氢键相互作用力,磺酰胺基团的这种独特的三点式相互作用有助于解释抑制剂中该极性基团的引入对于抑制活性的改善.构性关系还发现在苯并噻嗪环的C-2和C-5位掺入吸电子基团降低环电子密度,可以有效改善胆汁的转运识别,并同时减少代谢负产物的生成.20
在所选描述符中,nhet(分子中所含N,O和S等杂原子数目)、noxy(分子中O原子数目)和nsulph(分子中S原子数目)这三个简单性质描述符间接表征了抑制剂分子中极性基团(如羟基或甲基磺酰胺基等)与NS5B发生静电相互作用及形成氢键的能力;亲水性指数(Hiwpl)描述抑制剂分子与NS5B酶的亲水疏水相互作用;两个量子化学描述符,分子最大负电荷(AQ,min)和分子平均负电荷(Mnc)可能与苯并噻嗪环的电子离域化及C-2,C-5位上吸电基团的引入有关;S(1)(羟基H原子电拓扑态指数)和QH,Max(分子中氢原子上的最大正电荷)给出氢键给体的质子提供能力.从上面的讨论可以看出,两种方法所选择的描述符能够反映抑制剂分子的电荷分布、氢键相互作用、分子连接性、亲水疏水相互作用等性质.

表2 LSRA和GA-PLS特征选择方法选取的分子描述符Table 2 Molecular descriptors selected from the LSRAand GA-PLS feature selection methods
3.2 逐步回归分析法所选描述符的建模回归结果
我们采用逐步回归分析法所选的6个最优描述符分别建立多元线性、偏最小二乘和支持向量机回归模型,训练集(含45个化合物)用来训练和优化模型,测试集(含44个小分子抑制剂)评价回归模型的预测能力.训练得到的多元线性回归方程为:pIC50=0.329×nhet+0.383×noxy-0.16723.903×Mnc+0.051×Hiwpl-5.588,0.931,s2=0.144,F=99.473,为调整相关系数平方,s2为回归分析的标准偏差平方,即回归分析方差;F为回归分析的显著性检验.图S1(Supporting Information)为多元线性回归模型对于训练集和测试集的实验与预测pIC50数值对比图,模型对于训练集、测试集及整个数据集的均方误差(MSE)分别为0.121、0.122和0.122,相应的相关系数分别为0.970、0.958和0.965.偏最小二乘方法抽选出3个主成分,训练得到的回归方程为:pIC50=0.2153×nhet+0.3652×noxy-0.1463×Mnc+0.1519×Hiwpl-7.3150s2=0.166,F=85.454,N=45,图S2为偏最小二乘回归模型对于训练集和测试集的实验与预测pIC50数值对比图,模型对于训练集、测试集及整个数据集的MSE分别为0.140、0.122和0.131,相应的相关系数分别为0.965、0.958和0.961.由于影响NS5B酶抑制剂活性的分子特征极其复杂,并不是所有的分子描述符都与活性存在绝对的线性关系,为了与上述线性回归建模方法做比较,基于相同的最优描述符子集,我们使用遗传算法组合支持向量机方法建立非线性QSAR模型.首先,我们在训练集上使用GASVM方法同时优化SVM模型的三个参数,当SVM模型参数取值为:C=7.7387,σ=0.3546,ε=0.04664时,回归模型给出最好的留一法交叉验证误差(MSECV=0.135304);然后,我们采用这组优化参数建立SVM模型.图S3为SVM回归模型对于训练集和测试集的实验与预测pIC50数值对比图,模型对于训练集、测试集及整个数据集的MSE分别为0.113、0.108和0.111,相应的相关系数分别为0.972、0.962和0.968.在上述三种方法建立的QSAR模型中,支持向量机给出最好的回归模型,而多元线性回归给出预测效果最好的线性回归模型,三种机器学习模型对抑制剂活性的预测结果详见表S1.
3.3 遗传算法组合偏最小二乘法所选描述符的建模回归结果
我们采用GA-PLS所选的7个最优描述符子集分别建立偏最小二乘、多元线性和支持向量机回归模型.训练得到的多元线性回归方程为:pIC50=0.207×nhet+0.295×nsulph+0.304×noxy-0.035×S(1)+F=76.566,N=45,图S4为多元线性回归模型对于训练集和测试集的实验与预测pIC50数值对比图,模型对于训练集、测试集及整个数据集的MSE分别为0.131、0.243和0.186,相应的相关系数分别为0.967、0.918和0.946.偏最小二乘方法抽选出3个主成分,训练得到的回归方程为:pIC50=0.1667×nhet+0.5267×nsulph+0.2915×noxy+0.0220×S(1)+8.1236×QH,Max-8.7093×AQ,min+0.0827×Hiwpl-3.7535,R2=0.933,R2adjust=0.920,s2=0.163,F=73.606,N=45,图S5为偏最小二乘回归模型对于训练集和测试集的实验与预测pIC50数值对比图.模型对于训练集、测试集及整个数据集的MSE分别为0.134、0.114和0.124,相应的相关系数分别为0.966、0.960和0.964.基于相同的最优描述符子集,使用遗传算法组合支持向量机方法建立非线性QSAR模型,当SVM模型参数取值为:C=11.620579,σ=0.182558,ε=0.224924时,回归模型给出最好的留一法交叉验证误差(MSECV=0.145978),图S6为SVM回归模型对于训练集和测试集的实验与预测pIC50数值对比图,模型对于训练集、测试集及整个数据集的MSE分别为0.123、0.118和0.121,相应的相关系数分别为0.970、0.958和0.965.在上述三种方法建立的QSAR模型中,支持向量机回归模型对于训练集和整个数据集给出最好的预测结果;两种线性回归模型中,偏最小二乘模型对测试集和整个数据集给出最好的预测结果,而多元线性回归模型对于训练集给出最好的预测效果,三种机器学习模型对抑制剂活性的预测结果详见表S2.
4 结论
本工作研究了89个苯并异噻唑和苯并噻嗪类抗HCV抑制剂的定量构效关系.线性逐步回归分析和遗传算法组合偏最小二乘方法被用来选取最优描述符子集,对于如上两种特征选择方法所选描述符,我们分别采用多元线性回归、偏最小二乘、遗传算法组合支持向量机方法建模并得到了比较满意的预测结果.在采用LSRA所选描述符建立的三个QSAR模型中,支持向量机给出最好的回归模型,多元线性回归给出预测效果最好的线性模型;在采用GA-PLS所选描述符建立的三个QSAR模型中,支持向量机同样给出最好的回归模型,而偏最小二乘回归给出效果最好的线性模型.研究结果表明,非线性支持向量机方法的建模回归效果好于传统的多元线性回归和偏最小二乘方法;采用LSRA和GA-PLS特征选择方法所选描述符建立的模型都得到比较满意的回归效果,这表明两种特征选择方法都能筛选合适的描述符子集用于QSAR建模;多元线性回归和偏最小二乘方法建立的线性QSAR方程可以直观指示在生物抑制活性确定中发挥重要作用的小分子结构和物理化学特征信息,这些信息可以进一步指导及辅助基于配体和受体的抗HCV药物设计.
Supporting Information: The plots of experimental vs computational lg(109/IC50)values are given in Figs.S1-S6.The information of the investigated dataset is provided in Tables S1 and S2.This information is available free of charge via the internet at http://www.whxb.pku.edu.cn.
(1) Choo,Q.L.;Weiner,A.J.;Overby,L.R.;Bradley,D.W.;Houghton,M.Science 1989,244,359.doi:10.1126/science.2523562
(2)(a)Lauer,G.M.;Walker,B.D.N.Engl.J.Med.2001,345,41.doi:10.1056/NEJM200107053450107(b)Di Bisceglie,A.M.Lancet 1998,351,351.(c)Alter,M.J.;Kruszon-Moran,D.;Nainan,O.V.;McQuillan,G.M.;Gao,F.;Moyer,L.A.;Kaslow,R.A.;Margolis,H.S.N.Engl.J.Med.1999,341,556.
(3)Manns,M.P.;McHutchison,J.G.;Gordon,S.C.;Rustgi,V.K.;Shiffman,M.;Reindollar,R.;Goodman,Z.D.;Koury,K.;Ling,M.H.;Albrecht,J.K.Lancet 2002,347,975.
(4) (a)Koch,U.;Narjes,F.Curr.Top.Med.Chem.2007,7,1302.doi:10.2174/156802607781212211(b)Rönn,R.;Sandström,A.Curr.Top.Med.Chem.2008,8,533.(c)Zapf,C.W.;Bloom,J.D.;Levin,J.I.Ann.Rep.Med.Chem.2007,42,281.
(5) Appel,N.;Schaller,T.;Penin,F.;Bartenschlager,R.J.Biol.Chem.2006,281,9833.doi:10.1074/jbc.R500026200
(6) Ni,Z.J.;Wagman,A.S.Curr.Opin.Drug Discov.Dev.2004,7,446.
(7) Beaulieu,P.L.;Bos,M.;Bousquet,Y.;Fazal,G.;Gauthier,J.;Gillard,J.;Goulet,S.;LaPlante,S.;Poupart,M.A.;Lefebvre,S.;McKercher,G.;Pellerin,C.;Austel,V.;Kukolj,G.Bioorg.Med.Chem.Lett.2004,14,119.doi:10.1016/j.bmcl.2003.10.023
(8) Stansfield,I.;Ercolani,C.;Mackay,A.;Conte,I.;Pompei,M.;Koch,U.;Gennari,N.;Giuliano,C.;Rowley,M.;Narjes,F.Bioorg.Med.Chem.Lett.2009,19,627.doi:10.1016/j.bmcl.2008.12.068
(9)Louise-May,S.;Yang,W.;Nie,X.;Liu,D.;Deshpande,M.S.;Phadke,A.S.;Huang,M.;Agarwal,A.Bioorg.Med.Chem.Lett.2007,17,3905.doi:10.1016/j.bmcl.2007.04.103
(10) Stankiewicz-Drogon,A.;Palchykovska,L.G.;Kostina,V.G.;Alexeeva,I.V.;Shved,A.D.;Boguszewska-Chachulska,A.M.Bioorg.Med.Chem.2008,16,8846.doi:10.1016/j.bmc.2008.08.074
(11) Bosse,T.D.;Larson,D.P.;Wagner,R.;Hutchinson,D.K.;Rockway,T.W.;Kati,W.M.;Liu,Y.;Masse,S.;Middleton,T.;Mo,H.;Montgomery,D.;Jiang,W.;Koev,G.;Kempf,D.J.;Molla,A.Bioorg.Med.Chem.Lett.2008,18,568.doi:10.1016/j.bmcl.2007.11.088
(12) Lü,W.J.;Chen,Y.L.;Ma,W.P.;Zhang,X.Y.;Luan,F.;Liu,M.C.;Chen,X.G.;Hu,Z.D.Euro.J.Med.Chem.2008,43,569.doi:10.1016/j.ejmech.2007.04.011
(13)Luan,F.;Liu,H.T.;Ma,W.P.;Fan,B.T.Euro.J.Med.Chem.2008,43,43.doi:10.1016/j.ejmech.2007.03.002
(14) Melagraki,G.;Afantitis,A.;Sarimveis,H.;Koutentis,P.A.;Markopoulos,J.;Igglessi-Markopoulou,O.Bioorg.Med.Chem.2007,15,7237.doi:10.1016/j.bmc.2007.08.036
(15) Su,L.;Li,L.;Li,Y.;Zhang,X.;Huang,X.;Zhai,H.Med.Chem.Res.2012,21,2079.doi:10.1007/s00044-011-9734-x
(16) deVicente,J.;Hendricks,R.T.;Smith,D.B.;Fell,J.B.;Fischer,J.;Spencer,S.R.;Stengel,P.J.;Mohr,P.;Robinson,J.E.;Blake,J.F.;Hilgenkamp,R.K.;Yee,C.;Adjabeng,G.;Elworthy,T.R.;Li,J.;Wang,B.;Bamberg,J.T.;Harris,S.F.;Wong,A.;Leveque,V.J.P.;Najera,I.;Pogam,S.L.;Rajyaguru,S.;Ao-Ieong,G.;Alexandrova,L.;Larrabee,S.;Brandl,M.;Briggs,A.;Sukhtankar,S.;Farrell,R.Bioorg.Med.Chem.Lett.2009,19,5652.doi:10.1016/j.bmcl.2009.08.022
(17) Hendricks,R.T.;Spencer,S.R.;Blake,J.F.;Fell,J.B.;Fischer,J.;Stengel,P.J.;Leveque,V.J.P.;Pogam,S.L.;Rajyaguru,S.;Najera,I.;Swallow,S.Bioorg.Med.Chem.Lett.2009,19,410.doi:10.1016/j.bmcl.2008.11.060
(18) deVicente,J.;Hendricks,R.T.;Smith,D.B.;Fell,J.B.;Fischer,J.;Spencer,S.R.;Stengel,P.J.;Mohr,P.;Robinson,J.E.;Blake,J.F.;Hilgenkamp,R.K.;Yee,C.;Adjabeng,G.;Elworthy,T.R.;Tracy,J.;Chin,E.;Li,J.;Wang,B.;Bamberg,J.T.;Stephenson,R.;Oshiro,C.;Harris,S.F.;Ghate,M.;Leveque,V.;Najera,I.;Pogam,S.L.;Rajyaguru,S.;Ao-Ieong,G.;Alexandrova,L.;Larrabee,S.;Brandl,M.;Briggs,A.;Sukhtankar,S.;Farrell,R.;Xu,B.Bioorg.Med.Chem.Lett.2009,19,3642.doi:10.1016/j.bmcl.2009.05.004
(19) Hendricks,R.T.;Fell,J.B.;Blake,J.F.;Fischer,J.P.;Robinson,J.E.;Spencer,S.R.;Stengel,P.J.;Bernacki,A.L.;Leveque,V.J.P.;Pogam,S.L.;Rajyaguru,S.;Najera,I.;Josey,J.A.;Harris,J.R.;Swallow,S.Bioorg.Med.Chem.Lett.2009,19,3637.doi:10.1016/j.bmcl.2009.04.119
(20) deVicente,J.;Hendricks,R.T.;Smith,D.B.;Fell,J.B.;Fischer,J.;Spencer,S.R.;Stengel,P.J.;Mohr,P.;Robinson,J.E.;Blake,J.F.;Hilgenkamp,R.K.;Yee,C.;Zhao,J.;Elworthy,T.R.;Tracy,J.;Chin,E.;Li,J.;Lui,A.;Wang,B.;Oshiro,C.;Harris,S.F.;Ghate,M.;Leveque,V.J.P.;Najera,I.;Pogam,S.L.;Rajyaguru,S.;Ao-Ieong,G.;Alexandrova,L.;Fitch,B.;Brandl,M.;Masjedizadeh,M.;Wua,S.Y.;de Keczer,S.;Voronin,T.Bioorg.Med.Chem.Lett.2009,19,5648.doi:10.1016/j.bmcl.2009.08.023
(21) Todeschini,R.;Consonni,V.Handbook of Molecular Descriptors;Wiley-VCH:New York,2000.
(22)Xue,Y.;Li,Z.R.;Yap,C.W.;Sun,L.Z.;Chen,X.;Chen,Y.Z.J.Chem.Inform.Comp.Sci.2004,44,1630.doi:10.1021/ci049869h
(23)Tan,N.X.;Rao,H.B.;Li,Z.R.;Li,X.Y.SAR QSAR Environ.Res.2009,20,27.doi:10.1080/10629360902724085
(24) http://www.models.kvl.dk/source/GAPLS/index.asp,accessed June 2008.
(25) Leardi,R.;Boggia,R.;Terrile,M.J.Chemom.1992,6,267.
(26) Leardi,R.J.Chemom.1994,8,65.
(27) Burbidge,R.;Trotter,M.;Buxton,B.;Holden,S.Comput.Chem.2001,26,5.doi:10.1016/S0097-8485(01)00094-8
(28) Cherkassky,V.;Ma,Y.Selection of Meta-parameters for Support Vector Regression.Proceedings of the International Conference onArtificial Neural Networks,Madrid,Spain,Aug 28-30,2002.
(29)Hao,M.;Li,Y.;Wang,Y.;Zhang,S.Anal.Chim.Acta 2011,690,53.doi:10.1016/j.aca.2011.02.004
(30) Rainville,F.M.D.;Fortin,F.A.;Gardner,M.A.;Parizeau,M.;Gagné,C.DEAP:APython Framework for Evolutionary Algorithms.In EvoSoft Workshop,Companion Proc.of the GeneticandEvolutionaryComputationConference,July 07-11,2012.
(31)Chang,C.C.;Lin,C.J.LIBSVM:ALibrary for Support Vector Machines,2001.Software available at http://www.csie.ntu.edu.tw/-cjlin/libsvm,accessed Jun 2008.