含临床病毒株聚合酶逆转录酶区的乙肝病毒DNA稳定复制细胞系的构建
向明确,蔡雪飞,张文露,黄爱龙,胡接力
1重庆医科大学 重庆市第九人民医院感染科,重庆 400700
2重庆医科大学感染性疾病分子生物学教育部重点实验室,重庆 400016
乙肝病毒 (hepatitis B virus,HBV)耐药是目前慢性乙型肝炎治疗中的重要问题。HBV耐药突变主要发生在逆转录聚合酶 (reverse transcriptase,RT)区,例如RT M204I或M204V能大大降低病毒对拉米夫定(lamivudine,LVD)的敏感性[1];N236T 或 A181V 则与阿德福韦 (adefovir,ADV)耐药相关[2-3];LVD耐药突变背景 (M204V+L180M)加上T184、S202或M250相应突变,将导致HBV对恩替卡韦 (entecavir,ETV)抵抗[4-5]。
体外药敏实验 (或表型分析)是确定HBV RT特定突变与耐药间关系的关键环节。由于缺乏很好的体外HBV细胞感染系统,目前HBV体外药敏分析大多依赖这样一种方式:将合适的HBV DNA重组体转染肝癌细胞系,让其在体外复制HBV DNA,再用Southern blot检测不同浓度药物作用下HBV DNA复制量变化[6]。进行HBV体外表型分析时,需从细胞内分离纯化HBV复制中间体,并排除转染入细胞的质粒HBV DNA对后续检测干扰。但以往研究发现,转染入细胞的质粒DNA在纯化过程中往往难以有效及稳定地去除,导致影响后续Southern blot检测效果。本研究将一源于临床病毒株的HBV RT区克隆到HBV 1.1倍体质粒上,使其稳定转染肝癌细胞系,获得了HBV DNA的体外稳定复制。由于该体系免去了瞬时转染步骤,因而可以彻底去除转染质粒干扰,有助于提高HBV体外表型分析的准确性。
材料和方法
材料 血清来源于1例30岁男性慢性乙型肝炎患者,该患者为核苷 (酸)类似物初治患者,治疗开始时,谷丙转氨酶 (alanine aminotransferase,ALT)108 U/L,血清 HBV DNA 8.9×107copies/ml,HBeAg阳性,接受10 mg/d ADV治疗24周后,血清HBV DNA为 6.5×104copies/ml,ALT为 39 U/L。质粒p1818-LL为本课题组构建[7],含有 B基因型 HBV 1.1倍基因组DNA,位于巨细胞病毒极早期启动子(cytomegalovirus immediate-early promoter,CMV)下游,可支持HBV DNA体外复制。高纯度病毒核酸提取试剂盒、地高辛标记检测试剂盒为Roche公司产品;胶回收纯化试剂盒为QIAGEN公司产品;高保真DNA聚合酶 (PrimeStar HS DNA Polymerease)购自Takara生物技术公司;内切酶 Dpn I、Sma I及Proteinase K、Dnase I为Promega公司产品;表面抗原ELISA检测试剂盒为上海科华生物技术公司产品;real-time PCR试剂SsoFast EvaGreen Supermix为Biorad公司产品,所用荧光染料为Eva Green;real-time PCR仪型号为Bio-rad公司IQTM5。
细胞系 HepG2细胞为本室保存,源于美国ATCC(American Type Culture Collection),用含10%(体积分数)胎牛血清 (fetal bovine serum,FBS)的MEM于37℃,5%(体积分数)CO2条件下培养。
引物 由英骏 (上海)生物公司合成,引物序列及其在HBV基因组上的对应位置见表1。
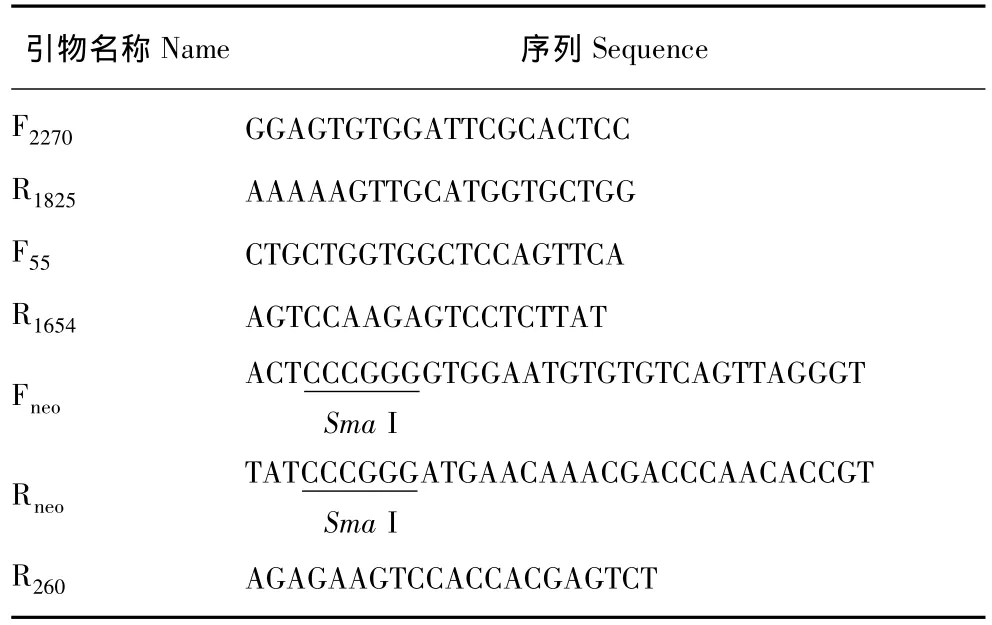
表1 引物及序列Table 1 Sequences of primers
血清HBV DNA片段扩增 用血清DNA提取试剂盒从患者血清中提取HBV DNA,具体操作按说明书进行。用引物F2270和R1825扩增HBV DNA片段,参数:预变性94℃ 2 min,循环条件:94℃ 15 s,52℃10 s,72℃ 2 min 10 s,共35个循环。以该PCR产物为模板,用引物F55和R1654做巢式PCR,扩增片段55~1654,参数:预变性94℃ 2 min,循环条件:94℃ 15 s,60℃ 10 s(-0.5℃ /cycle),72℃ 1 min 20 s,10个循环;94℃ 15 s,55℃ 10 s,72℃ 1 min 20 s,25 个循环。PCR产物纯化后测序。
55~1654片段置换反应 用QIAGEN胶回收试剂盒纯化上述PCR片段,电泳定量。取纯化后的55~1654片段800 ng做FSR,将源于患者的HBV DNA片段置换到HBV复制质粒pLL对应位置。55~1654片段置换反应 (fragment substitution reaction,FSR)原理见文献 [7]。具体如下:建立FSR反应体系:dNTPs 0.2 mmol/L,55~1654片段800 ng,pLL 50 ng,PrimeStar DNA 聚合酶 0.5 μl,5 × buffer 10 μl,总体积50 μl。反应参数:预变性94℃ 2 min;循环条件:94℃ 15 s,55℃ 10 s,72℃ 4 min 30 s,共18 个循环,反应产物纯化后,取部分产物用Dpn I 37℃消化4 h,取消化产物1.5 μl电转化JM109感受态细胞,然后挑克隆,提取质粒并测序鉴定,鉴定正确的质粒命名为p11。
质粒p11-neo构建 用引物Fneo及Rneo从pEGFP-N1扩增新霉素抗性基因表达片段,包括SV40启动子、新霉素抗性基因及PolyA信号序列,长度约1.4 kb。该片段用Sma I酶切后克隆到p11质粒中HBV DNA下游,候选克隆经酶切及测序鉴定,正确者命名为p11-neo。
细胞内HBV病毒DNA分析 HepG2细胞用含有10%胎牛血清的MEM培养基在37℃、含5%CO2的孵箱中培养。转染前20 h,将HepG2细胞接种于6孔板,密度约为7×105/孔,用无抗生素培养基培养过夜。第2天用LipofectamineTM2000转染细胞,转染后第5天,按文献方法从HepG2细胞中提取细胞质核心颗粒 HBV DNA[7],然后用 Southern blot检测HBV DNA:样品经1%(质量分数)琼脂糖凝胶电泳后,转印至带正电荷尼龙膜,烤干固定后进行预杂交、杂交 (地高辛标记的HBV DNA探针)、洗脱、检测,具体操作按产品说明书进行。
稳定复制HBV细胞株的构建 将p11-neo用Not I酶切线性化后转染HepG2细胞 (3.5 cm细胞培养皿),转染后第3天将细胞传入10 cm培养皿,并用1000 mg/L G418进行筛选3周,此时将剩余细胞用胰酶消化,并以2~3个细胞/孔的密度接种到96孔板,继续以500 mg/L G418筛选培养2~3周,每周更换培养基1次,待细胞克隆扩增后用real-time PCR检测培养上清液中HBV DNA。real-time PCR所用引物为F55及R260,反应条件:预变性95℃ 5 min。循环条件:95℃ 15 s,63℃ 30 s(-1℃ /cycle),72℃ 20 s,10个循环;94℃ 15 s,51℃ 30 s(本步检测信号),72℃ 20 s,30个循环。DNA定量标准品为p11梯度稀释品,标准品DNA浓度为6×102~6×106copies/μl。real-time PCR初筛出的克隆继续用ELISA检测上清液中HBsAg表达情况,阳性者继续扩大培养后,用Southern blot检测细胞内HBV复制中间体。
结 果
患者血清HBV DNA片段PCR扩增及克隆 本研究的这名患者是一项ADV临床药物试验的参与者,治疗24周后,病毒滴度较前期有一定程度下降,为6.5×104copies/ml。首先用巢式PCR从血清HBV DNA扩增出片段nt55~1654(图1B),测序结果显示,该片段编码的氨基酸序列中 (结果未显示),未发现已报道的ADV耐药相关突变。接下来,将nt55~1654克隆到 HBV 1.1倍体质粒 pLL上。pLL是一个可支持HBV体外复制的重组体,含有B基因型HBV DNA 1.1倍体 (图1A)。为了保证克隆了患者HBV DNA的质粒可复制,需将该nt55~1654替换原质粒上的nt55~1654片段,而保留1.1倍体的其他部分,以保证聚合酶、核心蛋白及表面蛋白的正常表达。由于该片段两侧并无合适酶切位点,本研究采用以往建立的FSR方法 (图1C)将该片段成功替换到pLL上。
新霉素抗性表达片段克隆 为了后续建立稳定HBV复制细胞系,需在p11质粒上引入抗性筛选基因。从pEGFP-N1上扩增新霉素抗性表达片段,包括SV40启动子、新霉素抗性基因和PolyA序列,并将该片段克隆到p11上HBV 1.1倍体下游的Sma I位点。图2为克隆初筛及酶切鉴定结果,1~3号克隆均含有符合预期大小的插入片段,测序结果证实克隆成功。

图1 患者血清HBV DNA片段PCR扩增及克隆Fig 1 Cloning of the DNA fragment amplified from the patient serum HBV DNA

图2 p11-neo克隆构建Fig 2 Cloning of the neomycin resistance gene expressing fragment to p11
含临床病毒株RT区的HBV 1.1倍体可在体外复制 为验证含有患者HBV DNA片段的p11-neo是否支持体外复制,将质粒转染HepG2细胞后,分离纯化细胞质核心颗粒HBV DNA,并用Southern blot检测。结果显示,与阳性对照pLL类似,p11-neo转染细胞后可产生单链DNA(ssDNA)及少量双链线性DNA(dslDNA)。尽管在分离纯化HBV复制中间体过程中采用DNase I去除质粒DNA,但在Southern blot检测结果中仍可见残余质粒 (图3),且这些质粒信号较强,对真正HBV复制中间体信号形成一定干扰。

图3 Southern blot检测HBV体外复制Fig 3 Detection of HBV replicative intermediates by Southern blot analysis
real-time PCR结合ELISA快速初筛稳定复制HBV的细胞克隆 在证明p11-neo可支持HBV复制后,将其转染HepG2细胞以构建稳定复制细胞系。因可复制HBV的细胞能将病毒颗粒分泌到胞外,据此利用real-time PCR初筛细胞克隆。为了使病毒富集从而便于检测,在检测前1周不更换培养基。检测时,直接从96孔板中取培养上清液2 μl进行realtime PCR。定量曲线显示扩增效率偏低 (75.9%)(图4)。根据定量结果,选择上清液HBV DNA浓度较高 (>2.8×105copies/ml)的5个细胞克隆进一步做ELISA鉴定。ELISA结果显示,这5个细胞克隆中有2个 (克隆3-4和3-10)培养上清中HBsAg表达为强阳性 (D450nm分别为2.213、2.106,阴性对照为0.077),另3个克隆为阴性或弱阳性。
HBV稳定复制细胞系的鉴定 为最终确定细胞系是否可复制HBV,将候选细胞克隆扩大培养,然后提取细胞质核心颗粒HBV DNA,并用Southern blot法进行检测。细胞克隆3-10及3-4(图5及未显示的结果)均可见明显复制中间体ssDNA,虽然dlsDNA及rcDNA不明显,但仍足以证明存在HBV复制。值得注意的是,在样本检测泳道,背景较干净,较高分子量区域未见含有HBV DNA的质粒或基因组信号 (图 5)。
讨 论
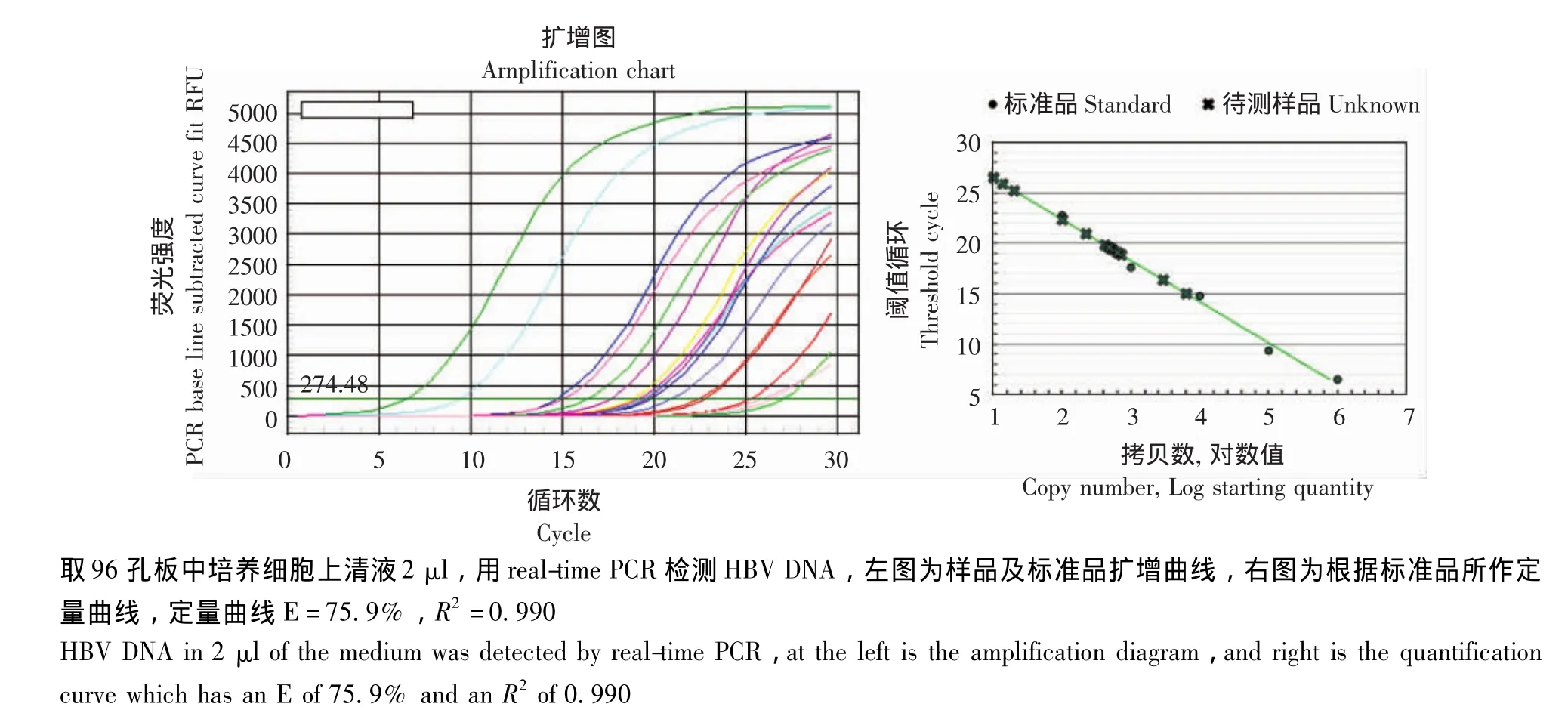
图4 real-time PCR初筛HBV稳定复制细胞系Fig 4 Screening for stable HBV replication cell lines with real-time PCR
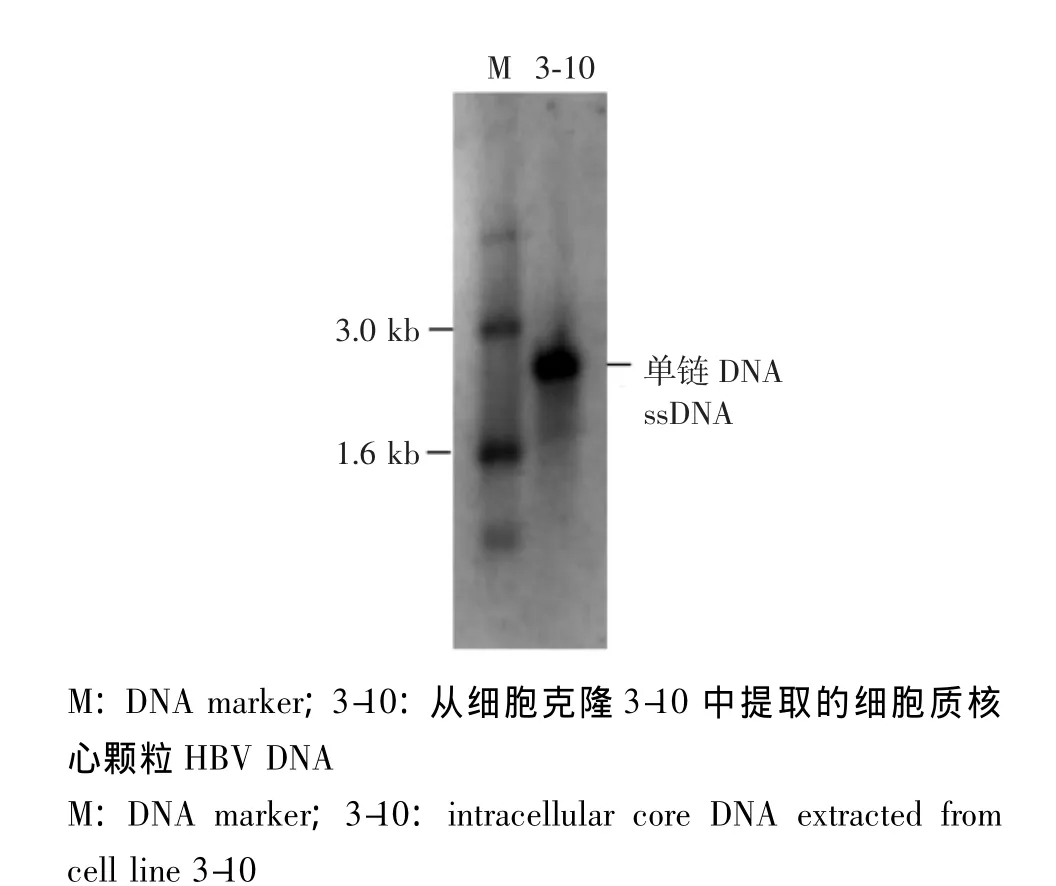
图5 Southern blot检测细胞系3-10中HBV DNAFig 5 Detection of HBV DNA extracted from cell line 3-10
体外表型分析是HBV耐药突变研究中常用方法。本课题组前期研究中发现,用转染HBV DNA重组体的方法做表型分析时,结果常不够稳定,原因有两个:(1)HepG2细胞较难转染,瞬时转染效率较不稳定;(2)由于转染的质粒量较大 (微克),尽管纯化HBV复制中间体时要用DNase I消化,质粒DNA仍不易彻底去除,且不同批次实验中,质粒去除程度也有差异,质粒中HBV序列会对目标信号造成不同程度干扰从而影响结果准确性 (这也是realtime PCR并不适合用来做此类检测的重要原因,因为无法区分质粒DNA和复制中间体)。本研究将含有患者HBV RT的HBV 1.1倍重组体稳定转染到HepG2细胞,可以解决以上两种问题,因为稳定复制HBV的细胞系无需再考虑转染效率及去除质粒的问题。结果显示,在检测3-10细胞系中HBV复制中间体时,的确未发现质粒或基因组DNA干扰现象。
在构建稳定复制HBV细胞株时,本研究用realtime PCR方法初筛细胞克隆,这有助于较快速鉴定出阳性克隆。若用ELISA初筛,则出现假阳性克隆的可能性较大,因为质粒的整合位置可能是随机的。有的细胞虽可表达HBsAg,但并不复制HBV,可能是由于质粒整合到基因组时,保持了S启动子及下游S开放阅读框 (open reading frame,ORF)的完整性,但其他病毒关键成分 (如聚合酶、核心蛋白等)表达遭到破坏。直接在细胞培养上清液中检测HBV DNA可以避免由于整合部位错误导致的假阳性问题,若确定上清液中有HBV DNA,则基本上可判定该细胞克隆能复制HBV。本研究利用real-time PCR初筛,将候选克隆范围从96孔板规模的数十个迅速缩小到数个,大大降低了后期细胞克隆扩增筛选的工作量,挑选上清液HBV DNA定量较高且ELISA结果阳性的2株细胞扩大培养,结果证明,这2株细胞均可稳定复制HBV。笔者的经验显示,在用real-time PCR初筛克隆时,需注意细胞数量不能太少,且检测前1周不宜更换培养基以富集上清液中的病毒颗粒,另外,由于real-time PCR的灵敏度高,需注意防范污染问题。
临床HBV病毒株稳定复制细胞系可改善表型分析实验效果,但其构建时间周期较长,笔者认为可能更适合于用来对初步阳性结果的进一步确认,例如先通过瞬时转染获得临床病毒株的初步药物敏感数据,若怀疑为耐药表型,则可构建稳定复制细胞株做进一步鉴定,这样可能有助于获得更可靠的结果。
[1]Allen MI,Deslauriers M,Andrews CW,et al.Identification and characterization of mutations in hepatitis B virus resistant to lamivudine.Lamivudine Clinical Investigation Group[J].Hepatology,1998,27(6):1670-1677.
[2]Angus P,Vaughan R,Xiong S,et al.Resistance to adefovir dipivoxil therapy associated with the selection of a novel mutation in the HBV polymerase [J].Gastroenterology,2003,125(2):292-297.
[3]Villeneuve JP,Durantel D,Durantel S,et al.Selection of a hepatitis B virus strain resistant to adefovir in a liver transplantation patient[J].J Hepatol,2003,39(6):1085-1089.
[4]Tenney DJ,Levine SM,Rose RE,et al.Clinical emergence of entecavir-resistant hepatitis B virus requires additional substitutions in virus already resistant to Lamivudine[J].Antimicrob Agents Chemother,2004,48(9):3498-3507.
[5]Tenney DJ,Rose RE,Baldick CJ,et al.Two-year assessment of entecavir resistance in Lamivudine-refractory hepatitis B virus patients reveals different clinical outcomes depending on the resistance substitutions present[J].Antimicrob A-gents Chemother,2007,51(3):902-911.
[6]Durantel D,Brunelle MN,Gros E,et al.Resistance of human hepatitis B virus to reverse transcriptase inhibitors:from genotypic to phenotypic testing[J].J Clin Virol,2005,34(Suppl 1):S34-S43.
[7]Hu JL,Cui J,Guo JJ,et al.Phenotypic assay of a hepatitis B virus strain carrying an rtS246T variant using a new strategy[J].J Med Virol,2012,84(1):34-43.

