偶联钠离子转运草酰乙酸脱羧酶研究进展
姜巨全,黄海鹏,孟 婧,胡宝忠
(东北农业大学生命科学学院,大豆生物学教育部重点实验室,哈尔滨 150030)
细菌在细胞质膜上进行能量转化过程中,质子是主要偶联离子[1-2]。许多初级钠离子泵借助膜内外钠离子梯度行使功能同时偶联质子转运[3]。次级钠氢逆向转运蛋白则会以跨膜质子电化学梯度为驱动力确立钠离子浓度梯度[4]。偶联钠离子转运脱羧酶作为一个酶家族,在细菌质膜上催化脱羧反应进行能量转化同时,偶联钠离子、质子跨膜运输,属于初级钠离子泵。该酶家族主要包括:草酰乙酸脱羧酶(Oxaloacetate decarboxylase)[5-6]、甲基丙二酰辅酶A脱羧酶(Methylmalonyl-CoA decar⁃boxylase)[7]、戊烯二酰辅酶A脱羧酶(Glutaconyl-CoA decarboxylase)[8]、丙二酸盐脱羧酶(Malonate decarboxylase)[9]。其中草酰乙酸脱羧酶是该羧酶家族中第一个被发现具有初级钠离子泵活性脱羧酶[10]。
草酰乙酸脱羧酶分为细胞质型和质膜型两种。前者不具有偶联钠离子转运功能,首先在溶壁微球菌(Microcococcus lysodeikticus)中被发现[11],并在乳球菌属(Lactococcus)[12-13]、假单胞菌属(Pseudomonas)[14]及棒状杆菌属(Corynebacterium)[15-16]等多个细菌属中相继被鉴定。后者具有偶联钠离子转运功能,最早在产气克莱勃氏杆菌(Klebsiella aerogenes)中被发现,其生理学基本特征被初步鉴定[10],继而在其他肠道致病细菌如霍乱弧菌(Vibrio cholerae)[17]、鼠伤寒沙门氏菌(Salmonella typhimurium)[18]等中被发现和鉴定。本实验室在松嫩平原盐碱地中分离得到一株中度嗜盐盐单胞菌新种并将其命名Halomonas songnenensis,利用基因功能互补与基因文库技术相结合方法,从该菌基因组上克隆得到一个编码偶联钠离子转运草酰乙酸脱羧酶基因簇oadGAB,目前正在对该酶活性以及钠离子转运机制进行鉴定。目前为止,肺炎克雷伯菌(Klebsiella pneumoniae)中草酰乙酸脱羧酶偶联钠离子转运脱羧反应机制已被详细地进行分析。本文综述具有偶联钠离子转运草酰乙酸脱羧酶生理学基本特征及其亚基结构,以及拓扑异构学、氨基酸定点突变等方法所获得该酶钠离子转运脱羧反应机制研究进展。
1 草酰乙酸脱羧酶在柠檬酸发酵途径中关键作用
1.1 柠檬酸发酵途径
柠檬酸通过偶联钠离子输出被摄入菌体,在柠檬酸裂解酶催化作用下分解为草酰乙酸和醋酸[19]。草酰乙酸被质膜上草酰乙酸脱羧酶脱羧,生成丙酮酸,同时脱羧反应偶联两个钠离子排出到细胞外。丙酮酸被进一步分解为乙酰辅酶A和甲酸,紧接着乙酰辅酶A反应形成乙酰磷酸。乙酰磷酸最后生成醋酸,反应过程中催化ADP生成ATP。在这个反应途径中,用于生物合成反应NADH合成尤为重要。肺炎克雷伯菌生长在高氧化柠檬酸基质中时必须为生物合成反应合成NADH,而这在柠檬酸循环中是不可行,因为α酮戊二酸脱氢酶合成会被缺氧抑制,但它具有独特的NADH生物合成途径:甲酸氢裂解酶产生氢气,然后经一个膜上氢化酶催化氢气和NAD+,生成NADH[20]。在柠檬酸发酵途径中,每消耗1 mol柠檬酸,生成一个1 mol ATP,同时行成一个钠离子电化学梯度。之后,这种电化学梯度被电中性柠檬酸钠:氢同向转运蛋白利用,驱动向胞内运送柠檬酸。
1.2 基因组成
柠檬酸发酵途径所必需基因在肺炎克雷伯菌基因组上共同组成一个基因簇[21-22],正向组成包括编码柠檬酸钠:氢同向转运蛋白citS基因,以及跟随在其后面由编码草酰乙酸脱羧酶oadGAB基因和编码一个双组份调控系统citA/B基因。反方向组成包括编码柠檬酸裂解酶连接酶citC基因和编码柠檬酸裂合酶三个亚基citDEF基因以及编码一个参与柠檬酸裂合酶生物素合成酶citG基因[23]。有研究指出肺炎克雷伯菌(Klebsiella pneumoniae)中有关柠檬酸发酵途径基因在不同菌株中呈现出遗传基因多样性,而该多样性与不同生长环境存在营养物质多样性关系很大[24]。
2 草酰乙酸脱羧酶亚基
2.1 亚基组成
草酰乙酸脱羧酶由三个不同亚基α、β、λ以1∶1∶1 比例组成[25-26]。如图 1 所示,外围α亚基(63.5 ku)包含两个不同结构域,即N端羧基转移酶结构域和C端生物素结合结构域[5]。一个富含脯氨酸和丙氨酸肽链连接着生物素在这两个结构域间置换。肽链上距C端35个氨基酸残基处,生物素以共价键连接在一个赖氨酸残基上。草酰乙酸脱羧酶β亚基(44.9 ku)呈强疏水性,紧密结合在质膜上[6]。γ亚基(8.9 ku)由两个结构域组成,N端锚定在质膜上,由一个脯氨酸和丙氨酸链与亲水C端结构域相连接[27]。C端包含有一个多组氨酸结构,为绑定在C端二价锌离子提供配体[28-29]。由于γ亚基不仅与水溶性α亚基蛋白相互作用,同时也与质膜上β亚基蛋白相互协调,γ亚基对草酰乙酸脱羧酶多亚基复合体稳定性具有重要作用[29]。
草酰乙酸脱羧酶可以被分离纯化,提取方法大体分两步,先利用去垢剂从膜上溶解下草酰乙酸脱羧酶蛋白,然后通过相应抗生素蛋白琼脂糖柱进行亲和色谱层析[25]。肺炎克雷伯菌草酰乙酸脱羧酶三亚基复合体,通过诱导表达已在大肠杆菌中被成功合成,为利用突变方法对其进行研究提供便利,并且双亚基复合体αγ和βγ也已在试验中得到克隆、表达和分离[29]。
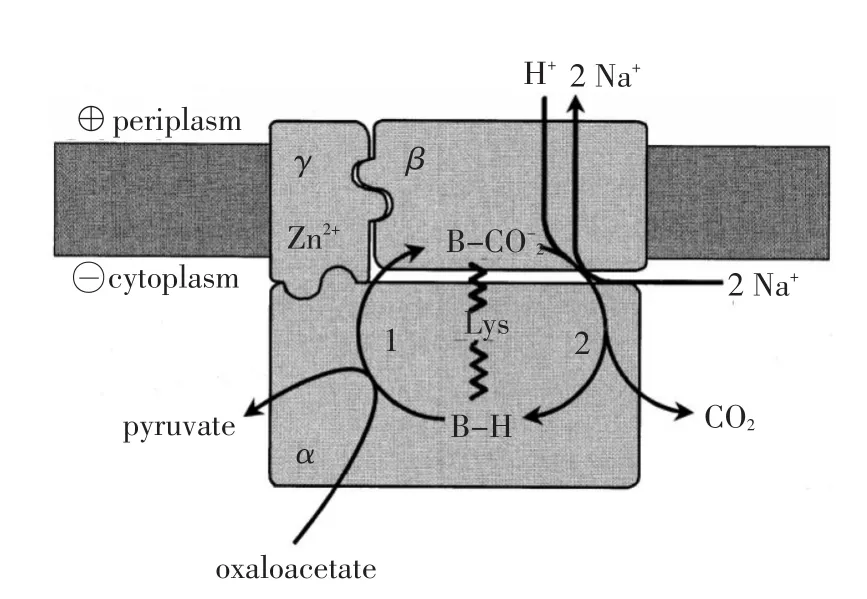
图1 草酰乙酸脱羧酶全景几何构型及其酶促反应特征Fig.1 Overall geometry of the oxaloacetate decarboxylase and features of the catalytic reactions
2.2 亚基作用
草酰乙酸脱羧酶催化反应循环始于草酰乙酸被结合到α亚基羧基转移酶结构域上[25],随后,草酰乙酸羧基被转移到生物素上,经过富有韧性脯氨酸丙氨酸链置换作用,羧化生物素从α亚基上羧基转移酶位点移动到β亚基上脱羧酶位点[30]。在羧基生物素脱羧过程中,一个由膜外导入质子被消耗,同时两个钠离子被泵入周质空间[31]。这一系列生物化学反应与其蛋白构象变化紧密相关。例如,在两个起催化作用结构域间置换生物素组分可能负责开启和关闭钠离子通道,这些构象变化在空间和时间上与生化反应协调一致,对整个脱羧反应具有至关重要作用。
3 Zn2+在羧基转移酶催化反应中作用
草酰乙酸脱羧酶中,每1 mol脱羧酶都含有1 mol紧密结合在γ亚基上Zn2+[32]。Zn2+在羧基转移酶催化反应时起到重要作用[33]。它协调作用于草酰乙酸上碳、氧原子,使氧原子因吸引与其相邻碳原子所共用电子而带负电,从而促进草酰乙酸上C-C基断裂,分解生成丙酮酸和羧化生物素[32]。将草酰乙酸脱羧酶α亚基置于含14C标记草酰乙酸环境中,尝试通过检测含14C标记羧基生物素蛋白量监控羧基转运反应。但是,羧基转运反应速率过低,以致于无法准确地监测羧基转移酶生理学活性。当加入γ亚基后,羧基转运反应速率大大提高,羧基转移酶活性可被监测到[29]。在γ亚基D62A和H77A突变株中,脱羧酶中Zn2+含量降到野生型35%和10%,当突变删除γ亚基C末端残基H82和P83时,酶中Zn2+含量降低5%。当相对应Zn2+含量减少时,这些突变株中草酰乙酸脱羧酶活性丧失[34]。在霍乱弧菌中,当突变猜测Zn2+离子配体γ亚基A17、H207、H209时,脱羧酶完全丧失活性[35]。这些结果一致说明γ亚基同它Zn2+在羧基转运催化机制中起着重要作用。
4 膜上β亚基拓扑异构学分析
膜蛋白拓扑学分析是描述一个跨膜蛋白跨膜片段数量、方向以及C端与N端分布。草酰乙酸脱羧酶β亚基是由433个氨基酸残基组成疏水性蛋白,疏水性分析显示,它具有9至10个跨膜结构。在推测跨膜区中,选择多个位点打断目蛋白肽链,使之与碱性磷酸酶或β-半乳糖苷酶形成融合蛋白,通过检测融合点位置分析整个蛋白跨膜情况。
4.1 β亚基上主要螺旋蛋白及组成
β亚基拓扑模型显示,它具有一个N末端在膜内细胞质一侧,一个C末端在膜外周质一侧,整个蛋白折叠在一起镶嵌在膜上,存在三个跨膜N末端螺旋,并由一个较大胞质环连通着螺旋Ⅱ和螺旋Ⅲ。进一步拓扑学分析表明,β亚基C末端部分由6个螺旋折叠形成,它N末端和C末端之间蛋白部分呈现出一个不规则折叠,为Ⅲa区域[36]。
4.2 β亚基上具有重要作用Ⅲa跨膜域
氨基酸残基179~189之间肽段构成一个环(见图2),该肽段氨基酸残基并不是完全保守,因为其中部分氨基酸残基在相关脱羧酶家族成员中并不存在。拓扑学分析表明,这一肽段应该定位在膜周质空间,这就排除Ⅲa区域会折叠成两个跨膜螺旋可能。有研究暗示,Ⅲa肽段是从靠近周质空间膜表面嵌入质膜中,但并没有完全穿透膜至靠近细胞质一侧膜表面(见图2)[36]。Ⅲa跨膜域极特殊重要性,不仅在于其C端在膜上特殊折叠方式,更是因为其包含一个重要氨基酸残基D203,该残基定位于草酰乙酸脱羧酶β亚基中由7个连贯非变异氨基酸残基构成最保守区域内(见图2)[30]。
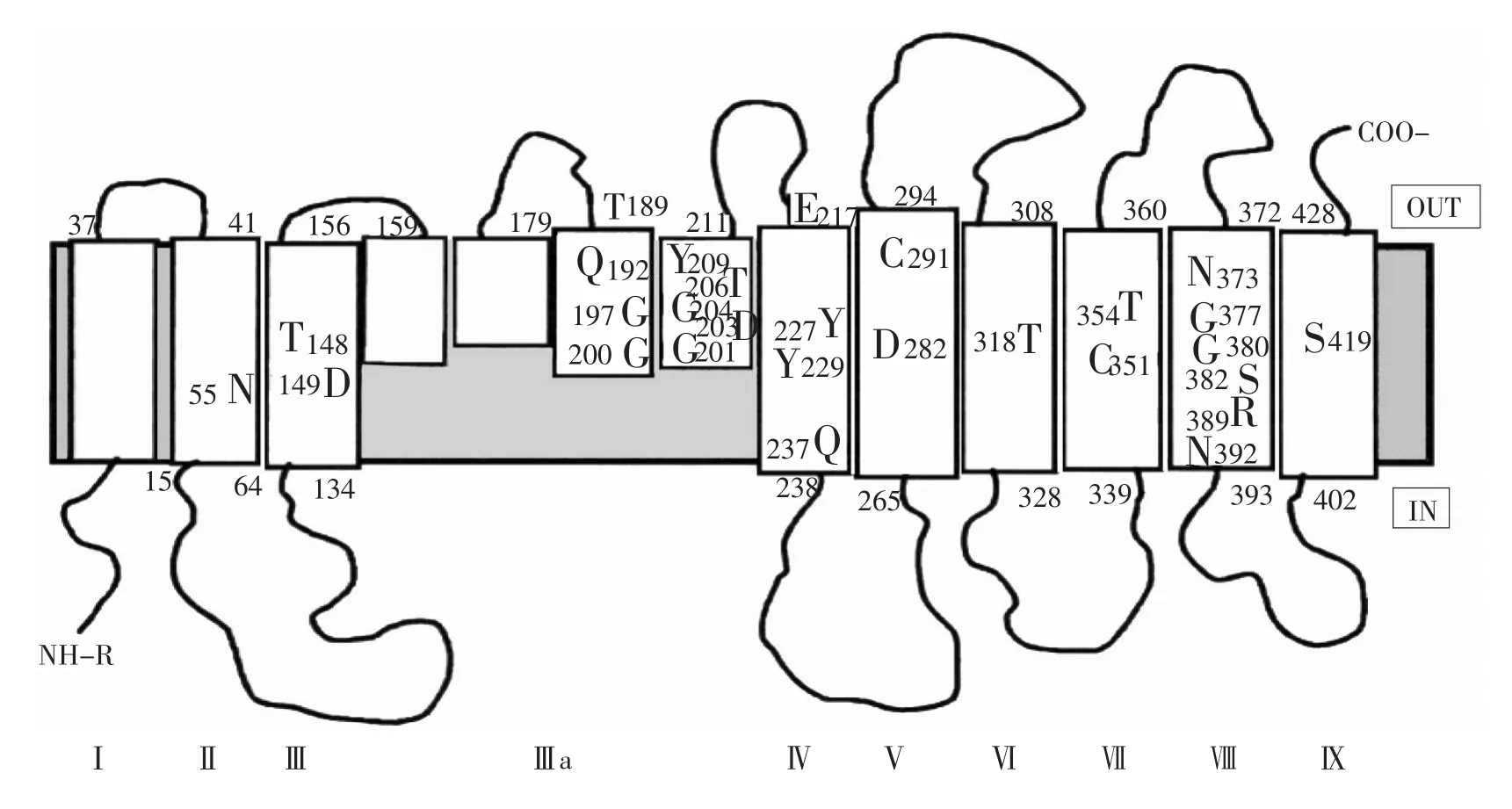
图2 草酰乙酸脱羧酶β亚基内特定位点氨基酸突变Fig.2 Location of the amino acids within the β subunit of oxaloacetate decarboxylas which have been changed by site-directed mutagenesis
5 基于定点突变β亚基功能研究
为鉴定草酰乙酸脱羧酶β亚基上重要氨基酸残基功能,可以使用氨基酸定点突变方法,通过研究突变株特性来分析突变意义。β亚基肽链上保守氨基酸残基被认为在功能上具有重要作用,尤其是镶入膜上Ⅲa区域极性氨基酸残基,它们可能是钠离子和氢离子跨膜运输最主要参与者。
5.1 β亚基上主要突变位点及钠离子泵活性鉴定
图2简要标注β亚基(OadB)上所有被突变过氨基酸残基[10,16,22]。其中,氨基酸残基D203、Y229、G377、S382中任何一个突变都导致草酰乙酸脱羧酶活性完全丧失,N373、R389突变大幅度地降低脱羧活性,而在其他位点如T148、G200、Y227、G380等突变并未明显影响草酰乙酸脱羧酶活性。D203(Ⅲa区域)[10]、Y229(螺旋Ⅳ)[22]、和G377或S382(螺旋Ⅷ)[16]等突变可使草酰乙酸脱羧酶活性降低,甚至完全丧失。
5.2 D203对β亚基功能作用
首个被鉴定出对于酶活性不可缺少功能性氨基酸残基是Ⅲa区域D203,即将203处保守天冬氨酸转为E、N、Q,均导致草酰乙酸脱羧酶完全丧失活性,并且突变后酶同时丧失初级钠离子泵活性[23]。钠离子泵活性是通过与大肠杆菌EP432(被敲出钠氢逆向转运蛋白基因大肠杆菌突变株菌株,不能在含盐350 mmol·L-1pH6葡萄糖矿物盐培养基上生长)功能互补试验来检测。野生型草酰乙酸脱羧酶基因转化EP432后,因具有初级钠离子泵活性可恢复其在高盐环境中生长能力[16]。然而,D203突变株草酰乙酸脱羧酶缺失初级钠离子泵活性,故不能与EP432进行功能互补恢复其在高钠离子浓度下生长。
草酰乙酸脱羧酶α亚基催化羧基转移酶活性,不会被β亚基D203突变所影响[13],这个酶可快速地催化14CO2从(4-14C)草酰乙酸上转移到共价生物素组分上[10]。因D203突变彻底敲出羧基生物素脱羧活性,生成含14C标记羧基生物素即使在钠离子环境中也很稳定。而在野生型酶中,羧基生物素酶会被立即脱羧而不能形成一个稳定中间产物。因此,D203对羧基生物素脱羧反应必不可少,而钠离子转运是与此偶联。D203可能提供一个双重作用,为转运溶解钠离子而提供结合位点和作为质子运送载体,引导质子反方向运动到达催化反应位点以备羧基生物素脱羧反应[10]。
5.3 S382对β亚基功能作用
羧基生物素脱羧反应及钠离子泵也同样依靠螺旋Ⅷ上S382。当这个氨基酸残基被突变为A、C、E、N或Q,即使在保留羧基转运活性情况下,草酰乙酸脱羧酶和钠泵活性也会完全消除[16]。而当将S382突变为保守T时,草酰乙酸脱羧酶会保持脱羧活性和钠离子泵活性,在S382突变为D时,这些活性会降低。突变株表现出酶活力对于研究S382在钠泵及羧基生物素脱羧中作用有重要指导意义。
所有在位点382可接纳氨基酸残基都携带一个羟基组分或一个酸性位点(S、T或D),这个结构可以使其转运质子到催化反应位点,从而启动羧基生物素脱羧反应[16]。位点382也可能是钠离子结合位点,因为当S382→E时,延长同方向氨基酸残基侧链,可能会通过扰乱这一范围中无机盐离子可溶性协调,使得钠离子不能结合,从而导致此突变株丧失活性。所以,S382可能具有双重作用:当可溶性盐离子被由胞质运入膜时,作为离子结合位点;而当质子被引导反方向运送由周质至膜上催化位点时,作为氢结合位点。所以,S382同D203具有相似功能,而且这两个氨基酸残基都可能是膜上钠氢转运网络重要部分。为起到质子传导功能,S382富含羟基侧链必须要在质子化和去质子化间转变。在水环境中,丝氨酸羧基组分PK>13,因此在生理学条件下不会发生去质子化[16]。然而,就如同丝氨酸蛋白酶对丝氨酸作用一样,去质子化可以在酶中疏水空间内,通过组氨酸和天冬氨酸成对作用来完成。
5.4 β亚基上R389功能推测
负责从β亚基上S382接收质子,可能是羧基生物素配体R389。R389氨基酸残基处在两个螺旋上,绕开S382朝向膜胞质一侧,这两个氨基酸残基都将它们侧链暴露在同一方向。R389非常保守,S382和R389在传导质子路径方面,有密切作用相互关系[17]。R389→K突变株表现出与野生型几乎相同草酰乙酸脱羧酶活性,同时也具有钠泵活性。在pH 9时,R389→C活性会降低,而当降低到野生型最适pH 6.5时,酶活性得以恢复[25]。当pH值由最适pH值提高两个单位时,对比R389→A、R389→L突变株与野生型酶活性,推测质子可能由S382经过R389传递给羧基生物素,从此处开启这个酸性复合物脱羧反应[16]。
5.5 β亚基上保守Y227和Y229重要性
β亚基上有两个保守酪氨酸残基,是在螺旋Ⅳ上Y227和Y229。如果将酪氨酸227突变成丙氨酸,则草酰乙酸脱羧酶活性从45 U·mg-1(野生型)降到0.5 U·mg-1,而突变株Y227→C和Y227→F酶活力更高一些,其酶活力分别降到12和9 U·mg-1[22]。因此,Y227对于保证酶高效催化作用是十分重要。而酪氨酸229对于酶重要性表现在,即使用苯基丙氨酸对酪氨酸做很保守交换,都会完全使其失去草酰乙酸脱羧酶活性和钠泵活性[37]。
6 能量偶联分子模型
图3为草酰乙酸脱羧酶能量偶联机制工作模型[31,38]。在这个模型中,β亚基上标示两个不同钠离子结合位点,一个位于D203,另一个位于S382。在Ⅲa部分中,螺旋Ⅳ和螺旋Ⅷ排列成一个膜上疏水空间,为钠离子和氢离子相互反向跨膜移动提供通道[39-40]。这个离子通道被预测拥有两种不同构象:构象1是使钠离子结合位点朝向细胞周质,而构象2则是当一阶段催化反应完成后,钠离子结合位点朝向胞质。
6.1 伴随构象变化离子运输途径
在α亚基上,催化反应循环始于羧基转移酶催化草酰乙酸提供羧基给生物素组分进行羧化反应。在图3A中,羧基生物素被氨基酸残基递送到并结合在β亚基上脱羧酶催化位点。跨膜螺旋Ⅷ上,接近于胞质膜表面R389对碱性离子有较强吸引力,它不仅对羧基生物素与催化位点结合起到促进作用,同时还对此时构象1起到稳固作用。S382是质子传递指挥网络一部分,这个网络还包含Y229、R389、H2O和羧基生物素[37]。当第一个钠离子结合到Asp203上后,第二个钠离子结合到S382,如图3B所示。此时,R389同羧基生物素协同催化作用S382,降低S382pK值,如同丝氨酸蛋白酶上组氨酸和天冬氨酸共同催化丝氨酸一样,使其更易去质子化[37]。钠离子由胞质膜通道接近S382,在β亚基中形成疏水环境下,取代S382侧链羟基上氢,重新形成离子对。这种取代会引起质子传递网络中氢重排,最后将一个质子传递给羧基生物素来催化这个酸性不稳定混合物立即脱羧。
如图3C所示,这一系列质子传递最终会引起整个蛋白模型构象变化,模型转化为构象2,胞质膜通道关闭,周质膜通道打开。一个周质膜外质子由此通道进入,将最先结合在接近周质膜表面D203上,使其释放第一个钠离子,随后被传送到S382,释放第二个钠离子,从而恢复S382羟基组分。D203和S382上钠离子被释放后,经过通道,到达膜外周质[40]。在所预测分子模型中,D203和S382是质子转移途径中心和枢纽,这同样与定点突变结果相一致,即二者之中任何一个氨基酸残基突变,脱羧活性将完全丧失[30-31]。由图3D→A所示,紧接着,α亚基上羧基转移酶作用草酰乙酸加工生成羧基生物素将重新结合到β亚基上,模型恢复到稳定构象1,开始一个新反应循环。
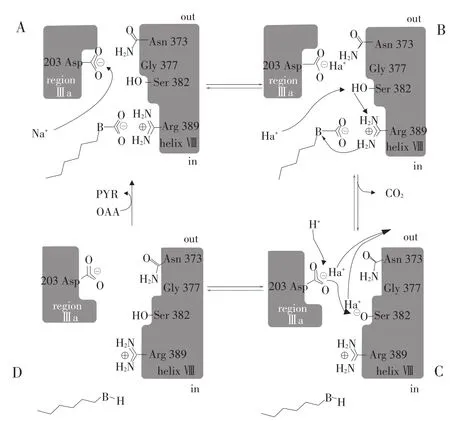
图3 羧基生物素脱羧基反应过程偶联钠氢离子跨膜运输机制分子演示模式图Fig.3 Model for coupling Na+and H+movements across the membrane to the decarboxylation of carboxybiotin
6.2 草酰乙酸脱羧酶能量偶联分子机制特点
在草酰乙酸脱羧酶钠离子转运反应循环中,有两个钠离子被由胞质运送至细胞周质,一个来自细胞周质质子在羧基生物素脱羧时被利用[41]。氨基酸残基N373可以看作是D203配体,两个残基都位于周质膜表面附近,它可能会协调促进D203对钠离子结合,N373→L、N373→D突变株都只保留很弱草酰乙酸脱羧酶活性。将R389突变为中性氨基酸A或L后,可以检测到酶活性降低,但不会完全丧失,即使R389→D突变株也会保留一部分活性[31]。
这个离子转运机制与所验证一个分子草酰乙酸对应两分子钠离子外排[42],同时会有一个分子质子运至膜内是一致[30]。这个模型更揭示催化过程中化学和数量上对应关系,同时说明生物素羧化和脱羧反应是钠离子跨膜转运所必需。草酰乙酸脱羧反应对钠离子的依赖性在此也得到证实,因为S382上质子需要提供给脱羧反应,而只有侵入钠离子能替换下这个质子[41]。运用此模型也可以对草酰乙酸脱羧酶活性会被高钠浓度和高pH值抑制作出解释,因为在此条件下,很难从低亲和力位点(膜上钠离子亲和位点)转换到钠离子,整个反应过程会变得迟缓[30]。
7 问题与展望
草酰乙酸脱羧酶钠泵偶联机制主要特点是利用嵌入膜氨基酸残基S382、D203-N373作为结合位点,及与其作用密切相关氨基酸残基共同形成钠离子、质子传递网络,引导钠离子和氢相对方向定向运动。由赖氨酸残基连接生物素将草酰乙酸脱羧及羧基生物素脱羧这两部分反应在生化反应能量传递上,及空间转移上有机地结合在一起。能量偶联分子模型被用以描述这一直接偶联机制。在草酰乙酸脱羧酶β亚基中,钠离子结合触发质子反向跨膜运动到达催化位点,在这里它们被消耗在催化反应中。
利用基因定点敲除技术,对突变处为D203、S382、Y229突变株研究从侧面验证反应机制模型,证明这些残基组成质子转运通道对催化作用至关重要。随着研究深入,在分子结构层面,对于某些氨基酸残基作用,存在许多新问题。如S382可以被D取代,但却不能被N、E或Q取代,由于其必须为钠离子提供结合位点,所以是否是在其链延伸方向上,氨基酸残基侧链长度起到至关重要作用;G377也是β亚基上一个关键残基,它的突变株完全失去脱羧酶活性,但目前对其研究较少,只是认为其在结构上起重要作用。在生化反应层面,以分子结构研究为基础,使通过对基因序列改变来正向调节脱羧酶活性及对钠离子运输能力成为可能。草酰乙酸脱羧酶作为重要初级钠离子泵之一,随着对其偶联钠离子转运脱羧机制研究不断深入,将为对其他钠离子初级泵及次级泵研究提供更广阔视野。同时,利用草酰乙酸脱羧酶钠离子转运特性,对其异源表达以改造菌体耐盐能力,将在构造耐盐工程菌株中产生重要作用。
[1] Mulkidjanian AY,Dibrov P,Galperin MY.The past and present of sodium energetics:May the sodium-motive force be with you[J].Biochim Bioph Acta,2008,177:985-992.
[2] Li T,Huo L,Pulley C,Liu A.Decarboxylation mechanisms in bio⁃logical system[J].Bioorg Chem,2012,43:2-14.
[3] Batista A P,Marreiros B C,Pereira M M.The role of proton and sodium ions in energy transduction by respiratory complex I[J].IUBMB Life,2012,64:492-498.
[4] Padan E,Schuldiner S.Na+/H+antiporters,molecular devices that couple the Na+and H+circulation in cells[J].J Bioenerg Biomem,1993,25:647-669.
[5] Schwarz E,Oesterhelt D,Reinke D,et al.The sodium ion translo⁃cating oxalacetate decarboxylase of Klebsiella pneumoniae.Se⁃quence of the biotin-containing alpha-subunit and relationship to other biotin-containing enzymes[J].J Bio Chem,1988,263:9640-9645.
[6] Woehlke G,Wifling K,Dimroth P.Sequence of the sodium ion pump oxaloacetate decarboxylase fromSalmonella typhimurium[J].J Bio Chem,1992,267:22798-22803.
[7] Grove T L,Benner J S,Radle M I,etal.A Radically different mechanism forS-adenosylmethionine-dependent methyltransfer⁃ases[J].Science Magazine,2011,332:604-607.
[8] Braune A,Bendrat K,Rospert S,et al.The sodiumion translocat⁃ing glutaconyl-CoA decarboxylase from Acidaminococcusfermen⁃tans:cloning and function of the genes forming a second operon[J].Mol Microbiol,1999,31:473-487.
[9] Berg M,Hilbi H,Dimroth P.Sequence of a Gene Cluster from Malonomonas Rubra encoding components of the malonate decar⁃boxylase Na+pump and evidence for their function[J].Eur J Bio⁃chem,1997,245:103-115.
[10] Dimroth P.A new sodium-transport system energized by the de⁃carboxylation of oxaloacetate[J].FEBS Lett,1980,122:234-236.
[11] Krampitz LO,Werkman CH.The enzymic decarboxylation of oxa⁃loacetate[J].J Bio Chem,1941,35:595-602.
[12] Sender PD,Martıín MG,Peirú S,et al.Characterization of an oxa⁃loacetate decarboxylase that belongs to the malic enzyme family[J].FEBS Lett,2004,570:217-222.
[13] Augagneur Y,Garmyn D,Guzzo J.Mutation of the oxaloacetate decarboxylase gene of Lactococcus lactis subsp.lactis impairs the growth during citrate metabolism[J].J Appl Microbiol,2008,104:260-268.
[14] Narayanan BC,Niu WL,Han Y,et al.Structure and function of PA4872 from Pseudomonas aeruginosa,a novel class of oxaloace⁃tate decarboxylase from the PEP mutase/isocitrate lyase superfam⁃ily[J].Biochemistry,2008,47:167-182.
[15] Ran T,Wang Y,Xu D,Wang W.Expression,purification,crystal⁃lization and preliminary crystallographic analysis of Cg1458:a novel oxaloacetate decarboxylase from Corynebacterium glutami⁃cum[J].Acta Cryst,2011,67:968-970.
[16] Klaff l S,Eikmanns B J.Genetic and functional analysis of the sol⁃uble oxaloacetate decarboxylase fromCorynebacterium glutami⁃cum[J].J Bacteriol,2010,192:2604-2612.
[17] Dahinden P,Pos K,Dimroth P.Identif i cation of a domain in the a-subunit of the oxaloacetate decarboxylase Na+pump that accom⁃plishes complex formation with the γ-subunit[J].FEBS J,2007,272:846-855.
[18] Wifling K,Dimroth P.Isolation and characterization of oxaloace⁃tate decarboxylase ofSalmonella typhimurium,a sodium ion pump[J].Arch Microbiol,1989,152:584-588.
[19] Saladino R,Brucato J R,Sio A D,et al.Photochemical synthesis of citric acid cycle intermediates based on titanium dioxide[J].As⁃trobiology,2011,11:815-824.
[20] Steuber J,Krebs W,Bott M,et al.A Membrane-Bound NAD(P)+-Reducing hydrogenase provides reduced pyridine nucleo⁃tides during citrate fermentation by Klebsiella pneumoniae[J].J Bacteriol,1999,181:241-245.
[21] Bott M,Dimroth P.Klebsiella pneumoniae genes for citrate lyase and citrate lyase ligase:localization,sequencing,and expression[J].Mol Microbiol,1994,14:347-356.
[22] Bott M,Meyer M,Dimroth P.Regulation of anaerobic citrate me⁃tabolism in Klebsiella pneumoniae[J].Mol Microbiol,1995,18:533-546.
[23] Schneider K,Dimroth P,Bott M.Biosynthesis of the prosthetic group of citrate lyase[J].Biochemistry,2000,39:9438-9450.
[24] Chen Y T,Liao T L,Wu K M.Genomic diversity of citrate fermen⁃tation in Klebsiella pneumoniae[J],BMC Microbiol,2009(9):1471-2180.
[25] Dimroth P,Thomer A.Subunit composition of oxaloacetate decar⁃boxylase and characterization of the a chain as carboxyltransferase[J].Eur J Biochem,1983,137:107-112.
[26] Dimroth P,Thomer A.Dissociation of the sodium-ion-translocat⁃ing oxaloacetate decarboxylase of Klebsiella pneumoniae and re⁃constitution of the active complex from the isolated subunits[J].Eur J Biochem,1988,175:175-180.
[27] Dahinden P,Pos K M,Dimroth P.Identification of a domain in the α-subunit of the oxaloacetate decarboxylase Na+pump that accomplishes complex formation with the β-subunit[J].FEBS J,2005,272:846-855.
[28] Dimroth P,Thomer A.The sodium ion pumping oxaloacetate de⁃carboxylase of Klebsiella pneumoniae Metal ion content[J].FEBS Lett,1992,300(1):67-70.
[29] Berardino M D,Dimroth P.Synthesis of the Oxaloacetate decar⁃boxylase Na+pump and its individual subunits in Escherichia Coli and analysis of their function[J].Eur J Biochem,1995,231:790-801.
[30] Berardino M D,Dimroth P.Aspartate 203 of the oxaloacetate de⁃carboxylase beta-subunit catalyses both the chemical and vectori⁃al reaction of the Na+pump[J].Embo J,1996,15:1842-1849.
[31] Jockel P,Schmid M,Steuber J,Dimroth P.A Molecular coupling mechanism for the oxaloacetate decarboxylase Na+pump as in⁃ferred from mutational analysis[J].Biochemistry,2000,39:2307-2315.
[32] Espariz M,Repizo G,Blancato V,et al.Identif i cation of malic and soluble oxaloacetate decarboxylase enzymes in Enterococcus faecalis[J].FEBS J,2011,278:2140-2151.
[33] Hall P R,Zheng R,Antony L,et al.Transcarboxylase 5S struc⁃tures:assembly and catalytic mechanism of amultienzyme com⁃plex subunit[J].EMBO J,2004,23:3621-3631.
[34] Schmid M,Wild M R,Dahinden P,et al.Subunit γ of the oxaloac⁃etate decarboxylase Na+pump:interaction with other subunits/do⁃mains of the complex and binding site for the Zn2+metalion[J].Biochemistry,2002,41:1285-1292.
[35] Studer R,Dahinden P,Wang W W,et al.Crystal Structure of the Carboxyltransferase domain of the oxaloacetate decarboxylase Na+Pump from Vibrio cholerae[J].J Mol Biol,2007,367(2):547-557.
[36] Jockel P,Berardino M D,Dimroth P.Membrane topology of the β-subunit of the oxaloacetate decarboxylase Na+pump from Kleb⁃siella pneumoniae[J].Biochemistry,1999,38:13461-13472.
[37] Jockel P,Schmid M,Choinowski T,et al.Essential role of tyro⁃sine 229 of the oxaloacetate decarboxylase β-subunit in the ener⁃gy coupling mechanism of the Na+pump[J].Biochemistry,2000,39:4320-4326.
[38] Pinner E,Kolter Y,Padan E,et al.Physiological role of NhaB,a specific Na+/H+antiporter in Escherichia coli[J].J Biol Chem,1993,268:1729-1734.
[39] Wild MR,Pos K M,Dimroth P.Site-directed sulfhydryl labeling of the oxaloacetate decarboxylase Na+pump ofKlebsiella pneu⁃moniae:helix VIII comprises a portion of the sodium ion channel[J].Biochemistry,2003,42:11615-11624.
[40] Schmid M,Vorburger T,Pos K M,et al.Role of conserved resi⁃dues within helicesⅣ and Ⅷ of the oxaloacetate decarboxylase β subunit in the energy coupling mechanism of the Na+pump[J].Eur J Biochem,2002,269:2997-3004.
[41] Saylor B T,Reinhardt L A,Lu Z,et al.A structural element that facilitates proton-coupled electron transfer in oxalate decarboxyl⁃ase[J].Biochemistry,2012,51:2911-2920.
[42] Dimroth P,Thomer A.On the mechanism of sodium ion transloca⁃tion by oxaloacetate decarboxylase of Klebsiella pneumoniae[J].Biochemistry,1993,32:1734-1739.

