白花马蹄莲诱导胡萝卜软腐果胶杆菌ubiG基因的克隆与功能分析
刘怀阿, 苏建坤, 刘 琴, 李 婷, 杨钟灵, 蒋 欢,3
(1.江苏里下河地区农业科学研究所,江苏 扬州 225007;2.南京农业大学植物保护学院,农作物生物灾害综合治理教育部重点实验室,江苏 南京 210095;3.重庆市涪陵区农业科学研究所,重庆 涪陵 408000)
马蹄莲(Zantedeschia spp.)是天南星科马蹄莲属多年生草本球根花卉植物,原产于南部非洲,被分为两类[1-2]。一类为 Zantedeschia,如白花马蹄莲(Z.aethiopica),该类植物地下茎为肉质根状,具有较强抗寒性,对软腐病具有一定的耐性。另一类为Aestivae,常被称为彩色马蹄莲,该类植物花色多样,地下茎肉质块状耐寒性较弱,需要休眠,易发细菌性软腐病[3]。
引起马蹄莲细菌性软腐病的病原菌主要为胡萝卜软腐果胶杆菌胡萝卜亚种(Pectobacterterium carotovorum subsp.carotovorum,1988年前为 Erwinia carotovora subsp.carotovora,下文简称 Pcc)[4-9]。Pcc寄主范围广泛,包括马蹄莲、结球甘蓝、胡萝卜、黄瓜、辣椒、芜青、莴苣和马铃薯等[10]。这种土传兼性厌氧性细菌在马蹄莲生长期和种球贮藏期侵染。发病初期叶片退绿黄化,呈水浸状坏死,随后发病加重,出现软腐,发出恶臭,严重时整个植株在几天之内死亡[3,11]。Pcc引起的软腐病一直是植物病理学研究的热点课题,特别是在其全基因序列公布后[12],与其致病相关的研究更有所增加[13-14]。
植物病原菌的致病过程是病原菌与寄主相互作用的过程,该过程由一系列致病因子参与。为了研究根瘤菌与甘草之间的共生固氮机制,Cai等人[15]运用甘草种子浸提物诱导中慢生型天山根瘤菌结瘤基因的表达,从5 000多个转座子插入突变株中获得8株受寄主诱导的突变株,鉴定出了中慢生型天山根瘤菌和其宿主甘草在共生固氮结瘤早期的信号交流中发挥作用的基因,并且提出了根瘤菌的LysE系统和其宿主植物分泌物中刀豆氛酸的互作模型。
为探寻Pcc的致病相关基因在致病过程中与寄主之间的关系,本研究拟利用转座子随机插入法构建Pcc突变体文库,通过寄主白花马蹄莲的组织提取液诱导Pcc中某些基因的表达,筛选获得受寄主诱导,且与致病性相关的突变株,进而对这些基因的功能进行研究,旨在为进一步研究Pcc的致病机制以及制定病害防控策略提供参考。
1 材料与方法
1.1 菌株、质粒、引物、培养基与植物材料
胡萝卜软腐果胶杆菌胡萝卜亚种 Pcc菌株PccS1,由本实验室分离和鉴定[6]。其他菌株与质粒见表1,所需引物见表2。胡萝卜软腐果胶杆菌及其相关突变株均用2YT培养基,28℃培养。E.coli菌株用LB培养基,37℃培养,试验所需限制性内切酶、连接酶和rTaq酶均购自TaKaRa公司。

表1 供试菌株和质粒Table 1 Strains and plasmids used in this study

表2 试验所用引物Table 2 The primers used in this study
白花马蹄莲(Zantedeschia aethiopica)、黄花马蹄莲(Z.elliotiana)均为本研究室引进品种,温室栽培;大白菜(Brassica pekinensis)为南京市场销售产品早熟五号。
白花马蹄莲(Z.aethiopica)组织提取液(ZaE),参照Aretusa等[18]介绍方法制备。挑选长势、大小一致的白花马蹄莲叶片10 g,75%酒精表面消毒,加入50 ml去离子水研磨,纱布过滤2次,取滤液,6 000 r/min、4℃离心10 min,离心 4~5次,取上清,用0.22 μm细菌过滤器过滤,得20%的叶片提取液,分装,-20℃保存备用。
1.2 PccS1突变体文库的构建以及目的菌株的筛选
将PccS1在含利福平(Rif)100 μg/ml的2YT固体培养基上划线,28℃下约17 h后,挑去单菌落在28℃、2YT液体培养基中,220 r/min振荡,过夜培养后,按1∶100转接到2YT(100 μg/ml Rif)液体培养基,28 ℃、220 r/min振荡培养至 OD600为0.6 ~0.8,分装至2 ml灭菌离心管,8 000 r/min离心,弃上清,用不含抗生素的2YT液体培养基漂洗3次,去除残留抗生素,用100 μl液体2YT培养基悬浮,获得受体菌;以E .coli SM10λpir(pJZ260,含有mariner转座子,南京农业大学生命科学院朱军教授提供)为供体菌,制备方法与供体菌相同,但液体培养基中添加抗生素为氨苄青霉素(Amp)100 μg/ml和氯霉素(Cm)100 μg/ml。将灭菌滤膜(孔径 0.22 μm,直径25 mm)置于2YT平板,在每张滤膜上分别滴加25 μl受体菌和供体菌,充分混合,37℃培养1 h后,转入28℃培养12~14 h。50 μl受体菌和供体菌分别滴加滤膜,作为对照。1 ml液体2YT培养基洗涤滤膜,洗涤液8 000 r/min离心3 min,适量2YT培养基悬浮菌体,分别涂布于2YT平板[含100 μg/m Rif+100 μg/ml Cm+40 μg/ml卡拉霉素(Km)+3%ZaE],28℃下倒置培养2~3 d,挑取单菌落,分别一一对应接种在100 μg/ml Rif+100 μg/ml Cm+40 μg/ml Km+3%ZaE(下文简称2YT-A)平板和100 μg/ml Rif+100 μg/ml Cm+40 μg/ml Km(下文简称2YT-B)平板上,只在2YT-A平板上能生长的菌株,可以初步确定该菌株为受白花马蹄莲组织液诱导、Km基因得到表达的突变株。
1.3 受马蹄莲组织提取液诱导的突变株生长曲线的测定
接种突变株(接种量1%)至两瓶50 ml MMX液体培养基(40 μg/ml Km)中,一瓶加入 3%ZaE,而另一瓶加入相同体积的无菌水做对照,28℃、200 r/min培养。每隔4 h测定菌体浓度(OD600)。
1.4 转座子插入位点的鉴定及序列分析
突变株、pUC19分别用SalⅠ酶切后,连接获得重组质粒,转化大肠杆菌DH5α感受态细胞,在LB(100 μg/ml Cm)固体平板上筛选转化子,挑选阳性克隆进行验证后,用转座子两端引物(Km-1和137-1)进行测序并拼接。测序结果用 NCBI上的BLASTx进行序列比对。
1.5 ubiG缺失突变体和互补菌株的构建及验证
1.5.1 ubiG缺失突变体的构建及验证 设计引物,通过PCR从ubiG、ubiG上下游以及Km片段的5'端和3'端扩增得到 U1(367 bp)、U2(438 bp)和Km(1 514 bp)DNA片段。纯化后分别用EcoRⅠ/HindⅢ、HindⅢ/KpnⅠ和KpnⅠ/XbaⅠ进行双酶切,回收,连接自杀载体pEX18GM构建重组载体,导入E.coli BW20676中获得供体菌。供体菌与受体菌PccS1两亲结合,得到一次交换子,一次交换子220 r/min振荡4 h,菌液涂布于含有10%蔗糖的2YT固体平板上筛选得到突变体。以UF1和UR2为上下游引物扩增突变株中2 319 bp的片段和野生型中1 525 bp的片段。
1.5.2 互补菌株的构建及验证 利用广宿主载体pBBR1-MCS5构建ubiG基因的互补载体。设计引物HUF和HUR从PccS1中扩增ubiG基因片段和其启动子片段(1 315 bp)。HindⅢ和XbaⅠ双酶切回收后与载体pBBR1-MCS5连接得到重组载体,导入E.coli BW20676中获得供体菌,与受体菌△ubiG两亲结合,获得互补菌株△ubiG(ubiG)。用载体上的引物GmF和GmR扩增互补菌株约500 bp的片段,以及引物HUF和HUR扩增目的片段,验证互补菌株。
1.6 缺失突变株和互补菌株的表型及活性分析
1.6.1 致病性测定 将 PccS1、△ubiG和△ubiG(ubiG)分别在含有相应抗生素的液体2YT培养基中,28℃、220 r/min振荡培养14~17 h,无菌水悬浮并调整OD600至0.6,取5μl接种于黄花马蹄莲(Z.elliotiana)和白花马蹄莲(Z.aethiopica)叶片和大白菜叶柄上(接种前用75%酒精表面消毒)。试验重复3次,置于25℃保湿培养,观察发病情况。
1.6.2 生长曲线测定 将PccS1、△ubiG和△ubiG(ubiG)分别在含有相应抗生素的液体2YT培养基中,28℃、220 r/min振荡培养14~17 h,无菌水悬浮并调整OD600至0.6,然后按1∶100的比例接种于2TY培养基中,每小时测定1次菌液OD600值。试验重复3次。
1.6.3 沉降性测定 将 PccS1、△ubiG和△ubiG(ubiG)分别于28℃、220 r/min振荡培养至OD600为2.0,取出后竖直静置24 h,观察各菌株的菌体状态。试验重复3次。
1.6.4 生物膜测定 主要参照Toole等[19]的方法,将PccS1、△ubiG和△ubiG(ubiG)分别于28℃、220 r/min振荡培养14~17 h,无菌水悬浮并调整 OD600为1.0,按1∶100比例转接到小试管中,28℃、220 r/min振荡培养至OD600为2.0,吸尽菌液,用等体积的无菌水清洗2次,加入等体积的1%结晶紫染色20 min,吸尽结晶紫,无菌水清洗3次,观察试管壁上是否有一圈紫色物质。用1 ml 90%乙醇溶解,575 nm测量吸光值。试验重复3次。
1.6.5 抗生素耐受性测定 所用抗生素包括四环素、氯霉素、头孢霉素、羧苄青霉素、壮观霉素、硫酸链霉素、硫酸新霉素以及氨苄青霉素,均配制为100 mg/ml,制 备 抗 生 素 终 浓 度 分 别 为 5 μg/ml、10 μg/ml、20 μg/ml、40 μg/ml、50 μg/ml、60 μg/ml、70 μg/ml、80 μg/ml、90 μg/ml和 100 μg/ml的 2YT 平板培养基。若菌株抗性大于100 μg/ml,则再根据具体抗性浓度而设置抗生素终浓度。
将PccS1、△ubiG和△ubiG(ubiG)分别于28℃、220 r/min振荡培养14~17 h,无菌水悬浮并调整OD600为1.0,各取2.5 μl接种于各浓度的抗生素平板上,28℃中培养24 h后观察菌落的生长情况。
1.6.6 胞外酶活性检测 蛋白酶检测培养基为LB培养基中加入1%高温灭菌的脱脂牛奶。参照Yi等[20]制备果胶酶酶和纤维素酶检测培养基。
将待测菌株分别在含有相应抗生素的液体2YT培养基中,28℃、220 r/min振荡培养14~17 h,无菌水悬浮并调至OD600为2.0,取2.5 μl分别接种于各检测平板上,28℃培养48 h。通过观察不同胞外酶检测平板上降解圈的大小来判断菌株胞外酶活性。在蛋白酶检测平板上可直接观察菌落周围透明降解圈的大小。果胶酶平板的观察:加入7.5%醋酸铜染色1 h。纤维素酶平板的观察:先加入0.2%刚果红染色20 min,再加入1 mol/L NaCl漂洗15 min,最后用1 mol/L HCl着色5 min,根据降解圈的大小比较突变菌株产生胞外酶的差异。
1.6.7 菌体形态和鞭毛观察 PccS1和突变株△ubiG分别用含相应抗生素的2YT平板纯化,28℃培养20 h,单菌落制成菌悬液,PTA负染,日立H-7650透射电镜观察菌体形态及鞭毛。
2 结果
2.1 彩色马蹄莲组织提取液诱导突变株(Km表达)的筛选
本研究中mariner转座子上含有没有启动子的卡那霉素抗性基因(Km),当它转座插入到某个启动子后面或基因的内部,Km的表达就会受到该启动子的调控。挑取在2YT-A平板上生长的单菌落分别点在2YT-A平板和2YT-B平板上。能生长的就是Km基因得到表达的突变株,即卡那霉素抗性基因插在组成型表达的基因内或者是被ZaE诱导表达的基因内。
通过此方法筛选获得1株能够诱导Km抗性基因表达的突变株,对突变株进行生长速率测定,发现突变株在添加Km的培养基和添加ZaE+Km的培养基中的菌体浓度(OD600)在生长20 h后发生明显差异,突变体在未加ZaE的培养基中的OD600值持续在1.0左右,而在加入ZaE的培养基中可持续生长(图1)。
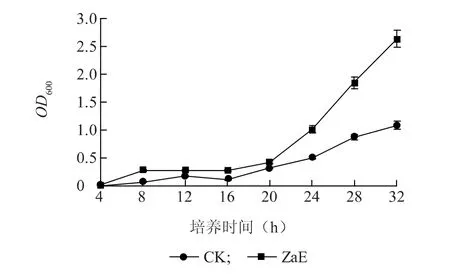
图1 马蹄莲组织提取液(ZaE)对突变株的诱导Fig.1 Induction of mutant by Zantedeschia aethiopica extraction
2.2 转座子插入位点序列分析
通过亚克隆技术鉴定转座子的插入位点。将测序所得序列拼接,找到1个720 bp的开放阅读框(ORF)。Blast分析发现,转座子插入位点位于辅酶Q氧甲基转移合成酶(Ubiquinone biosynthesis O-methyltransferase)编码基因ubiG。转座子插在了该阅读框的199 bp与200 bp之间。该基因全长720 bp,与 P.carotovorum subsp.carotovorum 菌株 PC1 有99%的同源性。
2.3 ubiG基因缺失突变体和互补菌株的构建及验证
2.3.1 突变体△ubiG的构建及验证 自杀重组质粒pEXubiG经EcoR I和Xba I双酶切验证,结果见图2,两个片段分别为载体pEX18GM和目的片段。验证重组载体构建成功后,通过两亲交配得到目标突变体。利用引物UF1和UR2对目标突变体PCR验证,突变体可扩增获得2 319 bp片段,野生株扩增获得1 525 bp片段(图3)。
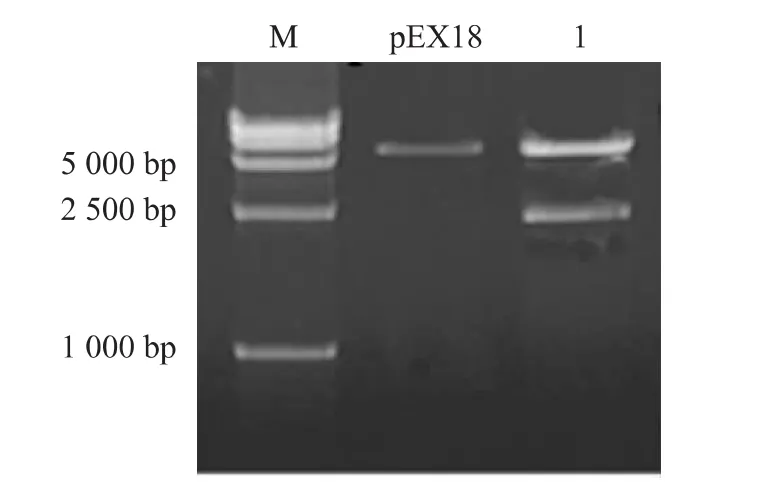
图2 重组质粒经EcoR I和Xba I双酶切验证Fig.2 Digestion of the recombinant plasmid with EcoR I and Xba I

图3 缺失突变体用引物UF1和UR2进行PCR验证Fig.3 PCR identification of the deletion mutants with primers UF1 and UR2
2.3.2 互补菌株的构建及验证 将ubiG基因和启动子片段连接到载体pBBR1-MCS5上,得到重组载体。重组载体经HindⅢ和Xba I双酶切验证(图4),导入突变株△ubiG,得到互补菌株△ubiG(ubiG)。互补菌株经引物GmF、GmR(约500 bp)和HUF、HUR(1 315 bp)验证正确(图 5)。

图4 重组载体经HindⅢ和Xba I双酶切验证Fig.4 Digestion of the recombinant vector with Hind Ⅲ and Xba I
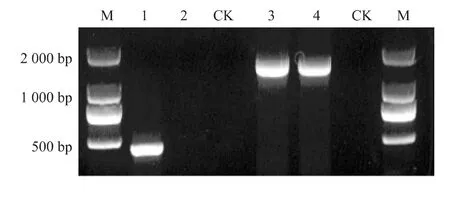
图5 △ubiG(ubiG)用引物 GmF、GmR和 HUF、HUR进行PCR验证Fig.5 PCR identification of strain △ubiG(ubiG)with primers GmF,GmR and HUF,HUR
2.4 缺失突变株和互补菌株的表型及活性分析
2.4.1 致病性 致病性测定结果显示,突变株在不同寄主上的致病性均减弱,从图6可以得出黄花马蹄莲(Z.elliotiana)和白花马蹄莲(Z.aethiopica)上野生型的病斑面积为突变株的2倍左右,致病性减弱不明显,而在大白菜上野生型的病斑面积为突变株的5倍左右。互补菌株使致病性在大白菜上有一定程度的恢复,病斑面积约为野生型的80%,而在黄化马蹄莲和白色马蹄莲上致病性恢复不明显。
2.4.2 生长曲线 为了解ubiG基因缺失是否影响细胞的正常生长,测定了野生型菌株 PccS1、突变株△ubiG和互补菌株△ubiG(ubiG)在2TY液体培养基上的生长速率,并绘制了生长曲线,发现突变株在迟缓期的生长稍慢于野生型,野生型在4 h左右开始生长,而突变株在5 h开始生长。突变株在对数期的生长速率相对于野生型较平缓。互补菌株没有恢复野生型的生长速率,与突变株基本相似(图7)。

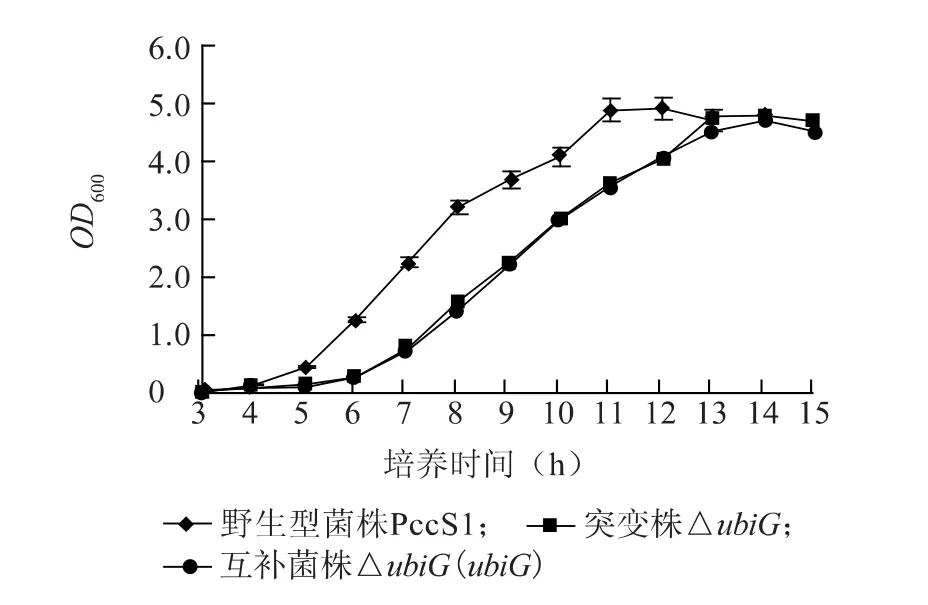
图7 突变株△ubiG在2YT培养基中的生长速率Fig.7 Growth rate of ubiG mutant strain in 2YT medium
2.4.3 沉降性 为研究ubiG基因的缺失是否影响PccS1的运动能力,测定了PccS1、△ubiG和△ubiG(ubiG)的沉降性。将浓度基本一致的菌株PccS1、△ubiG和△ubiG(ubiG)竖直放置于试管架上,室温下静置24 h后观察发现,突变株△ubiG沉降速度明显快于野生型PccS1和互补菌株△ubiG(ubiG),△ubiG菌体聚集于试管底部,菌液上层澄清,相比之下,PccS1和△ubiG(ubiG)菌液浑浊,菌体均匀分散于培养基中。
2.4.4 生物膜 为研究ubiG基因缺失是否影响Pcc的生物膜形成能力,分别定性和定量测定了PccS1、△ubiG以及△ubiG(ubiG)的生物膜产生量。同时在紫外分光光度计575 nm下对生物膜的产生进行定量测定,结果见图8。可以看出,△ubiG与PccS1相比,生物膜产量减少到野生型的1/2左右,互补菌株生物膜的产量稍低于野生型。

图8 突变株△ubiG生物膜的定性和定量检测Fig.8 Qualitative and quantitative detection of biofilm of ubiG mutant strain
2.4.5 抗生素耐受性 从不同浓度的抗生素平板上观察发现,ubiG基因的缺失增强了PccS1对硫酸链霉素(Sm)和硫酸新霉素(Neo)的抗性,Sm对PccS1的最低抑菌浓度为20 μg/ml,而Sm对△ubiG的最低抑菌浓度为60 μg/ml,△ubiG对Sm的抗性是野生型菌株的3倍,互补菌株抗性同突变株。
Neo对PccS1的最低抑菌浓度同样为20 μg/ml,但是对△ubiG的最低抑菌浓度为 550 μg/ml,△ubiG对Neo的抗性是野生型菌株的27.5倍,互补菌株抗性同突变株。
2.4.6 胞外酶 从3种胞外酶检测培养基上观察发现,突变株△ubiG在蛋白酶检测平板上无降解水解圈,而在纤维素酶和果胶酶检测平板上水解圈大小与野生型差异不明显。互补菌株△ubiG(ubiG)对合成胞外蛋白酶能力没有恢复。
2.4.7 菌体形态以及鞭毛观察 在透射电镜下观察突变株的菌体形态及鞭毛(图9)发现,ubiG基因突变株能够正常形成4~6根周生鞭毛,与野生型没有差异,菌体形态也与野生型无差异,都呈杆状。

图9 野生型PccS1和突变株△ubiG的鞭毛及菌体形态观察Fig.9 Flagellum and cell shapes of PccS1 and △ubiG
3 讨论
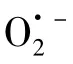
培养基中添加马蹄莲组织提取液,插入突变株生长明显快于不添加寄主组织提取液的对照,说明寄主提取液中可能存在某种物质能够诱导转座子插入基因的表达增强,也可能是因为寄主组织提取液提供突变株一个更好的生长环境,使得突变株在添加提取液后能够更好的生长。所以要探究马蹄莲组织是如何诱导ubiG基因表达,还需要进一步研究。
ubiG基因的缺失减弱了PccS1对不同寄主的致病力,同时,又影响了菌株的正常生长,所以致病性的减弱可能是由于缺失突变株生长不良而引起的。ubiG基因的缺失还加快了菌体的沉降速率,表明菌体运动能力降低。运动性是Pcc中的重要致病因子,Mijan Hossain等[23]对 Pcc菌株 PC1 的 fiiC 和motA基因进行敲除后,对大白菜上的致病性减弱,其中fiiC基因缺失后,鞭毛消失,丧失运动性;而motA基因缺失,菌株虽丧失运动性,但鞭毛依然存在,说明Pcc鞭毛介导的是运动性而非鞭毛本身影响了病原菌对寄主的毒力。所以ubiG基因缺失导致运动能力的降低,可能是导致病原菌致病性减弱的原因。
ubiG基因缺失突变株对不同抗生素的耐受性结果表明,ubiG基因缺失增强了对硫酸链霉素和硫酸新霉素的抗性,特别是硫酸新霉素,突变株对其耐受水平是野生型的27.5倍。硫酸链霉素和硫酸新霉素都属于氨基糖苷类抗生素,主要作用于细菌核糖体的30亚基,从而抑制细菌蛋白质的合成,并破坏细胞膜的完整性。链霉素和新霉素是防治革兰氏阴性细菌引起的植物病害的重要抗生素。随着单一药剂的长期大量使用,已经引起了病原菌对链霉素的抗药性[24],但是还没有报道过对硫酸新霉素产生抗药性的植物病原细菌。细菌对抗生素产生耐受性的原因主要有:(1)减少药物吸收或药物在细菌体内的积累;(2)细菌产生钝化酶使药物失活[25]。Yang等[26]在对Pcc各亚种链霉素抗性突变株的研究中,发现突变株的胞外多糖产量明显增多,胞外多糖是细菌生物被膜形成所必需的,而生物膜是细菌耐药性产生的重要机制之一,但是本研究发现ubiG突变株的生物膜产量比野生型少,故ubiG基因缺失突变株对两种抗生素的抗性增强可能是其他原因引起。但本研究基因敲除插入了Km抗性基因,能够引起突变株对链霉素和硫酸新霉素产生抗性,但是突变株对链霉素和硫酸新霉素的抗性存在较大差异,因此,ubiG基因缺失是否对链霉素和硫酸新霉素抗性产生有关,尚需进一步研究。
病原细菌的蛋白酶可能与提供病原菌蛋白质合成所需氨基酸或者降解植物抗性相关蛋白质有关,Pcc中则至少存在一种蛋白酶(PrtW),prtW基因缺失严重影响病原菌的毒力[27]。本研究发现ubiG基因缺失导致突变菌株蛋白酶活力丧失,所以,可以推测ubiG基因缺失可能间接影响蛋白酶编码基因的表达,进而影响Pcc的致病性。
突变株的互补菌株对大白菜的致病性菌体的沉降速率生物膜的产量能够部分恢复,而其他表型恢复不明显,可能与载体和病原菌基因组的遗传表达系统之间存在差异相关。
综上所述,利用寄主组织提取液并结合转座子随机插入法研究Pcc与寄主的互作是可行的。ubiG基因与Pcc致病相关的研究未见报道,本研究证实该基因与Pcc的毒力相关,它可能不直接参与致病,但在致病过程中起调控作用,通过影响菌株的运动能力以及胞外蛋白酶的活性间接影响致病性。
[1]仇 硕,张翠萍,李秀娟,等.几种植物生长调节物质诱导彩色马蹄莲抗细菌性软腐病初探[J].江苏农业科学,2012,40(6):106-109.
[2]黄作喜,林忠全,齐泽民.彩色马蹄莲种球越冬与贮藏技术[J].江苏农业科学,201l,39(6):296-297.
[3]SNIJDER R C,VAN TUYL J M.Evaluation of tests to determine resistance of Zantedeschia spp.(Araceae)to soft rot caused by Erwinia carotovora subsp.carotovora[J].European Journal of Plant Pathology,2002,108:565-571.
[4]SNIJDER R C,LINDHOUT P.Genetic control of resistance to soft rot caused by Erwinia carotovora subsp.carotovora in Zantedeschia spp.(Araceae),section Aestivae[J].Euphytica,2004,136:319-325.
[5]易建平,戚龙君,许志刚,等.马蹄莲细菌性软腐病的鉴定[J].植物检疫,2002,16(1):8-10.
[6]谷春艳,范加勤,杨 雪,等.彩色马蹄莲细菌性软腐病菌的鉴定及其群体感应淬灭的研究[J].南京农业大学学报,2009,32(3):71-77.
[7]HAUBEN L,MOORE ERB,VAUTERIN L,et al.Phylogenetic position of phytopathogens within the Enterobacteriaceae[J].System Application Microbiology,1998,21:384-397.
[8]WRIGHT P J,BURGE G K.Irrigation,sawdust mulch,and enhance(R)biocide affects soft rot incidence,and ower and tuber production of calla[J].New Zealand Journal of Crop and Horticultural Science,2000,28:225-231.
[9]FUNNELL K,AMACKAY B R.Directions and challenges of the New Zealand calla industry,and the use of calcium to control soft rot[C]//SHEEN T F,CHEN J J,YANG T C,et al.The international symposium on development of bulbous flower industry.Taiwan:TSIPS,1999.
[10]贾云鹤,张俊华,崔崇士.软腐欧文氏菌致病性的研究进展[J].东北农业大学学报,2007,38(1):113-118.
[11]WRIGHT P.A soft rot of calla(Zantedeschia spp.)caused by Erwinia carotovora subsp.carotovora[J].New Zealand Journal of Crop and Horticultural Science,1998,26:331-334.
[12]BELL K S,AVROVA A O,HOLEVA M C,et al.Sample sequencing of a selected region of the genome of Erwinia carotovora subsp.atroseptica reveals candidate phytopathogenicity genes and allows comparison with Escherichia coli[J].Microbiology,2002,148:1367-1378.
[13]CHARKOWSKI A O.The soft Ewinia[M]//GNANAMANICKAM S S.Plant-associated bacteria.Netherlands:Springer,2006.
[14]DONG Y H,WANG L H,XU J L,et al.Quenching quorum sensing dependent bacterial infection by an N-acyl homoserine lactonase[J].Nature,2001,411:813-817.
[15]CAI T,CAI W,ZHANG J,et al.Host legume-exuded antimetabolites optimize the symbiotic rhizosphere[J].Molecular Microbiology,2009,10:1-11.
[16]HOANG T T,KARKHOFF-SCHWEIZER R R,KUTCHMA A J,et al.A broad-host-range Flp-FRT recombination system for sitespecific excision of chromosomally-located DNA sequences:application for isolation of unmarked Pseudomonas aeruginosa mutants[J].Gene,1998,212:77-86.
[17]KOVACH M E,ELZER P H,HILL D S,et al.Four new derivatives of the broad-host-range cloning vector pBBR1MCS,carrying different antibiotic-resistance cassettes[J].Gene,1995,166:175-176.
[18]ARETUSA A E,SILVA L P,PEREIRA J L,et al.In vivo proteome analysis of Xanthomonas campestris pv.campestris in the interaction with the host plant Brassica oleracea[J].Federation of European Microbiological Societies,2008,1:167-174.
[19]TOOLE G A,KOLTER R.Initiation of biofilm formation in Pseudomonas fluorescens WCS365 proceeds via multiple,convergent signaling pathway:a geneticanalysis[J].Molecular Microbiology,1998,28:449-461.
[20]YI X,YAMAZAKI A,BIDDLE E,et al.Genetic analysis of two phosphodiesterases reveals cyclic diguanylate regulation of virulence factors in Dickeya dadantii[J].Molecular Microbiology,2010,77(3):787-800.
[21]LI H Y,WANG J S.Release of active oxygen species from phytopathogenic bacteria and their regulation[J].Chinese Science Bulletin,1999,44(1):71-75.
[22]CORT S A,IMLAY A J.Balance between endogenous superoxide stress and antioxidant defence[J].Bacteriol,1998,180(6):1402-1410.
[23]MIJAN HOSSAIN M D,SHIBATA S,AIZAWA S I,et al.Motility is an important determinant for pathogenesis of Erwinia carotovora subsp.carotovora[J].Physiological and Molecular Plant Pathology,2005,66:134-143.
[24]XU Y,ZHU X F,ZHOU M G,et al.Status of streptomycin resistance development in Xanthomanas oryzar pv.oryzae and Xanthomonas oryzae pv.oryzicola in China and their resistance characters[J].J Phytopathol,2010,158:601-608.
[25]MINGEOT-LECLERCQ M P,GLUPCZYNSKI Y,TULKENS P M.Aminoglycosides:activity and resistance[J].Antimicrob A-gents Chemother,1999,43(4):727.
[26]YANG B H,BRAND J M,GRAY J S S,et al.Extracellular polysaccharides of modified strains of Eewinia spp.[J].Carbohydate Research,2001,333:295-302.
[27]MARITS R,KOIV V,LAASIKE,et al.Isolation of an extracellular protease gene of Erwinia carotovora subsp.carotovora strain SCC3193 by transposon mutagenesis and the role of protease in phytopathogenicity[J].Microbiol,1999,145:1959-1966.

