石蒜属植物SCoT-PCR 反应体系构建及优化
姜小凤, 高燕会, 童再康, 黄春红
(浙江农林大学 亚热带森林培育国家重点实验室培育基地, 浙江 临安311300)
石蒜属Lycoris 植物具有极大观赏价值和药用价值。 全世界石蒜属植物共有20 多种, 主要分布于亚洲, 而中国是石蒜属植物的分布中心, 资源优势明显, 有16 种1 变种, 占全属的75%, 其中12 种为中国特有, 主要分布于江苏、 浙江、 安徽等地[1]。 对石蒜属植物的研究主要集中在分类学、 细胞遗传学、育种学、 分子生物学、 药用及药理几个方面。 近年来, 石蒜属植物在分子生物学方面的研究较多, 邓传良等[2-4]利用随机扩增多态性DNA 标记(random amplified polymorphic DNA, RAPD)对长筒石蒜Lycoris longituba 3 个居群的遗传多样性及分化程度进行了研究, 表明长筒石蒜种内具有较高的遗传多样性[2-4]。袁菊红等[5]用简单序列重复区间(inter-simple sequence repeat, ISSR)和RAPD 标记分别对不同采集地的37 份石蒜属植物的遗传多样性进行检测。 时剑等[6]构建并优化了中国石蒜的微卫星DNA(simple sequence repeat, SSR) 体系, 但这些标记在石蒜属植物的研究中存在一些不足, 如RAPD 标记的影响因素较多,稳定性和重复性差; 随机扩增长度多态性(amplified fragment length polymorphism, AFLP)标记对DNA 纯度和内切酶的质量要求很高且费用昂贵; SSR 标记虽为共显性标记, 但其引物的开发和设计耗时费力。因此, 为了更好地对石蒜属植物种质资源的遗传多样性和种质鉴定进行深入研究, 我们采用Collard 等[7]和Mackill[8]在水稻Oryza sativa 上基于单引物扩增反应(single primer amplification reaction, SPAR)提出的一种新型目的基因分子标记——目标起始密码子多态性(start condon tardeted polymorphism, SCoT)分子标记, 该标记具有操作简单、 多态性高﹑重复性好﹑引物通用性高、 可获得丰富的遗传信息且能跟踪性状等优点, 有效补充了ISSR 标记和RAPD 标记的不足[9]。 目前, SCoT 标记已成功应用于光稃稻Oryza glaberrima[7], 花生Arachis hypogaea[10], 甜橙Citrus sinensis[11], 柑 橘Citrus reticulata, 龙眼Dimocarpus longana[12-13], 甘蔗Saccharum officenarum[14]等的种质资源的遗传多样性分析, 但在石蒜属植物上遗传多样性以及种质鉴定等方面未见报道。 为此, 本研究以石蒜属植物为材料, 建立并优化石蒜属植物SCoTPCR 反应体系, 旨在为石蒜属种质资源的遗传多样性分析及遗传改良奠定基础。
1 材料和方法
1.1 试验材料
本研究所用的石蒜属植物种植于浙江农林大学遗传学科种质资源圃, 来源于湖南、 江西、 云南、 江苏、 浙江、 安徽及国外等, 取正常生长的嫩叶, 于-20 ℃冰箱保存备用。 共用34 份材料:1~3 为栽培石蒜Lycoris radiata, 4~6 为栽培稻草石蒜L. straminea, 7~9 为栽培玫瑰石蒜L. rosea, 10~11 为野生江苏石蒜L. houdyshelii, 12~13 为野生乳白石蒜L. albiflora, 14~15 为野生红蓝石蒜L. haywardii, 16~18 为栽培换锦花L. sprengeri, 19~21 为野生长筒石蒜L. longituba, 22~24 为栽培中国石蒜L. chinensis, 25~27 为栽培忽地笑L. aurea, 28~29 为来自日本的香石蒜L. incarnata, 30~31 为来自日本的夏水仙L.squamigera, 32~34 为石蒜与忽地笑的杂交种。
1.2 主要试剂
进行聚合酶链式反应(PCR)的各种试剂: Taq DNA 聚合酶, dNTPs, M. DL 3 000 标记均购自生工生物工程(上海)有限公司, SCoT 引物根据Collard 等[7]和Mackill[8]公布的引物, 由南京金斯瑞生物科技有限公司合成。
1.3 试验方法
1.3.1 石蒜属植物基因组DNA 提取与检测 石蒜属植物基因组DNA 提取采用改良的十六烷基三甲基溴化铵法(CTAB)[13], 用NanoDrop 微量分光光度计(ND-1000)测定DNA 的浓度和质量, 并取适量样品, 于10.0 g·kg-1琼脂糖凝胶电泳检测, 紫外凝胶成像系统Gene Genius Bio Imaging Ststem (BioRad)下观察并照相, 其余DNA 样品于-20 ℃冰箱中保存备用。
1.3.2 正交试验设计方案 以第23 份材料中国石蒜DNA 为模板, P40 为扩增引物。 针对影响PCR 反应的5 个要素: DNA, 引物, dNTPs, Mg2+和Taq DNA 聚合酶, 采用正交设计L25(56)对SCoT-PCR 反应体系进行优化。 设计水平5 个·因素-1(表1), 重复3 次·处理-1(表2)。PCR 扩增体系为20.0 μL, PCR 扩增反应程序: 94 ℃预变性5min;94 ℃变性35 s, 47~59 ℃(根据各引物的溶解温度Tm值)退火35 s,72 ℃复性90 s, 35 个循环; 72 ℃延伸10 min, 8 ℃保存[15], 扩增反应产物于10.0 g·kg-1的琼脂糖凝胶电泳检测并用凝胶成像系统观察拍照。
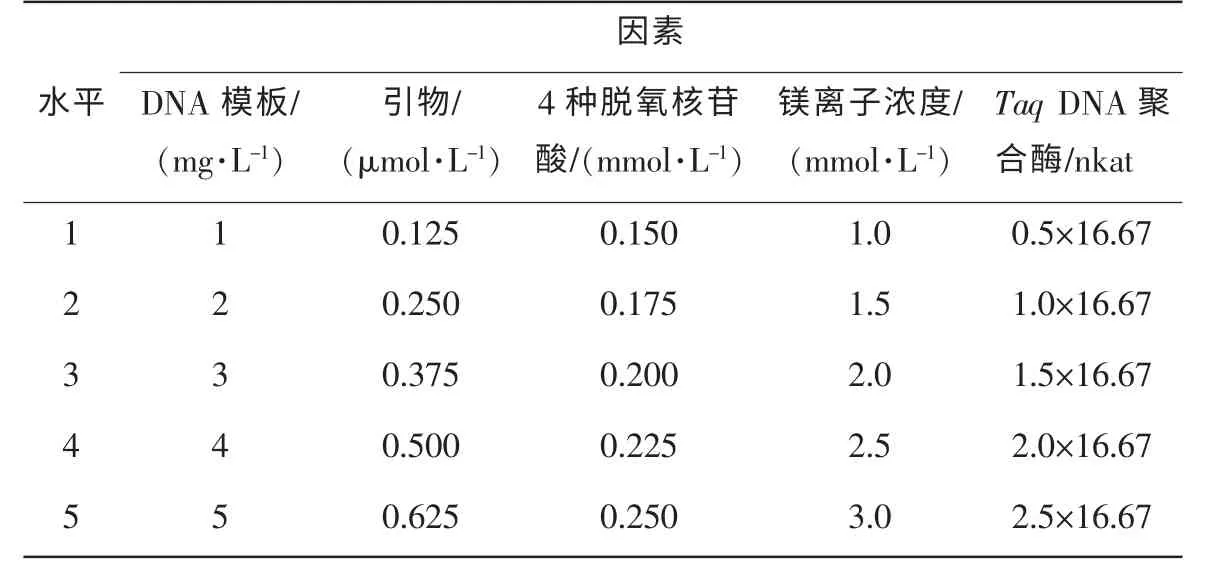
表1 正交试验的因素及水平Table 1 Factors and levels of orthogonal design

表2 L25(56)正交设计表Table 2 Orthogonal design with L25(56)
1.3.3 单因素试验 根据正交设计试验筛选出的最佳反应体系设计单因素试验, 对各个因素进行逐一优化(表3)。
1.4 退火温度优化
在最佳反应体系的基础上, 在温度梯度PCR仪上设置12 个温度梯度(47~59 ℃)对所用引物的退火温度进行优化, 扩增产物于10.0 g·kg-1的琼脂糖凝胶电泳检测。

表3 SCoT-PCR 扩增影响因子的梯度试验Table 3 Gradient experiment of factors influencing SCoT-PCR
1.5 数据统计与处理
参照桂腾琴 等[16]的方法, 根据PCR 扩增产物谱带的强度及清晰度、 杂带的多少和背景的深浅对正交试验各个处理依次打分, 将条带清晰、 丰富、 稳定的最佳处理记 “25” 分, 最差的记“1” 分。 不同的引物, 不同的模板以及不同的重复分别独立统计, 计算每个处理的平均分得分, 依据平均得分对试验结果进行分析, 列出正交试验的极差分析表以进行直观分析, 利用SPSS 17.0 对结果进一步进行方差分析, 综合2 种分析方法确定最佳组合。
1.6 反应体系的稳定性和通用性检测
选取引物P40 对34 份石蒜属植物DNA 模板进行扩增; 同时随机选取7 条引物对中国石蒜的DNA 进行扩增, 扩增产物用10.0 g·kg-1的琼脂糖凝胶电泳检测以检测优化的SCoT-PCR 反应体系的通用性、 稳定性和可靠性。
2 结果与分析
2.1 正交试验结果分析

图1 SCoT-PCR 反应体系正交实验结果Figure 1 Results of SCoT-PCR system with orthogonal design
2.1.1 直观分析 由正交试验扩增结果见图1。 25 个组合中23 个有谱带产生, 其中1 号和12 号组合没有扩增出产物或产物极少, 4 号、 5 号、 7 号、 11 号和16 号扩增谱带清晰、 丰富。 依据谱带的强弱、清晰度、 杂带的多少以及背景的强弱对PCR 扩增结果依次打分。 条带数量丰富、清晰的最佳产物记25 分, 最差的计1 分。每次处理及重复分别独立统计, 25个组合的分数依次为2.00,5.33,12.67,19.67,18.33,15.67,20.67,15.67,3.67,13.67,21.67,1.00,17.33,13.33,7.67,18.67,8.00,16.67,16.33,7.33,17.00,20.00,12.00,10.33,10.33。再根据平均分算出每个因素同一水平下的试验值之和抑制常数Ki以及每一因素水平下的数据平均值ki, 并求出同一因素不同水平间平均值的极差R, 对正交试验结果进行直观分析(表4)。 根据极差大小可以判断因素对反应体系的主次影响顺序。 R 越大, 表示该因素的水平变化对试验指标的影响越大, 因素越重要。 由以上分析可见, 因素影响主次顺序为Mg2+>Taq DNA 聚合酶>引物>dNTPs>DNA 浓度, Mg2+对PCR 反应体系影响最大, 为主要因素, DNA 浓度影响最小。 而根据各因素各水平的平均值可以确定优水平, 进而选出优组合。 每一因素水平下的数据平均值ki反映了影响因素各水平对反应体系的影响情况, ki值越大, 反应水平越好。 由表4 可以知道: SCoT-PCR 反应中5 个影响因素的最佳反应水平组合并没有在正交组合中出现, 但与分值最高的几个组合都比较接近。
2.1.2 方差分析 虽然直观分析具有直观简单、 计算量小等优点, 但直观分析法不能估计误差的大小,不能精确地估计各因素的试验结果影响的重要程度, 而方差分析可以弥补直观分析的这些不足, 得出更准确的试验结果[17]。 应用SPSS 17.0 软件对试验结果进行方差分析(表5), 结果表明: 镁离子浓度和Taq DNA 聚合酶酶量对试验结果有显著影响, DNA 模板浓度、 引物浓度和dNTPs 浓度对试验结果影响不显著, 各因素对试验结果影响的主次顺序与直观分析结果一致。 图1 和图2 分别为5 个因素对试验结果影响的趋势图, 由图1~2 可知: 该PCR 反应体系的最佳组合也与直观分析相一致。 经多重考虑分析得出石蒜属植物SCoT-PCR 反应体系的最优组合为: DNA 模板2.000 mg·L-1, 引物0.125 μmol·L-1, dNTPs 0.200 mmol·L-1, Mg2+3.000 mmol·L-1, Taq DNA 聚合酶1.0 × 16.67 nkat。
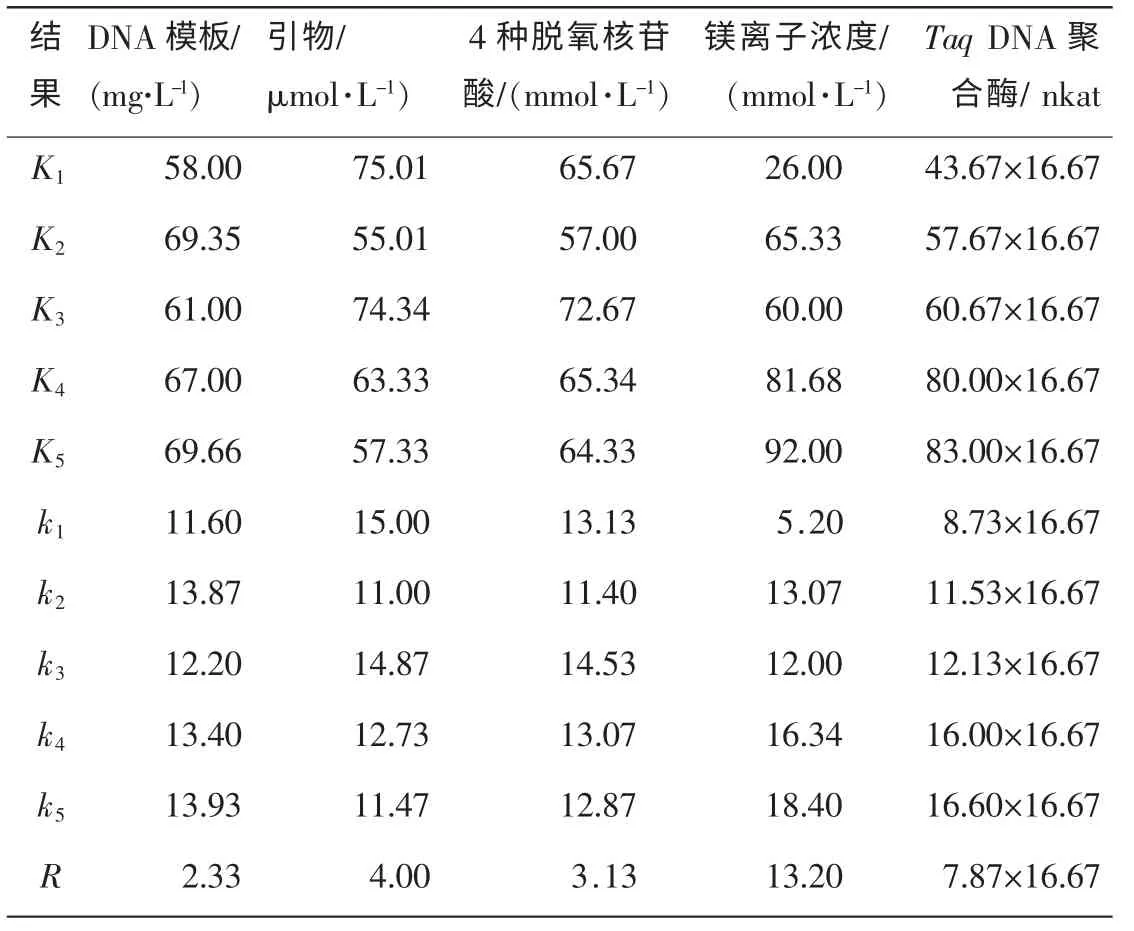
表4 正交试验直观分析Table 4 Intuitive analysis of orthogonal design

图2 DNA, 引物, dNTPs, Mg2+和Taq DNA 聚合酶5 个因素的效应Figure 2 Curve effect of DNA, primer, dNTPs, Mg2+and Taq DNA polymerase
2.2 单因素试验结果分析
PCR 反应对DNA 模板的纯度要求不高, 但DNA 模板的用量过大会对Mg2+的有效浓度起到干扰作用, 会降低特异性扩增效率, 增加非特异性产物或者使扩增失败, 而过少则可能扩增不出条带或条带太弱不利于分析[15]。一般情况下, 100.0 μL 体系中加入100.0 ng 的量即可。 由图3 可以看出: DNA 模板5 个浓度都能扩增出丰富的谱带, 但当质量浓度为2.000 mg·L-1时, 扩增谱带的质量最好, 该结果与正交试验的结果相吻合。
引物是PCR 反应的原料, 引物浓度过高会扩增出非特异性谱带, 引物浓度达到0.375 μmol·L-1时即出现特异性谱带。 由图3可以看出: 该SCoT-PCR 反应体系中最佳引物浓度为0.125 μmol·L-1。

表5 应用SPSS 17.0 进行方差分析Table 5 Analysis of variances by SPSS 17.0
dNTPs 浓度直接影响PCR 扩增, dNTPs与Taq DNA 聚合酶竞争Mg2+, 当dNTPs 浓度较低时, 会影响合成效率, 直接影响扩增产物的浓度, 甚至引起产物单链化而影响扩增 效 果[18]。 由 图4 可 看 出, 当dNTPs 浓度 为0.150 mmol·L-1和0.175 mmol·L-1时,扩增出的条带明显比高浓度少, 经综合分析比较, 得出0.200 mm·L-1为该反应体系中dNTPs 的最适合浓度。
Taq DNA 聚合酶在合成新DNA 链时,要求有游离的Mg2+。 Mg2+浓度太低无PCR 产物产生, 太高会导致非特异的产物产生[19]。 由 图4 可 以 看 出: 当Mg2+浓 度 为1.000 mmol·L-1时, 没有PCR 产物产生, 当Mg2+浓度为3.000 mmol·L-1时, 产生的谱带清晰、 稳定, 是该反应体系中的最优浓度。 而Taq DNA 聚合酶在5 个浓度下均能产生清晰稳定的谱带, 在考虑经济费用及试验质量的前提下, 将反应体系中的酶浓度定为1.0 × 16.67 nkat。

图3 DNA 模板浓度和引物浓度对SCoT-PCR 反应的影响Figure 3 Effect of DNA template concentration and primer concentration on SCoT-PCR

图4 dNTPs 浓度、 Mg2+浓度和Taq DNA 聚合酶浓度对SCoT-PCR 反应的影响Figure 4 Effect of dNTPs concentration、 primer concentration and Taq DNA polymerase concentration on SCoT-PCR
石蒜属植物SCoT-PCR 反应的最优体系为: DNA 模板2.000 mg·L-1, 引物0.125 μmol·L-1, dNTPs 0.200 mmol·L-1, Mg2+3.000 mmol·L-1, Taq DNA 聚合酶1.0×16.67 nkat。
2.3 SCoT-PCR 最佳退火温度选择
PCR 扩增反应过程中, 退火温度是影响PCR 产物特异性的重要因素, 退火温度的高低直接影响引物与模板DNA 的特异性结合, 不同的引物退火温度可能不同。 随机选择P13, P15, P16, P38, P39,P40, P57 等7 条SCoT 引物(表6) 进行12 个不同退火温度梯度试验, 其中引物P40 的扩增结果见图5。 在12 个退火温度中, 退火温度过低, 其扩增出的条带较少, 背景较强, 当退火温度接近最适温时,扩增的条带清晰, 丰富, 背景较弱。 而退火温度过高, 则引物与DNA 模板的特异性结合减弱, 扩增产物减少, 条带不清晰, 即温度过高或过低扩增效果均不理想, 因此, 确定引物P40 的最佳退火温度为57.9 ℃。
2.4 SCoT-PCR 反应体系及反应参数的稳定性和通用性检测
一方面, 引物选取P40, DNA 模板选取石蒜属植物不同种的34 份材料进行PCR 扩增; 另一方面,DNA 模板选取第23 号材料中国石蒜的DNA, 引物选取退火温度试验中的7 条 (分别为P13, P15,P16, P38, P39, P40, P57)进行PCR 扩增, 对优化后的SCoT-PCR 反应体系进行稳定性检测, 结果均能获得清晰可辨、 多态性丰富的条带 (图6~7), 说明该优化体系适用于石蒜属植物的遗传多样性的分析。

表6 7 条SCoT 引物Table 6 Seven SCoT primers

图5 不同退火温度下SCoT-PCR 的扩增结果Figure 5 Result of SCoT-PCR amplification at different annealing temperatures
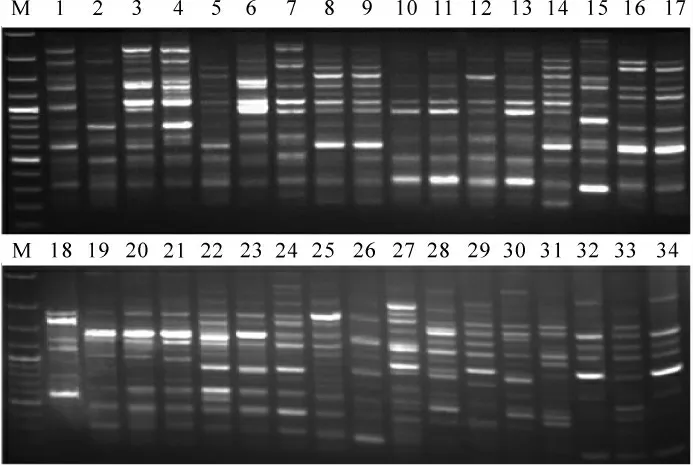
图6 34 份石蒜材料的SCoT-PCR 扩增结果Figure 6 SCoT-PCR amplification results in 34 Lycoris samplings
3 讨论
3.1 SCoT 标记的特点
SCoT 标记作为一种新型的分子标记, 已成功应用于花生, 葡萄Vitis vinifera, 龙眼, 枇杷Eriobotrya japonica[20],牡丹Paeonia suffruticosa[21], 芒果Mangifera indica[22-23]和柑橘[24]等作物的遗传育种和系统学分类研究, 可知该标记在物种间的通用性较好。 SCoT 标记的引物为单引物, 引物长度适中, 为18 个碱基的核苷酸。 在扩增时, 可以同时结合在双链DNA 的正义链与反义链上, 从而扩增出引物之间的区域, 其错配的几率小, 产生的条带丰富, 重复性好[11]。 且其PCR 产物可直接用琼脂糖凝胶电泳进行检测, 其操作简单, 用时短, 成本低; 而聚丙烯酰胺凝胶电泳操作复杂, 耗时, 成本较高且增加了影响因素[25]。 如果在用琼脂糖凝胶电泳检测时, 没有呈现明显的差异性, 可改用聚丙烯酰胺凝胶进行电泳, 为我们的研究提供更多的选择。 SCoT 标记的不足主要在于其PCR 体系偏小且该标记为显性标记, 不能区分杂合子与纯合子, 即不能进行杂交种的鉴定。

图7 7 条引物的SCoT-PCR 扩增结果Figure 7 SCoT-PCR amplification results in 7 primers
3.2 SCoT 标记在石蒜属中应用的优势
由于石蒜属植物种类丰富, 分布广泛, 且一些形态相似的种其分布区域又存在重叠现象, 因此使该属植物的物种鉴别、 亲缘关系研究以及分类等问题复杂化。 之前关于石蒜属植物的分子生物学研究中,不同的分子标记其研究的结果存在一定的差异性, 对于该属植物的亲缘关系一直没有定论。 在育种方面, 我们一直致力于选育矮干, 花型优美, 花色较纯的品种, 而SCoT 标记作为一种可以追踪性状且通用性良好的新型标记, 可为石蒜属植物观赏性状的分子关联技术及杂交育种提供一种新的方法, 且可对之前石蒜属植物的遗传多样性分析进行补充。
3.3 结论
本研究采用正交设计和单因素试验2 种方法对影响SCoT-PCR 反应的5 个主要因素进行了优化, 首次建立了适用于石蒜属植物遗传多样性分析的SCoT-PCR 扩增体系, 其PCR 最佳组合为: DNA 模板2.0 mg·L-1, 引物0.125 μmol·L-1, dNTPs 0.200 mmol·L-1, 镁 离 子(Mg2+)3.000 mmol·L-1, Taq DNA 聚合酶1.0×16.67 nkat。 对石蒜属植物SCoT-PCR 反应体系影响最显著的因素是Mg2+浓度和Taq DNA 聚合酶浓度, 而dNTPs 浓度、 引物、 DNA 模板用量对该体系的扩增结果无显著影响, 与赵瑞强等[15]在铁皮石斛Dendrobium officinale 上的研究结果基本一致。 退火温度也是SCoT-PCR 反应中的重要影响因子, 在本研究中得出每个引物的最适退火温度在其(Tm±5 )℃范围内, 与最初设计引物的标准一致。 本研究将优化的SCoT-PCR 体系在石蒜属不同种及杂交种上进行通用性及稳定性验证, 均得到了特异性丰富的谱带,即表明ScoT 标记适于石蒜属种质资源的遗传多样性分析。
[1] 裴鉴, 丁志遵. 中国植物志: 第16 卷 [M]. 北京: 科学出版社, 1985: 16 - 27.
[2] 邓传良, 周坚.长筒石蒜花被片DNA 的提取及ISSR 体系的建立[J].江西农业大学学报, 2005, 27(2): 257- 261.DENG Chuanliang, ZHOU Jian. A study on the perianth dna extraction and optimization of ISSR reaction system in Lycoris longituba [J]. Acta Agric Univ Jiangxi, 2005, 27(2): 257 - 261.
[3] 邓传良, 周坚, 卢龙斗, 等. 长筒石蒜种质资源的RAPD 及ISSR 研究[J]. 云南植物研究, 2006, 28(3): 300-304.DENG Chuanliang, ZHOU Jian, LU Longdou, et al. Study on germplasm resources of Lycoris longituba(Amarylliadaceae)by RAPD and ISSR [J]. Acta Bot Yunnan, 2006, 28(3): 300 - 304.
[4] 邓传良, 周坚. 石蒜属植物分支系统学分析[J]. 植物研究, 2005(4): 393 - 399.DENG Chuanliang, ZHOU Jian.A cladistic analysis of Lycoris(Amaryllidaceae)[J].Bull Bot Res, 2005(4):393-399.
[5] 袁菊红, 孙视, 彭峰, 等. 石蒜属植物遗传多样性的ISSR 和RAPD 标记比较研究[J].中草药, 2007, 38(10):1555-1561.YUAN Juhong, SUN Shi, PENG Feng, et al. Comparison between ISSR and RAPD markers in genetic diversity of plants in Lycoris Herb.[J]. Chin Trad Herb Drugs, 2007, 38(10): 1555-1561.
[6] 时剑, 童再康, 黄华宏, 等. 中国石蒜SSR 体系的建立及性状对应分析[J]. 园艺学报, 2011, 38(3): 571-578.SHI Jian, TONG Zaikang, HUANG Huahong, et al. Establishment of SSR molecular marker system and analysis of geneticcharacter in Lycoris chinensis[J]. Acta Hort Sin, 2011, 38(3): 571-578.
[7] COLLARD B C Y, MACKILL D J. Start codon targeted(SCoT)polymorphism: a simple, novel DNA marker technique for generating gene-targeted markers in plants [J]. Plant Mol Biol Rep, 2008, 27: 86-93.
[8] XIONG Faqian, TANG Ronghua, CHEN Zhongliang, et al. SCoT: a novel gene targeted marker technique based on the translation start codon [J]. Mol Plant Breed, 2009, 7: 635-638.
[9] 张君玉, 郭大龙, 龚莹, 等.葡萄目标起始密码子多态性反应体系的优化[J].果树学报, 2011, 28(2): 209-214.ZHANG Junyu, GUO Dalong, GONG Ying, et al. Optimization of start codon targeted polymorphism PCR(SCoT-PCR)system in Vitis vinifera[J]. J Fruit Sci, 2011, 28(2): 209-214.
[10] 熊发前, 蒋菁, 钟瑞春, 等.目标起始密码子多态性(SCoT)分子标记技术在花生属中的应用[J]. 作物学报, 2010, 36(12): 2055-2061.XIONG Faqian, JIANG Jing, ZHONG Ruichun, et al. Application of SCoT molecular marker in genus Arachis[J].Acta Agron Sin, 2010, 36(12): 2055 - 2061.
[11] 蒋巧巧, 龙桂友, 李武文, 等. SCoT 结合克隆测序鉴别湖南甜橙变异类型[J]. 中国农学通报, 2011, 27(6): 148-154.JIANG Qiaoqiao, LONG Guiyou, LI Wuwen, et al. Identification of genetic variation of sweet orange clones from Hunan by using SCoT markers and sequence analysis [J]. Chin Agric Sci Bull, 2011, 27(6): 148-154.
[12] 陈虎, 何新华, 罗聪, 等. 龙眼24 个品种的SCoT 遗传多样性分析[J]. 园艺学报, 2010, 37(10): 1651-1654.CHEN Hu, HE Xinhua, LUO Cong, et al. Analysis on the genetic diversity of 24 Longan (Dimocarpus longan)accessions by SCoT markers[J]. Acta Hort Sin, 2010, 37(10): 1651-1654.
[13] 陈虎, 何新华, 罗聪, 等.龙眼ScoT-PCR 反应体系的优化[J]. 基因组学与应用生物学, 2009, 28(5): 970-974.CHEN Hu, HE Xinhua, LUO Cong, et al. The optimization of SCoT-PCR system of Longan(Dimocarpus longan)[J].Genom Appl Biol, 2009, 28(5): 970-974.
[14] 陈香玲, 李杨瑞, 杨丽涛, 等. 低温胁迫下甘蔗抗寒相关基因的cDNA-SCoT 差异显示[J]. 生物技术通报,2010(8): 120-124.CHEN Xiangling, LI Yangrui, YANG Litao, et al. cDNA-SCoT differentia display of cold resistance related genes in sugar cane under low temperature stress[J]. Biotechnol Bull, 2010(8): 120-124.
[15] 赵瑞强, 章晓玲, 高燕会, 等. 铁皮石斛SCoT-PCR 反应体系构建及优化[J]. 核农学报, 2012,26(4): 648-655.ZHAO Ruiqiang, ZHANG Xiaoling, GAO Yanhui, et al. Establishment and optimization of SCoT-PCR reaction system for Dendrobium officinale[J]. J Nucl Agric Sci, 2012, 26(4): 648-655.
[16] 桂腾琴, 孙敏, 乔爱民, 等. 正交设计优化果梅ISSR 反应体系[J]. 果树学报, 2009, 26(1): 108-112.GUI Tengqin, SUN Min, QIAO Aimin, et al. Optimization of an ISSR reaction system of Japanese apricot(Prunus mume) based on orthogonal design [J]. J Fruit Sci, 2009, 26(1): 108-112.
[17] 李云雁, 胡传荣. 试验设计与数据处理[M]. 2 版. 北京: 化学工业出版社, 2008: 146.
[18] 马明, 杨克强, 刘晓菊, 等. 核桃Juglans regia SRAP 标记反应体系建立的研究[J] . 山东农业大学学报: 自然科学版, 2007, 38(2): 189-192.MA Ming, YANG Keqiang, LIU Xiaoju, et al.Establishment of stap marker reaction system in walnut[J]. J Shandong Agric Univ Nat Sci Edi, 2007, 38(2): 189-192.
[19] REN Yu, WANG Deyuan, ZHANG Yindong, et al.Optimization of SRAP-PCR in hot pepper(Capsicum annuum L.)[J]. Mol Plant Breed, 2004, 2(5): 689-693.
[20] 韩国辉, 汪卫星, 向素琼, 等.多倍体枇杷SCoT 分析体系的建立与优化[J].果树学报, 2011, 28(3): 433-437.HAN Guohui, WANG Weixing, XIANG Suqiong, et al. Establishment and optimization of SCoT system in polyploidy loquats [J]. J Fruit Sci, 2011, 28(3): 433-437.
[21] 侯小改, 王娟, 贾甜, 等. 牡丹SCoT 分子标记正交优化及引物筛选[J]. 华北农学报, 2011, 26(5): 92-96.HOU Xiaogai, WANG Juan, JIA Tian, et al. Orthogonal optimization of SCoT-PCR system and primer screening of tree peony [J]. Acta Agric Boreali-Sin, 2011, 26(5): 92-96.
[22] LUO Cong, HE Xinhua, CHEN Hu, et al. Analysis of diversity and relationships among mango cultivars using start codon targeted(SCoT) markers [J]. Biochem Systd Ecol, 2012, 38(6): 1176-1184.
[23] LUO Cong, HE Xinhua, CHEN Hu, et al. Genetic diversity of mango cultivars estimated using SCoT and ISSR markers [J]. Biochem Syst Ecol, 2011, 39(4/6): 676-684.
[24] 韩国辉, 向素琼, 汪卫星, 等. 柑橘SCoT 分子标记技术体系的建立及其在遗传分析中的应用[J]. 园艺学报, 2011, 38(7): 1243-1250.HAN Guohui, XIANG Suqiong, WANG Weixing, et al. Establishment and application of SCoT molecular marker system for Citrus [J]. Acta Hort Sin, 2011, 38(7): 1243-1250.
[25] 陈香玲, 李杨瑞, 杨丽涛, 等. cDNA-SCoT 基因差异表达2 种电泳方法的比较研究[J].生物技术通报, 2010(10): 93-95.CHEN Xiangling, LI Yangrui, YANG Litao, et al. Comparison of two electrophoresis methods in cDNA-SCoT gene differential expression [J]. Biotechnol Bul, 2010(10): 93-95.

