嗜纤维梭菌纤维体研究进展
李俊霞 张日俊 陈东东
(中国农业大学动物科技学院,北京 100193)
大多数植物细胞壁含有约15%-40%的纤维素、30%-40%的半纤维素、几丁质和20%的木质素[1]。这些组分难以被畜禽降解,但是可以被某些微生物产生的相应的酶系所降解。借助于这些酶的作用,植物细胞中的淀粉、蛋白质和脂肪等营养物质得以释放,从而提高植物饲料的消化利用率。微生物产生的能降解植物细胞壁的酶系分为两类,第一类为好氧真菌和细菌产生的游离纤维素酶系、半纤维素酶系和一些辅酶;第二类为某些厌氧微生物产生的大分子量胞外多酶复合体(分子量650 kD-2.5 MD)[2],称为纤维体(cellulosome)。
纤维体的基本组件是非催化活性的脚手架蛋白(scaffoldin)和多个具有催化活性的糖基水解酶亚单位。脚手架蛋白上含有多个黏附域(cohesin modules),每个酶亚单位上也都含有坞因子(dockerin domain),酶亚单位通过各自的坞因子和脚手架蛋白上相应的黏附域特异性相结合,充分发挥各酶之间的协同作用,高效降解植物细胞壁。因此,纤维体是一个完美组合的实体,而非各个组分的简单聚集,其降解细胞壁的效率远远高于几个游离酶的协同。纤维体高效降解细胞壁的特性引起了广大研究者的兴趣。
目前仅在一些厌氧细菌中得到了纤维体,尤其是梭菌和瘤胃微生物,如Bacteroides cellulosolvens(溶纤维拟杆菌)[3],Clostridium cellulovorans(嗜纤维梭菌)[4],C. thermocellum(热梭菌)[5],C.cellulolyticum(解纤维梭菌)[6],C. josui(约氏梭菌)[7],C. papyrosolvens(溶纸莎草梭菌)[8],Ruminococcus flavefaciens(生黄瘤胃球菌)[9],R. albus(白色瘤胃球菌)[10]。最近,有报道称在厌氧真菌Piromyces equi中也发现了纤维体,但是并未发现编码脚手架蛋白基因的存在。在产生纤维体的细菌中,C.cellulovorans分离自木浆粗纤维纸,是一种产孢、嗜中温的革兰氏阳性菌,它产生大量的纤维素酶,能降解晶型纤维素[11]。本研究就针对C.cellulovorans纤维体的结构、功能、基因簇和基因工程方面的研究进展进行综述,以期为其他纤维体的研究提供思路及构建特殊功能的mini-cellulosome、纤维体在生物技术上的应用奠定理论基础。
1 Clostridium cellulovorans纤维体的组成
嗜纤维梭菌纤维体依附在细胞表面,分子量大约为1×106Da,有至少10个亚单位组成。其中一个重要的亚单位是脚手架蛋白CbpA,并且只含有1个CbpA[12],分子量为189 kD。脚手架蛋白不具有催化活性,但是含有多个功能域,模型见图1。
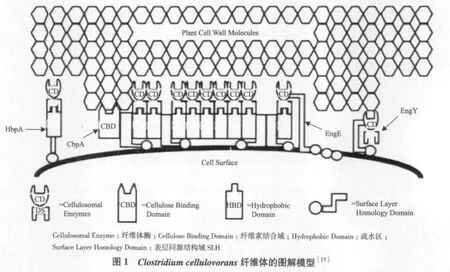
1.1 脚手架蛋白的结构和功能
嗜纤维梭菌纤维体的SDS-PAGE电泳分析结果显示,纤维体存在多个亚单位,包括3个主要的条带170 kD(P170),100 kD(P100)和70 kD(P70),其他的小条带分子量范围在35 kD-120 kD之间[11]。羧甲基纤维素酶活性分析显示,只有P170蛋白没有催化活性。P170蛋白具有多重功能,除了结合多酶亚单位外,还可以将纤维素的结晶型排列方式转变成易于被酶亚单位降解的非晶型排列,使得整个纤维体能有效降解结晶型纤维素,而纤维体上单个游离酶只能降解可溶性和非结晶纤维素[11]。
通过反相PCR得到了编码P170蛋白的全部基因序列,此基因被命名为cbpA,编码的蛋白称为脚手架蛋白CbpA,其分子量为189 kD[14]。cbpA基因的N端编码信号肽,成熟蛋白的N端存在1个纤维素结合域CBD,4个表层同源结构域SLH[15],9个黏附域或称酶结合域(也称疏水区)[14]。CBD能结合不同形式的纤维素,但是相对于非晶型纤维素来说,此CBD更偏向于结合结晶型纤维素。CBD能结合和晶型纤维素结构相似的几丁质。CBD对纤维体和降解基质之间的结合发挥着重要作用。针对C.cellulovorans而言,纤维体上的4个表层同源结构域也称亲水区。另外,在研究EngE的结构域时发现3个重复的类-SLH域串联位于内切葡聚糖酶基因engE的N端,内切葡聚糖酶通过此结构域既和脚手架蛋白CbpA相连又结合在细菌表面。9个黏附域高度保守、同源,各个酶亚单位通过坞因子-黏附域特异结合在脚手架蛋白上。Park等[16]研究黏附域1、6与两个酶亚单位P100、P70之间的亲和性,结果显示黏附域6比黏附域1更高效的结合这两种酶亚单位,说明黏附域结合酶亚单位的能力不同。但是关于C. thermocellum和C. cellulolyticum纤维体的相关研究结果表明,不同黏附域和酶亚单位上坞因子的结合能力相同。另外一项相关研究是体外克隆C.cellulovorans的黏附域,结果发现克隆出的黏附域同样可以与纤维体的酶亚单位相结合,说明了酶亚单位上的坞因子是黏附域和酶亚单位结合的关键性因素。根据这几项研究结果推测,黏附域和酶亚单位的结合能力可能取决于酶亚单位上坞因子的某些特性。如坞因子的大小和空间结构等,不同的产纤维体菌种,其坞因子的这类特性不同。关于黏附域的功能,Park和Doi还发现了黏附域不仅结合酶亚单位,而且不同的黏附域之间也能互相结合,这就使得不同CbpA之间通过黏附域聚集从而形成多聚纤维体。
纤维体上除CbpA非酶蛋白外,还存在另外一个非酶蛋白HbpA。其分子量为24.94 kD,含有一个SLH和一个黏附域,其黏附域和CbpA的黏附域相似。研究结果表明,HbpA既可以通过其上的SLH连接到C. cellulovorans的细胞壁上,又可结合纤维体酶和多糖底物,如纤维素、几丁质和木聚糖[17]。
1.2 纤维体纤维素酶和非纤维体纤维素酶
C. cellulovorans能合成纤维体纤维素酶和非纤维体纤维素酶两种,非纤维体酶基因上不存在坞因子序列,而纤维体酶一般含有坞因子序列(22个氨基酸的重复序列)。C. cellulovorans的纤维体酶基因大部分位于纤维体基因簇上,编码的纤维体酶包括1个外切葡聚糖酶(ExgS)、5个内切葡聚糖酶(EngH、EngK、EngL、EngM和EngN)和1个甘露聚糖酶(ManA)。除了上述7种酶组分,纤维体酶还包括2个内切葡聚糖酶EngB、EngE[18](同时也具有木聚糖酶活性)、1个果胶酸酯酶(EngY-PelA)[19]和1个木聚糖酶(XynA)[20],只是编码这几种酶的基因位于染色体的其他位置而非纤维体基因簇上,并且这几个酶基因互不相连。最近,Han[21]又分离和表达了一种新的木聚糖酶基因xynB。另外,Jeon等[22]检测到一种新的内切葡聚糖酶EngZ,但此内切酶具有很强的降解结晶纤维素活性。所有酶亚单位上的坞因子高度保守。除了坞因子,每个酶亚单位还包含催化域和信号肽,但是CBD和类Ig域并非每个酶亚单位都有。C. cellulovorans纤维体酶亚单位的种类繁多使得合成的纤维体能有效降解植物细胞壁的所有组分纤维素、木聚糖、甘露寡糖和果胶。
C. cellulovorans不仅能合成纤维体纤维素酶,还能产生游离纤维素酶——EngD(内切葡聚糖酶、木聚糖酶)和EngF(内切葡聚糖酶、纤维糊精酶)。engD和engB基因有较高的同源性[23]。
2 纤维体基因簇
C. cellulovorans纤维体基因簇含有9个相连基因,顺序为cbpA-exgS-engH-engK-hbpA-engL-manA-engM-engN,总基因长22 kb(图2)。此纤维体基因簇的5'端固定有非纤维体操纵子,由nifV-orf1-sigX-regA基因组成,3'端跟随的是与转座酶基因(trp)、苹果酸酯酶基因(mle)有同源性的非纤维体基因[24]。最近研究者还发现了engL基因簇并对其进行了测序,结果显示,engL基因簇包括5个不同的开放阅读框,包含编码ManA的基因序列[25]。除了C. cellulovorans纤维体,在C. cellulolyticum和C. josui纤维体中还发现了纤维体基因簇,但并未在C.thermocellum中发现纤维体基因簇,因此研究者推测,染色体上的纤维体基因簇是嗜温梭状芽孢杆菌的典型特点。
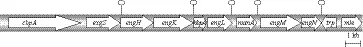
图2 Clostridium cellulovorans纤维体基因
3 纤维体基因的表达调控
研究者通过将C.cellulovorans培养在不同的基质中研究纤维体上的纤维素酶基因和半纤维素酶基因表达的调控因素[26,27]。当培养基中含有纤维二糖和纤维素中,纤维素基因(engE)和半纤维素基因(xynA)共表达;基质中纤维素、木聚糖和果胶的存在,使得纤维体中大部分的基因得到表达;而将纤维二糖和果糖作为碳源的时候,cbpA、engH、manA、engE和xynA的表达减弱;将碳源换为葡萄糖、半乳糖、麦芽糖和蔗糖等可溶单糖或二糖的时候,cbpA、engH、manA、engE和xynA基本不表达。xynA基因和pelA基因只有基质中分别含木聚糖和果胶的时候才会特异性表达。根据这些结果研究者推测出纤维素酶基因和半纤维素酶基因之间存在协同表达,在纤维素酶基因表达过程中可能存在一种降解物阻遏调控机制。
Cho等[28]的研究结果显示,当碳源为微晶纤维素和AXP(微晶纤维素-木聚糖-果胶,3∶1∶1)时,EngE、ExgS、EngK、XynB和ManA都表达,但是,EngY只在AXP培养基中才能检测到。Han等[29]还观察了C.cellulovorans培养在不同基质中纤维体与非纤维体酶的mRNA和蛋白表达的变化,以此理解纤维体和非纤维体水解酶之间的协同作用。结果发现,在多种碳源底物(纤维素、果胶、木聚糖、纤维二糖和玉米纤维)中纤维体和非纤维体酶都有协同活性。与非纤维体酶相比,碳源对纤维体的蛋白质组学影响更大。纤维素、果胶和木聚糖的混合物可以诱导多种酶表达。
4 Clostridium cellulovorans纤维体基因工程研究进展
在过去几年中研究者已对纤维体中纤维素酶各亚单位基因engB、engE、engH、engK、engL、engM、engY、exgS、木聚糖酶基因xynA、甘露聚糖酶基因manA及果胶酶基因pelA进行了克隆和测序,并已经研究不同组分之间对降解植物细胞壁的协同效应,包括纤维素酶亚单位之间的协同[30]、纤维素酶亚单位和半纤维素酶亚单位之间的协同[31,32]及纤维体纤维素酶与非纤维体纤维素酶之间的协同[33]。在这些协同效应的研究中,构建mini-cellulosome是一种有效的方法。如,Jeson等[34]设计mini-celluloso-me研究EngE和ManB对半乳甘露聚糖降解的协同作用。这些研究也为体外构建重组高活性的或有特定功能的mini-cellulosome做足了准备。minicellulosome是目前的研究热点,是由mini-CbpA和一种或几种不同的酶组合成的小复合物。mini-CbpA上的黏附域可以是种特异性的,也可以是来自不同种的黏附域,种特异性的黏附域只能结合来自同种的酶,而后者的黏附域可以结合那些种的同源酶。mini-cellulosome和 mini-CbpA示意图分别见图3,图4。当脚手架蛋白CbpA基因在E. coli中表达时,得到的是不同大小的蛋白片段(9个),因为CbpA上的9个黏附域两两之间被切割开,所以得不到完整的CbpA蛋白。因此,Murashima等[35]分别将engB基因克隆转入Bacillus subtilisWB800菌株,将minicbpA基因转化至E. coli中表达。最后,将纯化的含有坞因子的活性木聚糖酶和mini-CbpA两部分体外聚集构成mini-cellulosome。在随后的研究中,Cho等[36]又将这两部分的基因串联融合,使得两种基因在Bacillus subtilisWB800共表达以进行体内聚集构建mini-cellulosome。这种体内体外构建minicellulosome的方法为得到高活性的多功能纤维素酶提供思路。Lilly等[37]实现了mini-cellulosome在酿酒酵母系统中的成功表达,增加了成功构建具有纤维素水解活性的啤酒酵母菌株的可行性。Hyeon等[38]构建了酿酒酵母工程菌,异源表达C.cellulovorans的mini-CbpA和C.thermocellum的内切葡聚糖酶CelE基因,在体内聚集成mini-cellulosome,同时在表达的葡萄糖苷酶的协助下发酵纤维质生产乙醇,实现了一步加工纤维质的目的,这种途径将大大降低生产成本。为了相同的目的,Jeon等[39]构建酿酒酵母工程菌,只是简单的表达C.cellulovorans的EngD和Saccharomycopsis fibuligera的β-葡萄糖苷酶基因,没有构建mini-cellulosome。
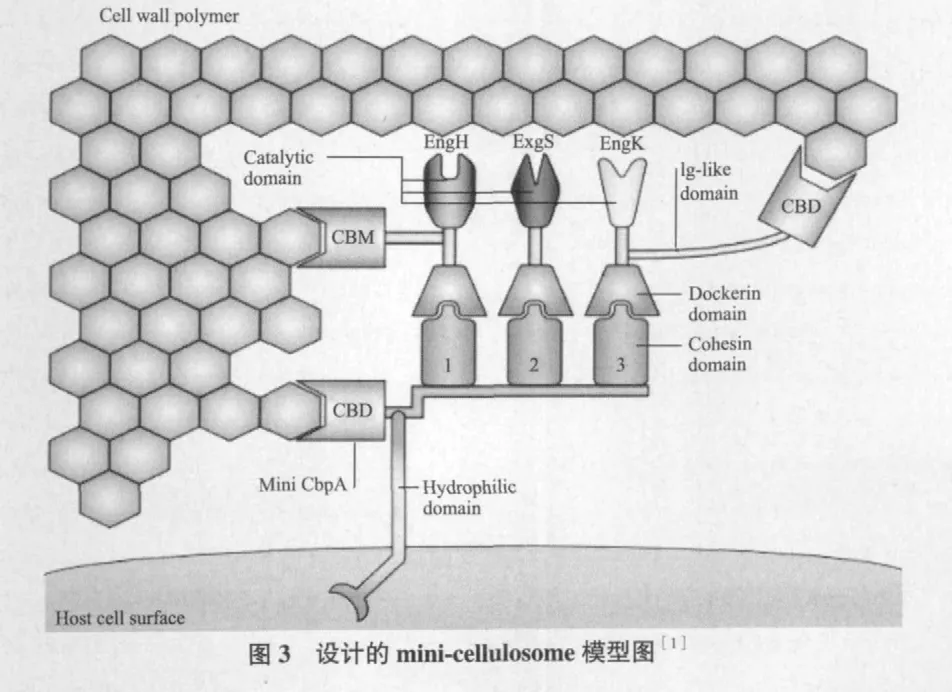
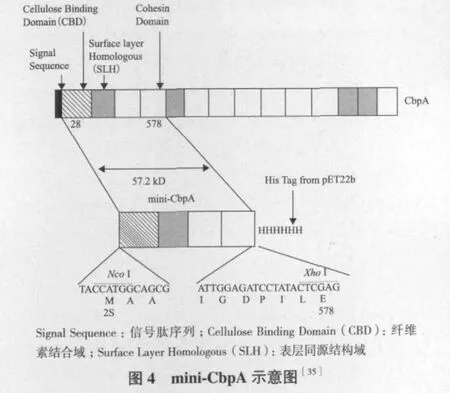
研究者还对改善纤维体酶的生物学特性进行了相关研究。如Murashima[40]用“分子育种”技术将engB基因和engD基因体外重组,得到了热稳定性比两种母本酶都提高并且活性不变的酶分子。Lee[41]将engD基因和Clostridium thermocellum的纤维体纤维素酶基因celE体外重组,大大提高了内切葡聚糖酶EngD的热稳定性和水解活性。另外,为搞清纤维体水解多糖的分子机制,研究者最近对几种梭菌进行了全基因组分析。Tamaru等[42]完成了C.cellulovorans743B(ATCC35296)的全基因组测序工作。基因组分析结果显示,C.cellulovorans基因组包括57个纤维体基因和大量的编码非纤维体酶基因[43]。纤维体基因不仅编码纤维素酶、脂肪酶和肽酶,还发现了两种新的脚手架蛋白CbpB和CbpC。另外,C.cellulovorans基因组分析结果显示,纤维体中还含有3个蛋白酶/肽酶抑制剂——cyspin 1、cyspin 2和 cyspin 3。Meguro等[44]的研究结果首次展示了3个蛋白酶/肽酶抑制剂的功能,即保护纤维体和产纤维体的微生物免遭植物蛋白酶的降解,保证纤维体对植物的降解顺利进行。
5 小结
研究者对嗜纤维梭菌纤维体系统进行近20年的研究成果为其他纤维体的研究提供了基本信息。归因于纤维体中糖基水解酶的多样性、水解酶之间的协同作用和纤维体与底物的紧密黏合,纤维体能高效降解植物细胞壁中的纤维素、半纤维素和几丁质等组分。对纤维体的基础研究会促进生物反应器和生物传感器的发展。纤维体的应用可提高饲料的消化利用率,还可将纤维素类物质转变为可发酵糖,以此产生可再生燃料如乙醇,为人们寻找再生性能源提供途径。提高纤维体的产量和纤维体的水解效率等都将会扩展纤维体的应用范围,但是如何提高目前还没有取得进展,有待于深入研究。
[1] Doi RH, Kosugi A. Cellulosomes:plant-cell-wall-degradingenzyme complexes[J].Nat Rev Microbiol, 2004, 2(7):541-551.
[2] Doi RH, Kosugi A, Murashima K, et al. Cellulosomes from mesophilic bacteria[J] . J Bacteriol, 2003, 185(20):5907.
[3] Ding SY, Bayer EA, Steiner D, et al. A scaffoldin of theBacteroides cellulosolvenscellulosome that contains 11 type II cohesins[J] .Journal of Bacteriology, 2000, 182(17):4915.
[4] Sleat R, Mah RA, Robinson R. Isolation and characterization of an anaerobic, cellulolytic bacterium,Clostridium cellulovoranssp.nov[J] . Appli Environ Microbiol, 1984, 48(1):88.
[5] Lamed R, Setter E, Bayer EA. Characterization of a cellulose-binding,cellulase-containing complex inClostridium thermocellum[J] .Journal of Bacteriology, 1983, 156(2):828.
[6] Pages S, Belaich A, Belaich JP, et al. Species-specificity of the cohesin-dockerin interaction betweenClostridium thermocellumandClostridium cellulolyticum:Prediction of specificity determinants of the dockerin domain[J] . Proteins, 1997, 29(4):517-527.
[7] Kakiuchi M, Isui A, Suzuki K, et al. Cloning and DNA sequencing of the genes encodingClostridium josuiscaffolding protein CipA and cellulase CelD and identification of their gene products as major components of the cellulosome[J] . J Bacteriol, 1998, 180(16):4303.
[8] Pohlschroder M, Leschine SB, Canale-Parola E. Multicomplex cellulase-xylanase system ofClostridium papyrosolvensC7[J] .Journal of Bacteriology, 1994, 176(1):70.
[9] Ding SY, Rincon MT, Lamed R, et al. Cellulosomal scaffoldinlike proteins fromRuminococcus flavefaciens[J] . Journal of Bacteriology, 2001, 183(6):1945.
[10] Ohara H, Karita S, Kimura T, et al. Characterization of the cellulolytic complex (cellulosome) fromRuminococcus albus[J] .Biosci, Biotechnol Biochem, 2000, 64(2):254-260.
[11] Doi RH, Tamaru Y. TheClostridium cellulovoranscellulosome:an enzyme complex with plant cell wall degrading activity[J] .Chemical Record, 2001, 1(1):24-32.
[12] Fontes CM, Gilbert HJ. Cellulosomes:highly efficient nanomachines designed to deconstruct plant cell wall complex carbohydrates[J] . Annu Rev Biochem, 2010, 79:655-681.
[13] Doi RH. Cellulases of mesophilic microorganisms:cellulosome and noncellulosome producers[J] . Annals of the New York Academy of Sciences, 2008, 1125(1):267-279.
[14] Shoseyov O, Takagi M, Goldstein MA, et al. Primary sequence analysis ofClostridium cellulovoranscellulose binding protein A[J] .Proc Nat Acad Sci USA, 1992, 89(8):3483.
[15] Goldstein MA, Takagi M, Hashida S, et al. Characterization of the cellulose-binding domain of theClostridium cellulovoranscellulosebinding protein A[J] . J Bacteriol, 1993, 175(18):5762.
[16] Park JS, Matano Y, Doi RH. Cohesin-dockerin interactions of cellulosomal subunits ofClostridium cellulovorans[J].Journal of Bacteriology, 2001, 183(18):5431.
[17] Matsuoka S, Yukawa H, Inui M, et al. Synergistic Interaction ofClostridium cellulovoransCellulosomal Cellulases and HbpA[J] .Journal of Bacteriology, 2007, 189(20):7190.
[18] Tamaru Y, Doi RH. Three surface layer homology domains at the N terminus of theClostridium cellulovoransmajor cellulosomal subunit EngE[J] . J Bacteriol, 1999, 181(10):3270.
[19] Tamaru Y, Doi RH. Pectate lyase A, an enzymatic subunit of theClostridium cellulovoranscellulosome[J] . Proceedings of the National Academy of Sciences, 2001, 98(7):4125.
[20] Kosugi A, Murashima K, Doi RH. Xylanase and acetyl xylan esterase activities of XynA, a key subunit of theClostridium cellulovoranscellulosome for xylan degradation[J].Applied and Environmental Microbiology, 2002, 68(12):6399.
[21] Han SO, Yukawa H, Inui M, et al. Isolation and expression of the xynB gene and its product, XynB, a consistent component of theClostridium cellulovoranscellulosome[J] . Journal of Bacteriology, 2004, 186(24):8347.
[22] Jeon SD, Yu KO, Kim SW, et al. The processive endoglucanase EngZ is active in crystalline cellulose degradation as a cellulosomal subunit ofClostridium cellulovorans[J].New Biotechnology, 2012, 29(3):365-371.
[23] Foong F, Doi R. Characterization and comparison ofClostridium cellulovoransendoglucanases-xylanases EngB and EngD hyperexpressed inEscherichia coli[J] . J Bacteriol, 1992, 174(4):1403.
[25] Tamaru Y, Karita S, Ibrahim A, et al. A large gene cluster for theClostridium cellulovoranscellulosome[J] . Journal of Bacteriology,2000, 182(20):5906.
[26] Han SO, Yukawa H, Inui M, et al. Regulation of expression of cellulosomal cellulase and hemicellulase genes inClostridium cellulovorans[J] . Journal of Bacteriology, 2003, 185(20):6067.
[27] Bayer EA, Belaich JP, Shoham Y, et al. The cellulosomes:multienzyme machines for degradation of plant cell wall polysaccharides[J] . Annual Review of Microbiology, 2004, 58:521-554.
[28] Cho W, Jeon SD, Shim HJ, et al. Cellulosomic profiling produced byClostridium cellulovoransduring growth on different carbon sources explored by the cohesin marker[J] . Journal of Biotechnology,2010, 145(3):233-239.
[29] Han SO, Cho HY, Yukawa H, et al. Regulation of expression of cellulosomes and noncellulosomal (hemi) cellulolytic enzymes inClostridium cellulovoransduring growth on different carbon sources[J] . Journal of Bacteriology, 2004, 186(13):4218.
[30] Murashima K, Kosugi A, Doi RH. Synergistic effects on crystalline cellulose degradation between cellulosomal cellulases fromClostridium cellulovorans[J] . J Bacteriol, 2002, 184(18):5088.
[31] Murashima K, Kosugi A, Doi RH. Synergistic effects of cellulosomal xylanase and cellulases fromClostridium cellulovoranson plant cell wall degradation[J] . J Bacteriol, 2003, 185(5):1518.
[32] Koukiekolo R, Cho HY, Kosugi A, et al. Degradation of corn fiber byClostridium cellulovoranscellulases and hemicellulases and contribution of scaffolding protein CbpA[J] . Applied and Environmental Microbiology, 2005, 71(7):3504.
[33] Kosugi A, Murashima K, Doi RH. Characterization of two noncellulosomal subunits, ArfA and BgaA, fromClostridium cellulovoransthat cooperate with the cellulosome in plant cell wall degradation[J] . Journal of Bacteriology, 2002, 184(24):6859.
[34] Jeon SD, Yu KO, Kim SW, et al. A celluloytic complex fromClostridium cellulovoransconsisting of mannanase B and endoglucanase E has synergistic effects on galactomannan degradation[J].Applied Microbiology and Biotechnology, 2011, 90:565-572.
[35] Murashima K, Chen CL, Kosugi A, et al. Heterologous production ofClostridium cellulovoransengB, using protease-deficientBacillus subtilis, and preparation of active recombinant cellulosomes[J] .Journal of Bacteriology, 2002, 184(1):76.
[36] Cho HY, Yukawa H, Inui M, et al. Production of minicellulosomes fromClostridium cellulovoransinBacillus subtilisWB800[J] .Applied and Environmental Microbiology, 2004, 70(9):5704.
[37] Lilly M, Fierobe HP, Van Zyl WH, et al. Heterologous expression of aClostridiumminicellulosome inSaccharomyces cerevisiae[J] .Fems Yeast Research, 2009, 9(8):1236-1249.
[38] Hyeon J, Yu KO, Suh DJ, et al. Production of minicellulosomes fromClostridium cellulovoransfor the fermentation of cellulosic ethanol using engineered recombinantSaccharomyces cerevisiae[J] .Fems Microbiology Letters, 2010, 310(1):39-47.
[39] Jeon E, Sung EL, Park BS, et al. Cellulosic alcoholic fermentation using recombinantSaccharomyces cerevisiaeengineered for the production ofClostridium cellulovoransendoglucanase and Saccharomycopsis fibuligeraβ-glucosidase[J].Fems Microbiology Letters,2009, 301(1):130-136.
[40] Murashima K, Kosugi A, Doi RH. Thermostabilization of cellulosomal endoglucanase EngB fromClostridium cellulovoransby in vitro DNA recombination with non-cellulosomal endoglucanase EngD[J] . Molecular Microbiology, 2002, 45(3):617-626.
[41] Lee CY, Yu KO, Kim SW, et al. Enhancement of the thermostability and activity of mesophilicClostridium cellulovoransEngD by in vitro DNA recombination withClostridium thermocellumCelE[J] . J Bioscience and Bioeng, 2010, 109(4):331-336.
[42] Tamaru Y, Miyake H, Kuroda K, et al. Genome sequence of the cellulosome-producing mesophilic organismClostridium cellulovorans743B[J].Journal of Bacteriology, 2010, 192(3):901.
[43] Tamaru Y, Miyake H, Kuroda K, et al. Comparative genomics of the mesophilic cellulosome-producingClostridium cellulovoransand its application to biofuel production via consolidated bioprocessing[J].Environmental Technology, 2010, 31(8-9):889-903.
[44] Meguro H, Morisaka H, Kuroda K, et al. Putative role of cellulosomal protease inhibitors inClostridium cellulovoransby their gene expression and measurement of the activities[J].Journal of Bacteriology, 2011, 193(19):5527-5530.

