1,3-丙二醇氧化还原酶及其工程菌研究进展
王飞,徐浪,杨泽茜,赵博凯,邓文颖,林静,王旭,齐向辉
(江苏大学食品与生物工程学院,江苏镇江212013)
1,3-丙二醇氧化还原酶及其工程菌研究进展
王飞,徐浪,杨泽茜,赵博凯,邓文颖,林静,王旭,齐向辉
(江苏大学食品与生物工程学院,江苏镇江212013)
作为合成许多缩聚物单体的1,3-丙二醇(1,3-propanediol,1,3-PD)是本世纪具有广阔市场潜力的化工原料,在化工、医药、食品等领域具有广泛的应用.介绍了1,3-丙二醇氧化还原酶(1,3-propanedioldehydrogenase, PDOR,EC 1.1.1.202)在1,3-PD生产菌代谢途径中的作用,着重综述了PDOR的基因克隆表达情况及其性质特点,分析了PDOR的晶体结构,同时对1,3-PD生物法生产中工程菌的研究进展进行了详细的介绍,并展望了生物法生产1,3-PD的前景,该文有助于加深对PDOR及其工程菌的了解,使其得到更多研究者的重视.
1,3-丙二醇;1,3-丙二醇氧化还原酶;克隆表达;晶体结构;工程菌
化学法是生产1,3-丙二醇(1,3-propanediol,1,3-PD)的主要方法,但其具有污染环境、生产成本高、产品分离困难等缺点,生物法以其清洁生产、利用可再生资源、有利于可持续发展等优势,逐渐成为国内外的研究热点,日后也必将取代化学法来生产1,3-PD[1].生物法生产1,3-PD需要酶的参与,而1,3-丙二醇氧化还原酶(1,3-propanedioldehydrogenase,PDOR,EC 1.1.1.202)是1,3-PD生产的关键酶和限速酶,对1,3-PD的生成起着至关重要的作用.基于此,本文介绍了PDOR的基因克隆表达情况、酶的性质特点和晶体结构,同时详细介绍了工程菌的相关研究情况.
1 PDOR在1,3-PD生产菌代谢途径中的作用
1,3-PD生产菌生物转化的天然底物是甘油,不能利用糖类等廉价的碳源来直接生产1,3-PD.研究至今所发现的1,3-PD生产菌主要包括肠道菌属的克雷伯氏菌(Klebsiella pneumoniae)、弗氏柠檬酸菌(Citrobacter freundii)、产气肠杆菌(Enterobacter aerogenes)和絮凝肠细杆菌(Enterobacter agglomerans);乳杆菌属的布氏乳杆菌(Lactobacilli buchneri)和短乳杆菌(Lactobacilli brevis);梭菌属的丁酸梭菌(Clostridium butyricum)和产气荚膜梭菌(Clostridium perferingens)等[2],其中K.pneumoniae、C.freundii和C.butyricum具有较高的1,3-PD产率,因而得到较多的关注.
在微生物体内甘油代谢生产1,3-PD主要涉及两步酶反应[3]:①甘油脱水酶(glyceroldehydratase, GDHt)转化甘油为中间产物3-羟基丙醛(3-hydroxypropionaldehyde,3-HPA);②PDOR在NADH的作用下催化3-HPA生成终产物1,3-PD(见图1).有研究显示,在1,3-PD工程菌的发酵过程中,3-HPA往往会大量积累,同时还会生成多种酸性物质,使PDOR的催化活力受到严重影响,而PDOR活力的下降又会使3-HPA进一步积累,形成恶性循环,并使发酵菌株的生长产生不可逆停止;另外,由于发酵液中的1,3-PD浓度积累会反馈抑制PDOR的催化活力,影响终产物1,3-PD的积累浓度,最终严重影响1,3-PD的产量[4].因此作为1,3-PD生产的关键酶和限速酶的PDOR,对1,3-PD的生成起着至关重要的作用.
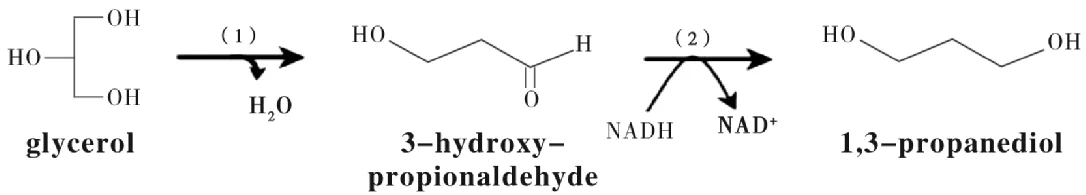
图1 甘油代谢生产1,3-PD原理Fig.1 Themetabolic pathway ofglycerol to 1,3-PD
2 PDOR基因的克隆表达
PDOR的编码基因属于1,3-PD生产菌的被称为dha的调节子系统(见图2),在C.freundii、C.butyricum等菌中由dhaT基因编码.1995年,Daniel等首次克隆表达了C.freundii的PDOR基因dhaT,基因长度为1 164 bp,编码387个氨基酸残基,亚基相对分子质量为41.324 kD;dhaT基因通过T7 RNA启动子在大肠杆菌中的高效表达的活力是野生菌的274倍;该酶与第三类醇脱氢酶有很高的同源性.Luers等建立了巴斯德梭菌(Clostridium pasteurianum)菌株的基因组文库,通过使用C.freundii中的dhaT基因作为异源探针进行菌落杂交,从而成功地克隆了C.pasteurianum的PDOR基因dhaT,基因长度为1 158 bp,编码385个氨基酸残基,亚基相对分子质量为41.776 kD;该基因与来源于C.freundii中的dhaT基因有89.8%的同源性,也属于醇脱氢酶家族中的第三类成员.随着研究的进一步开展, Nakamura等发现了来自于大肠杆菌的由yqhD基因编码的非特异性氧化还原酶是PDOR的同功酶,该酶催化3-HAP的效率较PDOR有明显的提高,Seo等[5]研究发现用yqhD构建的重组E.coli在分批发酵过程中具有较高的甘油转化率;另外yqhD基因也被Dupont公司利用来构建相关的高性能基因工程菌.
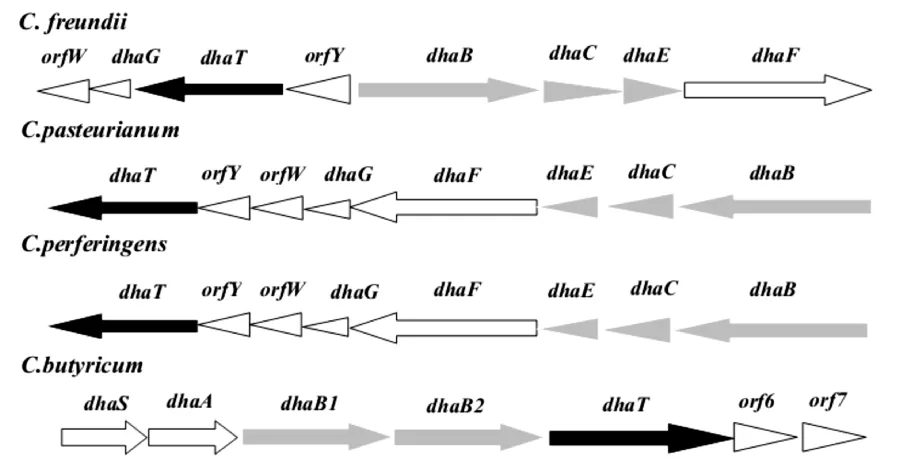
图2 dhaT在dha调节子中位置Fig.2 Geographic schematic diagram of dhaT in dha regulon
3 PDOR的性质特点
1987年,Jonhnson等首次从K.pneumoniae厌氧发酵甘油生产1,3-PD中纯化得到PDOR,推断该酶为同型八聚体或同型六聚体,亚基相对分子质量为45±3 kD;此酶以1,3-PD和NAD+为底物的Km值分别为18mmol/L和0.13mmol/L;其活性可被二价阳离子的螯合物强烈抑制,因形成α-α’联吡啶配体而使酶失活,而通过凝胶过滤,添加Fe2+、Mn2+可使其恢复活性.Talarico等从罗伊氏乳杆菌(Lactobacillus reuteri)中纯化得到PDOR;推断该酶为同型四聚体,亚基相对分子质量为41~46 kD;此酶以NAD+为底物的Km值为12mmol/L;酶的最适温度为37℃,最适pH值为7,比活力为7.3U/mg;K+是该酶的激活因子.随后研究者从L.brevis、L.buchneri等菌中分别纯化得到了PDOR,不同微生物体内的PDOR的性质如表1.

表1 不同微生物来源的1,3-丙二醇氧化还原酶的性质Tab.1 The propertiesofdifferentmicrobialsource1,3-propanedioldehydrogenase
4 PDOR的晶体结构
研究者于2000年开始研究PDOR的晶体结构,Rondon等首次解析了海栖热袍菌(Thermotoga maritime)的PDOR晶体结构.随后Schwarzenbacher等[6]利用多波长不规则反射理论在解析度为1.3×10-10m的条件下进一步确定了T.maritima0902(TM0902)的PDOR晶体结构,TM0902单体由359个多肽残基组成,由13个α螺旋、6个310螺旋和8个β折叠构成,三种结构所占的比例依次为48.7%、7.2%、12.3%,每个单体具有2个明显的结构域,Fe2+结合在这两个结构域形成的裂缝中间,因此该酶属于铁依赖型PDOR.另有研究显示TM0902的PDOR蛋白质序列与K.pneumoniae(PDBNo.:Q59477)和C.freundii(PDBNo.:P44513)分别有32%和47%的相似性.同年Sulzenbacher等[7]首次解析了由yqhD编码的PDOR同功酶的晶体结构,该酶是由2个单体构成的二聚体,二聚体间的结构与T.maritima的PDOR相似;其中每个单体由387个多肽残基组成,含有Rossmann折叠和α螺旋构成的2个结构域,结构域含有NADP辅酶的结合位点.2007年Marcal等结晶了K.pneumoniae的PDOR,并于2009年解析了该晶体结构(PDB code:3BFJ,2.7×10-10m),单体由13个α螺旋和8个β折叠构成,含有两个结构域,该结构与第三类Fe-NAD的乙醇脱氧酶类似.Jeyakanthan等[8]研究发现嗜热菌(Aquifex aeolicus)VF5的PDOR晶体结构与T.maritime和K.pneumoniae的蛋白质序列有34%~35%的相似性.上述研究为进一步了解该酶的分子催化机制奠定了坚实的基础.
5 工程菌的研究进展
在能源危机的当今社会,采用价格低廉的原材料生产1,3-PD是生物法的发展趋势.因为1,3-PD生物转化的天然底物是甘油,所以可以以非粮食物质如木薯、淀粉等为原料快捷简便地通过葡萄糖→甘油的途径来获得甘油,如美国杜邦公司开展了以可发酵的碳源为底物,用基因工程菌通过“一步法”(葡萄糖→甘油→1,3-PD)生产1,3-PD的研究,1,3-PD的最高产量为135g/L[9-10];另外近年来随着生物柴油产量的增加(预计到2020年,年产生物柴油将达到900万t),副产物甘油也迅速增加,因为在生物柴油的生产过程中会生成10%的副产物甘油,这导致甘油的价格迅速下滑,因此以粗甘油为底物来生产1,3-PD也是发展的趋势.1,3-PD生物法生产中面临着产量、产率和发酵率低的问题,但是这些问题有望通过代谢工程和基因工程来进行改善和解决.
首先,可以通过去除副产物或者氧化途径的方法来改善代谢途径以提高1,3-PD的产量和产率, K.pneumoniae HR526代谢过程中产生大量的乳酸盐、乙醇、丁二酸等,可以去除这些副产物来降低其对NADH的消耗;Xu等[11]通过敲除编码乳酸脱氢酶的基因ldhA使1,3-PD的产量和产率分别达到102 g/L和2.13g/(L·h).在去除氧化途径的研究中,Horng等[12]敲除了编码甘油脱氢酶的基因dhaD和编码二羟基丙酮激酶的基因dhaK,消除氧化途径,增强还原途径,使TC100 1,3-PD的产量比野生型K.pneumoniae提高了2.06倍,TC102GDHt和PDOR的活力比TC101分别增加了6.09和6.46倍.其次,在发酵过程中1,3-PD和3-HPA的积累均会影响1,3-PD的产量,而Otte等[13]利用DNA shuffling技术获得耐酸的突变株,其中两个突变株有较高的甘油转化率,1,3-PD的产量分别为78.5 g/L和85 g/L,比野生菌高1倍.另外,通过提高还原途径关键酶的活性来提高1,3-PD的产量,也可以用yqhD编码的PDOR的同功酶代替PDOR[5].同时,改变PDOR辅酶的选择性,也可以提高1,3-PD的产量,Ma等[14]应用理性蛋白设计来改善PDOR同功酶的特性使其可以利用NADH和NADPH两种辅酶因子来生产1,3-PD,这样由磷酸戊糖途径(PPP)和三羧酸(TCA)循环产生的NADPH就得到了较好的应用.K.pneumoniae重组菌中NAD+依赖型乙醛脱氢酶的过量表达可以促进氧化还原的平衡,该菌在分批发酵过程中,不仅产生大量的1,3-PD,而且不产生除醋酸盐之外的其他副产物[15].底物对1,3-PD的产量也有一定的影响,通常情况下,粗甘油会降低1,3-PD的产量,而Hiremath等研究发现C.butyricum(VPI1718)和K.pneumoniae在利用来源于麻风树生物柴油制备过程中的粗甘油生产1,3-PD时,不仅不会影响1,3-PD的产量,反而比其利用纯甘油来生产1,3-PD时更有效[16].
研究人员在了解PDOR特性的基础上构建由甘油到1,3-PD的基因工程菌以提高1,3-PD的产量[17].有研究表明,增加1,3-PD生产菌中PDOR的基因拷贝数,可以增加酶的活力,有助于降低3-HPA积累并增加1,3-PD的摩尔产量.Zhang等[18]在扩增yqhD基因的同时又扩增了C.freundii的GDHt基因dhaB,并将其克隆到温度诱导型pHsh质粒中,构建了重组的E.coli pHsh-yqhD-dhaB.在最佳条件下进行发酵,甘油、酵母提取物和CoB12的加入量分别为61.8 g/L、6.2 g/L和49mg/L,发酵30 h后使1,3-PD的产量达最高值43.86 g/L.Zhao等[19]构建了重组的K.pneumoniae工程菌,使PDOR基因和GHDt基因得以共表达,研究显示,3-HPA的浓度降低了73.6%,虽然发酵液中的1,3-PD浓度没有非常显著的变化,但1,3-PD的摩尔产量却由50.6%上升至64.0%,同时发酵液中的琥珀酸、乙醇和乳酸等副产物分别下降了47.4%、50.6%和51.8%,这样有利于1,3-PD后期的分离纯化.诸葛斌等为优化K.pneumoniae1,3-PD的合成途径,利用PCR技术扩增出yqhD基因,并从K.pneumoniae中扩增出2.66 kb的甘油脱水酶基因dhaB,构建了产1,3-PD关键酶基因的串联载体pEtac-dhaB-tac-yqhD,并将其转入到K.pneumoniae中,重组载体得到了表达.通过初步发酵,重组后K.pneumoniae1,3-PD的产量不仅比原始菌提高20%左右,而且副产物乙酸和丁二醇产量还分别下降30%左右.Vaidyanathan等[20]通过实现yqhD基因在L.reuteri中的表达,使PDOR及其同功酶可以协同利用辅酶NADPH和NADH,增加了PDOR的整体催化活力,从而使3-HPA降低了25%,1,3-PD生产率增加了34%,摩尔产量增加了13%.在提高1,3-PD工业化生产的研究中,Tang等[21]的研究值得一提,研究人员扩增出yqhD基因同时又扩增了C.butyricum的CoB12不依赖型GDHt的编码基因dhaB1、dhaB2,并将其克隆到温度敏感型pBV220质粒中,构建了重组E.coli.通过两阶段高密度发酵, 1,3-PD的最高产量可达104.4 g/L.这些研究均为1,3-PD的工业化生产提供了有研究价值的基因工程菌.
6 展望
由1,3-PD和对苯二甲酸(PTA)缩聚而成的聚对苯二甲酸丙二醇酯(PTT)纤维是Shell公司开发的一种性能优异的聚酯类新型纤维,其集各种优良适用性能于一体,成为当前国际上开发的热门高分子材料,在地毯纺织、装饰材料和工程塑料等领域具有广泛的应用.据预测,到2020年全球包括非纤维应用在内的PTT需求将超过150万t/a.随着石油等非再生资源日益减少,环境压力的增加,应用生物法生产1,3-PD成为发展的必然趋势.因此,研究关键酶的相关特性,据此构建高质量的工程菌,并运用分子生物学、途径工程等现代生物技术提高1,3-PD的发酵水平,采用价格低廉的碳源或粗甘油作为代谢底物,可为工业化生产提供有力的支撑.
参考文献:
[1]KaurG,Srivastava A K,Chand S.Advances in biotechnologicalproduction of1,3-propanediol[J].BiochemicalEngineering Journal, 2012,64:106-118.
[2]Ringel A K,Wilkens E,Hortig D,et al.An improved screening method for microorganisms able to convert crude glycerol to 1,3-propanedioland to toleratehigh productconcentrations[J].Applied Microbiologyand Biotechnology,2012,93(3):1049-1056.
[3]Dobson R,Gray V,Rumbold K.Microbial utilization of crude glycerol for the production of value-added products[J].Journal of IndustrialMicrobiology&Biotechnology,2012,39(2):217-226.
[4]Dabrowski S,Pietrewicz K D,Zablotna E,et al.1,3-propanediol production by Escherichia coli expressing genes of dha operon from Clostridium butyricum 2CR371[J].Acta Biochimica Polonica,2012,59(3):357-361.
[5]Seo JW,Seo M Y,Oh B R,et al.Identification and utilization of a 1,3-propanediol oxidoreductase isoenzyme for production of 1,3-propanediol from glycerol in Klebsiella pneumoniae[J].Applied Microbiology and Biotechnology,2010,85(3):659-666.
[6]Schwarzenbacher R,von Delft F,Canaves JM,et al.Crystal structure of an iron-containing 1,3-propanediol dehydrogenase(TM0920)from Thermotoga maritima at 1.3resolution[J].Proteins:Proteins:Structure,Function,and Bioinformatics,2004,54(1):174-177.
[7]Sulzenbacher G,Alvarez K,van den Heuvel R H,et al.Crystal structure of E.coli alcohol dehydrogenase YqhD,evidence of a covalentlymodified NADPcoenzyme[J].JournalofMolecular Biology,2004,342(2):489-502.
[8]Jeyakanthan J,Thamotharan S,Panjikar S,et al.Expression,purification and X-ray analysis of 1,3-propanediol dehydrogenase(Aq_1145)from Aquifex aeolicus VF5[J].Acta Crystallogr SectFStructBiolCrystCommun,2010,66:184-186.
[9]Nakamura C E,Whited G M.Metabolic engineering for the microbial production of 1,3-propanediol[J].Current Opinion Biotechnology,2003,14(5):454-459.
[10]Mendes FS,González-PajueloM,Cordier H,et al.1,3-Propanediolproduction in a two-step process fermentation from renewable feedstock[J].Applied Microbiology and Biotechnology,2011,92(3):519-527.
[11]Xu Y Z,Guo N N,Zheng ZM,et al.Metabolism in 1,3-propanediol fed batch fermentation by a D-lactate deficientmutant of Klebsiella pneumoniae[J].Biotechnology and Bioengineering,2009,104(5):965-972.
[12]Horng Y T,Chang K C,Chou T C,et al.Inactivation of dhaD and dhaK abolishes by-product accumulation during 1,3-propanediol production in Klebsiella pneumoniae[J].Journal of Industrial Microbiology&Biotechnology,2010,37(7):707-716.
[13]Otte B,Grunwaldt E,Mahmoud O,et al.Genome shuffling in Clostridium diolis DSM 15410 for improved 1,3-propanediol production[J].Applied and EnvironmentalMicrobiology,2009,75(24):7610-7616.
[14]Ma C,Zhang L,Dai J,et al.Relaxing the coenzyme specificity of1,3-propanedioloxidoreductase from Klebsiella pneumoniae by rationaldesign[J].JournalofBiotechnology,2010,146(4):173-178.
[15]Luo LH,Seo JW,Baek JO,et al.Identification and characterization of the propanediolutilization protein PduP of Lactobacillus reuteri for3-hydroxypropionic acid production from glycerol[J].Applied Microbiology Biotechnology,2011,89(3):697-703.
[16]Hiremath A,Kannabiran M,Rangaswamy V.1,3-Propanediol production from crude glycerol from jatropha biodiesel process[J]. New Biotechnology,2011,28(1):19-23.
[17]Leja K,Czaczyk K,Myszka K.The use ofmicroorganisms in 1,3-Propanediol production[J].African Journal of Microbiology Research,2011,5(26):4652-4658.
[18]Zhang X,LiY,Zhuge B,et al.Construction ofa novel recombinant Escherichia coli strain capable of producing 1,3-propanediol and optimization of fermentation parameters by statistical design[J].World Journal ofMicrobiology and Biotechnology,2006,22(9):945-952.
[19]Zhao L,Zheng Y,Ma X,et al.Effects of over-expression of glycerol dehydrogenase and 1,3-propanediol oxidoreductase on bioconversion of glycerol into 1,3-propandediol by Klebsiella pneumoniae under micro-aerobic conditions[J].Bioprocess and Biosystems Engineering,2009,32(3):313-320.
[20]Vaidyanathan H,Kandasamy V,Ramakrishnan GG,et al.Glycerol conversion to 1,3-Propanediol is enhanced by the expression ofa heterologousalcoholdehydrogenasegene in Lactobacillus reuteri[J].AMBExpress,2011,1:1-8.
[21]Tang X,Tan Y,Zhu H,et al.Microbial conversion of glycerol to 1,3-propanediol by an engineered strain of Escherichia coli[J]. Applied and EnvironmentalMicrobiology,2009,75(6):1628-1634.
(责任编辑:邓天福)
Advances on the 1,3-propanediol dehydrogenase and its engineering bacteria
Wang Fei,Xu Lang,Yang Zexi,Zhao Bokai,DengWenying,Lin Jing,Wang Xu,Qi Xianghui
(Schoolof Food and Biological Engineering,Jiangsu University,Zhenjiang212013,China)
As amonomer for polycondensations to produce polyesters,polyethers and polyurethanes,1,3-propanediol is an essential chemicalmaterial with a prosperousmarket potential in this century and has been widely used in the fields of chemical industry,medicine,food and so on.The role of 1,3-propanediol dehydrogenase in the metabolic pathway of 1,3-PD production strains was introduced in this paper.Then,molecular cloning,expression,properties and crystal structure of PDOR were emphatically reviewed,and the engineering bacteria of 1,3-PD was introduced in detail.At last,the microbial production of 1,3-PD was prospected.This study is helpful to understand PDOR and engineering bacteria so that it canmakemore researchers'attention to its application.
1,3-propanediol;1,3-propanediol dehydrogenase;cloning and expression;crystal structure;engineering bacteria
Q814
A
1008-7516(2013)03-0019-05
10.3969/j.issn.1008-7516.2013.03.004
2013-05-13
国家自然科学基金项目(21006041);江苏大学科研立项及百项本科创新项目(11A344,Y11A169,2012JSSPI TP1238,08JDG009)
王飞(1988-),女,河南新乡人,硕士研究生.主要从事微生物基因工程及代谢调控研究.
齐向辉(1976-),男,河北石家庄人,博士,副教授.主要从事微生物基因工程、代谢工程及调控研究.

