基质辅助激光解吸电离-串联飞行时间质谱鉴定鲨鱼硒结合蛋白
王 勇,李水明,何曼文
(1.深圳大学生命科学学院,深圳市海洋生物资源与生态环境重点实验室,广东 深圳 518060;2.深圳大学生命科学学院,深圳市微生物基因工程重点实验室,广东 深圳 518060)
质谱技术目前已成为研究蛋白质和其它生物分子的核心分析技术[1-5]。蛋白质组学研究中的一个关键问题是实验的可重复性,因此,前人在利用鸟枪法质谱平台进行肽段检测时,针对肽段鉴定的随机性和可预测性方面已开展较多工作[6]。值得注意的是,即使对于纯化的蛋白标准品或表达蛋白,仍很难有100%的鉴定覆盖率[7],这一现象除了与样品用量、实验操作和仪器灵敏度等有关外,还与数据解析相关[8]。目前应用最多的是针对串联质谱数据的数据库搜索算法,尽管这种方法的通量和可靠性可以满足生物学研究需要,并且在不断提高,但如果对质谱数据进行仔细分析和进一步实验验证,则有可能提高覆盖率,获得更多的结构信息。
硒 结 合 蛋 白 (selenium-binding protein,SBP)是一种特殊的含硒蛋白,因能结合硒而命名[9-13]。目前,硒结合蛋白的研究大多集中在模式生物,如人、鼠和拟南芥等[14],而对非模式生物研究较少。本工作前期克隆测序了白鳍鲨SBP基因,现利用基质辅助激光解吸电离-串联飞行时间质谱(MALDI-TOF/TOF)鉴定原核表达的鲨鱼硒结合蛋白,确定表达是否成功,同时讨论影响蛋白鉴定覆盖率的实验因素。
1 实验部分
1.1 仪器与试剂
4800MALDI TOF/TOF Analyzer基质辅助激光解吸电离-串联飞行时间质谱仪:美国AB Sciex公司产品;Milli-Q超纯水处理系统:美国Millipore公司产品;胰蛋白酶(质谱分析级):Promega公司产品;α-腈基-4-羟基肉桂酸:美国Sigma公司产品,使用前重结晶。
1.2 样品的还原、烷基化和酶解
经一维聚丙烯酰胺凝胶电泳分离后的目标条带脱色干燥后,用二巯基乙醇和碘乙酰胺烷基化30min,加入5μL 20mg/L质谱用测序级胰蛋白酶,4 ℃放置30min,再加入5μL 40 mmol/L NH4HCO3-10% 乙腈,37℃水浴8h。
1.3 质谱和数据分析条件
基质制备:将6g/Lα-腈基-4-羟基肉桂酸和2g/L柠檬酸氢二铵溶于10mL 50%乙腈(含0.1%三氟乙酸)中,基质溶液与蛋白酶解液按1∶1混合后,点于不锈钢靶板。采用正离子反射模式,一级质谱每张谱图累加800次,二级质谱累加1 200次,碰撞诱导解离条件为1kV碰撞能加空气碰撞。
数据库搜索条件:串联质谱数据搜库,信噪比设为5,子离子和母离子质量误差均设为0.3 u,Mascot软件分析,搜索数据库NCBI于2012年8月发布,甲硫氨酸氧化(oxidation)和半胱氨酸烷基化(carbamidomethyl)作为可变修饰,胰酶漏切位点数1。
2 结果与讨论
2.1 数据库检索结果
白鳍鲨硒结合蛋白的编码区基因序列提交到NCBI数据库(gi|350626507),Mascot得分为1 121分(肽段离子得分大于53为结果可信),说明原核表达成功,鉴定结果可证明确为该蛋白,其搜库结果示于图1。与其结构最为类似的是斑马鱼SBP,但只匹配了3个肽段,得分为237分。值得注意的是,m/z2 335.06离子的串联质谱也可以匹配为肽段FLHNPAATE(Methyl)GMVGCALGSSVFR(96分),但因得分相对较低,并经人工比对确认为半胱氨酸的丙酰胺化(141分)。在蛋白质组学的研究中,一般认为存在3个高置信度肽段即可认为鉴定了一个蛋白,但对于一个分子质量为52ku的原核表达蛋白鉴定而言,串联质谱只以35%的覆盖率鉴定12个肽段,详情列于表1。因此,本工作进一步探究了影响鉴定覆盖率的原因。

图1 白鳍鲨硒结合蛋白的Mascot搜库结果Fig.1 The database searching result of selenium-binding protein of whitetip reef shark

表1 白鳍鲨硒结合蛋白1的搜库鉴定结果Table 1 Peptides of selenium-binding protein 1identified by database searching
2.2 提高SBP鉴定覆盖率的研究
首先,分析了SBP胰酶水解液的一级谱图,发现除了m/z2 211.093离子外,其余的高丰度离子均被选择进行串联质谱分析并被匹配,说明蛋白分离较纯,其它杂质蛋白不干扰分析。由于在胰蛋白酶自切峰表中包含m/z2 211.105离子,所以常规条件下,m/z2 211.093因分子质量相近而未被选择进行串联质谱分析,但由于未观察到其它胰酶自身酶解肽段,并且经MS-digest软件分析发现,SBP也可产生质荷比为2 211.119的肽段,因此对该离子进行串联质谱分析,其质谱图示于图2。由图2发现,该肽段不是胰酶自切峰,而确为鲨鱼SBP的肽段NFLVDFGKEPDGPALAHEVR,该肽段中赖氨酸残基因与谷氨酸相邻而漏切[15]。由于只有在目标蛋白浓度较低时才可以检测到明显的胰酶自切峰,该结果提示在蛋白浓度较高时可不排除它们,以保证测定的完全。

图2 m/z 2 211.093离子的串联飞行时间质谱图Fig.2 MALDI-TOF/TOF spectrum of m/z 2 211.093
其次,目前蛋白质组学研究中多采用数据依赖型采集模式,即根据前体离子在一级谱中的丰度高低,决定是否选择其进行下一级分析[16],因此,一级谱中的很多低丰度离子未被选择,它们可能是噪音或其它蛋白质的污染,也可能是目标肽段,这可对所有可能的目标肽段离子进行串联质谱分析。例如,m/z833.39离子在一级谱中的相对强度只有1.35%,串联质谱的绝对强度小于500,但串联质谱几乎连续的y离子和丰富的a、b系列离子证明其为肽段GGDWAAEK,数据质量很好,可以用于从头测序,其质谱图示于图3,其中m/z159.08为色氨酸的亚胺离子。
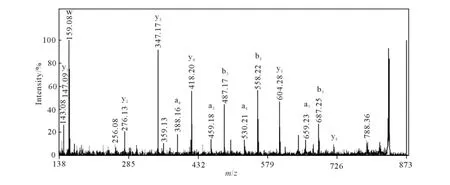
图3 m/z 833.40离子的串联飞行时间质谱图Fig.3 MALDI-TOF/TOF spectrum of m/z 833.40
由于串联飞行时间质谱的离子选择能力至少为3u,因此即使是对同一蛋白也可能产生混合谱[17]。m/z1 440.69离子的串联飞行时间质谱图示于图4,在图4中观察到了完整的y序列离子和部分b离子,搜库鉴定为肽段EEIVYVPC(Carbamidomethyl)IYR(理 论m/z1 440.719 2),但仔细比对发现还存在另一肽段MVGQVFIGGC(Carbamidomethyl)FVK(理论m/z1 441.733 0),主 要 观 察 到 了 该 肽 段 的y6*~y9*离子和甲硫氨酸及苯丙氨酸的特征亚胺离子,提示图4为这两个肽段组成的混合谱。最后,也有低丰度前体离子质量与酶解肽段的理论值接近但串联质谱不符的现象,说明单一利用肽指纹谱鉴定蛋白有可能产生肽段鉴定的假阳性。将串联质谱数据归纳整理后,发现鉴定肽段数明显增多,共检测出5个新肽段,162个氨基酸残基,鉴定覆盖率从原来的35%增加至51%,示于图5。这些新确定的肽段多以赖氨酸残基结尾(图5中划线部分),这与梁宋平等[18]报道的在MALDI电离条件下赖氨酸结尾肽段灵敏度低于精氨酸结尾肽段的结论一致。

图4 m/z 1 440.69离子的串联飞行时间质谱图Fig.4 MALDI-TOF/TOF spectrum of m/z 1 440.69

图5 白鳍鲨硒结合蛋白序列及所测肽段(加粗表示搜库结果)Fig.5 Protein sequence of selenium-binding protein of whitetip reef shark(Bold font means the database search result)
3 结论
本研究鉴定了原核表达的鲨鱼硒结合蛋白,对未匹配数据的分析表明:对一级质谱中的弱信号进行串联质谱分析亦可获得高置信度的序列信息,同时发现该目标蛋白含有与胰酶自切峰分子质量相近的肽段。通过人工解析提高了肽段鉴定的可信度和蛋白鉴定覆盖率,但覆盖率的进一步提高则需进行液相色谱分离。该结论对目前基于双向电泳分离的蛋白质鉴定和翻译后修饰研究具有参考作用。此外,在蛋白质的质谱鉴定中,由于实验操作或蛋白质结构的特异性而引起的胰蛋白酶漏切或不常见翻译后修饰也是影响质谱数据利用率的因素[19-20]。
[1] DOMON B,AEBERSOLD R.Mass spectrometry and protein analysis [J].Science,2006,312(5 771):212-217.
[2] 马海滨,张纪阳,刘 辉,等.蛋白质组学中质谱数据标准研究进展 [J].质谱学报,2011,32(3):129-137.MA Haibin,ZHANG Jiyang,LIU Hui,et al.Mass spectrometry data format standards in proteomics[J].Journal of Chinese Mass Spectrometry Society,2011,32(3):129-137(in Chinese).
[3] LI Q W,LU X Y,YOU Y,et al.Comparative proteomic analysis suggests that mitochondria are involved in autosomal recessive polycystic kidney disease[J]. Proteomics,2012,12 (15/16):2 556-2 570.
[4] KECK J M,JONES M H,WONG C C L,et al.A cell cycle phosphoproteome of the yeast centrosome[J].Science,2011,332(6 037):1 557-1 661.
[5] JUNG H R,PASINI D,HELIN K,et al.Quantitative mass spectrometry of histones H3.2and H3.3in Suz12-deficient mouse embryonic stem cells reveals distinct,dynamic post-translational modifications at Lys-27and Lys-36[J].Mol Cell Proteomics,2010,9(5):838-850.
[6] 徐长明,张纪阳,刘 辉,等.蛋白质组学质谱平台肽段可检测性预测研究进展 [J].分析化学,2010,38(2):286-292.XU Changming,ZHANG Jiyang,LIU Hui,et al.Advance of peptide detectability prediction mass spectrometry platform in proteomics[J].Chin J Anal Chem,2010,38(2),286-292(in Chinese).
[7] 柏兆方,林碧蓉,李 萍,等.超高效纳升液相色谱-电喷雾串联质谱鉴定重组人Ⅱ型肿瘤坏死因子受体-抗体融合蛋白[J].分析化学,2009,37(7):1 025-1 028 .BO Zhaofang,LIN Birong,LI Ping,et al.Characterization of recombinanthuman necrosis factor-α receptor IgG Fc fusion protein by nano ultra-high performance liquid chromatography-electrospray ionization mass spectrometry tandem mass spectrometry[J].Chin J Anal Chem,2009,37(7):1 025-1 028(in Chinese).
[8] BELL A W,DEUTSCHE W,AU C E,et al.A HUPO test sample study reveals common problems in mass spectrometry-based proteomics [J].Nature Methods,2009,6(6):423-430.
[9] HUANG K C,PARK D C,NG S K,et al.Selenium binding protein 1in ovarian cancer[J].Int J Cancer,2006,118(10):2 433-2 440.
[10] BROWN L M,HELMKE S M,HUNSUCKER S W,et al.Quantitative and qualitative differences in protein expression between papillary thyroid carcinoma and normal thyroid tissue [J].Mol Carcinog,2006,45(8):316-326.
[11] HE Q Y,CHEUNG Y H,LEUNG S Y,et al.Diverse proteomic alterations in gastric adenocarcinoma[J].Proteomics,2004,4(10):3 276-3 287.
[12] GUOAN C,HONG W,CHARLES T M,et al.Reduced selenium-binding protein 1expression is associated with poor outcome in lung adenocarcinomas[J].The Journal of Pathology,2004,202(3):321-329.
[13] JOSE R T,MATTHEW C,SUSAN G,et al.Selenium-binding protein-1in smooth muscle cells is downregulated in a rhesus monkey model of chronic allograft nephropathy[J].American Journal of Transplantation,2005,5(1):58-67.
[14] DUTILLEUL C,JOURDAIN A,BOURGUIGNON J,et al.The arabidopsis putative seleniumbinding protein family:Expression study and characterization of SBP1as a potential new player in cadmium detoxification processes [J].Plant Physiology,2008,147(1):239-251.
[15] THIEDE B,LAMER S,MATTOW J,et al.A-nalysis of missed cleavage sites,tryptophan oxidation andN-terminal pyroglutamylation after ingel tryptic digestion[J].Rapid Commun Mass Spectrom,2000,14(6):496-502.
[16] BERN M,FINNEY G,HOOPMANN M R,et al.Deconvolution of mixture spectra from ion-trap data-independent-acquisition tandem mass spectrometry[J].Anal Chem,2010,82(3):833-841.
[17] VESTAL M L,CAMPBELL J M.Tandem timeof-flight mass spectrometry[J].Methods in En-zymology,2005,402:79-107.
[18] CHEN P,NIE S,MI W,et al.De novo sequencing of tryptic peptides sulfonated by 4-sulfophenyl isothiocyanate for unambiguous protein identification using post-source decay matrix-assisted laser desorption/ionization mass spectrometry[J].Rapid Commun Mass Spectrom,2004,18(2):191-198.
[19] 王 勇,李水明,何曼文,等.蛋白质胰蛋白酶水解过程双位点漏切肽段的质谱鉴定 [J].质谱学报,2013,34(1):29-34 .WANG Yong,LI Shuiming,HE Manwen,et al.Identification of double missed cleavage sites tryptic peptides by mass spectrometry[J].Journal of Chinese Mass Spectrometry Society,2013,34(1):29-34(in Chinese).
[20] 王 勇,李水明,何曼文.溶菌酶基质辅助激光解吸电离-串联飞行时间质谱分析中的不常见修饰及脱水反应 [J].分析 化 学,2013,41 (4):494-499.WANG Yong,LI Shuiming,HE Manwen.Uncommon modifications and dehydration reaction in analysis of Lysozyme by matrix-assisted laser desorption ionization tandem time of-flight mass spectrometry[J].Chin J Anal Chem,2013,41(4):494-499(in Chinese).

