基于微卫星DNA标记的恒河猴遗传多样性研究
禹文海,和占龙,鲁绍雄,鲁帅尧,赵 远,杨凤梅,李艳艳,陈丽雄,王俊斌,沈 冬
(1.中国医学科学院北京协和医学院 医学生物学研究所,昆明 650118;2.云南农业大学动物科学技术学院,昆明 650201)
微卫星DNA(microsatellite DNA),也称为简单序列重复(simple sequence repeat,SSR)、短串联重复序列(short tandem repeat,STR),在生物基因组中一般以1~6个碱基为其核心序列、首尾相连组成串联核苷酸重复序列。由于核心序列重复数目的不同,产生了DNA多态性。微卫星DNA标记与其它分子标记相比,具有数量多、分布广、多态性丰富、共显性遗传、选择中性、易检测、重复性好、可提供高分辨率的遗传信息等特点,是一种理想的分子标记,已广泛应用于群体遗传结构研究和遗传多样性分析[1-5]。恒 河 猴 (Macaca mulatta),为 灵 长 目(Primates)猴科(Cercopithecidae)猕猴属的一个种,是非人灵长类中分布范围最广、数量最多的一个类群。国内外通过人工驯养繁殖,已建立了许多人工饲养繁殖种群,被广泛地应用到医学和生物学等研究领域。中国医学科学院医学生物学研究所全国医学灵长类研究中心开展人工饲养、驯化繁殖恒河猴已有50多年的历史,本研究从该中心人工饲养繁殖的2000余只恒河猴群体中随机抽取60只,利用19个微卫星DNA标记对该恒河猴群体进行遗传多样性检测,以期获得现有实验恒河猴种群的遗传概貌信息及建立一种对恒河猴群体有效的遗传监测方法,并对现有繁育方式作出评价,从分子遗传角度指导该恒河猴群体今后开展合理、科学的遗传繁育。
1 材料与方法
1.1 实验动物和基因组DNA提取
随机抽取人工饲养繁殖的普通级恒河猴共60只,雌雄各半,来源于中国医学科学院医学生物学研究所全国医学灵长类研究中心【实验动物生产许可证:(滇)SCXK2010—0006】。股静脉抽取全血2 mL,EDTA抗凝。采用 AXYGEN DNA提取试剂盒提取基因组DNA,详细操作步骤见说明书。
1.2 主要试剂和仪器
PCR 仪(BIO-RAD 公司,美国),垂直电泳槽、电泳仪和凝胶成像系统(北京六一仪器厂,中国);Sony数码相机(Sony公司,日本)。DNA提取试剂盒(爱思进生物技术有限公司,中国),2×PCR Master Mix、DL2000及聚丙烯酰胺凝胶电泳相关试剂(天根生化科技有限公司,中国)。
1.3 引物设计及PCR扩增
自 NCBI网站(http://www.ncbi.nlm.nih.gov/unists)查取19个微卫星 DNA标记,由生工生物工程(上海)有限公司合成,各微卫星标记在染色体上的分布等信息见表1。
PCR反应体积为 25 μL,引物 F和 R各(20 μM/L)1 μL,模板 DNA 1 μL,2 × PCR Master Mix 12.5 μL,ddH2O 9.5 μL。PCR 反应条件为 95 ℃ 预变性6 min;然后94℃变性1 min,54℃ ~65℃退火45 s,72℃延伸 1 min,35个循环;最后 72℃延伸10 min;4℃终止反应。
1.4 电泳及结果记录
PCR扩增产物经2%的琼脂糖凝胶电泳检测效果后,取4 μL与上样缓冲液混合后在8%的非变性聚丙烯酰胺凝胶上60V电泳16h,硝酸银染色后用氢氧化钠显色,条带清晰后用10%的乙酸终止,最后用凝胶成像系统和Sony数码相机双重照相。
1.5 统计分析
用BioRad凝胶成像系统对8%非变性聚丙烯酰胺凝胶电泳图带进行分析,确定其核苷酸条带分子量大小,以大小不同的扩增片段为不同的等位基因,按等位基因从小到大的顺序分别依次定名为A、B、C、D、E、F 等。用 POPGENE Version 1.32 软件计算出各座位的观察杂合度(Ho)、期望杂合度(He)、等位基因频率(gene frequency)、观察等位基因数(observed number of alleles,Na)、有效等位基因数(effective number of alleles,Ne)、香 隆 信 息 指 数(shannon’s information index,I)等指标,并根据Botstein等的公式利用 Excel计算多态信息含量(polymorphism information content,PIC)。
使用 POPGENE Version 1.32软件对各座位进行连锁分析,根据座位等位基因的数目多少,应用完全计数或马尔科夫链法检验群体的 Hardy-Weinberg遗传平衡状态。
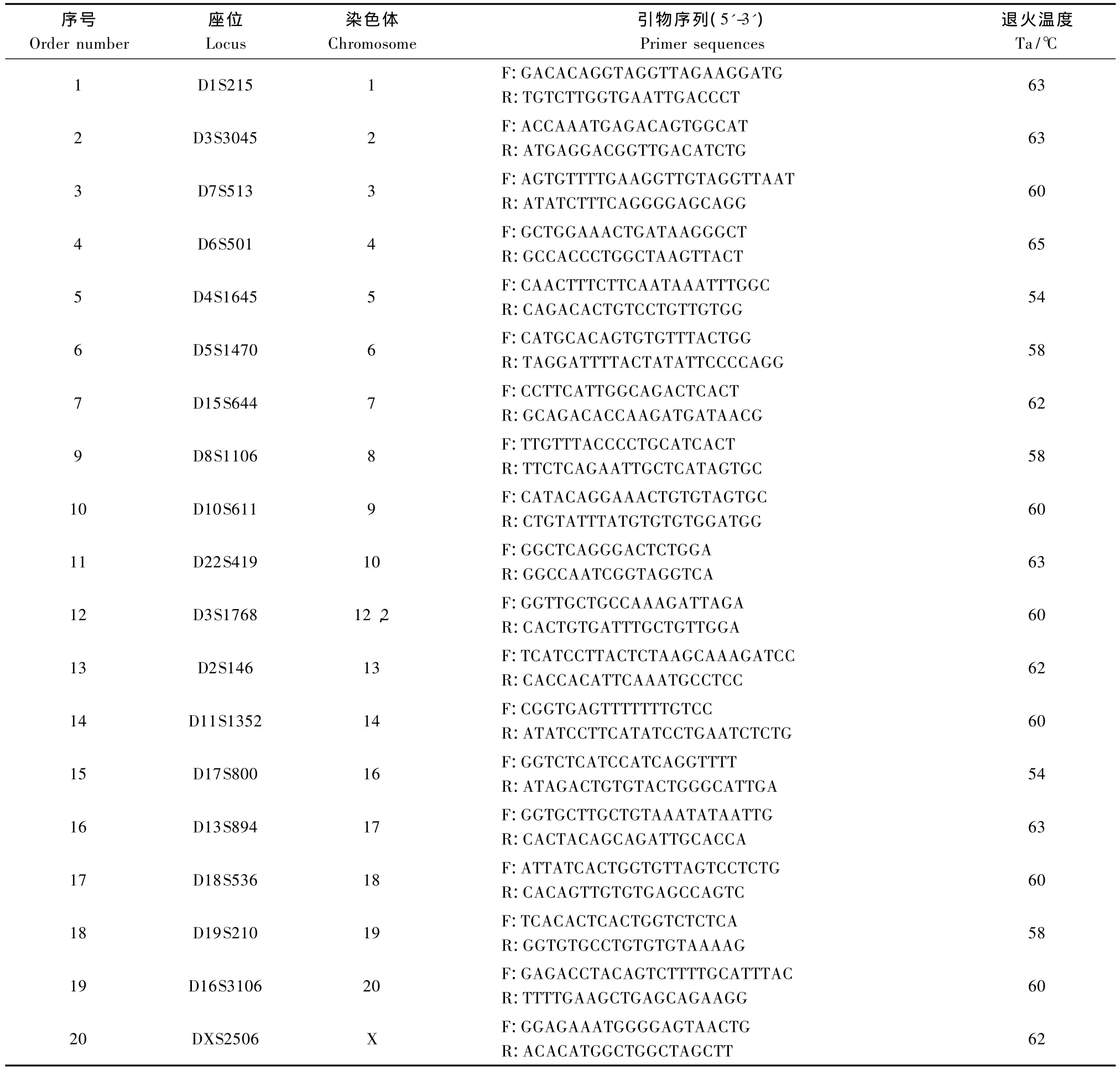
表1 微卫星DNA标记的相关信息Tab.1 The information ofmicrosatelliteDNA markers
2 结果
2.1 PCR扩增结果检测
对19个微卫星座位的PCR扩增产物进行8%非变性聚丙烯酰胺凝胶电泳,本文以D4S1645座位的电泳图谱为例说明(图1)。

注:M:DL2000 DNA分子量标准,1~22:样品编号。图1 D4S1645座位PCR扩增产物的聚丙烯酰胺凝胶电泳检测结果Note:M:DL2000 DNA marker,1 ~22:Number of samples.Fig.1 The PAGE image of PCR products at D4S1645 locus
2.2 群体遗传多样性分析
根据各微卫星座位的基因型,计算出各座位观察等位基因数及其频率、有效等位基因数、观察杂合度,结果见表2。19个微卫星座位均呈现出了多态性,共检测到了观察等位基因164个。各座位的观察等位基因数在6~11个之间,平均为8.6316个。各基因座位的有效等位基因数在 3.5727 ~8.9416个之间,平均为6.0709个。观察等位基因数最多的座位是 D4S1645、D5S1470和 D13S894,有11个等位基因;观察等位基因数最少的座位是D19S210和D16S3106,有6个等位基因。

表2 恒河猴群体19个微卫星座位的等位基因信息Tab.2 Allele information at 19 microsatellite loci of Macaca mulatta population

表3 恒河猴群体19个微卫星座位的遗传多态性Tab.3 The genetic diversity measured at 19 microsatellite loci of Macaca mulatta population
利用POPGENE version 1.32软件计算出各微卫星座位的观察杂合度、期望杂合度、香隆信息指数等指标,结果见表3。各个微卫星座位的观察杂合度在0.2560 ~ 0.6364 之间,群体平均值为 0.4309,其中,D5S1470座位最高,D19S210座位最低;期望杂合度在 0.7230 ~ 0.9010 之间,群体平均值 为 0.8270,D13S894座位最高,D16S3106座位最低;多态信息含量在 0.6771 ~ 0.8874 之间,群体平均值为 0.7979,D3S1768座位最高,D16S3106座位最低;香隆信息指数在 1.4249 ~ 2.2662 之间,群体平均值为 1.8901,D5S1470座位最高,D16S3106座位最低。
采用 POPGENE version 1.32软件计算恒河猴群体每个微卫星座位Hardy-Weinberg遗传平衡检验的精确P值,结果显示,本研究检测的19个微卫星座位均偏离 Hardy-Weinberg平衡(P <0.01)。
3 讨论
群体的遗传多样性通常用多个基因座位遗传多样性参数的平均值来描述,其重要指标主要有群体平均等位基因数、平均有效等位基因数、平均观察杂合度、平均期望杂合度、平均多态信息含量等[6]。本实验中各微卫星座位的观察等位基因数平均为8.6316个;有效等位基因数平均为6.0709个;多态信息含量平均值为0.7979,各微卫星座位多态信息含量均大于0.5,均属于高度多态性座位(当PIC>0.5时,为高度多态性座位);各个微卫星座位的观察杂合度群体平均值为0.4309;期望杂合度群体平均值为0.8270。由此可见,本实验所研究恒河猴群体等位基因数多、多态信息含量高、杂合度大,说明群体内基因型一致性差,遗传变异大,遗传多样性丰富。所研究恒河猴群体有如此丰富的遗传多样性,追溯原因有以下两点:一是恒河猴群体遗传基础广泛,他们的祖先来源于云南的镇沅、景东、楚雄、保山等多个地区;二是多年来开展的遗传繁育方式较合理,避免了近交系数的上升。
本研究检测的19个微卫星座位均偏离Hardy-Weinberg平衡(P<0.01),说明本研究检验的恒河猴群体存在杂合子缺失的现象。打破平衡的因素有突变、选择、随机遗传漂变、迁移和遗传异质性等,本研究19个微卫星座位均处于 Hardy-Weinberg不平衡状态,主要原因可能是群体处于非随机交配体制中,存在一定程度的人工选择。
微卫星DNA标记技术已在近交系大小鼠遗传监测、亲子鉴定及法医鉴定中得到了有效的应用[7-10]。本研究从大量的微卫星DNA座位中选择分布在不同染色体上的19个微卫星标记,利用这些微卫星引物对随机抽取的60只人工饲养繁殖的恒河猴个体进行PCR扩增,结果显示各微卫星座位均能够扩增出目的条带,电泳条带清楚可辨,呈现明显的DNA多态性,说明所选择的微卫星标记具有一定的代表性,能反映出个体间的遗传概貌,可以有效的进行恒河猴群的遗传质量监测。但本研究中样品较少,不足以完全反映出群体的遗传多样性,有必要进一步扩大样本量,并对实验方法进一步完善,建立标准的恒河猴遗传监测分子生物学方法,定时监测恒河猴群体遗传多样性状况,了解群体遗传多样性的变化趋势,从而指导人们进行合理的恒河猴遗传资源保护和利用。
[1]李瑞生,孙琪云,孙岩松,等.恒河猴群微卫星DNA多态性的分析[J].中国实验动物学报,2005,13(1):51 -54.
[2]皮道元,谢莉萍,卢晟盛,等.利用微卫星标记分析4个食蟹猴群体的遗传多样性[J].中国兽医学报,2011,31(3):444 - 448.
[3]Sodhi M,Mukesh M,Mishra BP,et al.Microsatellite analysis of genetic population structure of zebu cattle(Bos indicus)breeds from north-western region of India[J].Anim Biotechnol,2011,22(1):16 - 29.
[4]Korrida A,Jadallah S,Chbel F,et al.Patterns of genetic diversity and population structure of the threatened Houbara and Macqueen’s bustards as revealed by microsatellite markers[J].Genet Mol Res,2012,11(3):3207 - 3221.
[5]Velickovic N,Djan M,Obreht D,et al.Population genetic structure of wild boars in the West Balkan region[J].Genetika,2012,48(8):1007 - 1011.
[6]法兰克汉 R,巴卢J D,布里斯科D A(黄宏文,康明译).保育遗传学导论[M].北京:科学出版社,2005,47-62.
[7]郑龙,李建辉,王俊霞,等.微卫星DNA标记在近交系大鼠HFJ和MIJ遗传监测中的应用[J].中国实验动物学报,2012,20(2):32 - 36.
[8]王纯耀,杨卫红,宋国英,等.微卫星DNA在近交系小鼠遗传监测中的应用[J].中国兽医学报,2008,28(3):333-335.
[9]姜盼盼,张守纯,于宁,等.性染色体微卫星标记在亲缘鉴定中的应用[J].生命科学,2010,22(5):487 -491.
[10]Dettlaff - Kakol A, Pawlowski R.First Polish DNA“manhunt”—an application of Y-chromosome STRs[J].Int J Legal Med,2002,116(5):289 - 291.

