与人遗传病相关的DNA重复序列的体外核小体定位特性
柴 荣,赵宏宇,蔡 禄
(内蒙古科技大学数理与生物工程学院,内蒙古包头014010)
人们已经发现40 余种人类神经系统遗传疾病与DNA 重复序列扩增或删除有关[1],比如,(CTG)n·(CAG)n 的扩增或删除可能会引起脊髓小脑共济失调1、2、3、6、7、8、12 和17 型以及肌强直性营养不良1 型等,位于FRDA 基因1 号内含子上的GAA重复序列异常扩增会引起Friedreich 共济失调[2],ATXN10 基因含有较长的ATTCT 重复片段会导致脊髓小脑共济失调10 型[3],而肌强直性营养不良2 型则可能与(GCCT)n·(CAGG)n 扩增有关[4]。近十余年该领域的研究主要集中在重复序列形成的异常2 级结构以及遗传不稳定性[5],对于重复序列的核小体定位以及对染色质结构影响了解甚少。仅有少量工作表明,将重复序列纯化后,体外与组蛋白组装形成核小体的能力有所差异,发现CTG 重复片段容易形成核小体[6],ATTCT 重复序列对组蛋白的亲和性较强[7],而GAA 重复序列则很难形成核小体[8]。Widom 601 序列是较为稳定、容易形成核小体的序列[9]。本实验构建了含有601 序列的重组质粒pUC601 作为对照,并体外用盐透析法组装染色质及摸索微球菌核酸酶酶切条件。然后用本实验室构建的含有上述人遗传病相关重复序列的质粒体外组装染色质与微球菌核酸酶酶切分析方法,在质粒层次初步研究了重复序列核小体定位的特性,为该类疾病机理的理解奠定了一定的基础。
1 材料与方法
1.1 材料
1.1.1 试验菌株:大肠杆菌DH5α、质粒pUC19、pBS-601 由本实验室保存。重组质粒pUC(GAA)42、pUC(ATTCT)43和pUC(GCCT)18由本实验室构建,在质粒的多克隆位点分别含有重复序列(GAA)42、(ATTCT)43和(GCCT)18[10]。
1.1.2 试剂:质粒小提试剂盒(上海生工),微球菌核酸酶(MNase)、LD1000 标志、宽谱DNA 标志、蛋白酶K、限制性内切酶和T4 DNA 连接酶(TaKaRa公司)。
1.2 方法
1.2.1 含601 序列重组质粒的构建:用BamH Ⅰ和Pst Ⅰ双酶切pBS-601,分离199 bp的601 序列,连接到pUC19 质粒的BamH Ⅰ和Pst Ⅰ之间,转入E.coli DH5α 中,涂布在含氨苄青霉素的固体LB 平板上进行筛选,阳性克隆扩增后提取质粒,用BamH Ⅰ和Pst Ⅰ双酶切进行鉴定,将鉴定正确的质粒通过测序确认其完整性。
1.2.2 质粒提取:用质粒小提试剂盒小量提取质粒。按照参考文献中的方法大量提取质粒[11]。
1.2.3 组蛋白八聚体表达、纯化与复性:挑取含有H2A,H2B,H3,H4 基因的重组质粒的单菌落于25 mL 氨苄加氯霉素 LB 培养基中,37 ℃,220 r/min,过夜培养;取10 mL菌液加入到750 mL氨苄加氯霉素LB 培养基中;当OD600 达到0.4 ~0.6时,培养液中加入375 μL IPTG(1 mol/L)。37 ℃,220 r/min,培养3 ~4 h;收集培养液于4 ℃、4 000 ×g离心50 min,去上清,再用80 mL PBS 缓冲液充分重悬菌体沉淀,4 ℃、4 000 ×g离心30 min,去上清;将先前收的H2A、H2B、H3 和H4 菌液用100 mL洗脱缓冲液(50 mmol/L Tris,100 mmol/L氯化钠,1 mmol/L EDTA,1 mmol/L Benzamidine)重悬,再超声充分破碎细菌,4 ℃、23 000 ×g 离心20 min,去上清;用洗脱缓冲液+1% Triton-X 100 充分重悬沉淀,4 ℃、20 000 ×g离心10 min,去上清,重复此步;用洗脱缓冲液充分重悬沉淀,4 ℃、20 000 ×g离心10 min,去上清;用30 mL解折叠缓冲液(7 mol/L 盐酸胍,20 mmol/L Tris,10 mmol/L DTT)充分溶解沉淀,室温搅拌1 h 溶解;20 ℃、23 000 ×g离心20 min,保留上清,测定浓度;取5 mg的H2A,H2B,H3,H4 加入透析袋中,置于重折叠缓冲液(2 mol/L 氯化钠,10 mmol/L Tris,1 mmol/L EDTA)中透析,4 ℃搅拌12 h后,再换一次透析液,4 ℃透析24 h。将透析液取出,4 ℃、13 000 r/min,离心5 min,收集上清,再将样品浓缩至500 μL。过分子筛柱,收集片段;电泳检测。
1.2.4 体外组装核小体:将纯化的组蛋白八聚体与重组质粒以一定的比例加入到含有2 mol/L氯化钠的TE 缓冲液中混匀,总体系80 μL,然后加入透析管(Thermo,10 000 MWCO)中,放入含2 mol/L氯化钠的TE 透析液中透析16 h,在此过程中用恒流泵(HL-ZS,上海青浦西仪器厂)将TE 缓冲液匀速滴入透析液中,使其中氯化钠的浓度降到0.6 mol/L;之后将透析管转入到不含有氯化钠的TE 缓冲液中透析3 ~6 h。
1.2.5 MNase 酶切:取组装后含有4 μg DNA 的样品、微球菌核酸酶120 ~360 mU、10 × MNase 缓冲液,用灭菌水补齐到80 μL,37 ℃水浴,酶切5 ~15 min;等体积终止缓冲液(20 mmol/L EDTA,0.2 mol/L氯化钠,1% SDS,0.25 g/L甘油)终止反应,加入2 μL 100 g/L蛋白酶K,55 ℃反应30 min;等体积酚氯仿抽提,将上清转移到新管,加入2 倍体积无水乙醇,1/10 体积3 mol/L 的乙酸(pH 5.2),1 μL 20 g/L 糖原,-20 ℃ 沉淀过夜;4 ℃,12 000 r/min离心15 min,去上清;70%乙醇洗1 次,4 ℃,12 000 r/min离心15 min,室温晾干,20 μL TE缓冲液溶解。
1.2.6 琼脂糖凝胶电泳:微球菌核酸酶酶切处理后DNA 样品在1.3%的琼脂糖凝胶中电泳,电泳后EB染色,凝胶成像仪拍照观察分析。
2 结果
2.1 重组质粒的构建
重组质粒pUC-601 与重组质粒pUC(GAA)42、pUC(ATTCT)43、pUC(GCCT)18除了多克隆位点之间的目的片段不同外其余均相同(图1A)。将构建的pUC601 挑取两个亚克隆用BamH Ⅰ和Pst Ⅰ双酶切后可以得到2674bp和199 bp两条片段(图1B)。测序后其序列完全正确。
2.2 组蛋白的纯化与复性
纯化的单个组蛋白H2A(14 ku)、H2B(14 ku)、H3(15 ku)和H4(13 ku)(图2A),条带位置正确,纯度较高。复性后形成的组蛋白八聚体经AKTA 系统分子筛纯化后,可见样品C2 至C4 为纯度较高组蛋白八聚体(图2B),将回收的C2 至C4 样品混合测定浓度后,分装冻存于-80 ℃用于后续组装实验。
2.3 组蛋白与DNA 组装最适比例的确定
将组蛋白八聚体与重组质粒pUC601 分别以质量比为0.7、1.1 和1.5 3 个不同的比例体外组装形成染色质结构,透析后用360 mU的微球菌核酸酶进行酶切(图3)。从图中可以看出,组蛋白与DNA 的比例为1∶1时酶切可以形成明显的梯度条带,染色质结构较为均匀。所以后面的实验选择组蛋白与DNA 的质量比为1∶1进行组装。


2.4 最适酶量的确定
为了选择反应体系中微球菌核酸酶的酶量,将组蛋白与重组质粒pUC601 以1∶1比例进行体外组装染色质,透析后分别用120、240、360和480 mU的微球菌核酸酶进行酶切10 min(图4)。从图中可以看出用360 mU酶量切得到单个核小体的DNA 条带较明亮,且条带的梯度整齐,所以后续实验用360 mU的酶量来进行酶切。

图3 不同比例下pUC601 体外组装染色质微球菌核酸酶酶切结果Fig 3 Micrococcal nuclease digestion assay of pUC601 assembly chromatin in vitro in different ratio

图4 不同微球菌核酸酶酶量消化体外组装染色质的结果Fig 4 Micrococcal nuclease digestion assay of assembly chromatin in vitro in different enzyme amount
2.5 最适酶切时间的确定
微球菌核酸酶酶切反应对时间较为敏感,为了选择最佳酶切时间,将组蛋白与重组质粒pUC601以1∶1的比例体外组装形成染色质结构,透析后加微球菌核酸酶360 mU,分别酶切5、10 和15 min(图5)。从图中可以看出10 和15 min时的酶切效果基本一致,条带明亮整齐,梯度明显,因此选择10 min进行酶切。
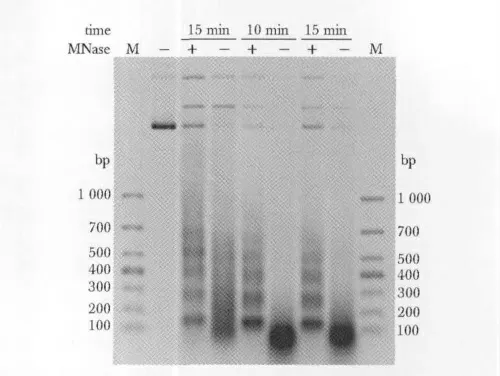
图5 不同酶切时间消化体外组装染色质的结果Fig 5 Micrococcal nuclease digestion assay of assembly chromatin in vitro in different digestion time
2.6 不同质粒形成核小体能力的比较
在以上组装及酶切条件下,分别将组蛋白与重组质粒pUC601、pUC(GAA)42、pUC(ATTCT)43和pUC(GCCT)18以1∶1比例体外组装形成染色质结构,用微球菌核酸酶360 mU、37 ℃酶切10 min(图6)。从图中可以看出在同样的条件下,质粒pUC(ATTCT)43酶切后形成的梯度条带位置靠上,可见明显的4 条带;质粒pUC(GAA)42的酶切后主要形成3条梯度的条带,且位置相对靠下;而pUC(GCCT)18介于pUC(ATTCT)43和pUC(GAA)42之间。
3 讨论
目前,染色质体外组装主要有两种方式:一种是采用盐透析的方法,另一种是依赖ATP 及蛋白因子(dNAP I 和ACF)进行的染色体体外组装[12]。而盐透析方法装配染色质系统内只含有组蛋白和DNA,不需要加入特殊的蛋白质,因此产生的染色质结构仅依赖于DNA 序列的核小体定位特性。实验以构建的重组质粒pUC601 为模板,用盐透析方法体外与组蛋白形成核小体结构,确定了微球菌核酸酶的酶切时间和酶量,然后用这个条件进一步比较了含有与人遗传病相关的3 种重复序列的质粒形成核小体能力的区别。

图6 不同质粒与组蛋白组装后酶切比较电泳结果Fig 6 Micrococcal nuclease digestion assay of assembly chromatin in vitro in different recombinant plasmids
可以看出在同样的条件下,质粒pUC(ATT CT)43酶切后形成的梯度条带位置靠上,可以看到明显的4 条带,相比其他质粒不容易被切开,说明染色质结构较为紧密;质粒pUC(GAA)42的酶切后主要形成3 条梯度的条带,且位置相对靠下,表明易被微球菌核酸酶消化,提示染色质结构相对比较松散;而pUC(GCCT)18体外组装形成染色质结构后酶切的难易程度介于pUC(ATTCT)43和pUC(GAA)42之间。而已有工作表明重复序列ATTCT 比GAA 容易形成核小体[7],这与本实验结果一致。实验构建的重组质粒除了多克隆位点插入的重复序列片段不同,其余侧翼序列均一致,所以其形成核小体结构能力不同的主要原因是由于插入了不同的重复序列片段。据此结果推测,GAA 重复序列本身对组蛋白的亲和力弱,而ATTCT 易形成核小体,所以含有ATTCT 重复序列片段的质粒比含有GAA 重复序列的质粒体外形成染色质结构要相对紧密,核小体的占据情况要稍高。但是对于重复序列是否能形成异常的二级结构以及其可能对染色质结构的影响,仍需要进一步的研究。本实验对于由DNA 重复序列引起的人遗传病致病机理在核小体及染色质水平的理解具有一定的意义。
志谢:组蛋白八聚体的表达、纯化与复性在中国科学院生物物理研究所李国红课题组完成,感谢李国红研究员给予的帮助与指导。
[1]Pearson CE,Nichol Edamura K,Cleary JD.Repeat instability:mechanisms of dynamic mutations[J].Nature Rev Genet,2005,6:729-742.
[2]Liu G,Chen X,Gao Y,et al.Altered replication in human cells promotes DMPK(CTG)(n)·(CAG)(n)repeat instability[J].Mol Cell Biol,2012,32:1618-1632.
[3]Votsi C,Zamba-Papanicolaou E,Georghiou A,et al.Investigation of SCA10 in the cypriot population:Further exclusion of SCA dynamic repeat mutations[J].J Neurol Sci,2012,323:154-157.
[4]Raheem O,Olufemi SE,Bachinski LL,et al.Mutant(CCTG)n expansion causes abnormal expression of zinc finger protein 9 (ZNF9)in myotonic dystrophy type 2[J].Am J Pathol,2010,177:3025-3036.
[5]Rajeswari MR.DNA triplex structures in neurodegenerative disorder,Friedreich's ataxia[J].J Bio Sci,2012,37:519-532.
[6]Godde JS,Wolffe AP.Nucleosome assembly on CTG triplet repeats[J].J Biol Chem,1996,271:15222-15229.
[7]Hagerman KA.,Ruan H,Edamura KN,et al.The ATTCT repeats of spinocerebellar ataxia type 10 display strong nucleosome assembly which is enhanced by repeat interruptions[J].Gene,2009,434:29-34.
[8]Mulvihill DJ,Nichol Edamura K,Hagerman KA,et al.Effect of CAT or AGG Interruptions and CpG Methylation on Nucleosome Assembly upon Trinucleotide Repeats on Spinocerebellar Ataxia,Type 1 and Fragile X Syndrome[J].J Biol Chem,2005,80:4498-4503.
[9]Ravindra D,Makde JR,England HP.Structure of RCC1 chromatin factor bound to the nucleosome core particle[J].Nature,2010,467:562-566.
[10]赵宏宇,蔡禄,赵秀娟,等.化学药物对与人遗传病相关的DNA 重复序列不稳定性的影响[J].生物技术通报,2010,11:153-161.
[11]J 萨姆布鲁克,DW 拉塞尔.分子克隆实验指南(第3版)[M].黄培堂等译.北京:科学出版社,2002:32-36.
[12]Wippo CJ,Korber P.In vitro reconstitution of in vivo-like nucleosome positioning on yeast DNA[J].Methods Mol Biol,2012,833:271-287.

