甲烷八叠球菌研究进展
杨 冰,卢向阳,田 云
(1.湖南农业大学生物科学技术学院,湖南 长沙410128;2.湖南省农业生物工程研究所,湖南 长沙410128)
用有机物生产可再生能源是可持续发展理念中最重要的方法之一,欧盟已提出到2020年欧洲能源必须有20%来源于可再生能源,而可再生能源的总预算中用有机物发酵产生的沼气占25%[1]。我国是一个能源消耗大国,可持续发展是我们必须坚持的发展战略之一。因此,用有机废弃物生产可再生能源也将是我国未来可再生能源研究的重点。
厌氧消化技术是应用得最广泛的将有机废弃物转化为甲烷的技术,它除了能稳定降解有机废弃物产生沼气外,还有生物量低、有机物装载率高、营养需求限度低、运行和维护费用低等优点[2]。
厌氧消化过程主要包括两个步骤:一是高分子有机物的分解;二是分解后物质的甲烷化。甲烷化是厌氧消化的限速步骤,主要靠产甲烷古细菌将厌氧消化产生的有机或无机化合物厌氧发酵生成甲烷。在有机物厌氧消化的甲烷化过程中,70%以上的甲烷来自乙酸的裂解[3,4],而目前所知,只有甲烷鬃毛菌(Methanosaeta)和甲烷八叠球菌(Methanosarcineae)能裂解乙酸产生甲烷[5]。但是当反应器中乙酸浓度超过3000mg·L-1时甲烷鬃毛菌就无法生长,而甲烷八叠球菌在乙酸浓度高达15 000mg·L-1的发酵液中仍能利用乙酸产生甲烷[6~8]。因此,甲烷八叠球菌在有机物厌氧消化中起着重要的作用。目前,欧美国家对甲烷八叠球菌的研究已进入分子机理研究阶段,但我国对甲烷八叠球菌研究的报道还比较少。
通过查阅国内外文献,作者在此对甲烷八叠球菌的特征和应用研究进展进行了综述。
1 甲烷八叠球菌的分类
甲烷八叠球菌属于古细菌域(Archaea)、广古菌门(Euryarchaeota)、甲烷球菌纲(Methanococci)、甲烷八叠球菌目(Methanosarcinales)、甲烷八叠球菌科(Methanosarcinaceae)、甲烷八叠球菌属(Methanosarcina)。1947年,荷兰学者Sehnellen[9]首次分离出了甲烷八叠球菌属并命名为Methanosarcina barkeri。随后各国科学家又相继分离到M.acetivorans、M.baltica、M.lacustris、M.mazei、M.semesiae、M.siciliae、M.thermophila、M.vaculolata等8个甲烷八叠球菌种[10]。
2 甲烷八叠球菌的特征
2.1 形态特征(图1)

图1 甲烷八叠球菌细胞的显微照片[11]Fig.1 Micrographs of Methano sarcineae[11]
甲烷八叠球菌通常是8个单细胞以图1a中的形式进行生长,它存在2种不同的形态:在淡水中生长时,以聚集形式存在,细胞外包裹着杂多糖基质(图1b);在高盐环境中生长时,则是以分散形式存在,没有胞外聚合物层(图1c)[11~13]。甲烷八叠球菌是唯一能够通过胞外多糖形成多细胞结构的古细菌,胞外多糖的形成是甲烷八叠球菌的一种自我保护机制,它能吸收水作为湿润剂,保持细胞内的水活度;同时也能减少扩散到细胞的氧,保护细菌免受氧的损害[12]。
2.2 代谢特征
甲烷八叠球菌是已知的唯一能够利用所有已知产甲烷途径(乙酸营养途径、氢营养途径和甲基营养途径)的产甲烷微生物菌属[14~16]。第一种途径是乙酸裂解生成甲烷,同时生成的CO与H2O反应生成CO2和H2;第二种途径是以CO2和H2为原料合成甲烷;第三种途径是以各种甲基化合物(甲醇、甲胺、甲基硫化物)为底物,在甲基转移酶的作用下生成甲烷[17]。如图2所示。
正是由于存在如此多的代谢途径,使甲烷八叠球菌成为厌氧消化中最重要的产甲烷菌群。

图2 甲烷生物合成途径[14]Fig.2 The synthetic pathways for methanogenesis[14]
2.3 基因组特征
2002年,M.acetivorans和M.mazei的全基因组测序完成[14,18];2007年又完成了 M.barkeri的基因组测序[19]。甲烷八叠球菌基因组特征比较见表1 。
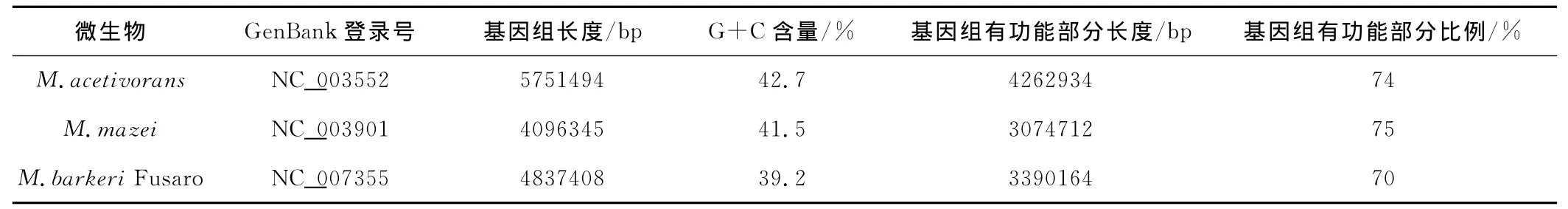
表1 甲烷八叠球菌基因组特征比较[19]Tab.1 Comparison of genomic features among Methanosarcinaspp.
由表1 可知,甲烷八叠球菌的基因组大小约为4.0×106~6.0×106bp,G+C含量在39%~43%之间,编码蛋白质的ORE约为3300~4600个,基因组有功能部分比 例 约 为70% ~75%[14,18,19]。随 着 这 3 个 菌种的全基因组测序的完成,人们对甲烷八叠球菌的遗传特征有了全面的认识,上述3种菌也因此成为研究产甲烷古细菌的模式菌种[20]。
甲烷八叠球菌的代谢路径多样,某一底物代谢路径相关基因的改变或敲除并不影响菌株在其它底物上的生长,这有利于用基因分析的方法研究其生长机制和产甲烷路径。针对这些种属的遗传工具也相继开发出来,包括对特定基因的定向突变和表达调控的工具如链霉菌属噬菌体φC31载体[21,22];许多代谢重建菌株,如 M.acetivorans iMB745[15]可用于假定的药物靶点的识别和设计利于生物燃料生产的菌株;同时M.barkeri Fusaro[12]已用于研究产甲烷古细菌的耐旱机制、M.mazei Go1[23]用于研究产甲烷古细菌的耐盐机制等。
3 甲烷八叠球菌在厌氧消化中的应用
3.1 产甲烷
厌氧消化虽然是目前被广泛应用的可再生能源生产的重要技术手段,但产甲烷菌群对不同环境的高度敏感性(pH值的改变、盐和有机物浓度的增加、负载率的增大、有毒物质的产生)易导致系统的崩溃[2,24,25]。这是由于,在消化过程中要维持不同微生物组合之间的微妙平衡,其中产甲烷菌是最易受到损害的,而甲烷八叠球菌通常能够耐受不同的逆境因子[26,27],对铵盐毒害、超负载率、高盐浓度、温度变化等逆境因子都有很强的抗性[28]。表2 是主要的产甲烷菌甲烷鬃毛菌和甲烷八叠球菌的的特点比较。

表2 甲烷鬃毛菌和甲烷八叠球菌的特点比较[28]Tab.2 Comparison of features between Methanosaetaand Methanosarcina[28]
由表2 可看出,无论是最大比生长速度(μmax)、半饱和常数(Ks)还是对各种逆境因子的耐受性,甲烷八叠球菌都比甲烷鬃毛菌要高得多。
3.2 水下阴极选择性增殖培养
厌氧消化中产甲烷菌的生长速度很慢,因此成为限制发酵速度的主要因素,但相比于其它产甲烷菌,甲烷八叠球菌的生长速度要快2~20倍[8]。所以加快产甲烷菌的生长速度并延长滞留时间,就能扩大厌氧消化器的处理能力。为了达到这一目的可在反应器中增设水下产氢阴极,选择性地加强氢营养型产甲烷菌的生长[7,29]。而甲烷八叠球菌既是氢营养型的又是乙酸营养型的,还能产生具有粘附作用的胞外多糖,能附着在阴极上生长[7,30],同时在阳极端产生氧气为水解微生物创造有氧条件,促进有机物的水解作用。通过在厌氧反应器中引入阴极(-0.8V)和阳极(+0.8V),可加快甲烷八叠球菌和水解菌的生长速度并延长滞留时间,从而提高沼气生产和反应器的稳定性,使甲烷的产率提高10%~25%[29]。
3.3 组合发酵
厌氧消化产甲烷主要靠两条途径:一是乙酸直接裂解生成CH4和CO2(乙酸营养型);二是利用H2还原CO2生成CH4(氢营养型)[31]。当反应器的氨浓度升高、pH值减小时,部分乙酸被互养乙酸氧化(Syntrophic acetate oxidizing,SAO)菌氧化为 CO2和 H2,而不是被乙酸营养型产甲烷菌直接转化为甲烷[32,33]。但是乙酸氧化生成CO2和H2在热力学上是非常不利的(△G0′=104.6kJ·mol-1),SAO细菌只有在 H2分压非常小(2.6~74Pa)时才能完成这一氧化过程,因此只有当互养乙酸氧化菌和氢营养型产甲烷(Hydrogenotrophic methanogenesis,HM)菌 耦 合(SAO-HM),乙酸的甲烷化才可以顺利进行[32,34]。研究发现[35],当反应器中总氨浓度上升到3000mg·L-1或 乙酸浓度达到3000mg·L-1时,厌氧消化系统的甲烷生成途径就会由乙酸营养型转变为氢营养型。
甲烷八叠球菌既可以进行乙酸营养型产甲烷又可以进行氢营养型产甲烷,如果以它们作为产甲烷菌群与SAO细菌耦合,基于以上原理,当反应器负载增大或由于突然的损害导致氨或乙酸浓度升高时,就不需要进行菌群的变化,而只需要甲烷八叠球菌改变生理特性,由乙酸营养变为氢营养就好,这样就缩短了反应器应对干扰的时间,提高了反应器的稳定性和处理能力[28]。
4 结语
古菌群对环境的敏感性易导致厌氧消化产甲烷反应系统崩溃,而甲烷八叠球菌因其遗传和代谢的特点成为弥补这些缺陷最有潜能的菌种。甲烷八叠球菌能够耐受高氨、高盐、高乙酸浓度,其独特的表面结构使可以在水下阴极上生长,与SAO细菌的耦合可以增强厌氧消化反应器的性能。
甲烷八叠球菌有如此多的优越性,可能是未来厌氧消化系统能源转化的基础,因此,可以利用分子生物学技术,如插入启动子、插入甲烷合成相关基因、敲除相关阻遏基因等,对它们进行进一步的改造,使它们能更好地应用于生产;同时还可以用组合菌研究的方法筛选可以与之耦合的最适SAO细菌;因甲烷八叠球菌Ks(200~280mgCOD·L-1)比较大,在发酵初期转化效率低,故也可以与Ks比较小的甲烷鬃毛菌(10~50mgCOD·L-1)组合来提高初期转化效率,以进一步发掘其产甲烷潜力,为厌氧消化技术的进步奠定基础。
但这些都还只是理论上的研究,目前并没有一款以甲烷八叠球菌为唯一产甲烷菌的厌氧消化器投入到实际生产中,因此这也是亟待进一步研究的方向。
[1]Holm-Nielsen J B,Al Seadi T,Oleskowicz-Popiel P.The future of anaerobic digestion and biogas utilization[J].Bioresource Technology,2009,100(22):5478-5484.
[2]Wijekoon K C,Visvanathan C,Abeynayaka A.Effect of organic loading rate on VFA production,organic matter removal and microbial activity of a two-stage thermophilic anaerobic membrane bioreactor[J].Bioresource Technology,2011,102(9):5353-5360.
[3]McCarty P L,Smith D P.Anaerobic wastewater treatment[J].Environmental Sci Technol,1986,20(12):1200-1206.
[4]Harper S R,Polhland F G.Recent developments in hydrogen management during anaerobic biological wastewater treatment[J].Biotechnol Bioeng,1986,28(4):585-602.
[5]Lattinga G,van Velsen A F M,Hobma S W,et al.Use of the upflow sludge blanket(USB)reactor concept for biological wastewater treatment[J].Biotechnol Bioeng,1980,22(4):699-734.
[6]Staley B F,de los Reyes F L,Barlaz M A.Effect of spatial differences in microbial activity,pH,and substrate levels on methanogenesis initiation in refuse[J].Applied and Environmental Microbiology,2011,77(7):2381-2391.
[7]Hao L P,Lu F,He P J,et al.Predominant contribution of syntrophic acetate oxidation to thermophilic methane formation at high acetate concentrations[J].Environmental Science and Technology,2011,45(2):508-513.
[8]Qu X,Vavilin V A,Mazeas L,et al.Anaerobic biodegradation of cellulosic material:Batch experiments and modelling based on isotopic data and focusing on aceticlastic and non-aceticlastic methanogenesis[J].Waste Management,2009,29(6):1828-1837.
[9]Sehnellen C G T P.Onderzoekingen over de methehnellen,Onderzoekingen over de methaangisting[D].Rotterdam:Technical University Delft,1947.
[10]Boone D R,Whitman W B,Rouvihre P.Diversity and taxonomy of methanogens[C].In Ferry J G(ed.),Methanogenesis:Ecology,physiology,biochemistry,and genetics[A].New York:Chapman & Hall,1993:35-80.
[11]Qu X,Mazeas L,Vavilin V A,et al.Combined monitoring of changes in delta13CH4and archaeal community structure during mesophilic methanization of municipal solid waste[J].FEMS Microbiol Ecol,2009,68(2):236-245.
[12]Anderson K L,Apolinario E E,Sowers K R,et al.Desiccation as a long-term survival mechanism for the archaeon Methanosarcina barkeri[J].Appl Environ Microbiol,2012,78(5):1473-1479.
[13]Sowers K R,Boone J E,Gunsalus R P.Disaggregation of Methanosarcina spp.and growth as single cells at elevated osmolarity[J].Appl Environ Microbiol,1993,59(11):3832-3839.
[14]Galagan J E,Nusbaum C,Roy A,et al.The genome of M.acetivorans reveals extensive metabolic and physiological diversity[J].Genome Res,2002,12(4):532-542.
[15]Benedict M N,Gonnerman M C,Metcalf W W,et al.Genomescale metabolic reconstruction and hypothesis testing in the methanogenic archaeon Methanosarcina acetivorans C2A[J].Bacteriol,2012,194(4):855-865.
[16]Welander P V,Metcalf W W.Loss of the mtr operon in Methanosarcinablocks growth on methanol,but not methanogenesis,and reveals an unknown methanogenic pathway[J].Proc Natl Acad Sci,2005,102(30):10664-10669.
[17]Vepachedu V R,Ferry J G.Role of the fused corrinoid/methyl transfer protein CmtA during CO-dependent growth of Methanosarcina acetivorans[J].Bacteriol,2012,194(16):4161-4168.
[18]Deppenmeier U,Johann A,Hartsch T,et al.The genome of Methanosarcina mazei:Evidence for lateral gene transfer between bacteria and archaea[J].Mol Microbiol Biotechnol,2002,4(4):453-461.
[19]Maeder D L,Anderson I,Brettin T S,et al.The Methanosarcina barkeri genome:Comparative analysis with Methanosarcina acetivorans and Methanosarcina mazei reveals extensive rearrangement within methanosarcinal genomes[J].Bacteriol,2006,188(22):7922-7931.
[20]Buan N,Kulkarni G,Metcalf W W.Genetic methods for Metha nosarcinaspecies[J].Methods Enzymol,2011,494:23-42.
[21]Apolinario E E,Jackson K M,Sowers K R.Development of a plasmid-mediated reporter system for in vivo monitoring of gene expression in the archaeon Methanosarcina acetivorans[J].Appl Environ Microbiol,2005,71(8):4914-4918.
[22]Guss A M,Rother M,Zhang J K,et al.New methods for tightly regulated gene expression and highly efficient chromosomal integration of cloned genes for Methanosarcinaspecies[J].Archaea,2008,2(3):193-203.
[23]Spanheimer R,Muller V.The molecular basis of salt adaptation in Methanosarcina mazei Go1[J].Arch Microbiol,2008,190(3):271-279.
[24]Chen Y,Cheng J J,Creamer K S.Inhibition of anaerobic digestion process:A review[J].Bioresource Technology,2008,99(10):4044-4064.
[25]Ma J X,Mungoni L J,Verstraete W,et al.Maximum removal rate of propionic acid as a sole carbon source in UASB reactors and the importance of the macro-and micro-nutrients stimulation[J].Bioresource Technology,2009,100(14):3477-3482.
[26]Shin S G,Zhou B W,Lee S,et al.Variations in methanogenic population structure under overloading of pre-acidified highstrength organic wastewaters[J].Process Biochemistry,2011,46(4):1035-1038.
[27]Thauer R K,Kaster A K,Seedorf H,et al.Methanogenic archaea:Ecologically relevant differences in energy conservation[J].Nature Reviews Microbiology,2008,6(8):579-591.
[28]De Vrieze J,Hennebel T,Boon N,et al.Methanosarcina:The rediscovered methanogen for heavy duty biomethanation[J].Bioresour Technol,2012,112:1-9.
[29]Sasaki K,Hirano S,Morita M,et al.Bioelectrochemical system accelerates microbial growth and degradation of filter paper[J].Applied Microbiology and Biotechnology,2011,89(2):449-455.
[30]Francoleon D R,Boontheung P,Yang Y,et al.S-layer,surfaceaccessible,and concanavalin A binding proteins of Methanosarcina acetivorans and Methanosarcina mazei[J].Journal of Proteome Research,2009,8(4):1972-1982.
[31]Demirel B,Scherer P.The roles of acetotrophic and hydrogenotrophic methanogens during anaerobic conversion of biomass to methane:A review[J].Reviews in Environmental Science and Biotechnology,2008,7(2):173-190.
[32]Hattori S.Syntrophic acetate-oxidizing microbes in methanogenic environments[J].Microbes and Environments,2008,23(2):118-127.
[33]Sasaki D,Hori T,Haruta S,et al.Methanogenic pathway and community structure in a thermophilic anaerobic digestion process of organic solid waste[J].Journal of Bioscience and Bioengineering,2011,111(1):41-46.
[34]Nettmann E,Bergmann I,Pramschufer S,et al.Polyphasic analyses of methanogenic archaeal communities in agricultural biogas plants[J].Applied and Environmental Microbiology,2010,76(8):2540-2548.
[35]Schnurer A,Nordberg A.Ammonia,a selective agent for methane production by syntrophic acetate oxidation at mesophilic temperature[J].Water Science and Technology,2008,57(5):735-740.

