实时逆转录聚合酶链式反应检测活的金黄色葡萄球菌*
张冲,刘祥,陈计峦
1(国家加工食品质量监督检验中心(天津),天津,300384)
2(新疆石河子大学食品学院,新疆 石河子,832000)
实时逆转录聚合酶链式反应检测活的金黄色葡萄球菌*
张冲1,2,刘祥1,,陈计峦2
1(国家加工食品质量监督检验中心(天津),天津,300384)
2(新疆石河子大学食品学院,新疆 石河子,832000)
针对基于DNA水平的聚合酶链式反应(DNA-PCR)检测食品中金黄色葡萄球菌时会出现假阳性结果的缺点,建立一种能够区分死、活金黄色葡萄球菌的检测方法。根据金黄色葡萄球菌的femA基因,自设计Taqman探针、引物,采用一步法逆转录聚合酶链式反应(RT-PCR),以femA mRNA为检测对象,发现只有活的金黄色葡萄球菌显阳性,死亡的则呈阴性;纯培养时,重复检测变异系数为0.15,灵敏度为9×102CFU/mL,检出限可达1/3 CFU/3 mL。实验表明,该研究所建立的一步法逆转录聚合酶链式反应检测法,不仅灵敏度高、特异性强,而且能够有效地区分死、活金黄色葡萄球菌。
逆转录聚合酶链式反应,金黄色葡萄球菌,死活菌,普通聚合酶链式反应
金黄色葡萄球菌是引起细菌性食物中毒的重要病原菌之一[1-3],据美国疾病控制中心报告,由金黄色葡萄球菌引起的食物中毒仅次于大肠埃希菌,居第2位,占细菌性食物中毒的33%[4];大约有10%~40%的人是该病原菌的携带者[5],约32%的食品中存有该菌,由此引起的食物中毒极为普遍[6]。因此,对金黄色葡萄球菌的实时监测尤为重要。
目前,实时荧光定量聚合酶链式反应(real-time PCR)已被认为是检测金黄色葡萄球菌的最可靠、高效、低耗费的方法[7],具有特异性强、灵敏度高、重复性好、定量准确、速度快等优点[8-9]。但国内外当前运用最多的实时 PCR法,大都是基于 DNA水平(DNA-PCR),虽然快速、灵敏,但由于细菌死亡后,DNA降解缓慢,能够稳定存在,容易产生假阳性结果[10],无法区分死、活细菌。与DNA相比,mRNA的稳定性较差,在细菌死亡后能够快速降解[11],而在细菌还具有活性时,就会产生mRNA。因此,选择恰当的mRNA作为靶点基因,运用逆转录聚合酶链式反应(RT-PCR)技术,便能够只检测到具有活性的细菌,避免死亡细菌假阳性的出现[12]
femA是金黄色葡萄球菌细胞壁合成的必备基因,其编码蛋白与细胞壁肽聚糖五甘氨酸桥合成有关,同时也是当前最重要的病原菌——耐甲氧西林金黄色葡萄球菌(MRSA)表达耐药特性的重要辅助基因[13-15]。目前,RT-PCR 已被成功地应用到金黄色葡萄球菌 gryB、hlgA、hlgB、hlgC、lukF、luk S、luk E、luk M、spa、seA、seB、seB、seD等基因的检测,均表现出较高的特异性与灵敏度[16-17]。由此,本研究选择金黄色葡萄球菌的特异性femA基因,自设计TaqMan探针和引物,采用一步法RT-PCR技术,拟建立一种能够区分死、活金黄色葡萄球菌的检测方法。
1 材料与方法
1.1 材料与仪器
金黄色葡萄球菌(Staphylococcus aureus)、副溶血弧菌(Vibrio parahaemolyticus)、大肠杆菌(O157:H7)[E.coli(O157:H7)]、空肠弯曲菌(Campylobacter jejuni)、霍乱弧菌(Vibrio cholerae)、小肠结肠炎耶尔森菌(Yersinia enterocolitica)、创伤弧菌(Vibrio vulnificus)、溶藻弧菌(Vibrio alginolyticus)、沙门氏菌(Salmonella)。上海之江生物科技Bio-Rad PCR仪,配有CFX96TMReal-Time System与C1000TMThermal Cycler,美国Bio-Rad公司;RNAprep pure培养细胞/细菌总RNA提取试剂盒,北京天根生化公司;PCR反应试剂,Methicillin-resistant Staphylococcus aureus(MRSA)Real Time PCR Kit上海之江生物科技;DNase I、RNase-free,美国Fermentas公司;以上试剂均为分析纯试剂。
1.2 引物与探针
本实验所用的引物与探针如表1所示。

表1 实验所用的引物与探针
1.3 实验方法
1.3.1 金黄色葡萄球菌培养
将活化后的菌液50 μL接种于3 mL LB培养液中,振荡培养3h,200 r/min,37℃。
1.3.2 DNA与RNA提取
分别按提取试剂盒说明书进行。
1.3.3 DNase I消化
按DNase I说明书进行。
1.3.4 一步法RT-PCR扩增体系
扩增体系共 40 μL,其中包括 10× Buffer4μL,MgCl23.2 μL,dNTP 0.8 μL,femA-f 0.5 μL,femA-r 0.5 μL,femA-p 0.1 μL,混合酶1μL,ddH2O 25.9 μL,RNA 提取液4 μL。
扩增参数为:45℃×10 min;95℃×15 min;94℃×20 s,55℃×20 s,72℃×30 s,40cycles;单点荧光在72℃;选用FAM通道检测。
1.3.5 DNA-PCR扩增体系
1.3.5.1 试剂盒法
扩增体系共40 μL,金黄色葡萄球菌荧光定量检测混合液 35.6 μL,Taq 酶 0.4 μL,DNA 提取液4μL。
扩增参数为:94℃×2 min;93℃×15 s,60℃×60 s,40cycles;单点荧光在60℃;选用HEX通道检测。
1.3.5.2 femA及gB探针法
扩增体系共 40 μL,其中包括 10× Buffer4μL,MgCl23.2 μL,dNTP 0.8 μL,femA-f/gB-f 0.5 μL,femA-r/gB-r 0.5 μL,femA-p/gB-p 0.1 μL,Taq 酶0.4 μL,ddH2O 26.5 μL,DNA 提取液4μL。
扩增参数为:95℃×3 min;94℃×20 s,55℃×20 s,72℃×30 s,40 cycles;单点荧光在 72℃;选用 FAM通道检测。
1.3.6 区分死活菌
吸取2管100 μL活化后的金黄色葡萄球菌液,一管设为活菌,不做任何处理;另一管设为死菌,80℃水浴 20 min[11,18]。提取后,采用一步法 RT-PCR 检测。
同时另取2管相同浓度的死、活菌直接提取DNA,然后采用常规荧光PCR(DNA-PCR)的方法检测。
比较2组扩增结果,若DNA-PCR扩增后活菌与死菌均为阳性,而RT-PCR扩增后活菌结果为阳性,死菌为阴性,则说明能够区分死活菌。
1.3.7 特异性检测
取上述菌种与金黄色葡萄球菌同时抽提RNA后,再进行femA RT-PCR检测,验证体系的特异性。若只有金黄色葡萄出现阳性结果,而其它致病菌显阴性,则说明该引物与探针具有良好的特异性。
1.3.8 重复性
分别取体积为 500 μL、100 μL、10 μL、1 μL 的菌液进行RNA抽提,然后采用RT-PCR扩增,每个体积重复6次。根据统计学原理,将每个体积所得数据相对标准偏差作为变异系数(CV),再求其平均值,若平均值小于0.5%,则重复性良好。
1.3.9 标准曲线制作
取1 mL活化后的金黄色葡萄球菌,然后按步骤提取RNA,将提取液先按10倍比例稀释,在稀释至10-3后,按5倍比例稀释至 10-6∶100、10-1、10-2、10-3、10-3/5、10-4、10-4/5、10-5、10-5/5、10-6,再进行RT-PCR扩增绘制标准曲线,并将定量结果与传统培养法比对。
1.3.10 检出限
在3 mL培养基中分别加入菌量100 CFU、10 CFU、1 CFU、1/3 CFU、1/5 CFU、1/10 CFU,200 r/min,37℃,增菌3 h后,RT-PCR扩增,观察其扩增曲线。
2 结果与分析
2.1 区分死活菌

图1 活菌与死菌的扩增曲线图
活菌与死菌经过不同方式扩增后的结果见图1。由于金黄色葡萄球菌死亡以后,femA DNA降解缓慢,所以DNA-PCR扩增时死菌、活菌均出现峰值,如图1-A所示;而femA mRNA稳定性较差,在金黄色葡萄球菌死亡后迅速降解,所以经过RT-PCR扩增后,活菌扩增曲线出现峰值,死菌的扩增曲线未出现峰值,如图1-B所示。数据显示,经RT-PCR扩增后,活菌的Ct值为31.38,呈阳性,死菌未检出,为阴性;而另一组经 DNA-PCR扩增后,活菌的 Ct值为20.27,死菌为20.83,两者均呈阳性。由此说明,本实验所建立的RT-PCR具有区分死、活细菌的能力,能够避免死菌假阳性的出现。
2.2 特异性
将其它菌种与金黄色葡萄球菌同时抽提RNA后,经RT-PCR扩增,结果显示,除金黄色葡萄球菌的检测结果为阳性外,其它致病菌菌的检测结果均为阴性。由此可见,本实验所用的femA引物与探针具有较高的特异性,能够避免其它致病菌的干扰。

图2 特异性扩增曲线
2.3 重复性
取4组不同体积的菌液,重复检测6次,结果见表2,4组体积的平均变异系数分别为0.18、0.12、0.15、0.13,平均变异系数为 0.15,小于 0.5,由此可以看出,该方法具有良好的重复性。
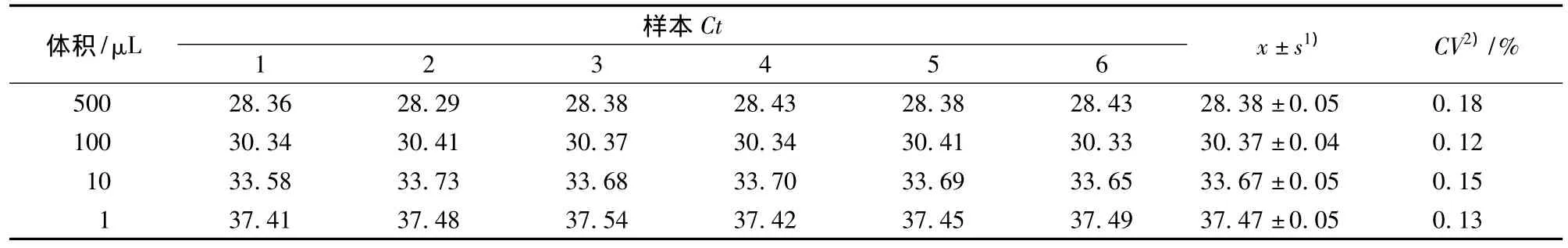
表2 RT-PCR检测结果
2.4 标准曲线
稀释后的样品经RT-PCR扩曾后,浓度在4.5×106、4.5×105、4.5×104、4.5×103、9×102的样品可形成较为理想扩增曲线,如图3-A,所以本方法的灵敏度为9×102CFU/mL。以Ct值为纵坐标,对数浓度为横坐标,绘制标准曲线,如图3-B。经对比,RT-PCR活菌定量结果与传统培养法结果吻合率为100%。
2.5 检出限
增菌3 h后,100 CFU、10 CFU、1 CFU、1/3 CFU、1/5 CFU、1/10 CFU RT-PCR扩增曲线如图4所示,100 CFU、10 CFU、1 CFU、1/3 CFU 呈阳性,其它均为阴性。因此,在纯培养时,该方法的最低检出限为1/3 CFU/3 mL。
3 讨论
3.1 阳性样本判定
本实验所建立的RT-PCR法,活菌显阳性,死菌呈阴性,即有扩增曲线出现的为金黄色葡萄球菌阳性样本,未出现扩增曲线的为金黄色葡萄球菌阴性样本。但由于细菌死亡后,DNA能够稳定存在,若消化不彻底,RT-PCR检测时仍然会出现死菌为阳性的结果,会严重影响检测结果的判定,如图5所示。因此,彻底消化抽提液中的残留DNA是区分死、活菌的关键。

图3 金黄色葡萄球菌标准曲线
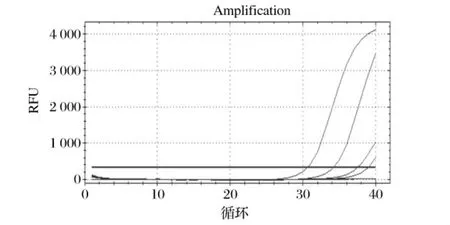
图4 RT-PCR检出限扩增曲线
已有研究表明,Ct值相差在3.1~3.6时,同一体系中,其浓度大约有10倍左右的差异。同一样本抽提液在不彻底消化时,经过RT-PCR与DNA-PCR扩增后,在Ct值上会形成一定的差异。因此,除上述判定方法外,还可以采用RT-PCR与DNA-PCR检测结果的差值(ΔCt)来判定是否为阳性样本[19]。
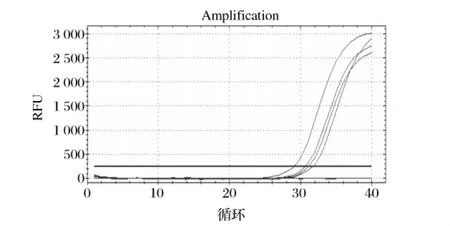
图5 未完全消化的RT-PCR扩增曲线
若同一样本RT-PCR的扩增Ct值与PCR的扩增Ct值相差大于4,则该样本为金黄色葡萄球菌阳性;若二者的Ct差值小于4,那么使用DNase I再消化1次,如果再次消化后,二者的Ct差值≥4,那么该样本为金黄色葡萄球菌阳性,反之则为金黄色葡萄球菌阴性。例如,3组样本经RT-PCR与DNA-PCR的扩增后,ΔCt分别为5.99、2.51、6.78,如表2,由于样本 1、3的ΔCt均大于4,样本2的ΔCt<4,那么可判定样本1、3为阳性;再取样本2,经二次消化后扩增,其ΔCt为0.04,如表3,由于二次消化后 ΔCt<4,那么样本2为阴性。由此,3组样本的最终判定结果为:1、3号样本为金黄色葡萄球菌阳性样本,2为金黄色葡萄球菌阴性样本,如表4所示。
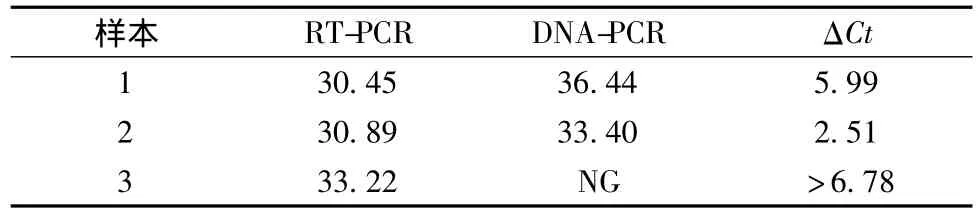
表2 RT-PCR与DNA-PCR扩增结果

表3 再次消化后的扩增结果

表43组样本判定结果
3.2 引物与探针
就大多数实时PCR法而言,引物、探针的序列和质量[20]是影响检测结果的重要因素。本实验以市售的荧光PCR检测试剂盒为标准,采用femA与gB引物、探针,对同一样本进行多次DNA-PCR检测,结果如表5所示。取前5个梯度的(107~103)3次检测Ct平均值为横纵坐标,数量级的对数值为横坐标,得到3条标准曲线,试剂盒:y=-3.356x+44.19,R2=0.998;femA:y=-3.147x+42.83,R2=0.995;gB:y=-3.362x+45.79,R2=0.987,如图7所示。若忽略实验操作误差的影响,将试剂盒的检测结果作为绝对准确定量,那么femA检测结果的R2=0.995×(1/0.998)=0.997,gB的 R2=0.987×(1/0.998)=0.989。

表3 不同的引物与探针检测结果
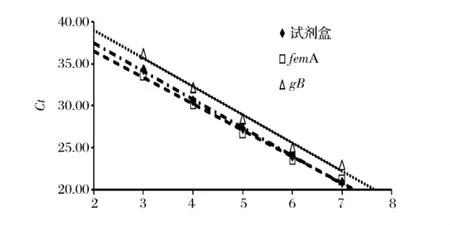
图6 试剂盒、femA、gB标准曲线
在采用RT-PCR对活菌的mRNA检测时,femA引物、探针能够区分死、活金黄色葡萄球菌,具有较高特异性及灵敏度(前文所示),而gB引物、探针几乎无法形成较完整的扩增曲线(结果未列出),难以区分死、活金黄色葡萄球菌。因此,合理的选择靶基因,筛选高效的引物、探针,是一个至关重要的过程,不同的引物与探针,对实验结果会有很大的影响[7-9,16-17]。同时,结果也说明,gB 引物、探针的准确性较差(R2<0.99),只适用于阳性样本的DNA检测,不适用于样本的准确定量,也无法用于活菌的mRNA检测;而本研究自设计的femA引物、探针在DNA定量准确性上面已经接近市售标准试剂盒水平,且在灵敏度上已优于市售试剂盒。
目前,实时PCR已经被认为是检测金黄色葡萄球菌最为高效、准确的方法[21],但由于基于DNA水平的常规PCR无法区分死、活菌,使其应用受到极大地限制。本研究以金黄色葡萄球菌femA基因为靶基因,选取其保守区域设计一对Taqman探针、引物,建立一了套基于RNA水平的PCR检测方法(RTPCR),不仅特异性强、灵敏度高,检出限可达1/3CFU/3 mL,而且能够有效的区分死、活金黄色葡萄球菌,具有较强的实际应用价值。同时,本文所采用的实时RT-PCR技术不仅能有效的区分死、活菌,而且还能够检测到处于活的非培养状态(VBNC)下的细菌,其检测结果可作为菌体活性评估的重要参照[22],是活菌检测的“金”标准[23],具有广阔的推广应用前景。
[1]Laupland KB,Gregson DB,Zygun DA,et al.Severe bloodstream infections:a population-based assessment[J].Crit Care Med,2004,32:992-997.
[2]Sharma M,Berriel-Cass D,Baran J Jr.Sternal surgical site infection following coronary artery bypass graft:prevalence,microbiology,and complications during a 42-month period[J].Infect Control Hosp Epidemiol,2004,25:468-471.
[3]Suzanne M Paule,Anna C Pasquariello,Richard B Thomson,et al.Real-time PCR can rapidly detect methicillinsusceptibleand methicillin-resistant Staphylococcus aureus directly from positive blood culture bottles[J].Microbiology and Infectious Disease,2005,124:404-407
[4]Unal S,R Masterton,H Goossens.Bacteraemia in Europeantimicrobial susceptibility data from the MYSTIC surveillance programme[J].Int J Antimicrob,2004,23(2):155-163.
[5]Ingeborg Hein,Angelika Lehner,Petra Rieck,et al.Comparison of different approaches to quantify Staphylococcus aureus cells by real-time quantitative PCR and application of this technique for examination of cheese[J].Applied and Environmental Microbiology,2001,67(7):3122-3126.
[6]Dinges M,Orwin PM,Patrick M.Schlievert exotoxins of Staphyococcus aureus[J].Clin Microbiol Rev,2000,13(1):16-34
[7]M Klotz,S Opper,K Heeg,et al.Detection of Staphylococcus aureus enterotoxins A to D by real-time fluorescence PCR assay[J].Journal of Clinical Microbiology,2003,41(10):4683-4687.
[8]A Ramesh,B P Padmapriya,A Chandrashekar.Applica-tion of a convenient DNA extraction method and multiplex PCR for the direct detection of Staphylococcus aureus and Yersinia enterocolitica in milk samples[J].Molecular and Cellular Probes,2002,16(8):307-314.
[9]Vincenzina Fusco,Grazia Marina Quero,Maria Morea.Rapid and reliable identification of Staphylococcus aureus harbouring the enterotoxin gene cluster(egc)and quantitative detection in raw milk by real time PCR[J].International Journal of Food Microbiology,2011,144(3):528-537.
[10]Keer J T,L Birch.Molecular methods for the assessment of bacterial viability[J].Journal of Microbiological Methods,2003,53(2):175-183.
[11]Corbette Roberts,Kelsi L Anderson,Paul M Dunman,et al.Characterizing the effect of the Staphylococcus aureus virulence factor regulator,SarA,on log-phase mRNA half-lives[J].Journal of Bacteriology,2006,188(7):2593-2603.
[12]Takayama K,S A Kjelleberg.The role of RNA stability during bacterial stress responses and starvation[J].Environ Microbiol,2000,2(4):355-365.
[13]Hegde SS,Shrader TE.FemABX family members are novel nonribosomal peptidyltransferases and important pathogen-specific drug targets[J].The Journal of Biological Chemistry,2001,276(10):6998-7003.
[14]Judith Hübscher1,Andrea Jansen,Brigitte Berger-Bchi,et al.Living with an imperfect cell wall:compensation of femAB inactivation in Staphylococcus aureus[J].BMC Genomics,2007,8(1):307.
[15]Thomas L C,Gidding H F,Ginn A N,et al.Development of a real-time Staphylococcus aureus and MRSA(SAM-)PCR for routine blood culture[J].Journal of Microbiological Methods,2007,68(2):296-302.
[16]Jovanka M Voyich,Michael Otto,Barun Mathema.Is panton-valentine leukocidin the major virulence determinant in community-associated methicillin-resistant Staphylococcus aureus disease?[J].The Journal of Infectious Diseases,2006,194(12):1761-1770.
[17]Regina Adriana de Oliveira Calsolari,Valéria Cataneli Pereir,Joo Pessoa Araújo Júnio.Determination of toxigenic capacity by reverse transcription polymerase chain reaction in coagulase-negative staphylococci and Staphylococcus aureus isolated from newborns in Brazil[J].Microbiology and Immunology,2011,55(6):394-407.
[18]Kelsi L Anderson,Corbette Roberts,Paul M Dunman,et al.Characterization of the Staphylococcus aureus heat shock,cold shock,stringent,and SOS responses and their effects on log-phase mRNA turnover[J].Journal of Bacteriology,2006,188(9):6739-6756.
[19]Narjol Gonzalez-Escalona,Thomas S Hammack,Mindi Russell,et al.Detection of Live salmonella sp.Cells in produce by a TaqMan-Based quantitative reverse transcriptase real-time PCR Targeting invA mRNA[J].Applied and Environmental Microbiology,2009,75(11):3714-3720.
[20]Caifu Chen,Dana A Ridzon,Adam J Broomer,et al.Real-time quantification of microRNAs by stem–loop RT– PCR[J].Nucleic Acids Research,2005,33(20):e179.
[21]Vincenzina Fusco,Grazia Marina Quero,Maria Morea.Rapid and reliable identification of Staphylococcus aureus harbouring the enterotoxin gene cluster(egc)and quantitative detection in raw milk by real time PCR[J].International Journal of Food Microbiology,2011,144(3):528-537.
[22]Matsuda K,Tsuji H,Asahara T,et al.Sensitive quantitative detection of commensal bacteria by rRNA-targeted reverse transcription-PCR [J].Appl Environ Microbiol,2007,73(1):37-39.
[23]Tania Nolan1,Rebecca E Hands2&Stephen A Bustin2.Quantification of mRNA using real-time RT-PCR [J].Nature Protocols,2006,1(3):1559-1582.
ABSTRACTThis research focuses on establishment of a way that distinguish dead Staphylococcus aureus from the viable to avoid false-positive results caused by the ordinary DNA-based polymerase chain reaction in food detection.Primer and probe are designed by ourselves.One-step reverse transcription polymerase chain reaction(RT-PCR)is applied to detect mRNA of Staphylococcus aureus femA.Results show that only living Staphylococcus aureus are positive and deaths are negative.In pure culture,the coefficient of variation is 0.15 by repeat detection,and the analytical sensitivity and detection limit of the assay are 9×102CFU/mL and 1/3 CFU/3mL respectively.Experiments have shown that the one-step reverse transcription-polymerase chain reaction assay established in this study is not only a rapid,sensitive and specific method but also can effectively distinguish the dead from the viable Staphylococcus aureus.
Key wordsStaphylococcus aureus,RT-PCR,dead and live bacteria,Ordinary PCR
Detection of Live Staphylococcus aureus Using Real-time Reverse Transcription-polymerase Chain Reaction
Zhang Chong1,2,Liu Xiang1,Chen Ji-luan2
1(National Quality Supervision and Inspection Center of processed foods(Tianjin),Tianjin 300384,China)
2(College of Food Science and Engineering in Shihezi University,Shihezi 832000,China)
硕士研究生(刘祥为通讯作者,E-mail:wenyang76@163.com)。
*国家质检总局科技项目(2010QK307),天津市质监局科技项目(10-11)
2011-12-06,改回日期:2012-02-14

