黑曲霉XZ-3S木聚糖酶Xyn ZF-1基因克隆和序列分析
付冠华,李 端,周晨妍
(新乡医学院生命科学技术系,河南省医学遗传学与分子靶向药物高校重点实验室培育基地,河南新乡 453003)
黑曲霉XZ-3S木聚糖酶Xyn ZF-1基因克隆和序列分析
付冠华,李 端,周晨妍*
(新乡医学院生命科学技术系,河南省医学遗传学与分子靶向药物高校重点实验室培育基地,河南新乡 453003)
利用RT-PCR技术从黑曲霉XZ-3S中扩增并克隆了木聚糖酶(Xyn ZF-1)基因的cDNA片段,测序结果表明,Xyn ZF-1基因核苷酸序列阅读框全长678bp,编码225个氨基酸,推测分子量为24.06ku,等电点为5.20。以XZ-3S基因组DNA为模板扩增Xyn ZF-1的DNA序列,该序列仅存在一个内含子,长度为68bp。成熟肽基因推导的氨基酸序列与其他微生物来源的木聚糖酶一级结构进行比较,同源性最高达到了89.47%。系统进化树分析该酶属于G/11族糖基水解酶。又进一步对Xyn ZF-1二级结构进行了分析,并通过SWISS-MODEL预测了该酶的三维结构。
黑曲霉,木聚糖酶,基因克隆,序列分析
Abstract:The cDNA of xylanase(Xyn ZF-1) gene was amplified from the Aspergillus niger XZ-3S total RNA by RT-PCR.The result showed that Xyn ZF-1 was 678bp in size and encoded 225 amino acids.The molecular weight and theoretical isoelectric point was 24.06ku and 5.20,respectively.The DNA of Xyn ZF-1 gene was amplified from the Aspergillus niger XZ-3S genmic DNA by PCR.The result confirmed that there was only one intron with the 68bp in length.The homology analysis of amino acid sequence shared 89.47%in base sequence with other microbial xylanase primary structure.Phylogenetic analysis showed that the enzyme belongd to glycosyl hydrolase family G/11.Besides,the secondary structure of Xyn ZF-1 was analysed,and threedimensional structure of Xyn ZF-1 was predicted by SWISS-MODEL.
Key words:Aspergillus niger;Xylanase;gene cloning;sequence analysis
木聚糖(Xylan)是植物半纤维素的主要成分,是仅次于纤维素的第二丰富的可再生资源。木聚糖结构十分复杂,它的完全降解需要多种水解酶的参与而共同完成,其中β-1,4-内切木聚糖酶(EC 3.2.1.8)能够以内切方式作用于木聚糖主链产生不同长度的木寡糖和少量的木糖,是木聚糖降解酶系中最关键的酶[1-2]。木聚糖酶被广泛地应用于造纸、食品、饲料等行业中,尤其是当今世界能源日益紧缺,如何有效利用资源越来越受到人们的关注[1,3]。根据木聚糖酶催化区域的氨基酸序列组成和疏水簇分析,可将其归为F/10和G/11两大家族,同一家族中的木聚糖酶催化区域具有较高的同源性。G/11木聚糖酶多为单结构域,相对分子质量一般在19~25ku,酶最适温度50~60℃,有一个β-胶状卷曲结构;F/10木聚糖酶则含有较多结构域,不仅有催化结构域,还存在纤维素结合结构域(Cellulose Binding Domain:CBD),相对分子质量一般大于30ku,酶最适温度60~80℃,有一个(αβ)8桶状结构[4-6]。Bernier等首次从Bacillus subtillis PAP II 5中分离得到木聚糖酶基因(Xyn)以来,Maconukul等[7-9]先后克隆并分析了来自细菌、放线菌、霉菌、酵母菌和藻类等300多种不同生物来源的木聚糖酶基因,其中上百种基因在不同宿主中进行了表达。张茂等[10]从黑曲霉GIM3.452中克隆得到木聚糖酶XynA的成熟肽编码序列,并将其片段插入真核表达载体,在脂质体介导下成功将重组表达载体转入PK15细胞,最终实现了异源基因分泌表达。黑曲霉XZ-3S是本实验室保藏的一株高产木聚糖酶菌株。本研究旨在克隆Xyn ZF-1基因,并对该基因序列及推导的氨基酸序列进行详尽的分析,为进一步研究该基因的生物学功能和工程菌的构建奠定理论基础。
1 材料与方法
1.1 材料与仪器
黑曲霉(Aspergillus niger)XZ-3S、质粒转化受体菌大肠杆菌(Escherichia coli)JM109 新乡医学院生命科学技术系酶与发酵工程实验室保存;TA克隆载体pUCm-T质粒、RNA分离TRIzol试剂、饱和酚、氨苄青霉素(Amp) Sangon公司;TaKaRa RNA PCR试剂盒(AMV)Ver.3.0、T4 DNA连接酶、Taq DNA聚合酶、蛋白酶K、IPTG、X-galTaKaRa生物工程(大连)有限公司;UNIQ-10柱式DNA胶回收试剂盒 生工生物工程(上海)有限公司;DNA MarkerMBI公司;蛋白胨、酵母粉、琼脂糖 BBI公司;溴化乙锭 Amresco公司;其他主要生化试剂 为进口产品或国产分析纯;P1、P2、P3引物 由北京鼎国昌盛生物技术有限公司合成。
L-Broth(LB) 蛋白胨10g/L,NaCl 10g/L,酵母粉5g/L,pH7.4;用于培养含Ampr质粒的E.coli时,添加Amp浓度为100μg/mL;LB固体培养基,添加20g/L的琼脂粉;黑曲霉液体发酵培养基 蛋白胨10g/L,酵母提取物5g/L,葡萄糖10g/L,200mL/L的玉米芯提取液[11],自然pH。发酵条件:28℃,110r/min,培养36h,收集菌体用于XZ-3S总RNA,总基因组DNA提取。
梯度PCR扩增仪C1000 Bio-Rad Laboratory;稳压稳流电泳仪(DYY-2) 北京市六一仪器厂;Touching 956数码相机凝胶成像系统 上海天呈科技有限公司;恒温振荡器(HZQ-F160A) 上海一恒科学仪器有限公司;低温恒温槽(DC-0506) 上海比朗仪器有限公司;高速冷冻离心机(Neofuge 23R) 上海力申科学仪器有限公司。
1.2 实验方法
1.2.1 PCR引物设计、合成 通过对GenBank中真菌木聚糖酶相关基因的比较,以起始密码子和终子密码子附近相似度较高序列为参照,设计了以下引物:
上游引物
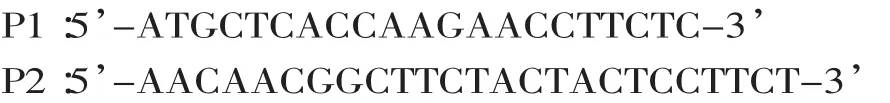
下游引物

1.2.2 黑曲霉总RNA和基因组DNA的提取 黑曲霉培养液经抽滤、无菌水洗涤菌体数次,采用TRIzol试剂一步法提取总RNA,具体操作参照TRIzol使用说明书。通过测定A260/A280,检测RNA提取的纯度。黑曲霉基因组DNA的提取参照文献[12]。
1.2.3 目的基因的扩增
1.2.3.1 RT-PCR扩增cDNA片段 在反应体系中以总RNA为模板,具体反应体系为:MgCl22μL,10×RT Buffer 1μL,RNase Free dH2O 3.75μL,dNTP Mixture 1μL, RNase Inhibitor0.25μL, AMV Reverse Transcriptase 0.5μL,Oligo dT-Adaptor Primer 0.5μL,总RNA 1μL,共10μL;反应条件为:50℃ 30min,99℃5min,5℃ 5min。具体反应液配制及反应条件参见TaKaRa RNA PCR Kit(AMV)Ver.3.0试剂盒说明书。
在PCR反应体系中,以cDNA第一条链为模板,具体反应体系为:5×PCR Buffer 10μL,灭菌蒸馏水28.75μL,TaKaRa Ex Taq® HS 0.25μL,5’端引物P1 0.5μL,3’端引物M13 0.5μL,逆转录产物 10μL,共50μL;反应条件为:94℃ 2min;94℃ 30s,52℃ 30s,72℃ 1min,35个循环;72℃延伸10min。扩增产物经1.0%的琼脂糖凝胶电泳鉴定。
1.2.3.2 PCR扩增基因组DNA片段 以基因组DNA为模板进行扩增,其反应体系组成为:10×PCR Buffer 2.5μL,dNTP 1μL,无菌水19μL,Taq酶0.5μL,5’端引物P1 0.5μL,3’端引物P3 0.5μL,DNA 1μL,共25μL;反应条件为:95℃ 10min;94℃ 30s,52℃ 45s,72℃1min,35个循环;72℃延伸10min。扩增完毕,取样用1.0%的琼脂糖凝胶电泳鉴定。
1.2.4 回收产物验证
1.2.4.1 总RNA扩增产物验证 以总RNA胶回收产物为模板,分别以P1/M13、P1/P3、P2/P3为引物,PCR扩增相应DNA片段。反应体系:DNA 1μL,10×PCR Buffer 2.5μL,dNTP 1μL,上游引物0.5μL,下游引物0.5μL,Taq酶0.5μL,无菌水19μL,共25μL;反应条件为:94℃ 2min;94℃ 30s,52℃ 30s,72℃ 1min,35个循环;72℃延伸10min。扩增完毕,分别取样用1.0%的琼脂糖凝胶验证扩增结果。
1.2.4.2 基因组DNA扩增产物验证 以基因组DNA胶回收产物为模板,分别以P1/P3、P2/P3为引物,PCR扩增相应DNA片段。PCR反应体系:DNA 1μL,10×PCR Buffer 2.5μL,dNTP 1μL,上游引物0.5μL,下游引物0.5μL,Taq酶0.5μL,无菌水19μL,共25μL;反应条件为:95℃ 8min;94℃ 30s,52℃ 45s,72℃ 1min,35个循环;72℃延伸10min。扩增完毕,分别取样用1.0%的琼脂糖凝胶验证扩增结果。
1.2.5 连接反应 连接体系:胶回收产物7μL,pUCm-T质粒1μL,10×T4 DNA Ligase Buffer1μL,T4 DNA Ligase 1μL,共10μL;连接条件:16℃过夜。
1.2.6 重组质粒的筛选及鉴定 各取5μL连接产物转化100μL感受态细胞JM109,然后涂在含Amp、IPTG、X-gal的LB琼脂平板上,37℃培养过夜。随机挑取白色克隆提取质粒,同时以蓝色质粒为对照。以白色重组质粒为模板做PCR(参照1.2.3),扩增、电泳回收目的片段,然后以胶回收产物为模板做PCR验证(参照1.2.4)。
1.2.7 序列测定及序列分析 将新鲜菌液送金唯智生物科技(北京)有限公司进行目的片段的双向测序,测序结果利用Vector NTI 9.0软件进行序列拼接;序列的同源性比较用DNAMAN5.0软件处理,并将翻译后的氨基酸序列递交至http://swissmodel.expasy.org/tools/中进行Xyn ZF-1的氨基酸序列分析、二级结构预测及三维结构的同源建模。
2 结果与讨论
2.1 木聚糖酶Xyn ZF-1 cDNA片段的扩增与克隆
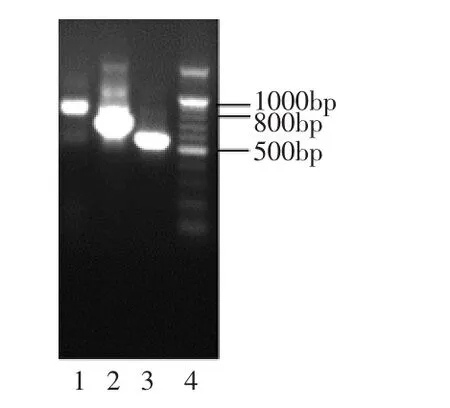
图1 Xyn ZF-1 cDNA的凝胶电泳Fig.1 Analysis of Xyn-ZF-1 cDNA fragment amplified by PCR
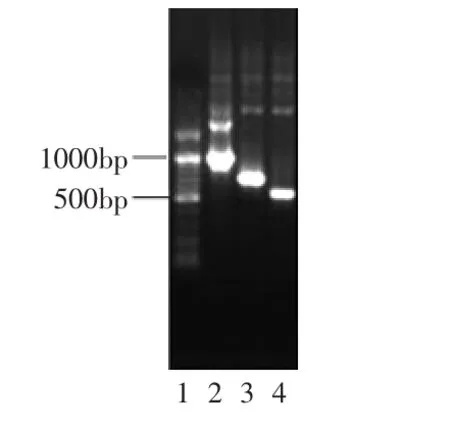
图2 pUCm-T-Xyn ZF-1阳性克隆的PCR验证Fig.2 Identification of the recombinant plasmid by PCR
根据引物设计时参照Genbank中基因的大小和PCR扩增结果(图1)割胶回收800bp左右的条带(图1第1泳道),利用T-A克隆策略将该cDNA片段直接克隆到pUCm-T vector中,连接转化E.coli JM109,蓝白斑筛选,挑取重组子,经PCR鉴定,证明所插入片段与扩增片段长度相同(图2),送样测定核苷酸序列。
2.2 木聚糖酶Xyn ZF-1 DNA片段的扩增克隆
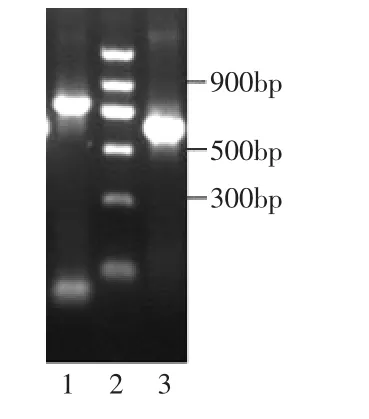
图3 Xyn ZF-1 DNA的凝胶电泳Fig.3 Analysis of Xyn-ZF-1 DNA fragment amplified by PCR
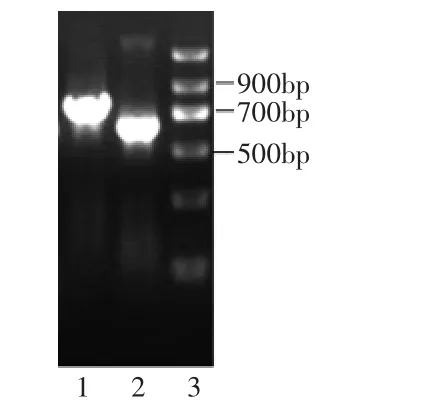
图4 pUCm-T-Xyn ZF-1阳性克隆的PCR验证Fig.4 Identification of the recombinant plasmid by PCR
根据目的基因的大小和PCR扩增结果(图3)割胶回收800bp左右的条带(图3第1泳道),利用T-A克隆策略将该cDNA片段直接克隆到pUCm-T vector中,连接转化E.coli JM109,蓝白斑筛选,挑取重组子,经PCR鉴定,证明所插入片段与扩增片段长度相同(图4),送样测定核苷酸序列。
2.3 木聚糖酶Xyn ZF-1 cDNA片段的序列分析

图5 黑曲霉木聚糖酶Xyn ZF-1 cDNA序列和推导的氨基酸序列Fig.5 Partial cDNA and deduced amino acid sequence of Xyn ZF-1 from Aspergillus niger
测序结果表明,RT-PCR产物全长确实为Xyn ZF-1 cDNA(图5),该cDNA包含Xyn ZF-1信号肽编码区,成熟肽编码区,3’非编码区和Poly(A)等核苷酸序列。该序列全长785bp(不包括Poly(A)),其中蛋白质编码区678bp,编码225个氨基酸,3’非编码区107bp。根据cDNA序列推导出该木聚糖酶的氨基酸序列,共225个氨基酸,推测分子量为24.06ku,等电点为5.20,分子式C1065H1577N283O349S4。
2.4 木聚糖酶Xyn ZF-1 DNA序列分析

图6 木聚糖酶Xyn ZF-1 DNA序列Fig.6 Xylanase Xyn ZF-1-encoding DNA
Xyn ZF-1序列包括起始位点、内含子和外显子等核苷酸序列,全长746bp。该DNA序列与对应的cDNA进行序列对比分析,由图6可知,在Xyn ZF-1 DNA编码区仅存在一个内含子,长度为68bp。
2.5 黑曲霉木聚糖酶Xyn ZF-1与其他木聚糖酶一级结构的同源性比较
将黑曲霉木聚糖酶Xyn ZF-1成熟肽完整的氨基酸序列与Genbank中其他微生物来源的木聚糖酶一级结构进行同源性比较。结果表明,它与G/11族的木聚糖酶具有较高的同源性。图7表明,Xyn ZF-1与Aspergillus usamii(Genbank登 录 号 :AEJ87263)、Chaetomium thermophilum(Genbank登录号:CAD48750)、Holomastigotoides mirabile(Genbank登录号:BAH56520)、Penicillium citrinum(Genbank登录号:BAE71133)4种微生物中的木聚糖酶的同源性比对结果分别为89.47%、60.77%、56.94%、64.11%。可见推导的木聚糖酶氨基酸序列与Aspergillus usamii序列相似性最高,达到了89.47%。
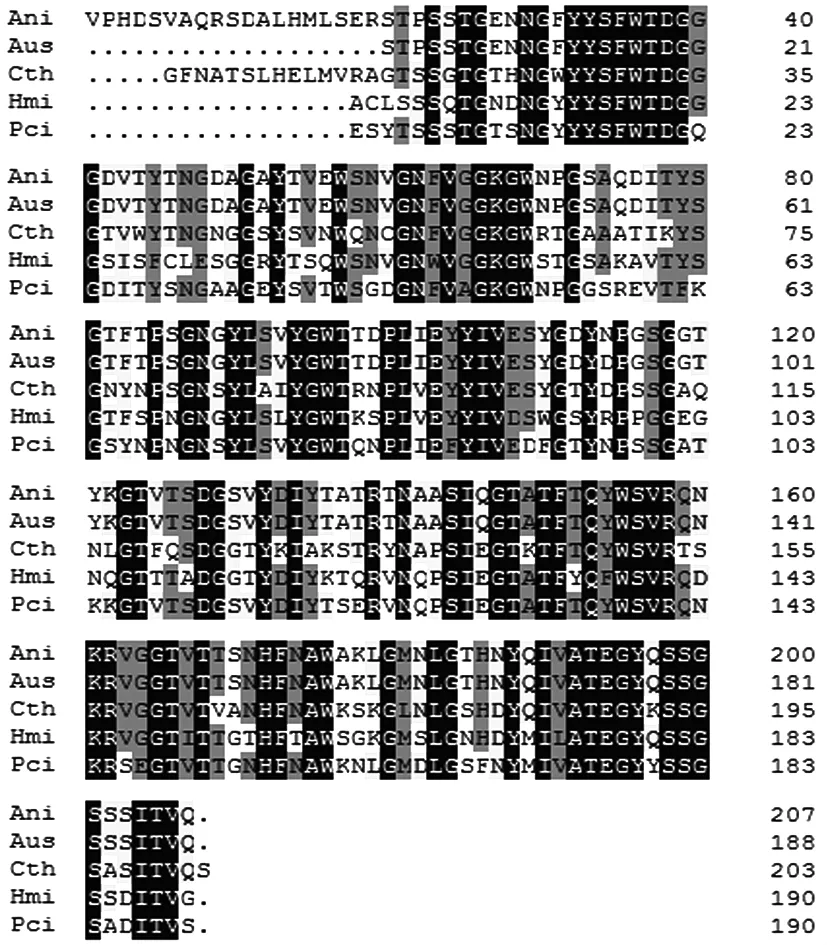
图7 微生物来源的木聚糖酶序列的多重比较Fig.7 Comparison of Xyn ZF-1to other microbial xylanase
2.6 木聚糖酶Xyn ZF-1的系统进化树分析
将Xyn ZF-1成熟肽完整的氨基酸序列与从Genbank中随机选取的13种木聚糖酶氨基酸序列进行系统进化树分析(图8),结果表明,它属于G/11族糖基水解酶。
2.7 木聚糖酶XynZF-1二级结构分析及三维结构建模

图8 木聚糖酶一级结构的系统进化树分析Fig.8 Phylogenetic tree of xylanase gene based on amino acid sequnce annalysis
通过CFSSP(Chou&;FasmanSecondary Structure Prediction Server)方法分析得知,Xyn ZF-1二级结构主要由α-螺旋,β-折叠和少量转角构成,所占比例分别为32.4%、46.7%和16.0%。三维结构建模显示Xyn ZF-1具有G/11木聚糖酶典型的β-胶状卷曲结构(图9)。

图9 通过SWISS-MODEL预测的Xyn ZF-1三维模型Fig.9 Three-dimensional structure of Xyn ZF-1 predicted by SWISS-MODEL
3 结论
3.1 以黑曲霉XZ-3S总RNA为模板,采用RT-PCR等技术分别扩增和克隆了木聚糖酶(Xyn ZF-1)cDNA的相关片段,经拼接获得Xyn ZF-1 cDNA的完整序列,该cDNA全长785bp包含了起始密码子、信号肽编码区、成熟肽编码区、终止密码子和3’端非编码区等核苷酸序列。
3.2 以黑曲霉XZ-3S基因组DNA为模板,采用PCR技术扩增和克隆了Xyn ZF-1的DNA相关片段,经拼接得到了Xyn ZF-1 DNA序列。该DNA全长746bp,包含起始密码子、1个内含子和2个外显子以及终止密码子等核苷酸序列。内含子长度为68bp,位于Gln93第3个和Asp94第1个碱基之间。
3.3 一级结构和系统进化树分析可知,该酶属于G/11族,且与G/11族中的木聚糖酶有较高的同源性。二级结构预测该酶由许多α-螺旋、β-折叠和少量转角构成。三维结构建模显示Xyn ZF-1具有G/11木聚糖酶典型的β-胶状卷曲结构。
[1]余元莉,李秀婷,宋焕禄,等.微生物木聚糖酶的研究进展[J].中国酿造,2009(2):1-4.
[2]Fan Zhang,Pengjun Shi,Yinguo Bai,et al.An acid and highly thermostable xylanase from Phialophora sp.G5[J].Appl Microbiol Biotechnol,2011,89,1851-1858.
[3]万红鬼,武振军,蔡恒,等.微生物发酵产木聚糖酶研究进展[J].中国生物工程杂志,2010,30(2):141-146.
[4]Sibtain Ahmed,Saba Riaz,Amer Jamil.Molecular cloning of fungal xylanase:an overview[J].Appl Microbiol Biotechnol,2009,84:19-35.
[5]刘亮伟,杨海玉,胡瑜,等.F/10木聚糖酶研究进展[J].食品与生物技术学报,2009,28(6):727-732.
[6]Motoki Wakiyama,Koji Yoshihara,Scchio Hayashi,et al.An extracellular endo-1,4-β -xylanase from Aspergillus japonic properties,and characterization of the encoding gene[J].Journal of Bioengineering,2010,109(3):227-229.
[7]Waconukul Rattiya,Patthra Pason,Khin Lay Kyu,et al.Cloning,Sequencing,and Expression of the Gene Encoding a Multidomain Endo-β-1,4-Xylanasefrom Paenibacillus curdlanolyticus B-6,and Characterization of the Recombinant Enzyme[J].J Microbiol Biotechnol,2009,19(3):277-285.
[8]周晨妍,符丹丹,邬敏辰.宇佐美曲霉木聚糖酶基因的克隆和序列分析[J].食品与发酵工业,2005,31(10):29-32.
[9]Junqing Wang, Huimin Zhang, Minchen Wu, et al.Cloning and sequence analysis of a novel xylanase gene,Auxyn10A,from Aspergillus usamii[J].Biotechnol Lett,2011,33:1029-1038.
[10]张茂,刘德武,林纯,等.黑曲霉木聚糖酶基因xynA的克隆及真核表达载体的构建[J].华中农业大学学报,2010,29(5):588-592.
[11]姚笛,马萍,王颖,等.响应面法优化玉米芯中木聚糖的提取工艺[J].食品科学,2011,32(8):111-115.
[12]Wendland J,Lengeler KB,Kothe E.An instant preparation method for mucleic acids of filamentous fungi[J].Fungal Genet Newslett,1996,43:54-55.
Cloning and sequence analysis of the gene encoding xylanase Xyn ZF-1 from Aspergillus niger XZ-3S
FU Guan-hua,LI Duan,ZHOU Chen-yan*
(Key Laboratory of Medical Genetics and Molecular Targeting Drugs,Xinxiang Medical College,Department of Life Science and Technology,Xinxiang 453003,China)
Q789
A
1002-0306(2012)16-0187-05
2012-01-11 *通讯联系人
付冠华(1986-),男,硕士研究生,研究方向:微生物酶工程。
河南省教育厅自然研究计划项目资助(2011A180026);河南省科技厅科技攻关项目(112102210299);新乡医学院研究生科研创新支持计划项目(YJSCX201104Z)。

