玉米丝黑穗病菌基因组SSR的信息分析
王士臻,张作刚,李新凤,高俊明,王建明
(山西农业大学 农学院,山西 太谷030801)
玉米丝黑穗病是玉米生产上危害极大的一种世界性土传真菌病害。在中国北方春玉米产区,其病害发生较为严重,每年因丝黑穗病危害减产达30万吨左右[1~3]。玉米丝黑穗病是由孢堆黑粉菌(Sporisorium reilianum)引起的,孢堆黑粉菌为二倍体,由23条染色体组成。随着其基因组测序的完成,产生的大量遗传信息对日后玉米丝黑穗的遗传分析起到十分重要的作用。特别是对开展孢堆黑粉菌的SSR信息分析及其引物开发和利用提供了重要的基础资料和遗传信息。
SSR(简单重复序列,simple sequence repeat)广泛存在于动植物基因组中,主要分布在其基因组的编码区和非编码区[4,5],由1~6个核苷酸重复序列组成[5],是一种以PCR反应为基础的分子标记技术,是在DNA复制过程中由于DNA聚合酶的滑 动 或 者 不 均 等 充 足 造 成 的[6,7]。 与 AFLP、RAPD等分子标记技术相比,SSR标记具有以下优点:(1)SSR在整个基因组中数量多,涵盖区域广;(2)包含的信息量高;(3)其遗传方式符合孟德尔遗传规律,呈共显性。因而SSR分子标记在基因定位、遗传多样性检测、品种鉴定以及分子辅助育种等方面得到了广泛的应用[8~14]。同时SSR标记还可以作为联系其遗传图谱、序列信息以及其最终的表现型差异三者的重要纽带[15]。
玉米丝黑穗病菌作为玉米的重要病原菌,其基因组测序的完成有助于对其基因组SSR序列进行信息分析。利用SSR分子标记技术对玉米丝黑穗病菌全基因组数据进行分析、开发都具有重要的理论和应用价值。因此对孢堆黑粉菌的基因组进行SSR分析,了解其基因组SSR位点的数量以及分布情况,利用对该基因组的分析数据开发SSR引物,无论对玉米丝黑穗病菌还是其相近的感染高粱的高粱丝黑穗病菌的遗传分析均有重要意义。
本文以生物信息学的方法在丝黑穗病菌的全基因序列中搜索SSR,对其基因组SSR进行分析,了解其分布规律及特点。这将为丝黑穗病菌基因组SSR标记的开发以及进一步研究提供理论基础和科学依据。
1 材料和方法
1.1 材料
试验于与2012年4~9月在山西农业大学植物病理实验室进行,以FASTA格式在NCBI(http://www.ncbi.nlm.nih.gov/)数据库中,下载孢堆黑粉菌(Sporisorium reilianum)的23条染色体基因组序列。
1.2 孢堆黑粉菌全基因组SSR的检索
根据SSR的特征,利用SSRHunter搜索孢堆黑粉菌基因组SSR序列。该程序类似一个在线的SSR 位 点 搜 索 程 序 SSRIT (http://www.gramene.org/db/markers/ssrtool)对孢堆黑粉菌进行基因组搜索,进行SSR搜索之前要对一些参数进行设置,包括:构成重复单元的核苷酸数以及相应的最少重复次数。
本试验对以上参数设定如下的设置:单核苷酸的重复次数≥16,二核苷酸重复次数≥8,三核苷酸重复次数≥5,四核苷酸重复次数≥4,五核苷酸重复次数≥3,六核苷酸重复次数≥3。对搜索出的SSR频率与长度进行统计与分析[1]。
2 结果与分析
2.1 孢堆黑粉菌SSR的重复类型和频率
孢堆黑粉菌的基因组信息共包含23条染色体(以2012年5月NCBI公布的数据为准),由于其所包含的信息量巨大,选取其中3条染色体即第1、2、3条染色体对其进行分析。由表1可以看出,3条染色体中共搜索到888条SSR,平均每条染色体出现296条SSR,其总长度为3.512kb,占这3条基因组染色体碱基数的0.068%,平均每6.50 kb中就分布有一个长度大于15bp的SSR序列。
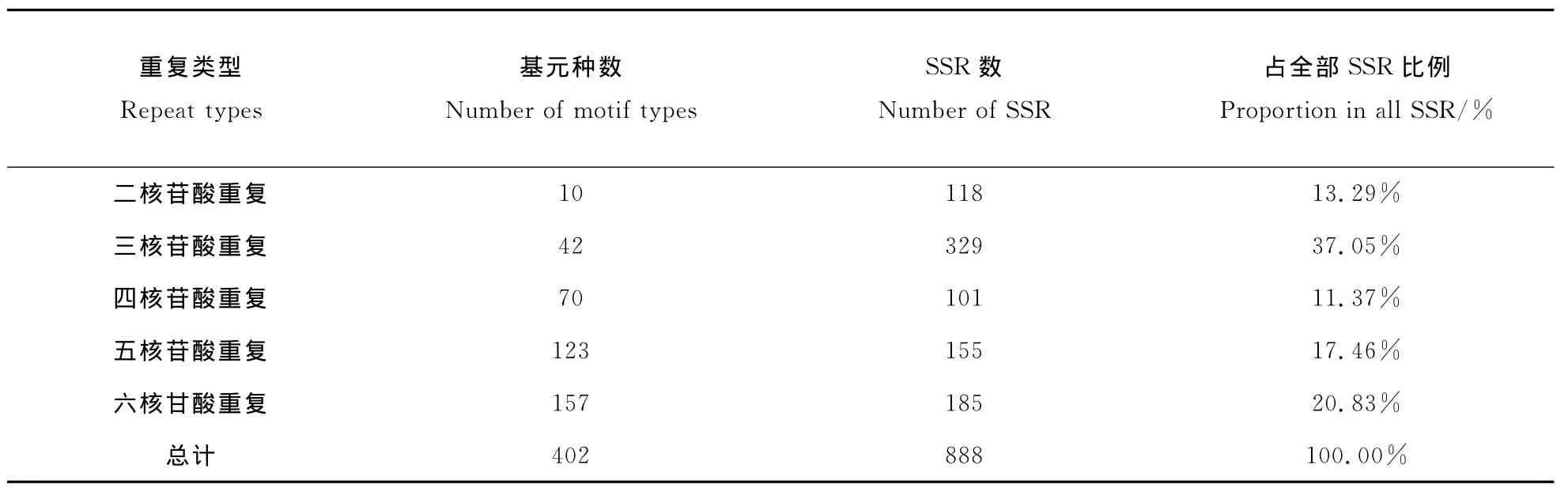
表1 孢堆黑粉菌SSR信息Table 1 The information of SSR in Sporisorium reilianum
2.2 孢堆黑粉菌SSR重复基元的分布特点
在孢堆黑粉菌基因组的3条染色体中,共发现888个SSR序列,其中三碱基重复序列数量最多,达到329个,六核甘酸重复为185个,五核苷酸重复为155个,这3种SSR共计达669个,占SSR总数的75.34%;四核甘酸重复最少为101个(表2)。从表2还可以看出,在孢堆黑粉菌基因组的各类SSR序列中,重复次数最多的是三核甘酸重复序列,其重复次多达23次,其次是二核苷酸重复,其重复次数为17次;而且,在各SSR序列中长度最大的SSR序列是三核甘酸重复序列,其长度达69bp。此结果与粗糙脉孢菌基因组中SSR序列的组成和分布情况甚为相似,在粗糙脉孢菌基因组中,重复次数最多的是三核甘酸碱基重复,达到145次;其次是二核苷酸碱基重复,重复127次;而且其长度最大的SSR序列也是三核苷酸重复序列,其长度为435bp。在二核苷酸重复中,SSR序列数量最多的为CT类,达33个,约占二核苷酸重复SSR序列总数的27.95%。其次是GA类,达30个,TC、AG类均为19个,GC类与AT类最少,仅有1个。

表2 孢堆黑粉菌基因组中SSR组成和频率分布Table 2 Distribution and frequency of SSR in genomic sequence of Sporisorium reilianum
3 结论与讨论
与其他的真核生物相比,真菌的基因组较为紧凑,因此,各种不同的重复序列的含量在基因组中一般占10%[3],其中简单重复序列的数量还不到整个基因组的1%[16]。在二核苷酸重复中,SSR序列重复最多的为CT,最少的为AT,这与粗糙脉孢菌基因组中SSR的基序组成相似[2],但与酵母基因组中SSR的组成与分布有显著差异,在酵母中,数量最多的二核苷酸碱基重复序列为AT[17]。通过分析可知在孢堆黑粉菌的3条染色体基因组中,三碱基重复序列数量最多,六核甘酸重复次之,四核甘酸重复最少,这与以往报道的稻瘟病菌(Magnaporthe grisea)有非常大的差异,在稻瘟病菌中最丰富的SSR是单碱基重复,其次是三碱基重复[2]。稻瘟病菌以及9种真菌基因组SSR位点中存在大量的单、二、三核苷酸短重复基元[2,18];本试验与陆景标等[19]对高粱的研究结果相似,这些物种的基因组SSR位点均是以四、五、六核苷酸长重复序列为主。因此,在不同真菌中其SSR组成存在着很大的差异。本研究以及以前对其它生物的研究结果均表明,大量的SSR基序分布在基因间区和蛋白质区。而与其他重复核苷酸系列相比,SSR数量更多分布更广,并且其覆盖性也很好。因此,将SSR作为分子标记应用,是具有其显著优点的[20]。本研究结果表明,SSR序列在这3条染色体中占有0.068%。可利用的SSR最小长度为15bp,这意味着平均每6.50kb中就分布有一个长度大于15bp的SSR序列。这一结果表明,虽然不同的真菌基因组中SSR的分布和密度有很大差异,但是玉米丝黑穗病菌基因组中仍然存在着丰富的SSR序列,可为SSR分子标记的利用提供相关信息。本文选取了孢堆黑粉菌基因组中的3条染色体进行分析,虽然未能对所有的遗传信息进行系统的分析,但是通过对这3条染色体的SSR分析,也能够在一定程度上反映出全部基因组SSR的信息,这为今后进一步分析SSR奠定了基础。
[1]杨素丽,明军,刘春,等.基于EST信息的百合SSR标记的建立[J].园艺学报,2008,35(7):1069-1074.
[2]李云成,李进斌.粗糙脉胞菌基因组中的微卫星序列的组成和分布[J].中国农业科学,2004,37(6):851-858.
[3]Barve M P,Haware M P,Sainani M N,et al.Potential of microsatellites to distinguish four races of Fusarium oxysporumf.sp.ciceri prevalent in India[J].Theoretical and applied genetics,2001,102:138-147.
[4]Gupta P K,Balyan H S,Sharma P C,et a1.Mierosatellite inplants:a new class of molecular markers[J].Current science,1996,70:45-54.
[5]Tautz D,Renz M.Simple sequence repeats are ubiquitous repetitive components of eukaryotic genomes[J].Nucleic acids research,1984,12:4127-4138.
[6]Wang Z,Weber J L,Zhong G,et al.Survey of plant short tandem DNA repeats[J].Theoretical and applied genetics,1994,88:1-6.
[7]Richards R I,Sutherland G R.Dynamic mutations:a new class of mutation causing human disease[J].Cell,1992,70(5):709-712.
[8]王爱德,李天忠,许雪峰,等.苹果品种的SSR分析[J].园艺学报,2005,32(5):875-877.
[9]高华,樊红科,万怡震,等.苹果栽培品种的SSR鉴定及遗传多样性分析[J].西北农业学报,2011,20(2):153-158.
[10]Zhang C Y,Chen X S,He T M,et al.Genetic structure of Malus sieversii population from Xinjiang,China,revealed by SSR markers[J].Journal of genetics and genomics,2007,34(10):947-955.
[11]程保山,万志兵,洪德林.35个粳稻品种SSR指纹图谱的构建及遗传相似性分析[J].南京农业大学学报,2007,30(3):1-8.
[12]Levinson G,Gutman G A.Slipped-strand mispairing:a major mechanism for DNA sequence evolution[J].Molecular biology and evolution,1987,4(3):203-221.
[13]Goul~ao L,Oliveira C M.Molecular characterization of cultivars of apple(Malus×domestica Borkh.)using microsatellite(SSR and ISSR)markers[J].Euphytica,2001,122:81-89.
[14]Gianfranceschi L,Seglias N,Tarchini R,et al.Simple sequence repeats for the genetic analysis of apple[J].Theoretical and applied genetics,1998,96:1069-1076.
[15]Di Gaspero G,Cipriani G,Adam-Blondon A F.Linkage maps of grapevine displaying the chromosomal locations of 420microsatellite markels and 82markers for R-gene candidates[J].Theor appl genet,2007,114:1249-1263.
[16]李成云,李进斌,周晓罡等.稻瘟病菌基因组中微卫星序列的频率与分布[J].中国水稻科学,2004,18(3):269-273.
[17]Benton G.Tandem repdats finder:A program to analyze DNA sequence[J].Nucleic acids research,1999,27:573-580.
[18]Haraoglu H,Lee C M Y,Meyer W.Survey of simple sequence repeats in complete fungal genomes[J].Molecular biology and evolution,2005,22(3):639-649.
[19]陆景标,李杰勤,卢杰,等.高粱非编码区SSR引物设计以及e-PCR的验证[J].种子,2010,29(9):1-6,12.
[20]李成云,罗韩喜,李进斌,等.稻瘟病菌无毒基因的分子标记[J].中国农业科学,2000,33(3):49-53.

