基于“树枝状”信号放大的电化学DNA生物传感器研究
朱进清,王 青,羊小海,王柯敏,杨丽娟,丁 静,周 冰
(湖南大学化学生物传感与计量学国家重点实验室,化学化工学院,生物纳米与分子工程湖南省重点实验室,湖南长沙410082)
0 引言
DNA分子的检测对于分子生物学、遗传学、临床诊断、基因治疗、药物的研制与开发、环境监测等方面的研究均具有十分重要的意义[1~2]。因此,发展高灵敏的检测DNA分子的方法成为研究的热点之一。其中,生物传感器技术由于检测快速灵敏、选择性好、稳定性高及较低的成本[3~4],成为检测DNA分子的重要手段。
近年来,随着科学研究的不断深入,有越来越多的问题需要高灵敏的检测技术来解答。如:对于疾病早期阶段的少量碱基突变DNA序列的检测。因此,如何提高检测灵敏度成为生物传感器技术发展的方向之一。由于纳米颗粒制备简单、易于修饰和保存,并具有良好的生物相容性与独特的光电性质,因此基于纳米颗粒的放大技术已被广泛应用于生化检测中。如Ding等[5]报道了一种采用阳极溶出CdS量子点的方法来放大检测DNA序列;Wang等[6]将二茂铁的衍生物巯基二茂铁与信号探针一起修饰到纳米金颗粒上,实现对目标物的放大检测。除此之外,有些研究工作基于纳米颗粒构建了二次放大的方法实现了对目标物的检测。如利用DNA功能化的纳米金颗粒与目标物、捕获探针形成“三明治”结构,再通过银增强[7]或金增长[8~9]的方法使信号进一步放大。Zhang等[10]将辣根过氧化物酶标记在纳米金颗粒上,通过酶催化放大与纳米颗粒放大相结合的方法实现了对目标物的二次放大检测。
该文发展了一种基于DNA功能化纳米金颗粒 “树枝”状结构增强信号的方法实现了目标DNA的检测。该方法中利用两种DNA功能化的纳米金颗粒进行两次“三明治”杂交,从而在电极表面自组装成“树枝”状结构,可灵敏地检测DNA。相比只利用纳米金颗粒放大的方法而言,其检测限降低了4倍。同时该传感器对含有错配位点的序列亦具有较好的选择性。该文构建的方法有望推广至小分子或者蛋白质等目标物的检测。
1 实验部分
1.1 主要试剂及仪器
氯金酸(HAuCl4·4H2O,分析纯,国药集团化学试剂有限公司),二水合柠檬酸三钠(Na3C6H5O7·2H2O,上海试剂一厂)用于纳米金颗粒的制备。 巯基己醇(97%,C6H14OS,MCH,Simga公司)用来封闭传感芯片,以减少表面的非特异性吸附。钌胺(RuHex,Sigma公司)用作电活性分子。实验中所用的DNA均购自大连宝生物有限公司,其碱基序列见表1。所有DNA均溶于10 mmol/L PBS 缓冲溶液(pH7.4,0.2 mol/L NaCl)中。如没有特别说明,其它试剂均为分析纯,实验用水均为 18.2 MΩ·cm 超纯水。

表1 所用DNA的序列Tab.1 Sequence of synthesized oligonucleotides used in the work
电化学工作站(CHI 660C型,上海辰华仪器公司)用于电化学测定,UV-1601型紫外-可见分光光度计(日本Shimadzu公司)用于紫外可见吸收光谱的测定。SB2200型超声清洗器(上海必能信超声有限公司)用于金电极和器皿的清洗。台式离心机(Beckman公司)用于纳米金颗粒的洗涤与纯化。
1.2 纳米金颗粒的制备与修饰
参照文献制备[11]和修饰[12]纳米金颗粒,并用紫外-可见分光光度计进行表征。
1.3 金电极的表面处理与修饰
在新处理好的金电极表面滴加20 μL 0.1 μmol/L巯基DNA1,室温下反应2 h之后用10 mmol/L PBS缓冲液反复冲洗。然后将20 μL 1 mmol/L MCH滴在金电极表面,室温反应1 h后用10 mmol/L PBS缓冲液反复冲洗。最后将电极表面用氮气吹干,备用。
1.4 杂交反应
首先,在巯基DNA1修饰的电极表面滴加10 μL不同浓度的cDNA与10 μL巯基DNA2修饰的纳米金颗粒(DNA2-AuNPs)的混合液,在 37°C下孵育2 h。之后用10 mmol/L PBS反复清洗,以除去未反应的目标cDNA和DNA2-AuNPs。然后将50 nmol/L cDNA与巯基DNA1修饰的纳米金颗粒(DNA1-AuNPs)于 37℃下孵育 30 min,8 000 r/min的速度离心30 min,重复洗涤两次后得到cDNA/DNA1-AuNPs。 取 20 μL cDNA/DNA1-AuNPs滴到电极表面,37℃下杂交 2 h。用10 mmol/L PBS反复清洗,以除去未反应的cDNA/DNA1-AuNPs;最后,电极表面用高纯氮气吹干,将20 μL 50 μmol/L的钌胺溶液滴加到电极表面,静电吸附平衡10 min后进行电化学测定。
1.5 电化学测定
所有电化学检测均在CHI 660C型电化学工作站(上海辰华仪器公司,上海)上进行,采用三电极体系:金电极为工作电极、铂电极为对电极、KCl饱和的饱和甘汞电极(SCE)为参比电极。计时电量测定前将电极浸入测量底液中允许钌胺嵌入富集10 min,测量底液是:50 μmol/L钌胺溶液 (10 mmol/L Tris-HCl,pH7.4), 测量前通入高纯氮气15 min以除去溶解氧;交流阻抗测定底液 是 : 5.0 mmol/L Fe(CN)63-/4-(10 mmol/L PBS,pH7.3,0.1 mol/L KCl)。
2 结果与讨论
2.1 实验原理
实验原理如图1所示。首先,将巯基DNA1修饰到金电极上作为捕获探针,当目标DNA存在时就会形成巯基DNA1、目标DNA与DNA2-AuNPs的“三明治”杂交结构。由于每个金纳米颗粒上修饰有多条DNA序列,可以通过静电吸附作用结合大量的钌胺分子,从而实现对目标DNA的第一次放大检测。然后,将cDNA/DNA1-AuNPs滴加到金电极表面,就会形成DNA2-AuNPs、cDNA与DNA1-AuNPs的第二个 “三明治”杂交结构。电极表面形成的这种DNA功能化纳米金颗粒“树枝”状结构能结合更多的钌胺分子,从而实现对目标DNA的二次放大检测。
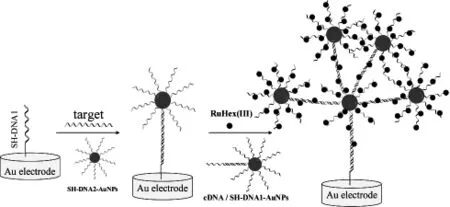
图1 功能化纳米金颗粒“树枝”状结构增强信号的方法检测DNA的原理图Fig.1 Schematic illustration of the dendritic amplification electrochemical biosensor
2.2 巯基DNA修饰的纳米金颗粒的表征
采用紫外-可见分光光度计表征了纳米金颗粒修饰巯基DNA前后的吸收光谱变化。由柠檬酸三钠还原法制备的纳米金颗粒,其最大吸收波长为518 nm,可推测其粒径约为13 nm[11]。经巯基DNA修饰后,溶液的最大吸收波长红移至524 nm,说明巯基DNA已修饰到纳米金颗粒上。根据文献[13]可知纳米金颗粒浓度约为2.5 nmol/L。
2.3 可行性考察
分别采用计时电量法和交流阻抗法对该方法的可行性进行了考察,结果如图2、3所示。图2为利用计时电量方法考察的结果。其中曲线a为巯基己醇修饰电极的计时电量曲线,修饰SHDNA1电极的计时电量响应如曲线b,根据文献[14]可计算电极表面修饰密度约为5.2×1012分子/cm2。加入0.1 nmol/L cDNA和DNA2-AuNPs后,计时电量响应由曲线b变化为曲线c,响应信号有明显增强,这是由于固定在电极表面的SHDNA2-AuNPs可嵌入的钌胺分子数量增多所致。当加入cDNA/DNA1-AuNPs后,计时电量响应由曲线c化为曲线d,响应信号进一步增强,说明在电极表面形成了“树枝”状结构之后,其嵌入的钌胺分子数量进一步增加,从而引起电化学响应信号进一步增强。
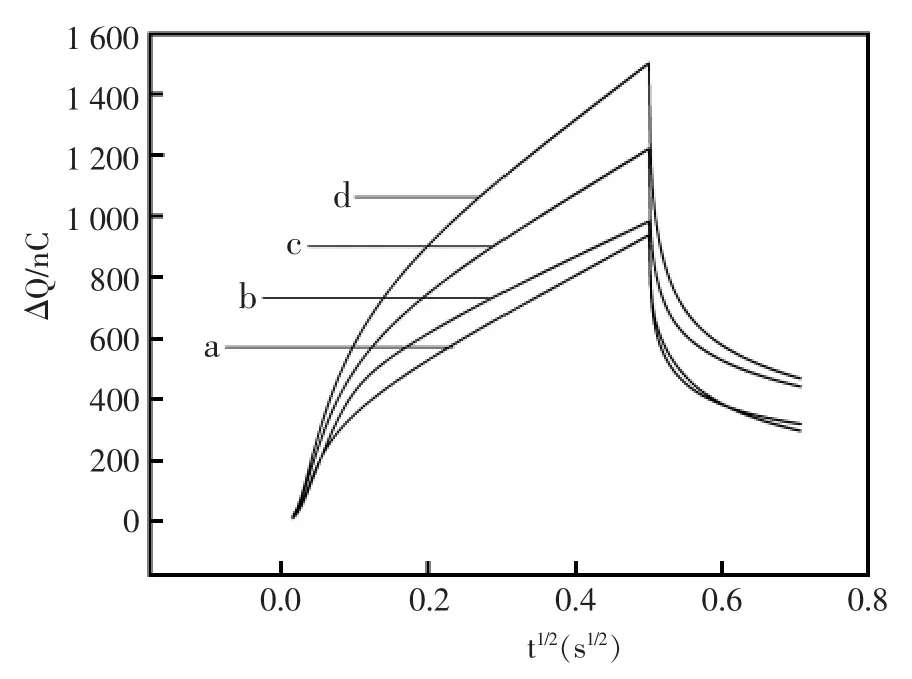
图2 传感器检测DNA可行性考察的计时电量曲线图(a)MCH 修饰的电极;(b)SH-DNA1/MCH 修饰的电极;(c)加入0.1 nmol/L cDNA,基于纳米金颗粒放大后的计时电量曲线;(d)基于功能化纳米金颗粒“树枝”状结构放大后的计时电量曲线Fig.2 Chronocoulometry responses of the electrochemical biosensor(a)MCH modified gold electrode;(b)SH-DNA1 and MCH modified gold electrode;(c)after treatment with Au nanoparticles amplification;(d)after treatment with dendritic amplification.The concentration of cDNA was 0.1 mol/L
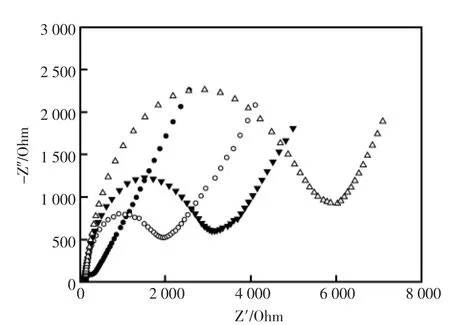
图3 法拉第阻抗图谱(a)裸电极;(b)巯基 DNA1 和 MCH 修饰的金电极;(c)加入0.1 nmol/L cDNA,基于纳米金颗粒放大后的交流阻抗曲线;(d)基于功能化纳米金颗粒“树枝”状结构放大后的交流阻抗曲线Fig.3 Faradic impedance spectra(a)of the bare gold electrode;(b)of SH-DNA1 and MCH modified gold electrode;(c)after treatment with Au nanoparticles amplification;(d)after treatment with dendritic amplification.The concentration of cDNA was 0.1 mol/L
然后采用阻抗法进行表征,结果如图3所示。其中a为裸金电极的法拉第阻抗图谱,其阻抗R很小(R=138.1 Ω)。当金电极表面修饰了巯基探针并用MCH封闭后,其阻抗R增大至1 588 Ω,这是由于电极表面形成了一层致密的分子层所致。当加入0.1 nmol/L cDNA与DNA2-AuNPs后,其阻抗值增加到2 559 Ω(如曲线c所示),这是由于电极表面形成“三明治”结构,电极表面引入更多的负电荷,进而对检测系统中的Fe(CN)63-/4-产生静电排斥作用所致;当加入cDNA/DNA1-AuNPs后,其阻抗值进一步增大至4 660 Ω(如曲线d所示),这是由于在电极表面形成了“树枝”状结构,电极表面引入的负电荷进一步增加,导致阻抗值也随之增加。
2.4 目标DNA的定量检测
利用计时电量法检测了一系列不同浓度的目标DNA分子,如图4A所示。可以看出随着目标DNA浓度的提高,计时电量信号逐步增强。以表面吸附钌胺发生还原反应所需的法拉第电量增量ΔQ对目标DNA浓度作图,得到两者之间的关系如图4B所示。 其中 ΔQ=Q1-Q2,Q1为目标DNA存在时的电量值,Q2为cDNA不存在时的计时电量值。图4B插图 为对应的电量浓度对数曲线。用纳米金颗粒放大的方法对目标DNA的线性回归方程为ΔQ=30.64Log(c)+398.15,相关系数为0.96,检测下限为0.52 pmol/L。“树枝”状结构放大的线性回归方程为ΔQ=42.0 Log(c)+556.15,相关系数为0.98,检测下限为0.13 pmol/L,使检测限降低了4倍。

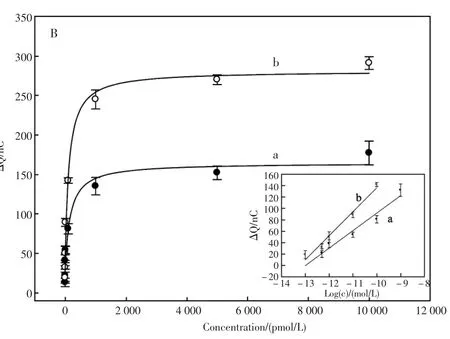
图4 不同浓度目标cDNA对应的的计时电量图(A).不同浓度cDNA对应的计时电量响应曲线,从上到下cDNA的浓度依次是10 000、5 000、1 000、100、10、1、0.5、0.1、0 pmol/L;(B)工作曲线。插图 为电量增量与cDNA浓度对数值的关系曲线,其中,a是用纳米金颗粒放大的响应曲线;b是用“树枝”状结构放大的响应曲线Fig.4 Chronocoulometry curves for electrodes with capture probe hybridized with target DNA at a series of concentrations(A).The concentration of cDNA was 0,0.1 pmol/L,0.5 pmol/L,1 pmol/L,10 pmol/L,0.1 nmol/L,1 nmol/L and 10 nmol/L,respectively.(B)Calibration plots of target DNA detection:(a)after treatment with Au nanoparticles amplification;(b).after treatment with dendritic amplification
2.5 选择性的考察
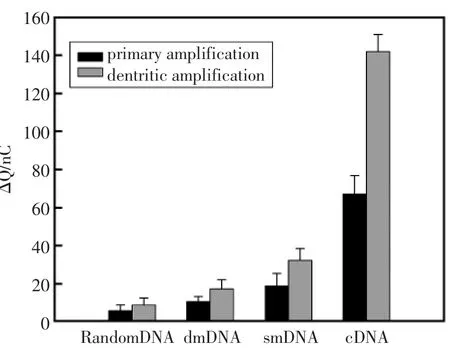
图5 选择性Fig.5 Selectivity
考察了该方法对含错配位点序列的选择性。结果如图5所示。其中目标DNA浓度均为0.1 nmol/L。可以看出,采用“树枝”状结构增强信号的电化学传感器检测时,smDNA所引起的计时电量响应信号是cDNA的23%,dmDNA所引起的计时电量响应信号是cDNA的12%,随机DNA所引起的计时电量响应信号是cDNA的6%。而采用纳米金颗粒增强信号的电化学传感器检测时,smDNA、dmDNA和随机DNA所引起的计时电量响应信号分别是cDNA的28%、16%和8%。因此,相对仅用纳米金颗粒增强信号的电化学传感器检测而言,“树枝”状结构增强信号的电化学传感器的碱基错配识别能力略有提高。
3 小结
构建了一种基于核酸适体和金纳米颗粒“树枝”状信号放大检测核酸分子杂交的电化学生物传感器。该传感器对DNA分子检测下限达到了0.13 pmol/L,对目标物的检测具有高灵敏度、高特异性的特点,有望拓展到小分子或其他目标物的检测中。
[1]Riccardi C D S,Kranz C,Kowalik J,et al.Hairpin DNA Switch for Ultrasensitive Spectrophotometric Detection of DNA Hybridization Based on Gold Nanoparticles and Enzyme Signal Amplification[J].Anal.Chem.,2007,80(1):237~245.
[2]Li J S,Chu X,Liu Y L,et al.Identification of RNA recognition elements in the Saccharomyces cerevisiae transcriptome[J].Nucleic.Acids.Res.,2005,33(19):168~176.
[3]Cho K,Lee Y,Lee C H,et al.Selective Aggregation Mechanism of Unmodified Gold Nanoparticles in Detection of Single Nucleotide Polymorphism[J].J.Phys.Chem.C.,2008,112(23):8 629~8 633.
[4]Algar W R,Krull U J.Toward a multiplexed solid-phase nucleic acid hybridization assay using quantum dots as donors in fluorescence resonance energy transfer[J].Anal.Chem.,2009,81(10):4 113~4 120.
[5]Ding C F,Zhang Q,Lin J M,et al.Electrochemical detection of DNA hybridization based on bio-bar code method[J].Biosens.Bioelectron.,2009,24(10):3 140~3 143.
[6]Wang J,Zhu X,Tu Q,et al.Capture of p53 by Electrodes Modified with Consensus DNA Duplexes and Amplified Voltammetric Detection Using Ferrocene-Capped Gold Nanoparticle/Streptavidin Conjugates[J].Anal.Chem.,2008,80(3):769~774.
[7]Xue X,Xu W,Wang F,et al.Multiplex Single-Nucleotide Polymorphism Typing by Nanoparticle-Coupled DNA-Templated Reactions[J].J.Am.Chem.Soc.,2009,131(33):11 668~11 669.
[8]Fan A,Lau C,Lu J.Hydroxylamine-amplified gold nanoparticles for the naked eye and chemiluminescent detection of sequence-specific DNA with notable potential for single-nucleotide polymorphism discrimination[J].Analyst.,2009,134(3):497~503.
[9]Strerath M,Marx A.Genotypingfrom genomic DNA to genotype in a single tube[J].Angewandte Chemie International Edition,2005,44(48):7 842~7 849.
[10]Zhang Y Y,Tang Z W,Wang J,et al.Hairpin DNA Switch for Ultrasensitive Spectrophotometric Detection of DNA Hybridization Based on Gold Nanoparticles and Enzyme Signal Amplification[J].Anal.Chem.,2010,82(15):6 440~6 446.
[11]Frens G.Controlled Nucleation for the Regulation of the Particle Size in Monodisperse Gold Suspensions[J].Nature Physics Science,1973,241:20~22.
[12]He L,Musick M D,Nicewarner S R,et al.Colloidal Au-Enhanced Surface Plasmon Resonance for Ultrasensitive Detection of DNA Hybridization[J].J.Am.Chem.Soc.,2000,122(38):9 071~9 077.
[13]Jin R,Wu G,Li Z,et al.What Controls the Melting Properties of DNA-Linked Gold Nanoparticle Assemblies[J].J.Am.Chem.Soc.,2003,125(6):1 643~1 654.
[14]Steel A B,Herne T M,Tarlov M J.Electrochemical quantitation of DNA immobilized on gold[J].Anal.Chem.,1998,70(22):4 670~4 677.

