粤蓝链霉菌中丝氨酸/苏氨酸蛋白激酶基因gra-orf7的功能初探
李 超, 胡建伟, 邓名荣, 朱红惠
(1.塔里木大学 塔里木盆地生物资源保护利用重点实验室, 新疆 阿拉尔 843300; 2.广东省微生物研究所,广东省菌种保藏与应用重点实验室,广东省微生物应用新技术公共实验室,广东省华南应用微生物重点实验室-省部共建国家重点实验室培育基地, 广东 广州 510070)
0 引 言
丝氨酸/苏氨酸蛋白激酶STPK(serine-threonine protein kinase)广泛存在于真核生物中,在ATP等磷酸供体的存在下,能特异地使底物蛋白中丝氨酸或苏氨酸残基上的羟基磷酸化而激活底物蛋白,参与细胞生长、形态发育及生理分化等多种重要的生命过程.目前研究显示STPK不仅存在于真核生物中,在原核生物中也同样存在,如黄色粘球菌(Myxococcusxanthus)[1],天蓝链霉菌(StreptomycescoelicolorA3(2))[2],无乳链球菌(Streptococcusagalactiae)[3]等.
链霉菌不仅能产生丰富的活性次级代谢产物,同时还具有复杂的发育周期.对链霉菌的代表种天蓝链霉菌A3(2)的基因组测序结果表明,该菌存在约44个可能的STPK[4],这提示STPK可能对该菌的形态发育、次级代谢调控等具有重要的影响.AfsK是发现最早、研究最深入的一个链霉菌STPK[5],它在天蓝链霉菌A3(2)中与次级代谢全局性调控蛋白AfsR组成双组份系统,调控放线紫红素(actinorhodin)、十一烷基灵菌红素(undecylprodigiosin)等的生物合成[6].进一步的研究发现,PkaG、AfsL等STPK也能使AfsR磷酸化[7],提示这些STPK都可能参与了次级代谢的调控.在灰色链霉菌(S.griseus)中存在与afsK高度同源的基因afsK2,该基因参与气生菌丝的形成,但对链霉素(streptomycin)的生物合成没有影响[8],这说明高度同源的STPK在不同链霉菌中可参与不同的生命活动,也从侧面反映了STPK在链霉菌的生命活动过程中扮演了多样化的角色.
榴菌素GRA(Granaticin)与放线紫红素同属苯并异色满醌BIQ(Benzoisochromanequinone)类抗生素,均具有抗菌、抗肿瘤等生物活性.早期从紫红链霉菌(S.violaceoruberTü22)中克隆测序了榴菌素的生物合成基因簇,发现其左翼存在一个可能编码STPK的基因gra-orf7,该基因与天蓝链霉菌A3(2)中的PkaA存在较高同源性[9],与此同时,在多个完成基因组测序的链霉菌,如委内瑞拉链霉菌(S.venezuelae)ATCC 10712、灰黄链霉菌(S.flavogriseus)ATCC 33331、灰色链霉菌灰色亚种(S.griseussubsp.griseus)NBRC 13350、阿维链霉菌(S.avermitilis)MA-4680、冰城链霉菌(S.bingchenggensis)BCW-1、疮痂病链霉菌(S.scabiei)87.22等中也发现同源基因.同源基因的普遍存在,提示其可能参与了重要的生命活动过程,但其具体生理功能如何,目前仍不明确.
近年,塔里木盆地生物资源保护利用重点实验室从热带原始森林土壤中分离获得一株链霉菌新种——粤蓝链霉菌(S.vietnamensis)[10],该菌能够产生榴菌素[11].前期,我们测定了该菌榴菌素生物合成基因簇的全长序列[12],并首次成功建立了榴菌素产生菌(粤蓝链霉菌)的有效遗传转化体系[13].本研究在此基础上,通过体内基因敲除,对gra-orf7这一可能的STPK基因的生理功能进行初步研究.
1 材料与方法
1.1 材料
1.1.1 菌株、质粒和引物
本研究所涉及的菌株,质粒和引物见表1.
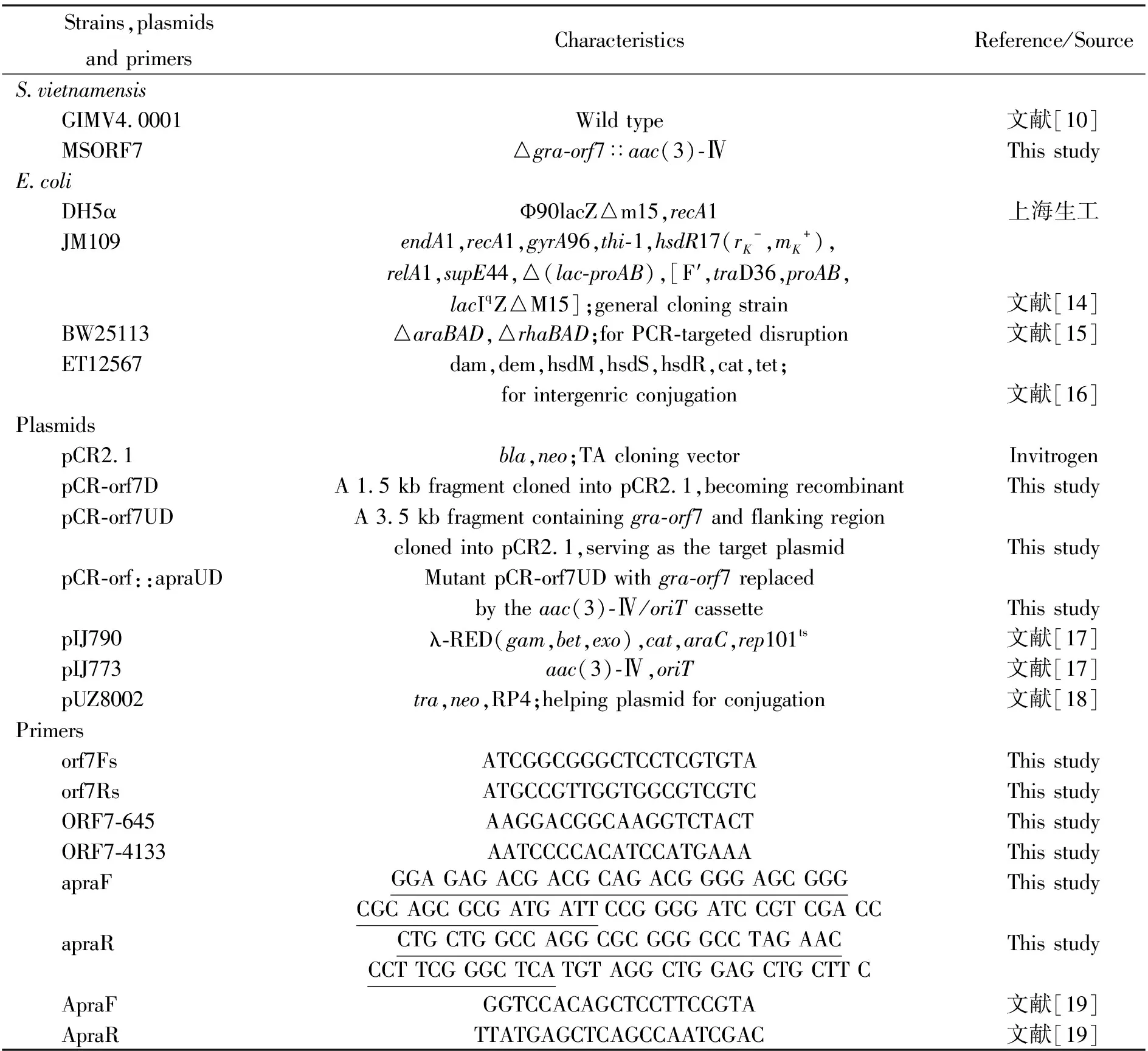
表1 参试菌株、质粒和引物
1.1.2 主要试剂与培养基
UNIQ-10柱式DNA胶回收试剂盒购自上海生工生物工程技术服务有限公司;pCR2.1 TA克隆试剂盒购自上海英骏生物技术有限公司;IPTG、X-Gal、限制性核酸内切酶等均购自宝生物工程(大连)有限公司;萘啶酮酸(Nalidixic acid,Nali)、阿普拉霉素(Apramycin,Apra)为Sigma产品;氨苄霉素(Ampicillin,Amp)、卡那霉素(Kanamycin,Kan)、氯霉素(Chloramphenicol,Cml)等均为常规产品.LB、SOB、YEME、YD、NA、2×YT、YMS、高氏一号等培养基均按标准配方配制.
1.2 gra-orf7基因下游未知序列的克隆与测序
前期GRA基因簇测序中已获得gra-orf7完整基因及下游部分序列,但已知的下游序列长度并不足以在基因敲除中作为同源臂.为了获得更长的下游序列,本研究对gra-orf7基因及其上、下游已知序列进行了同源性分析,通过BLAST比对,在同源性较高区域设计引物,以期获得下游未知序列.PCR扩增产物经纯化TA克隆到pCR2.1载体上,酶切验证后送上海英骏公司测序.
1.3 粤蓝链霉菌突变株的构建
大肠杆菌和链霉菌相关的分子生物学操作分别按照文献[20]和文献[21]进行.突变株MSORF7的构建采用PCR-targeting方法[18]进行,稍有改动如下:根据gra-orf7基因上下游已知序列设计引物ORF7-645和ORF7-4133,利用保真性能高的LA Taq酶从粤蓝链霉菌基因组中扩增出gra-orf7基因及其上下游共约3.5 kb的片段,TA克隆至载体pCR2.1,得到质粒pCR2.1-orf7UD,并将此重组质粒作为突变母体质粒转入E.coliBW25113/pIJ790感受态细胞中,获得转化子E.coliBW25113/pIJ790/pCR2.1-orf7UD.
利用引物对apraF/apraR(下划线部分与gra-orf7基因上下游序列同源)扩增pIJ773质粒上包含Apra抗性基因和转移起始序列约1.4 kb片段(aac(3)-IV/oriTcassette),PCR扩增产物纯化后用电击法转化至E.coliBW25113/pIJ790/pCR2.1-orf7UD感受态细胞.在30℃条件下,λ/RED重组系统将重组质粒pCR2.1-orf7UD上的orf7基因替换成aac(3)-IV/oriTcassette,从而获得pCR2.1-orf7::ApraUD.将质粒pCR2.1-orf7::ApraUD转化至E.coliET12567/pUZ8002,得到转化子E.coliET12567/pUZ8002/pCR-orf7::ApraUD,将其与粤蓝链霉菌孢子进行接合转移,在YD平板上筛选接合子.待YD平板上刚长出孢子时,用Apra和萘啶酮酸抗生素覆盖平板,直到平板上长出接合子.随机挑取若干接合子,点接转移至含有Apra和Kan以及只含Apra的两种NA平板上,挑取单一抗性(KanS、ApraR)的接合子,即双交换接合子.
1.4 粤蓝链霉菌突变株基因型验证及表型分析
1.4.1 突变株的基因型验证
得到双交换接合子后,提取其基因组DNA,利用PCR方法进行基因型验证.PCR采用4条引物ApraF、ApraR、ORF7-645和ORF7-4133等组合成的4对引物,即:ApraF/ApraR、ORF7-645/ORF7-4133、ApraF/ ORF7-645、ApraR/ ORF7-4133等进行验证.
1.4.2 突变株菌落形态分析
在多种培养基,如YD、YEME、高氏一号、MM、P2等中,观察突变株的菌落形态.取粤蓝链霉菌突变株与野生株甘油种各50μL,分别培养于5 mL含Apra的YEME液体培养中,30 ℃过夜培养.吸取1 mL菌液于1.5 mL无菌离心管,5 000 rpm离心,弃去上清,1 mL无菌水重悬菌体.用接种环进行划线.30 ℃培养3 d,观察菌落形态.
1.4.3 突变株孢子微观形态分析
扫描电镜观察粤蓝链霉菌孢子微观形态特征.YD培养基上培养7天的粤蓝链霉菌采用戊二醛固定法处理.将链霉菌孢子用3%戊二醛固定5 h,依次用PBS,30%乙醇,50%乙醇,70%乙醇,90%乙醇,无水乙醇处理,再用叔丁醇置换,冷冻干燥后,进行电镜观察.
1.4.4 突变株产色(榴菌素)能力分析
利用比色法对突变株的产色能力进行测定.挑取粤蓝链霉菌突变株与野生株单菌落,分别培养于5 mL的YEME液体培养基中,30 ℃过夜培养.吸取1 mL菌液于1.5 mL无菌离心管,5 000 rpm离心,弃去上清,用1 mL无菌水重悬菌体,采用比色法进行相对定量.以相同的接种量接于30 mL YEME液体培养基的250 mL三角瓶中,30 ℃,170 rpm条件下培养,每个处理3次重复.产色后,每小时取一次样.从培养液中吸取300μL菌液,12 000 rpm离心1 min,取上清,测定580 nm波长处的光吸收值.以时间(h)为横坐标,光吸收值(OD580)为纵坐标,绘制色素生产曲线.
2 结果与分析
2.1 gra-orf7基因下游序列的克隆与测序
对gra-orf7基因的BLAST分析表明,在多个完成基因组测序的链霉菌中存在同源基因,进一步的分析发现其上下游序列在委内瑞拉链霉菌、灰色链霉菌灰色亚种、冰城链霉菌等中也存在较高同源性,且该区域中相邻基因间相对位置相同(图1).将委内瑞拉链霉菌的SVEN-3648基因与灰色链霉菌灰色亚种、冰城链霉菌等的相应同源基因进行比较,发现该基因序列高度保守,多个区域序列同源率达100%.因此,在该3种链霉菌的序列一致区域设计了上游引物orf7Fs,与根据已知序列设计的下游引物orf7Rs组成引物对,以粤蓝链霉菌野生型菌株基因组为模板,进行PCR扩增(图1).电泳显示,扩增产物约为1.5 kb,与预期大小相近(图2a).将TA克隆后得到的重组质粒pCR-orf7D进行酶切验证(图2b),并送英骏公司测序.对测序结果分析表明,该扩增产物是粤蓝链霉菌榴菌素基因簇侧翼基因gra-orf7的下游区域.

图1 gra-orf7基因上、下游同源性比较及引物设计

图2 粤蓝链霉菌gra-orf7基因下游区域的PCR扩增及其重组质粒酶切鉴定
2.2 突变株的构建及基因型验证
Escherichia-Streptomyce属间接合转移操作之后大约12 h,在YD培养基平板上覆盖Apra和Nali.30 ℃培养2 d后,从中挑取的表现单一抗性表型(KanS、ApraR)接合子数目约占10%.双交换接合子经扩繁后,提取其基因组DNA,进行PCR验证.琼脂凝胶电泳检测显示708 bp(ApraF/ApraR)、3 511 bp(ORF7-645/ORF7-4 133)、1 861 bp(ApraF/ORF7-645)、2 358 bp(ApraR/ORF7-4 133)条带(图3),其中,M,分子量标准DS5000;1~4分别为ApraF/ApraR、ORF7-645/ORF7-4133、ApraF/ORF7-645、ApraR/ORF7-4133的PCR产物,结果符合预期,提示突变株构建成功.

图3 粤蓝链霉菌gra-orf7突变株基因型的PCR验证
2.3 突变株表型分析
2.3.1 突变株菌落形态分析
突变株在YD、YEME、高氏一号、MM、P2等培养基平板上,仍然能够产蓝色素,即榴菌素,且具有产孢的能力,菌落形态与野生株无显著差异(图4).
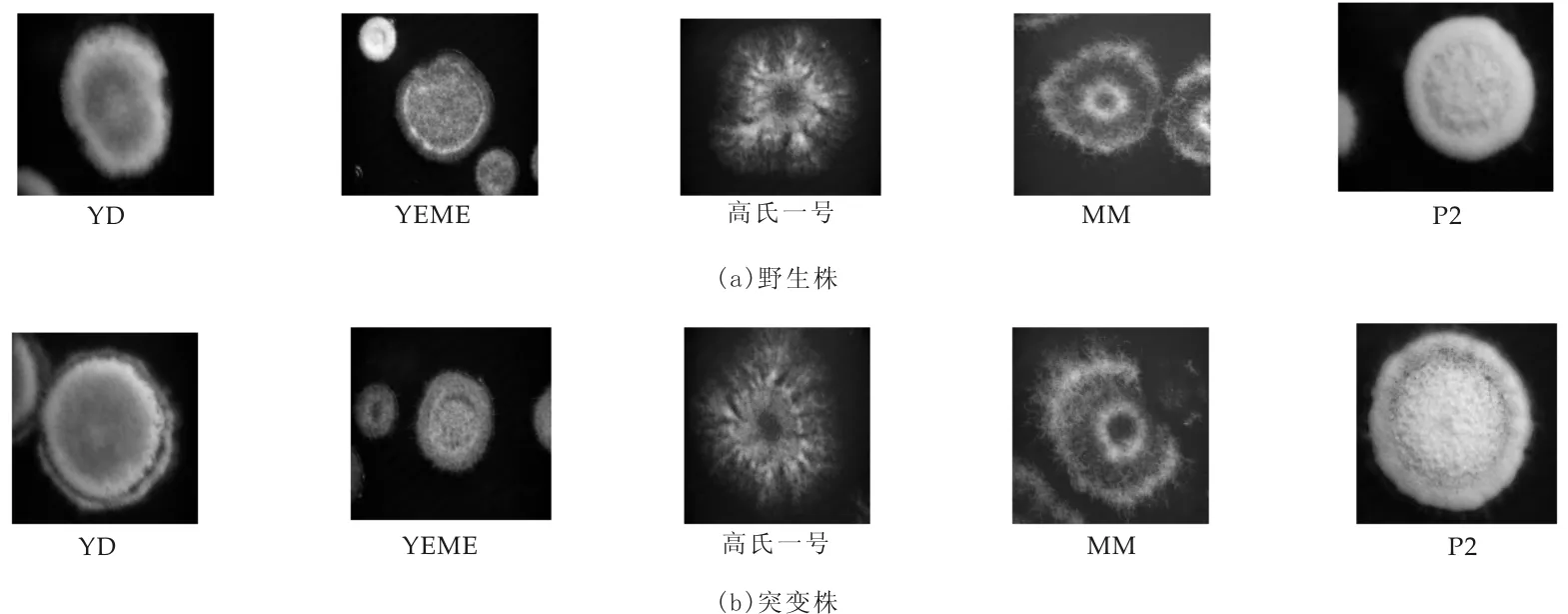
图4 粤蓝链霉菌突变株MSORF7与野生株菌落形态比较
2.3.2 突变株孢子微观形态分析
通过扫描电镜的观察,其孢子形态与野生株也基本一致(图5).

图5 扫描电镜对粤蓝链霉菌突变株与野生株孢子形态观察
2.3.3 突变株产色(榴菌素)能力分析
在YEME液体培养基中,对照野生株在培养到11 h时开始产生色素(榴菌素),而突变株约在12 h时才开始产色,但突变株在培养约19 h时,色素产量与野生株基本相同(图6).在多次独立实验中,均可观察到突变株较野生株延迟产色的现象,说明这一变化特征较为稳定.
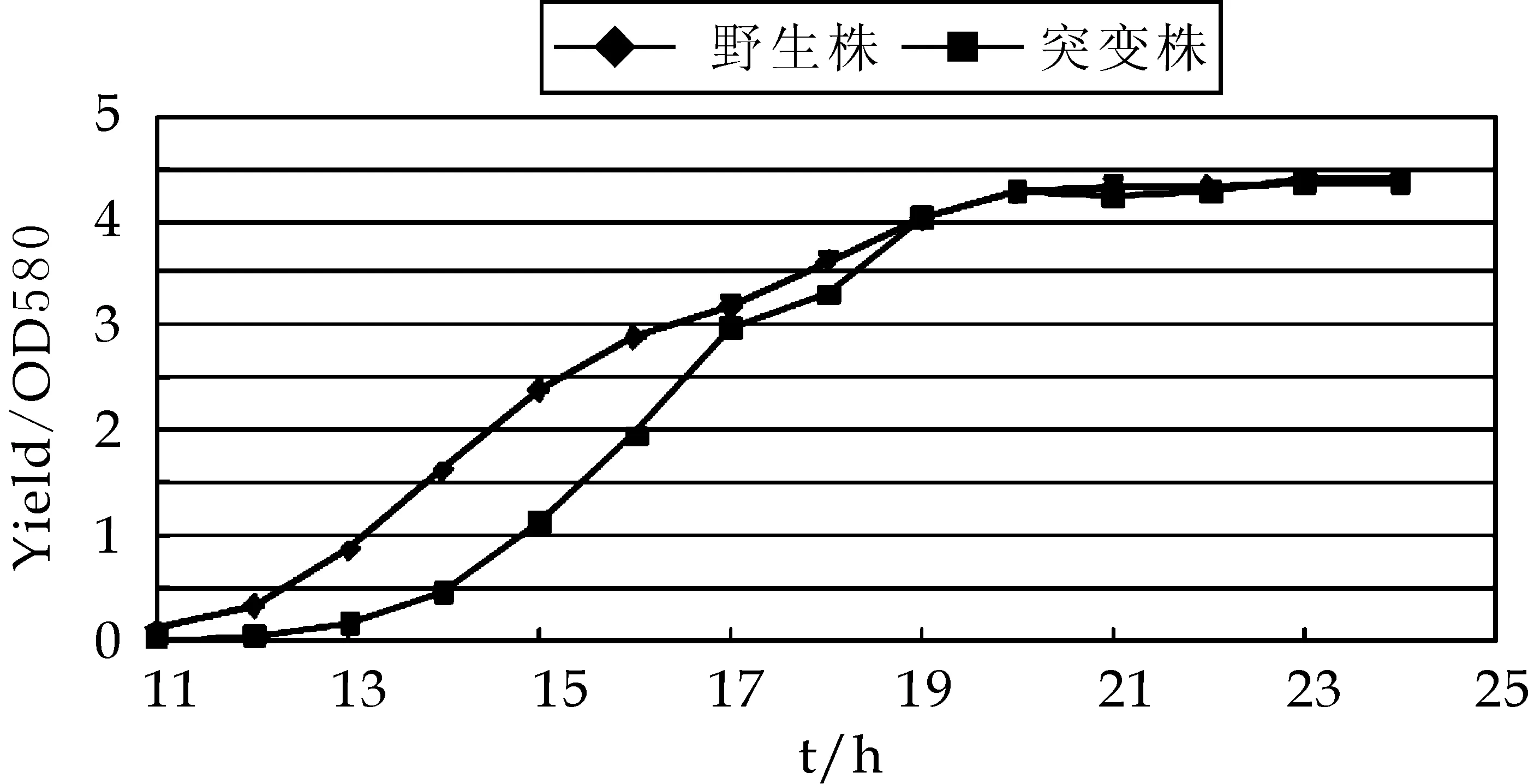
图6 粤蓝链霉菌gra-orf7突变株与野生株在YEME液体培养基产色比较
3 结论与探讨
基因敲除是研究目标基因功能的主要手段之一,本研究对粤蓝链霉菌中一个可能的丝氨酸/苏氨酸蛋白激酶(STPK)编码基因gra-orf7进行了体内敲除,PCR基因型验证结果显示,gra-orf7缺失突变株构建成功.突变株在菌落形态、产孢能力、孢子形态以及色素(榴菌素)最终产率等方面与野生株无显著差异,这些结果似乎暗示gra-orf7在粤蓝链霉菌的发育分化,和榴菌素的生物合成过程中并不扮演重要角色.然而,突变株的产色延迟现象在重复的独立实验中均能观察到,该表型变化应与gra-orf7基因的失活直接相关,这说明gra-orf7基因编码的STPK在粤蓝链霉菌中具有生物学功能.
第三,“免费退货政策”为那些无法亲身体验真实产品的网购者提供了一种安全感。在这种情况下,该政策为网络买家提供了精神和物质上的保障。这项政策鼓励更多的留学生进行网上购物,因为他们没有任何后顾之忧。最后一点也极其重要,那就是隐私和安全问题已经成为网络消费者最关心的问题之一。根据图5的数据,84.9%的留学生认为安全是最重要的考虑因素。对于所有的电子零售商,应该加强他们的在线交易和支付系统,以防丢失隐私、信用卡交易和身份盗窃,并为所有在线购物者提供一个安全可靠的在线交易平台。最后,作者还建议政府、供应商、零售商和其他参与者,多方共同解决安全问题。因为依靠单方面的力量很难取得令人满意的结果。
突变株产色(榴菌素)的延迟,却并不能说明gra-orf7基因编码的STPK直接参与了榴菌素的生物合成.在许多抗生素生物合成基因簇中存在被称为抗生素合成“分子开关”的途径特异性调控基因[22],只有当途径特异性调控基因被激活后,抗生素的生物合成才会启动.在榴菌素的生物合成基因簇中也同样存在这种途径特异性调控基因gra-orf9[9].gra-orf7缺失突变株在产色后,无论是色素的累积速度和最终产量水平均与野生株无显著差别,这说明gra-orf7并没有直接参与榴菌素的生物合成.产色延迟可能是由于gra-orf7基因的失活,影响到了上游调控信号传导,导致榴菌素生物合成的途径特异性调控基因gra-orf9的激活滞后.
STPK是通过使目标蛋白磷酸化而激活目标蛋白的.在链霉菌中,不同的STPK可能能够激活同一个目标蛋白.例如,在天蓝链霉菌A3(2)中,次级代谢全局性调控蛋白AfsR是蛋白激酶AfsK的目标蛋白[5],但PkaG、AfsL等STPK也能使AfsR磷酸化[7].在本研究中,gra-orf7基因编码的STPK可能参与了与榴菌素生物合成有关的上游调控信号的传导,但其目标蛋白可能也能够被其它未知的STPK激活,形成某种补偿机制,使得gra-orf7基因的失活,并不能彻底阻断榴菌素的合成,而是延迟产生.
近期,也有研究者获得了与我们类似的结果[23].他们在研究天蓝链霉菌A3(2)的一个STPK基因pkaF时发现,pkaF过量表达突变株表现出生长延迟、完全丧失了产孢和产放线紫红素的能力,表明pkaF对天蓝链霉菌A3(2)的生长、发育分化和次级代谢有重要的影响;然而pkaF失活突变株在细胞生长、气生菌丝分化、产孢和放线紫红素的产生等方面与野生株并无差别,仅产孢和产放线紫红素的时间提早了约0.5天.造成这一结果的原因也可能与本研究类似,即该基因的目标蛋白还能够被其它未知的STPK激活,通过某种补偿机制,在该基因失活的情况下,菌株仍然能够维持大致正常的生长、发育、分化和次级代谢过程.也正是基于这个原因,虽然gra-orf7缺失突变株的各种表型与野生株无显著差别,但并不能排除gra-orf7参与粤蓝链霉菌发育、分化的可能.
链霉菌STPK早在1994年即被发现[24],此后许多可能的链霉菌STPK陆续被证实,在体外具有丝氨酸/苏氨酸残基磷酸化活性,然而其体内生理功能和作用机制被阐明的仅有AfsK等极少数STPK.基因敲除后,突变株的表型常常与野生株相近,是导致其体内真实生理功能难以被发现的重要原因.本研究虽然暗示了gra-orf7基因编码的STPK,在粤蓝链霉菌中可能参与了与榴菌素生物合成有关的上游调控信号的传导,但要揭示其生理功能和作用机制,仍有大量的工作需要展开.例如,体外丝氨酸/苏氨酸残基磷酸化活性的证实、体内过量表达研究、目标蛋白的捕获和鉴定等,这些均有助于我们深入了解gra-orf7基因在粤蓝链霉菌中的生理功能.目前,进一步的研究正在进行中.
参考文献
[1] Muňoz-Dorado J, Inouye S, Inouye M. A gene encoding a protein serine/threonine kinase is required for normal development of M. xanthus, a gram-negative bacterium[J]. Cell, 1991, 67(5): 995-1 006.
[3] Rajagopal L, Vo A, Silvestroni A, et al. Regulation of purine biosynthesis by a eukaryotic-type kinase in Streptococcus agalactiae[J]. Mol Microbiol, 2005, 56(5): 1 329-1 346.
[4] Bentley SD, Chater KF, Cerdeňo-Tarraga AM, et al. Complete genome sequence of the model actinomycete Streptomyces coelicolorA3(2)[J]. Nature, 2002, 41(7): 141-147.
[5] Umeyama T, Lee PC, Horinouchi S. Protein serine/threonine kinases in signal transduction for secondary metabolism and morphogenesis in Streptomyces[J]. Appl Microbiol Biotechnol, 2002, 59(4-5): 419-425.
[6] Horinouchi S. AfsR as an integrator of signals that are sensed by multiple serine/threonine kinases in Streptomyces coelicolor A3(2)[J]. J Ind Microbiol Biotechnol. 2003, 30(2): 462-467.
[7] Sawai R, Suzuki A, Takano Y, et al. Phosphorylation of AfsR by multiple serine/threonine kinases in Streptomyces coelicolor A3(2)[J]. Gene, 2004, 33(4): 53-61.
[8] 高 瑾,姜卫红,焦瑞身.原核生物中的信号传导与次生代谢[J].国外医药抗生素分册,2001,22(6):269-273.
[9] Koji Ichinose, David J Bedford, et al. The granaticin biosynthetic gene clusterof Streptomyces violaceoruber Tü22: sequence analysis and expression in a heterologous host[J]. Chemistry &Biology, 1998, (5): 647-659.
[10] Zhu HH, Guo J, Yao Q, Yang SZ, Deng MR, Phuong LTB, Hanh VT, Ryan MJ. Streptomyces vietnamensis sp. nov. , a streptomycete with violet blue diffusible pigment isolated from soil in Vietnam[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(8) : 1 770-1 774.
[11] 邓名荣,朱春华,郭 俊,朱红惠.粤蓝链霉菌中紫罗兰蓝色素成分的分离与鉴定[J].华南理工大学学报.2011,5(39):132-137.
[12] Deng MR, Guo J, Li X, Zhu CH, Zhu HH. Granaticins and their biosynthetic gene cluster from Streptomyces vietnamensis: evidence of horizontal gene transfer[J]. Antonie van Leeuwenhoek Journal of Microbiology, 2011, 100(4): 607-617.
[13] Deng MR, Guo J, Zhu HH. Streptomyces vietnamensis GIMV4.0001: a granaticin-producing strain that can be readily genetically manipulated[J]. The Journal of Antibiotics, 2011, 64(2), 345-347.
[14] Yanisch-Perron C, Vieira J, Messing J. Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mpl8 and pUC19 vectors[J]. Gene, 1985, 33(1): 103-119.
[15] Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(12): 6 640-6 645.
[16] MacNeil DJ, Gewain KM, Ruby CL, Dezeny G, Gibbons PH, MacNeil T. Analysis of Streptomyces avermitilis genes required for avermectin biosynthesis utilizing a novel integration vector[J]. Gene, 1992, 111(1): 61-68.
[17] Gust B, Challis GL, Fowler K, Kieser T, Chater KF. PCR-targeted Streptomyces gene replacement identifies a protein domain needed for biosynthesis of the sesquiterpene soil odor geosmin[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(4): 1 541-1 546.
[18] Paget MSB, Chamberlin L, Atrih A, Foster SJ, Buttner MJ. Evidence that the extracytoplasmic function sigma factor sigma E is required for normal cell wall structure in Streptomyces coelicolor A3(2)[J]. Journal of Bacteriology, 1999, 181(1): 204-211.
[19] 邓名荣,郭 俊,黄永恒,朱红惠.粤蓝链霉菌榴菌素生物合成基因orf20的功能[J].微生物学报, 2011,51(3):402-409.
[20] Sambrook J, Russell DW. Molecular cloning: a laboratory manual, 3rd edn[J]. New York: Cold Spring Harbor Laboratory, 2001,35(2):73-74.
[21] Kieser T, Bibb MJ, Buttner MJ, Chater KF, Hopwood DA. Practical Streptomyces Genetics, 2nd edn. Norwich[J]. John Innes Foundation, 2000,45(8):25-26.
[22] Arias P, Fernandez-Moreno MA, Malpartida F. Characterization of the Pathway-Specific positive transcriptional regulator for actinorhodin biosynthesis in Streptomyces coelicolor A3(2) as a DNA-Binding Protein[J]. Journal of Bacteriology, 1999, 181(22): 6 958-6 968.
[23] Oh EA, Chi WJ, Kim MS, Kang SS, Chun J, Hong SK. Characterization of the autophosphorylating kinase, PkaF, in Streptomyces coelicolor A3(2) M130[J]. Archives of Microbiology, 2011, 193(12): 845-856.
[24] Matsumoto A, Hong S-K, Ishizuka H, Horinouchi S, Beppu T. Phosphorylation of the AfsR protein involved in secondary metabolism in Streptomyces species by a eukaryotic type protein kinase[J]. Gene, 1994, 146: 47-56.

