人IgG四亚类对噬菌体展示Ig结合蛋白单结构域随机组合文库的体外进化筛选
祁培培,丁莹莹,吴莉莉,陈秋莉,王锦红,刘超,廖文婷,张婧,曹洁,潘卫
1 中国人民解放军第二军医大学微生物学教研室,上海 200433
2 安徽医科大学病理生理学教研室,安徽 合肥 230032
细菌免疫球蛋白结合蛋白 (Immunoglobulin (Ig)-binding proteins,IBPs) 是一类由细菌产生的特异结合宿主抗体的细菌免疫球蛋白结合蛋白,是细菌重要致病因子之一[1-3]。研究最多的 IBPs有金黄色葡萄球菌蛋白A (Staphylococcal protein A,SpA)、链球菌 (C 和 G 群) 蛋白 G (Streptococcal protein G,SpG) 和部分大消化链球菌表面的蛋白 L (Peptostreptococcus magnus protein L,PpL)[4-8]。研究表明,细菌IBPs均包含由多个序列高度同源的结合结构域头尾相连重复组成的抗体结合区,每个单结构域具有与全分子完全相同的结合特性,形成 Ig结合功能的基本单位。SpA含有5个高度同源的重复结构域,自N端起分别为E、D、A、B、C,单个结构域约58个氨基酸残基大小,形成3股反向平行排列α-螺旋的空间结构,与哺乳动物IgG抗体重链中的第2及第3恒定区 (CH2γ, CH3γ) 间的分界面结合,该结合以螺旋1和螺旋2与Fc的疏水作用为主并通过两个分子之间的4对氢键得到进一步稳定[9-11]。Protein A还可以结合由人VHⅢ基因家族编码的Fab片段,因此还能结合其他类型抗体[12-14]。SpG分子量为65 kDa,包含3个高度同源的Ig结合结构域B1、B2和B3[15](为区别于SpA中的B,以下统称G1、G2、G3),与SpA不同,其单结构域由4个串联的β折叠和1个α螺旋组成[16-17],也结合于IgGFc的CH2γ和CH3γ间的分界面上,与SpA的结合部位部分重叠。因其折叠结构含量很高,Protein G结合Fc位点位于其螺旋和连结螺旋至 β-折叠3的环区中[18]。此外,Protein G可以结合 IgG的Fab段,其识别部位定位于 γ链的第 1个恒定区(CH1γ),所以它仅结合IgG,并具有更高的亲和力[19]。以SpA为代表的IBP已被广泛用于抗体的纯化、制备与鉴定、酶联免疫吸附法检测与诊断、免疫沉淀试验、临床体外免疫吸附治疗,成为非常有价值的科研、诊断和治疗工具[20-22]。
SpA和 SpG的这些单结构域的核苷酸序列高度同源但又存在细微差异,这些差异是否对IgG的结合特性产生影响?SpA和SpG为什么需要多个单结构域的重复?这些问题尚没有明确答案。以金黄色葡萄球菌为例,该细菌广泛存在于自然界中,为多种动物体表正常菌群的组成菌种,同时也可在特定条件下感染致病。然而,人和其他动物IgG分子作为SpA和SpG作用的靶分子序列虽然同源,但进化差异已明显存在,并且在某些物种中存在多种亚类的IgG,人的IgG就存在四亚类。因此,理论上不同的IBP单结构域的重复可产生具有不同结合特性组合以应对不同物种、不同亚类IgG分子的这种多样性。根据这一思路,我们尝试应用基于噬菌体展示的分子进化方法对该问题进行探索性研究。先前,本实验室应用自主建立的基于噬菌体展示技术的分子进化平台,对噬菌体展示Protein A、Protein G、Protein L等IBPs的单结构域随机组合文库并进行体外进化筛选获得了 A-G2[23]、A-L[23](Protein L的B3结构域)、LD3[24]及LD5[24-25]等多种新型进化免疫球蛋白结合分子 (Novel evolved Ig-binding molecules,NEIBMs),其中LD3和LD5为天然IBPs中不存在的单结合结构域组合,这种组合被证实产生了天然 IBPs所没有的抗体结合特性,即对Ig Fabκ轻链和VHⅢ重链的双结合特性,该结合特性被证实大大提高了与IgM的结合力,并被成功地应用于HCV的IgM检测,充分显示了分子进化方法用于该研究的可行性。在本研究中,我们构建了以SpA的A、B、C、D、E 5个结构域和SpG的G2、G3结构域7个单结构域随机组合的噬菌体展示文库,并应用人IgG1、IgG2、IgG3和IgG4四亚类分子进行体外进化筛选,首次获得了一种与人IgG四亚类均具有结合优势的新型组合分子D-C。
1 材料与方法
1.1 材料
1.1.1 质粒载体和菌株
噬菌粒载体 pCANTAB5S由第二军医大学微生物学教研室保存[26];pCANTAB5S-SpA噬菌粒、pMD-18T-G2和pMD-18T-G3质粒分别克隆有 SpA的 EDABC五结构域基因 (GenBank Accession No. P02976)、SpG的B2和B3结构域的基因 (GenBank Accession No. P06654) 为本室构建保存;E. coli TG1和辅助噬菌体M13K07为本室保存。
1.1.2 试剂
限制性内切酶XbaⅠ、碱性磷酸酶 (CIP) 购自宝生物工程 (上海) 公司;高效连接液购自TOYOBO公司;质粒抽提试剂盒及DNA Taq酶购自上海申能博彩生物公司;质粒小提试剂盒和琼脂糖凝胶DNA回收试剂盒购自天根生化科技(北京) 有限公司;氨苄青霉素 (Amp) 与卡那霉素 (Kana) 均购自华美生物公司;人IgG1、2、3、4四亚类分子购自Sigma公司;鼠抗噬菌体酶联单抗 mouse anti-M13mAb-HRP购自 Amersham Pharmacia公司。
1.2 方法
1.2.1 引物设计
根据本室保存的碱基序列设计 12条引物,序列见表1。应用12条引物进行PCR扩增获得下游带有3个氨基酸大小的随机连接肽编码序列(NNSNNSNNS) 的A、B、C、D、E、G2、G3七个结构域的DNA片段,且片段两端均引入XbaⅠ酶切位点 (TCTAGA) 和53 bp保护序列。此外,用于PCR扩增检测噬菌粒pCANTAB5S中克隆片段及测序的上游引物pCANTAB5S-1序列为:5¢-CAACGTGAAAAAATTATTATTCGC-3¢,而下游引物pCANTAB5S-6序列为:5¢-GTAAATGAA TTTTCTGTATGAGG-3¢。以上引物均委托上海生工生物工程科技服务有限公司合成。
1.2.2 SpA和SpG七个单结构域随机组合噬菌体展示文库的构建
以pCANTAB5S-SpA噬菌粒为模板,分别以uA和dAD、uB和dB、uC和dC、uD和dAD、uE和dE为上下游引物,PCR扩增SpA的A、B、C、D、E五结构域片段;以uG2和dG2为上下游引物,分别以pMD-18T-G2、pMD-18T-G3质粒为模板,PCR扩增SpG的G2、G3结构域片段;为延长保护碱基以提高酶切效率,再以Sec为上下游引物,分别以上述7个单结构域片段的PCR产物为模板,PCR扩增得到在两端分别引入约50 bp个保护碱基的7个单结构域片段。将7个片段的PCR产物纯化后用XbaⅠ酶消化,与同样酶切并用CIP酶去磷酸化处理的pCANTAB5S连接后转化E. coli TG1超级感受态细菌 (其制备见文献[27])。分别取0.1、1、10 μL转化后的细菌涂布LB (含100 mg/L Amp) 平皿,并计算库容。随机挑取23个转化子对文库中片段插入情况进行 PCR鉴定,将插入多个结构域片段的转化子委托上海杰李生物技术有限公司 (以下简称上海杰李) 进行序列测定。剩余菌液继续加入10 mL 2×YT (Amp) 培养基,37 ℃、250 r/min振荡培养过夜。加 500 μL 1.3×1012TU/mL的M13K07辅助噬菌体,37 ℃、250 r/min振荡培养1 h,加入Kana后,37℃,250 r/min振荡培养8 h。1 000×g离心10 min,上清经0.22 μm滤膜过滤,即为展示SpA和SpG单结构域随机组合噬菌体文库,滴度测定和无菌实验见文献[28]和[29]。

表1 七个免疫球蛋白结合结构域片段的扩增引物Table 1 Primers for amplification of DNA fragments encoding seven Ig-binding domains of SpA and SpG
1.2.3 人IgG1、IgG2、IgG3和IgG4对噬菌体文库的体外分子进化筛选
应用人 IgG四亚类分别对上述构建的噬菌体文库进行4轮体外分子进化筛选,详细步骤参见文献[23]。根据本实验室工作基础,筛选过程中文库空噬菌体所占比例的减少和展示多结构域 (≥2个结构域) 噬菌体的增加情况是监测筛选是否有效的重要指标,但因原代噬菌体文库中零插入噬菌体比例很少,我们将原代库噬菌体与零插入噬菌体在相同滴度下等体积混匀作为筛选的初始文库 (经PCR鉴定,空噬菌体占74%,展示一个结构域的噬菌体占9%,展示多结构的域噬菌体占 17%)。此外,为深入了解整个进化过程中噬菌体克隆展示序列的变化情况,本研究从人IgG1、2、3、4四组的各代平板上的阳性单克隆中都分别随机挑选 10个插入多结构域片段的单克隆委托上海杰李进行序列测定;若有插入多结构域片段的单克隆总数不足10个,则将实际数目的单克隆进行测序。
1.2.4 ELISA鉴定优势组合分子噬菌体的结合活性
根据序列分析结果,将筛选获得的优势组合D-C的单克隆,划LB平板 (Amp),37 ℃过夜培养。为保证结果的真实可靠,我们随机挑取5个菌落分别接种于5管2×YT培养基 (Amp) 中,经M13KO7拯救,制备成5管具有相同展示物却分别来自不同单克隆的噬菌体,测定滴度后用作平行组实验。相同方法制备pCANTAB5S-SpA噬菌体和pCANTAB5S噬菌体,分别作为本实验阳性、阴性对照。取100 μL噬菌体加入用人IgG1包被的ELISA板条中,37 ℃、2 h后用PBST洗5次,加入鼠抗噬菌体酶联单抗结合,37 ℃、2 h后PBST洗10次,TMB显色后读取OD450数值,检测单克隆噬菌体与人IgG1的结合活性。同样的方法检测与人IgG2、3和4结合活性。
1.2.5 统计学处理
采用SPSS 17.0统计软件对ELISA检测数据进行统计学处理,计量资料多组间比较采用单因素方差分析,两两比较采用q检验,以P<0.05为有统计学意义。
2 结果
2.1 噬菌体展示SpA和SpG单结构域随机组合文库的构建
经PCR扩增得到SpA的A、B、C、D、E和SpG的G2、G3共7个IBP单结构域的DNA片段,纯化后经 XbaⅠ酶消化,得到两端带有XbaⅠ酶切位点的7个单结构域片段 (图1)。
将上述7个IBPs单结构域片段与经同样酶切并CIP处理后的噬菌粒pCANTAB5S连接,连接产物转化超级感受态细胞E. coli TG1,构建的SpA和 SpG单结构域随机组合文库的库容量即转化数为8.2×106CFU,原代噬菌体文库的滴度为1.3×1012TU/mL。
从转化平板上随机挑取23个转化子,对原代文库结构域片段插入情况的PCR检测结果为:零插入的转化子即5S载体自连的为3个,百分比为13%;插入单个结构域片段的为14个,占61%;插入2个的为4个,占17%;插入3个的为2个,占 9% (图 2)。原代文库的结构域片段插入率达87%。此外PCR鉴定的6个插入多个结构域片段的阳性克隆序列测定结果显示:在插入到噬菌粒上共14单结构域片段中,A:B:C:D:E:G2:G3个数比为2:2:1:2:0:4:3,虽然E未测出,但根据本实验室研究基础,原代库中插入的各片段具有随机性;在14单结构域片段中,插入片段的正反向之比为8:6,与 1:1相近,总插入片段的正反向也具有随机性;在 14条随机连接肽序列 (NNS)3中,第 1位N上 A:T:C:G为15:8:13:6,第2位N上A:T:C:G为7:9:14:12,所有S位上的G:C为26:16,均具有随机性 (表 2)。因此我们构建的文库符合体外进化筛选要求。
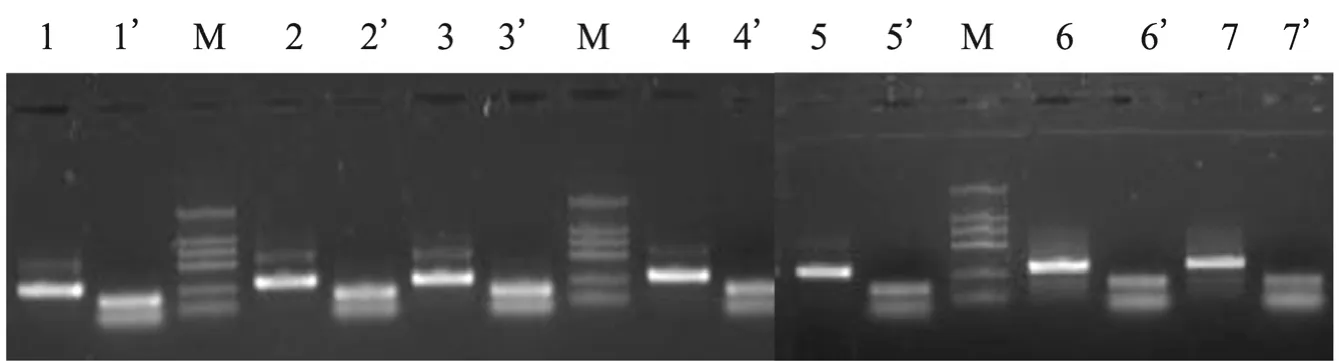
图1 七个SpA、SpGIg结合单结构域DNA片段的XbaⅠ酶消化电泳图Fig. 1 Electrophoretic profile of XbaⅠdigestion of seven DNA fragments of Ig-binding mono-domains of SpA and SpG. M: DL2000 DNA marker (2 000 bp, 1 500 bp, 1 000 bp, 750 bp, 500 bp, 250 bp, 100 bp); 1−7: DNA fragments of A, B, C, D, E, G2, G3 respectively; 1’, 2’, 3’, 4’, 5’, 6’, 7’: A, B, C, D, E, G2, G3 fragments after XbaⅠdigestion respectively.
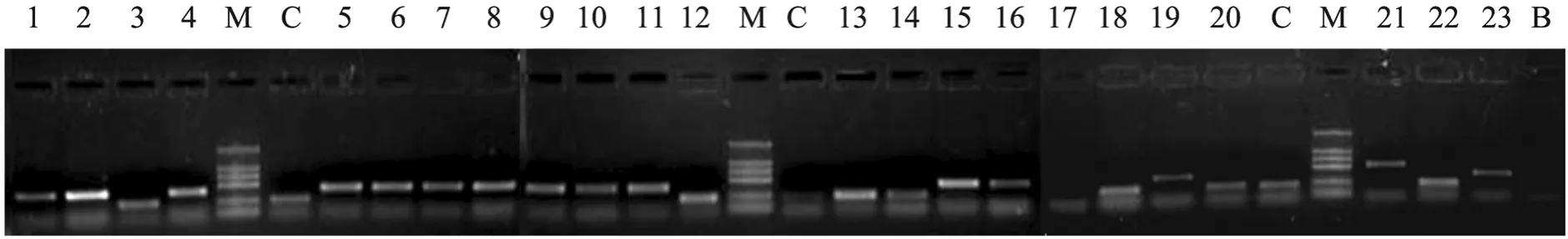
图2 SpA、SpG单结构域随机组合原代文库插入片段大小的PCR鉴定Fig. 2 Identification of the size of inserted fragments in the original library displaying randomly-rearranged various binding domains of SpA and SpG by PCR. M: DL2000 DNA marker (2 000 bp, 1 500 bp, 1 000 bp, 750 bp, 500 bp, 250 bp, 100 bp); 1−23: the number of 23 single clones; C: the phagemid pCANTAB5S for positive control; B: the negative control.
2.2 人IgG1、IgG2、IgG3和IgG4对噬菌体展示SpA和SpG单结构域随机组合文库的体外进化筛选
根据本实验室对同类文库的进化筛选经验[23-24],因IgG具有2条Fc链,可以同时结合2个单结构域,文库中展示两结构域噬菌体所占比例的增加和空噬菌体的减少情况是检测筛选是否有效的重要指标。从图3可知,在所有人IgG四亚类进化筛选中,展示两结构域片段克隆所占的比例持续稳定地增加,最后文库中基本全部进化为两结构域组合分子;相反地,零插入克隆和插入单个结构域片段克隆所占比例不断降低,最后基本被淘选消失,这种变化充分显示了筛选的有效性。R: reverse sequence of original sequence; 9n: the sequence of random linking peptides composed of nine nucleotides.
表2 SpA、SpG单结构域随机组合原代文库中噬菌体克隆插入片段的序列分析Table 2 Sequence analyses of inserted fragments on phage clones in the original library displaying randomly-rearranged various binding domains of SpA and SpG
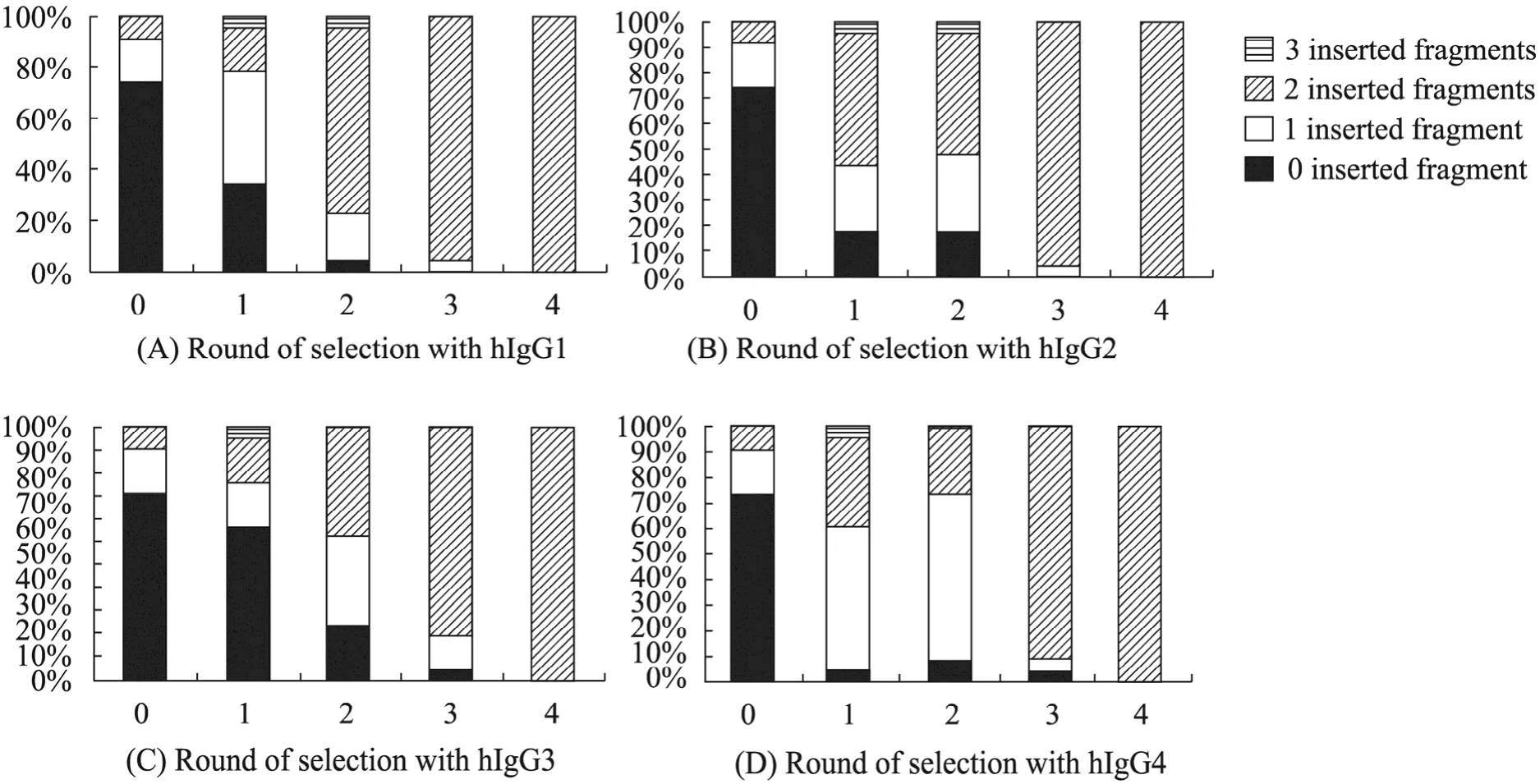
图3 经人IgG四亚类筛选后各代噬菌体文库中23个单克隆噬菌体插入片段的组成比例变化Fig. 3 Proportion of phage clones with different sizes of inserted fragments in 23 phage clones after each round of selection with four human IgG subclasses respectively (A−D).
为深入了解在整个进化过程中两结构域的序列变化情况,本研究从人IgG四亚类筛选的各代文库中都分别随机挑选10个插入多结构域片段的单克隆进行测序,而在IgG1组的F1代、IgG3组的F1和F2代、IgG4组的F1和F2代中因插入多结构域片段的单克隆数目较少,则按实际数目测序。测序结果显示:1) 第 1轮筛选中四亚类组共测得25种多结构域组合分子,其中能正确展示的均为正向插入的多结构域组合为17种,包括D9n2-C9n2(人IgG2组的第1轮筛选)。2) 第2轮筛选中四亚类组均出现一种新的组合分子D9n1-C9n1(以下简称D-C),然而所占的比例在四亚类中却差别很大,IgG1最高,占50%,IgG2和IgG4次之,均为30%,而IgG3最低,仅为10%。3) 人IgG四亚类组在第3、4轮筛选最终都得到相同的优势组合分子,D-C。第4轮筛选后的文库中随机挑取10个克隆除IgG3组出现了30%的A9n-G39n,其余均为D-C,见表3。

表3 人IgG1、IgG2、IgG3和IgG4诱导的进化筛选各轮获得展示片段的测序结果Table 3 Sequence analyses of inserted fragments on phage clones in the four rounds of post-selection libraries with four human IgG subclasses
2.3 ELISA检测 D-C噬菌体与人 IgG1、IgG2、IgG3和IgG4的结合优势
将D-C、pCANTAB5S-SpA和pCANTAB5S三组分别制备5个单克隆噬菌体,并将其滴度均调整为约 1.0×1012TU/mL后在相同条件下进行ELSIA比较3组噬菌体分别对人IgG四亚类的结合活性。ELISA结果显示: D-C噬菌体和SpA噬菌体对人 IgG四亚类的结合活性均远强于阴性对照组;D-C噬菌体对人IgG四亚类的结合活性又均强于SpA噬菌体,两组的差异有统计学意义(图4),且在人IgG四亚类中D-C噬菌体对IgG1的结合活性最强,IgG3组最弱。
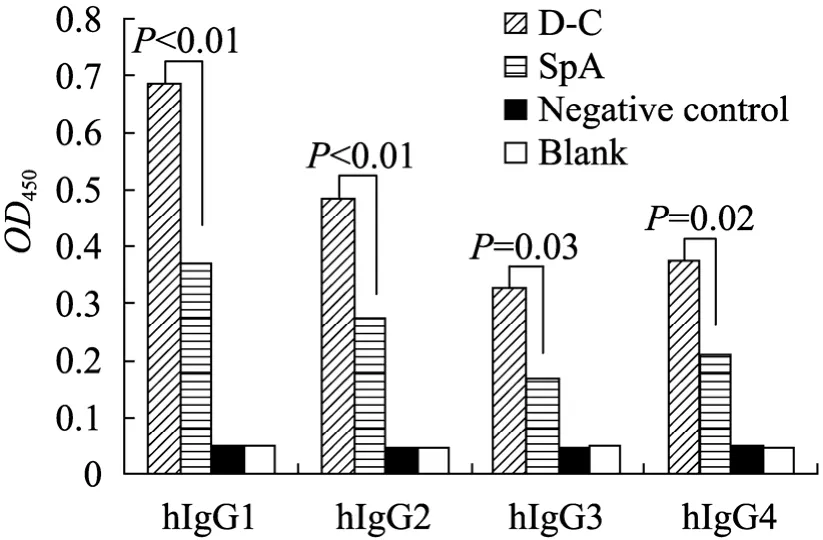
图4 ELISA比较 D-C噬菌体和 SpA噬菌体对人IgG1、IgG2、IgG3和IgG4的结合活性Fig. 4 Comparison of the binding activities of D-C and SpA phage clones with four human IgG subclasses respectively by ELISA.
3 讨论
SpA和 SpG是细菌表面与宿主抗体特异结合的蛋白,是极具价值的诊断、治疗及抗体制备工具。它们的各单结构分别以天然的组合形式与哺乳动物IgG的Fc结合。为探索SpA和SpG的各单结构域重组后能否获得对人 IgG各亚类优势结合的NEIBMs,本研究构建展示SpA、SpG单结构域随机组合的噬菌体展示文库,并应用人IgG四亚类分子进行体外进化筛选。我们预期能获得与各人 IgG亚类特异结合的不同的NEIBMs,然而,本研究的结果却与预期相反,4种人 IgG亚类所诱导的分子进化均得到了同一种优势分子,D-C组合。
我们所构建的文库库容为8.2×106CFU,原代噬菌体文库的滴度为1.3×1012TU/mL,对原始文库中随机挑取阳性单克隆的 PCR检测显示已插入结构域片段的阳性克隆总计达87%。插入多结构域片段的阳性克隆序列测定结果显示原始文库中的各片段插入情况、片段正反向比例、随机连接肽序列 (NNS)3均具有随机性。理论上,文库中两结构域组合的方式应为7×2×7×2=196,各单结构域随机连接肽理论上的组合方式应为(2×4×4)3×(2×4×4)3=1048576。可见,该文库虽然不能将所有的组合方式完全包含,但已远远超出了对 2个及 2个以上结构域组合充分展示的要求。这些结果表明我们所构建的文库在库容、随机性和多样性方面能够满足筛选要求。
根据本实验室对同类文库的IgG及IgG Fc筛选经验[23-24],因IgG具有两条Fc链,可同时结合两个单结构域,文库中展示两结构域噬菌体比例的增加情况是监测筛选是否有效的最重要的指标。在人四IgG亚类的进化筛选中,展示两结构域噬菌体的比例随着筛选的进行不断增多,筛选到第3、4代时,文库中基本已全部进化为两结构域组合分子,这种变化充分显示筛选的有效性。
该次筛选中我们还特别检测了两结构域组合分子组成和序列在进化过程中的变化情况。出乎我们意料的是,人 IgG1、IgG2、IgG3、IgG4筛选最终都得到具有相同的优势组合分子D-C,并且其随机连接肽序列也完全相同。该优势组合分子D-C均在第2轮筛选中出现,然而所占的比例在四亚类中却很大差别,IgG1最高,占50%,IgG2和IgG4次之,均为30%,而IgG3最低,仅为 10%。有意思的是,这样的差别与 phage ELISA所反映的各亚类IgG与D-C的结合能力完全相符。人IgG1、IgG2、IgG4诱导的进化3、4轮即已完全,D-C成为唯一的克隆,而 IgG3所诱导的筛选到第 4轮筛选时出现了 D-C和A9n-G39n两种优势分子,其中D-C占70%,为相对优势分子。另外,原代文库和四亚类的第1轮筛选后文库两个结构域组合中均未发现D-C,表明其在原代文库中未形成优势。同时四亚类的第1、2轮筛选我们发现至少存在E-G3、D-G2、C-C、A-B等28种不同的正确展示的两个结构域组合分子,显示了D-C的超强结合能力。的确,我们采用严格对照的噬菌体ELISA实验证明了 D-C噬菌体与人 IgG四亚类的结合均远远强于 SpA噬菌体。因此两结构域组合分子序列的动态变化情况很好地反映了筛选的有效性和D-C的意义。另外,在人IgG2组第1轮筛选中还出现了具有不同于D-C的随机连接肽序列的D9n2-C9n2,然而在后续测序中再未出现过,最终D-C成为四亚类筛选的优势组合分子,这说明随机连接肽的序列对D-C的结合功能有影响,的确,四亚类IgG最终筛选到的D-C随机连接肽序列完全一致,被严格选择。
上述结果均说明和证明了D-C与IgG的结合优势,然而,为什么D-C组合具有结合优势目前尚无明确解释。SpA中五结构域的天然组合顺序是E-D-A-B-C,D-C组合不存在其中。我们应用5种SpA的单结构域和2种SpG的单结构域共7种IgG Fc单结合结构域构建噬菌体随机组合文库,因此理论上能产生同时正确结合两条IgG Fc的双结构域正确组合为49种 (7×7),远远超过天然SpA和SpG所能产生出来的6种两个结构域组合 (SpG分子中3个结合结构域的组合顺序是G1-G2-G3,可产生2种组合)。此外,文库中两个结构域组合是通过3个氨基酸的随机连接肽连接,这种方式比天然分子的直接连接对结合IgG Fc双链更具有优势 (随机连接肽的序列对两个结构域组合的结合功能有影响,见上段讨论)。因此,产生出结合能力超过天然两个结构域组合的分子完全可能。金黄色葡萄球菌感染多种宿主时,SpA能与多物种、多亚类IgG结合,其5个单结构域的串联可大大扩展其结合谱,然而,由于基因容量的限制,使其无法容纳更多的串联体而限制其产生具有最强结合能力的组合。因此,我们所得到的在 SpA天然分子中不存在的具有强结合能力的D-C也具合理性。目前,我们正在进行原核表达D-C融合蛋白,以期在分子水平上进一步确证D-C的超强结合能力。
与其他3组相比,人IgG3组筛选中D-C富集偏慢,在第4轮筛选时除此还筛得一定比例的另一种组合分子A9n-G39n(30%)。噬菌体ELISA结果也显示D-C与IgG3的结合活性在四亚类中最弱。根据人IgG1 (Gene ID:3500)、IgG2 (Gene ID:3501)、IgG3 (Gene ID:3502) 和 IgG4 (Gene ID:3503) 重链区CH2-CH3的氨基酸序列比对结果 (图5),我们将SpA与人IgG的CH2-CH3区域的作用位点[30-31]进行标注 (阴影区域) 后发现与其他三亚类不同,人IgG3在第334、335位结合位点分别为R、F,我们推测其影响了IgG3的Fc构象以及与 SpA结合,同时也引起了上述IgG3与其他IgG组进化筛选的不同。
本研究用IgG四亚类对展示了SpA、SpG共 7个单结构域随机组合噬菌体文库成功地进行了体外分子进化筛选,得到了天然SpA分子中不存在的具有强结合活性的D-C组合,不仅可为IgG纯化、制备、检测等方面的应用提供了新的候选分子,还为细菌IBP结构功能的进一步研究提供了新的手段。

图5 人IgG1、IgG2、IgG3和IgG4四亚类Fc的CH2-CH3段序列比对分析Fig. 5 Alignment of amino acid sequences in CH2-CH3 of four human IgG subclasses. The binding sites of human IgG Fc with SpA are shaded.
REFERENCES
[1] Housden NG, Harrison S, Roberts SE, et al. Immunoglobulin-binding domains: protein L from Peptostreptococcus magnus. Biochem Soc T, 2003, 31(3): 716−718.
[2] Graille M, Stura EA, Housden NG, et al. Complex between Peptostreptococcus magnus protein L and a human antibody reveals structural convergence in the interaction modes of Fab binding proteins. Structure, 2001, 9(8): 679−687.
[3] Kumar A, Tassopoulos AM, Li Q,et al. Staphylococcus aureus protein A induced inflammatory response in human corneal epithelial cells. Biochem Biophys Res Commun, 2007, 354(4): 955−961.
[4] Atkins KL, Burman JD, Chamberlain ES, et al. S. aureus IgG-binding proteins SpA and Sbi: host specificity and mechanisms of immune complex formation. Mol Immunol, 2008, 45(6): 1600−1611.
[5] Nomellini JF, Duncan G, Dorocicz IR, et al. S-Layer-mediated display of the immunoglobulin G-binding domain of streptococcal protein G on the surface of Caulobacter crescentus: development of an immunoactive reagent. Appl Environ Microbiol, 2007, 73(10): 3245−3253.
[6] Goward CR, Scawen MD, Murphy JP, et al. Molecular evolution of bacterial cell-surface proteins. Trends Biochem Sci, 1993, 18(4): 136−140.
[7] Brockwell DJ, Beddard GS, Paci E, et al. Mechanically unfolding the small, topologically simple protein L. Biophys J, 2005, 89(1): 506−519.
[8] Stura EA, Graille M, Housden NG, et al. Protein L mutants for the crystallization of antibody fragments. Acta Crystallogr D Biol Crystallogr, 2002, 58(Pt 10 Pt 1): 1744−1748.
[9] Uhlén M, Guss B, Nilsson B, et al. Complete sequence of the staphylococcal gene encoding protein A. A gene evolved through multiple duplications. J Biol Chem, 1984, 259(3): 1695−1702.
[10] Lei HX, Wu C, Wang ZX, Zhou YQ, et al. Folding processes of the B domain of protein A to the native state observed in all-atom ab initio folding simulations. J Chem Phys, 2008, 128(23): 235105.
[11] Moks T, Abrahmsén L, Nilsson B, et al. Staphylococcal protein A consists of five IgG-binding domains. Eur J Biochem, 1986, 156(3): 637−643.
[12] Graille M, Stura EA, Corper AL, et al. Crystal structure of a Staphylococcus aureus protein A domain complexed with the Fab fragment of a human IgM antibody: structural basis for recognition of B-cell receptors and superantigen activity. Proc Natl Acad Sci USA, 2000, 97(10): 5399−5404.
[13] Sasso EH, Silverman GJ, Mannik M. Human IgM molecules that bind staphylococcal protein A contain VHIII H chains. J Immunol, 1989, 142(8): 2778−2783.
[14] Sasso EH, Silverman GJ, Mannik M. Human IgA and IgG F(ab’)2 that bind to staphylococcal protein A belong to the VHIII subgroup. J Immunol, 1991, 147(6): 1877−1883.
[15] Stone GC, Sjobring U, Bjorck L, et al. The Fc binding site for streptococcal protein G is in the Cγ2-Cγ3 interface region of IgG and is related to the sites that bind staphylococcal protein A and human rheumatoid factors. J Immunol, 1989, 143(2): 565−570.
[16] Gronenborn AM, Clore GM. Identification of the contact surface of a streptococcal protein G domain complexed with a human Fc fragment. J Mol Biol, 1993, 233(3): 331−335.
[17] Sloan DJ, Hellinga HW. Dissection of the protein G B1 domain binding site for human IgG Fc fragment. Protein Sci, 1999, 8(8): 1643−1648.
[18] Kato K, Lian LY, Barsukov IL, et al. Model for the complex between protein G and an antibody Fc fragment in solution. Structure, 1995, 3(1): 79−85.
[19] Watanabe H, Matsumaru H, Ooishi A, et al. Optimizing pH response of affinity between protein G and IgG Fc: how electrostatic modulations affect protein-protein interactions. J Biol Chem, 2009, 284(18): 12373−12383.
[20] Zhou J, Liao YX, Chen Z, et al. Development of a SPA-ELISA method for detecting anti-coronavirus IgG antibodies in serum samples from fulvous fruit bats. J South Med Univ, 2008, 28(5): 736−738.周杰, 廖玉学, 陈忠, 等. SpA-ELISA检测棕果蝠血清冠状病毒抗体的研究. 南方医科大学学报, 2008, 28(5): 736−738.
[21] Poullin P, Announ N, Mugnier B, et al. Protein A-immunoadsorption (Prosorba®column) in the treatment of rheumatoid arthritis. Joint Bone Spine, 2005, 72(2): 101−103.
[22] Hober S, Nord K, Linhult M. Protein A chromatography for antibody purification. J Chromatogr B Analyt Technol Biomed Life Sci, 2007, 848(1): 40−47.
[23] Yang H, Cao J, Li LQ, et al. Evolutional selection of a combinatorial phage library displaying randomly-rearranged various single domains of immunoglobulin (Ig)-binding proteins (IBPs) with four kinds of Ig molecules. BMC Microbiol, 2008, 8(1): 137.
[24] Jiang SH, Wang JF, Xu R, et al. Alternate arrangement of PpL B3 domain and SpA D domain creates synergistic double-site binding to VH3 and Vkappa regions of fab. DNA Cell Biol, 2008, 27(8): 423−431.
[25] Cao J, Chen QL, Zhang HQ, et al. Novel evolved immunoglobulin (Ig)-binding molecules enhance the detection of IgM against hepatitis C virus. PLoS One, 2011, 6(4): e18477.
[26] Xu R, Pan W, Shen YJ, et al. Construction of a novel phagemid pCANTAB5S. Acta Univ Med Anhui, 2004, 39(2): 83−86.徐容, 潘卫, 沈毅珺, 等. 新型噬菌体展示载体pCANTAB5S的构建. 安徽医科大学学报, 2004, 39(2): 83−86.
[27] Ge YB, Yang XF, Du ZM, et al. Constructing a phage-displayed random mutation library of HIV-1 Tat38-61 at the sites of 51 and 55 amino acids in basic region. Chin J Biotech, 2011, 27(5): 1755−763.葛宜兵, 杨旭芳, 杜哲明, 等. 噬菌体展示HIV-1 Tat38-61碱性区51和55位随机突变体文库的构建. 生物工程学报, 2011, 27(5): 755−763.
[28] Xu R, Shen YJ, Deng SH, et al. Phage display of random combinatorial libraries of Ig-binding mono-domains of protein a and protein l and Ig affinity screening. Prog Biochem Biophys, 2005, 32(6): 535−543.徐容, 沈毅珺, 邓松华, 等. 噬菌体展示 protein A及protein L Ig结合单结构域随机组合文库及Ig亲和筛选. 生物化学与生物物理进展, 2005, 32(6): 535−543.
[29] Shen YJ, Pan W, Xu Y, et al. Phage display of recombinant human lymphotoxin mutation libraries and receptor affinity screening. Prog Biochem Biophys, 2005, 32(1): 75−80.沈毅珺, 潘卫, 许燕, 等. 噬菌体展示重组人淋巴毒素突变体库及受体亲和筛选. 生物化学与生物物理进展, 2005, 32(1): 75−80.
[30] DeLano WL, Ultsch MH, de Vos AM, et al. Convergent solutions to binding at a protein-protein interface. Science, 2000, 287(5456): 1279−1283.
[31] Verkhivker GM, Bouzida D, Gehlhaar DK, et al. Monte Carlo simulations of the peptide recognition at the consensus binding site of the constant fragment of human immunoglobulin G: the energy landscape analysis of a hot spot at the intermolecular interface. Proteins, 2002, 48(3): 539−557.

