LDR-qPCR法检测miRNA122a的表达及其与乳腺癌雌激素调控的关系
恽 文, 季明华, 李文静, 李 建, 钟山亮, 唐金海,赵建华
微小RNA(miRNA)是一类新近发现的存在于真核细胞中的非编码小分子RNA,通过下调蛋白编码基因的表达而对不同细胞的生长、分化或发育过程起关键调控作用。分析细胞或组织样本中miRNAs的表达可为研究这类分子的生物学功能提供重要的信息。成熟miRNAs片段小,没有poly(A)尾巴,家族成员间通常只有一两个碱基的差别,并且在细胞中的表达水平普遍较低,这些特性给miRNA定量检测方法的建立带来了困难和挑战。为此,本文统一采用SYBR Green I的荧光模式,在Li等[1]报道的基础上建立一种新的基于连接酶依赖性反应的miRNA实时定量PCR方法(LDR-qPCR法),并与茎环逆转录的SYBR GREEN实时定量PCR法[2](茎环RT-qPCR法)进行比较,希望能为miRNA的研究提供敏感而准确的检测工具。目前miR-122的研究主要集中在肝脏疾病上,已被认为是一个调节肝脏代谢和发育的“肝特异性miRNA”。不过,最近Sun等[3]通过芯片筛选和RT-qPCR法确认,证实 miR-122a在乳腺癌干细胞中的表达水平较乳腺癌MCF-7细胞明显增高;靶基因预测分析显示其潜在靶点涉及肿瘤发生、DNA修复、细胞周期调控和信号转导等多个过程,提示miR-122a很可能是乳腺癌细胞、特别是癌干细胞的重要调控因子。已知乳腺癌是激素依赖性肿瘤,对雌激素的依赖性最大;其发生发展与内分泌功能失调密切相关,该过程有多种miRNAs参与,如 Wickramasinghe等[4]发现在乳腺癌MCF-7细胞中,雌二醇可抑制miR-21的表达,这种抑制作用可被内分泌治疗药物如他莫昔芬和氟维斯群等阻断。乳腺癌细胞miR-122a的表达是否也与雌激素调控有关,本文亦对此进行初步研究。
1 资料与方法
1.1 材料与试剂
人肝癌细胞株HepG2由南京大学医学院遗传系惠赠;人乳腺腺癌上皮细胞株MCF-7购自中科院上海细胞所;DMEM和无酚红DMEM购自GIBCO公司;胎牛血清(FBS)和活性碳处理的FBS购自Boind公司;Taq DNA连接酶和Taq酶购自NEB公司;Trizol、cDNA第一链合成试剂盒和SYBR Green Mix购自Biouniquer公司;17β-雌二醇购自sigma公司;引物和探针由上海闪晶生物公司合成,PAGE法纯化。
1.2 仪器与设备
荧光定量PCR仪:配有IQ5数据处理系统,美国Bio-Rad公司;超微量紫外分光光度计:配有NanoDrop2000软件,美国NanoDrop公司;凝胶成像仪:配有141万像素全自动数码凝胶成像分析系统,上海培清科技有限公司;琼脂糖水平电泳槽(中号):美国伯乐公司。
1.3 试验方法
1.3.1 总 RNA的提取 收集对数期 HepG2或MCF-7细胞,加Trizol提取总RNA;3%琼脂糖凝胶电泳检测其完整性,紫外分光光度仪测定其吸光度(A),A260/A280 为 1.8 ~2.0,纯度符合要求,以A260值计算核酸浓度。
1.3.2 探针和引物的设计 查询miRBase数据库获得成熟hsa-miR-122a序列:5'-UGGAGUGUGA↓CAAUGGUGUUUGU-3'(箭头为连接反应点)。按参考文献[1]设计一对含茎环结构的探针和一对引物。探针1的3'端13个碱基和探针2的5'端10个碱基与miR-122a全序列互补并相邻(粗体标出),探针1:5'-GGTATCCAGGGAAGTGGATACGAAGAAT GCACAAACACCATTG-OH-3'和探针 2:5'-PO4-TCACACTCCACGCTGTGTTAAAGTCGTACAGGTA TTACGACA-3';上游引物与探针1的部分序列重复,其序列为5'-GGGAAGTGGATACGAAGAATGC-3',下游引物与探针2的部分序列互补,其序列为5'-CGACTTTAACACAGCGTGGAG-3'。以 U6为内对照,同法设计,探针为5'-TATGGAACGCTTCACGAAT TTGCGTGTCATCCTTGCGCAGGGGCCATGCT-OH-3'和5'-PO4-AATCTTCTCTGTATC-GTTCCAATTTTAGTATATGTGCTGCCGAAGCGAAGA T3';上、下游引物分 别 为 5'-CTCGCTTCGGCAGCACA-3'和 5'-AACGCTTCACGAATTTGCGT-3'。
1.3.3 连接反应 取总 RNA 1 μg,加入 miR-122a或U6的探针1和探针2各25 fmol进行杂交,反应条件为65℃变性3min后缓慢地降至室温(约45 min);然后加入 1 ×Taq ligase buffer、1 μL RNasin 和
0.4 μL Taq DNA 连接酶,总体积 20 μL 进行连接反应,45℃温浴2 h后,90℃灭活10 min。
1.3.4 LDR-qPCR 法 以 miR-122a或 U6的连接产物为模板,加入相应上、下游引物进行实时定量PCR,反应体系25 μL,含模板2 μL、2 ×SYBR Green Mix 12.5 μL,上、下游引物(10 pmol/μL) 各 0.5 μL,去离子水 9.5 μL;反应条件为 95 ℃ 变性 10 min,95 ℃5 s,60 ℃30 s,72 ℃30 s,共 40 个循环,每个循环于72℃收集荧光;55℃ ~95℃每上升1℃收集一次荧光,绘制溶解曲线。同时设置HeGp2为阳性对照,水代替模板为阴性对照同步扩增。
1.3.5 茎环 RT-qPCR法 按参考文献[2]设计miR-122a的茎环逆转录引物:5'-GCGCGTGAGCAGGCTGGAGAAATTAACCACGCGCACAAACAC-3';U6特异逆转录引物的5'端不必添加延伸序列和茎环序列,且与下游引物序列相同:AACGCTTCACGAATTTGCGT。miR-122a上游引物:5'-TGGAGTGTGACAATGG-3',U6上游引物:5'-CGCAAGGATGACACG-3'。miR-122a下游引物:5'-GAGCAGGCTGGAGAA-3'。取总 RNA 1μg,利用 miR-122a 或U6的特异性茎环逆转录引物,逆转录合成cDNA;再以该 cDNA为模板,以 miR-122a或 U6的 PCR上、下游引物分别进行实时定量PCR,并计算miR-122a的相对表达量。
1.3.6 细胞培养及分组处理 (1)MCF-7细胞常规传代培养于含10%FBS的DMEM培养基(A1组)。(2)对数期MCF-7细胞经PBS洗涤后,加入含10%活性碳处理FBS的无酚红DMEM中进行激素饥饿培养48h(B1组)。(3)以DMSO为溶剂,将终浓度5 nmol/L的17β-雌二醇分别加入常规培养(A2组)和激素饥饿培养的MCF-7细胞中(B2组),孵育24 h。每组设置3份平行。计A1组为校准样本(miR-122a表达量为 1),2-ΔΔct法计算各组 miR-122a相对表达量。
1.3.7 LDR-qPCR方法学评价 (1)扩增产物特异性:结合熔解曲线、3%琼脂糖凝胶电泳和扩增产物测序,分析其特异性。(2)重复性:以 MCF-7和HeGp2细胞作为待测样本,每份样本从总RNA提取开始重复3次,以ΔCt=Ctu6-CtmiR-122a代表各反应管检测结果,评价检测反应的重复性。(3)检测范围和灵敏度:以MCF-7细胞为样本,10倍倍比稀释miR-122a的连接产物或cDNA,检测各稀释系列样本。各样本均设3个复管。比较LDR-qPCR法与茎环RT-qPCR法的线性检测范围和灵敏度。(4)相关性:对下述 A1、A2、B1、B2组 MCF-7细胞进行 miR-122a表达量的检测,分析LDR-qPCR法与茎环RT-qPCR法的相关性。
1.4 统计学分析
采用SPSS17.0软件进行Paired-t test和双变量相关性分析。以P<0.05为差异有统计学意义。
2 结果
2.1 方法学评价
2.1.1 特异性 LDR-qPCR法检测miR-122a或U6的熔解曲线为单一熔解峰,电泳为单一清晰条带,且峰值、电泳条带大小均与阳性对照一致,阴性对照无相应熔解峰和条带(图1);扩增产物测序与 hsamiR-122a公布序列一致。
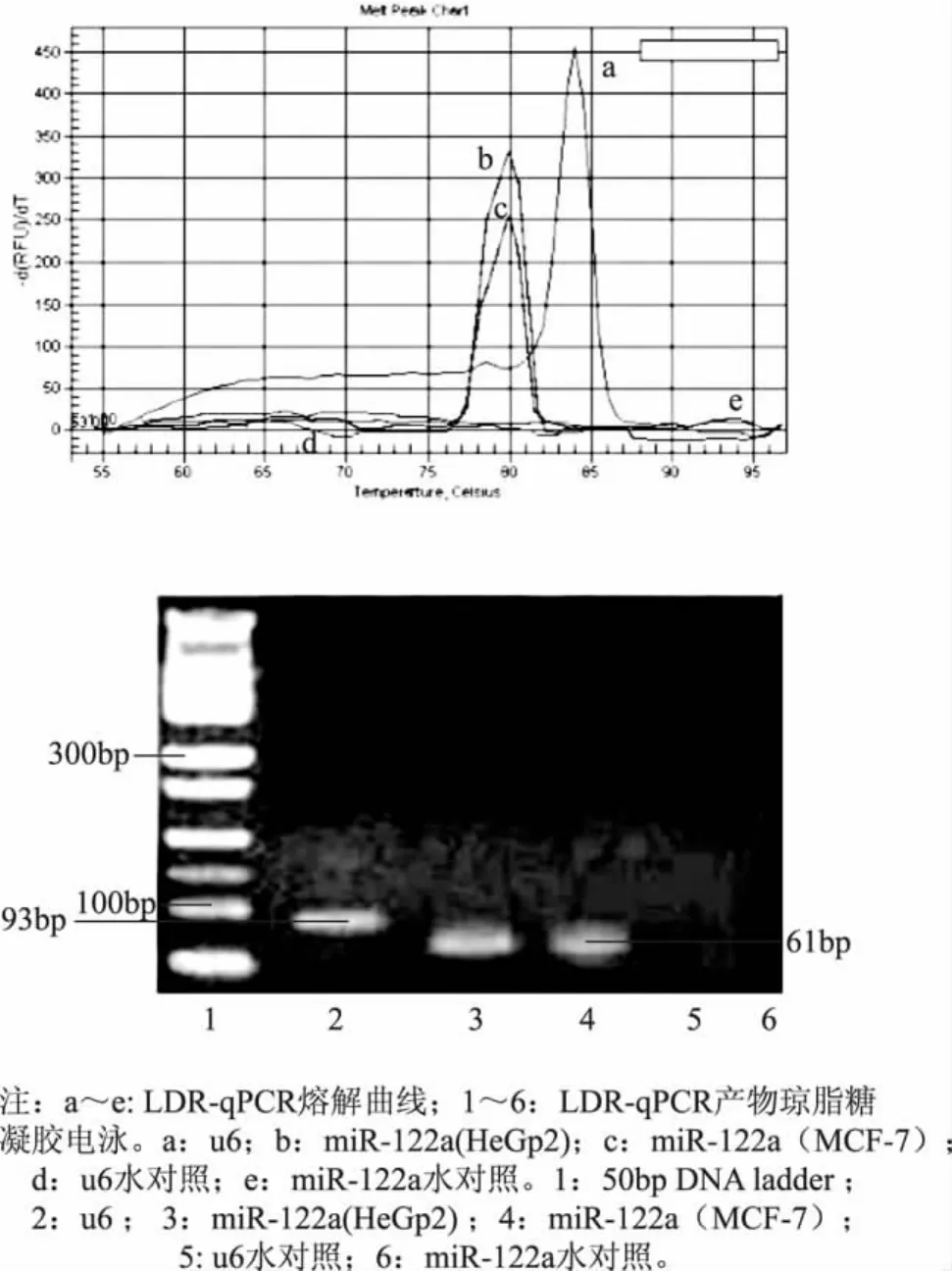
图1 LDR-qPCR法的扩增结果
2.1.2 重复性 LDR-qPCR法检测的批内变异系数均小于10%,与茎环RT-qPCR法基本相当(表1)。
表1 LDR-qPCR法检测miR-122a的重复性(± s,n=3)
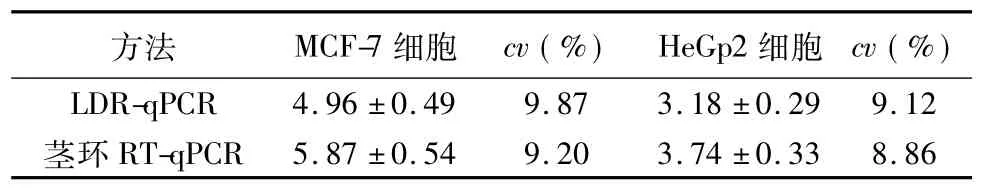
表1 LDR-qPCR法检测miR-122a的重复性(± s,n=3)
LDR-qPCR 4.96 ±0.49 9.87 3.18 ±0.29 9.12_茎__环RT-qPCR____5.87 ±0.54_______9.20________________________3.74_±0.338.86
2.1.3 检测范围和灵敏度 各稀释系列样本检测结果见图2。LDR-qPCR法与茎环RT-qPCR法检测miR-122a的检测最低限为10-4稀释度(RNA浓度为5 pg/μL),茎环 RT-qPCR 法为10-3稀释度(RNA浓度为50 pg/μL),前者比后者敏感10倍。
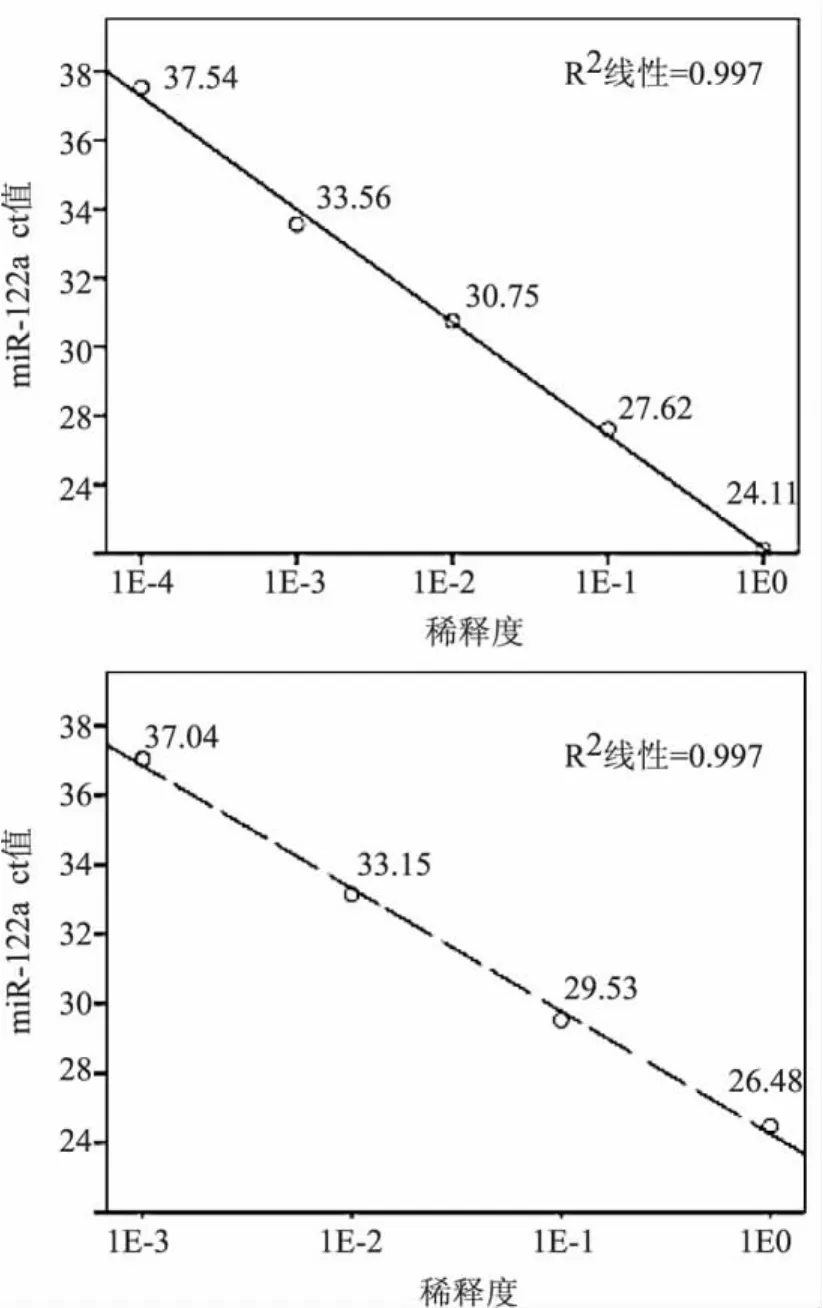
图2 LDR-qPCR法(上图)与茎环RT-qPCR法(下图)的线性检测范围
2.1.4 相关性 分别用LDR-qPCR法与茎环RT-qPCR法检测A1、A2、B1、B2组细胞 miR-122a表达量,二者结果具有良好的相关性(r=0.996,P=0.542)。
2.2 雌激素对miR-122a表达的调节
经LDR-qPCR法检测,miR-122a表达水平随着雌激素含量的增加而下调,呈负相关(r=-0.960)。激素饥饿培养的MCF-7中miR-122a表达上调,约为常规培养细胞的3.65倍;经5 nmol/L雌二醇处理后miR-122a的表达又出现明显下调(P=0.040),但未能回到常规培养细胞水平(图3)。茎环RT-qPCR法检测结果亦显示相似的变化趋势。
3 讨论
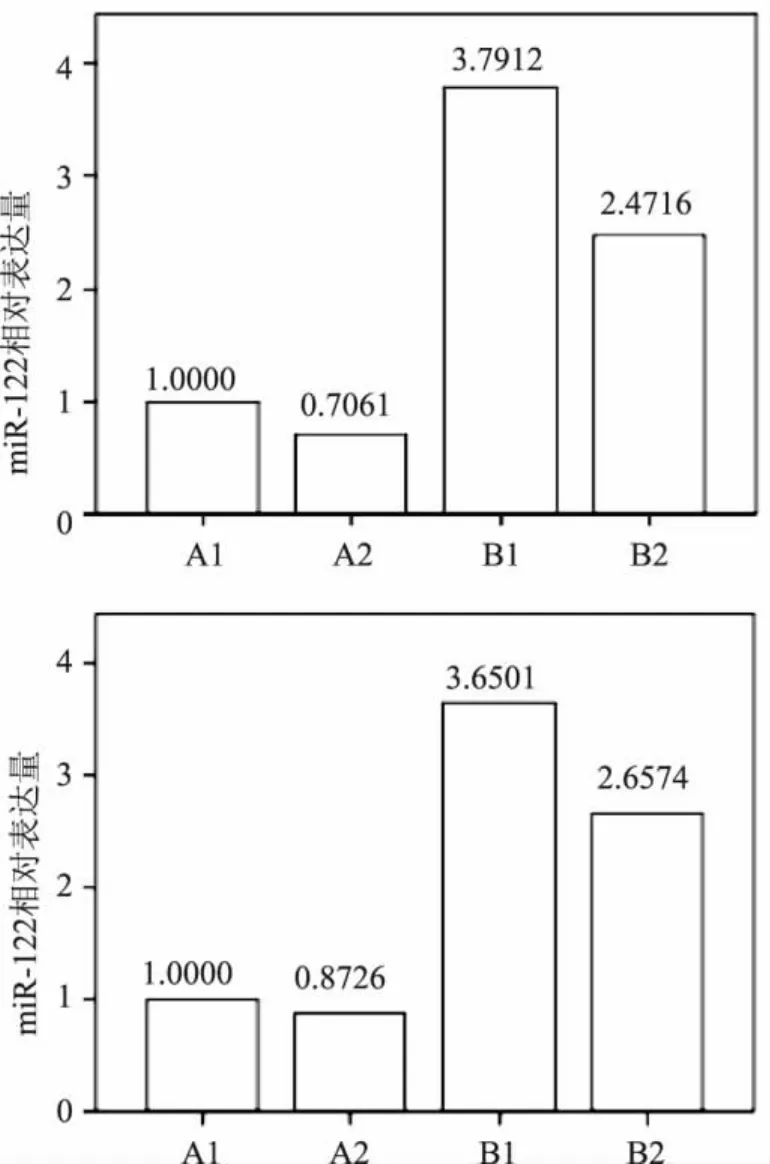
图3 LDR-qPCR法(上图)与茎环RT-qPCR法(下图)检测A1、A2、B1、B2组MCF-7细胞miR-122a表达的结果
miRNAs种类多、序列短且同源性高,实时定量PCR法需经过特殊的设计或样本处理才能用于miRNA的检测,其中Chen等[2]设计的茎环RT引物不仅解决了miRNAs短小难以扩增的问题,还可区分碱基差别在3'端的同家族成员,配合Taqman探针特异性高、费时短等优点,是目前检测miRNAs普遍采用的方法,但成本较高。在本研究中,我们建立了LDR-qPCR法,不需经过逆转录过程,仅依赖于一对茎环结构探针与靶miRNA的特异性杂交以及连接酶的忠实性连接,就能非常特异地识别出序列间的差异;两探针相连端茎环结构的设计能有效地防止靶miRNA序列不存在时探针的自身连接,避免假阳性结果[1]。扩增产物凝胶电泳和熔解曲线分析仅出现单一条带或预期熔解峰,测序分析为miR-122a序列,均证明其特异性。如此,在后续的实时定量PCR过程中只需采用通用型荧光燃料SYBR Green I即可进行信号检测,大大降低了成本。同时,我们的结果还显示此方法重复性好,与茎环RT-qPCR法检测结果具有良好的相关性(r=0.996),且检测范围更宽、灵敏度更高。
2002年Lagos-Quintana等[5]对小鼠不同组织中的miRNAs进行鉴定和分析,发现在其肝脏组织中miR-122高度特异表达,并克隆出其完整的碱基序列,同时在小鼠的肝脏中还克隆出miR-122的亚型,分别是miR-122a和miR-122b,两者只是在3'末端分别多一个碱基U或A。随后的研究发现,miR-122/122a在人类肝脏中也高度表达,可通过调控其靶基因的表达参与肝脏的分化和功能维护;实际上最为关键的似乎是调节肝脏胆固醇和脂肪酸的代谢[6];而在肝癌组织或肝癌来源的各种细胞株中表达下调,是肝癌的一个潜在生物标志[7]。不过,有关miR-122在肝外肿瘤或细胞株中的表达和功能研究还鲜有报道。本研究发现乳腺癌细胞MCF-7也能表达miR-122a,且其表达量与雌激素水平呈负相关性:利用活性碳处理的FBS从培养基中去除类固醇激素后发现,无激素培养MCF-7细胞表达miR-122a明显上调,而加入雌二醇后miR-122a的表达又受到抑制,表现为一定程度的下调。据报道17β-雌二醇、胰岛素以及类胰岛素生长因子可作为分裂素促进乳腺癌细胞生长,其机制部分与调节脂肪酸合成有关[8],而miR-122是脂肪酸代谢的主要调控因子,由此,我们推测:miR-122可能在无激素环境下对乳腺癌细胞的生长和生存起重要作用,其中雌激素是关键调节因子之一,值得进一步研究。
综上所述,本研究成功建立了检测miR-122a的LDR-qPCR法,此方法特异性高,且检测范围和灵敏度均优于茎环RT-qPCR法,有推广应用前景;同时我们的研究还发现乳腺癌细胞也表达miR-122a并受雌激素的调节,开辟了miR-122在肝肿瘤外的功能研究,而其具体的功能及激素调节机制还有待阐明。
[1]Li J,Yao B,Huang H,et al.Real-time polymerase chain reaction microRNA detection based on enzymatic stem-loop probes ligation[J].Anal Chem,2009,81(13):5446-5451.
[2]Chen C,Ridzon DA,Gugler KJ,et al.Real-time quantification of microRNAs by stem-loop RT-PCR[J].Nucleic Acids Res,2005,33(20):e179.
[3]Sun J,Lian R,Qiu J,et al.Microarray-based analysis of microRNA expression in breast cancer stem cells[J].Exper Clin Cancer Res,2010,29:174.
[4]Wickramasinghe NS,Manavalan TT,Dougherty SM,et al.Estradiol downregulates miR-21 expression and increases miR-21 target gene expression in MCF-7 breast cancer cells[J].Nucleic Acids Res,2009,37(8):2584-2595.
[5]Lagos-Quintana M,Rauhut R,Yalcin A,et al.Identification of tissue-specific microRNAs from mouse[J].Curr Biol,2002,(9):735-739.
[6]Esau C,Davis S,Murray SF,et al.miR-122 regulation of lipid metabolism revealed by in vivo antisense targeting[J].Cell Metab,2006,3(2):87-98.
[7]Diao S,Zhang JF,Wang H,et al.Proteomic identification of microRNA-122a target proteins in hepatocellular carcinoma[J].Proteomics,2010,10(20):3723-3731.
[8]Menendez JA,Lupu R.Fatty acid synthase and the lipogenic phenotype in cancer pathogenesis[J].Nat Rev Cancer,2007,7(10):763-777.

