大肠杆菌不同SOD调控序列红色荧光蛋白报告基因载体的构建及功能鉴定
翁丛丛,方益民,黄润巧,林冬冬,张洪勤,施孟如
(温州医学院,浙江 温州 325035,1.生物系;2.生物学教学中心)
大肠杆菌不同SOD调控序列红色荧光蛋白报告基因载体的构建及功能鉴定
翁丛丛1,方益民1,黄润巧1,林冬冬1,张洪勤2,施孟如2
(温州医学院,浙江 温州 325035,1.生物系;2.生物学教学中心)
目的:通过将大肠杆菌BL21中MnSOD、FeSOD以及Cu/ZnSOD调控序列(包括启动子,-3518~+15 bp)与红色荧光蛋白(RFP)连接,构建成含SOD调控序列的红色荧光蛋白报告基因,用以研究不同SOD启动子对RFP的调控作用。方法:采用重组PCR技术,分别构建以MnSOD、FeSOD、Cu/ZnSOD启动子驱动的RFP报告基因载体,并将构建的融合基因通过T克隆转化入大肠杆菌,通过不同浓度Cd2+作用,分别诱导RFP表达。结果:正确构建了三种SOD-RFP融合基因,重组PCR结果与测序结果完全一致;该报告基因在室温静息状态下,红色荧光有微弱表达,经Cd2+诱导后,红色荧光亮度明显增强,其中MnSOD调控序列驱动的RFP表达最为明显。结论:该报告基因载体的成功构建,为研究SOD的基因表达调控机制和研制检测环境中污染物的微生物传感器提供了重要基础和工具。
超氧化物歧化酶;调控序列;重组PCR;红色荧光蛋白;基因,报告;大肠杆菌
当处于极端温度、重金属等环境中,生物体氧自由基产生过多或对氧自由基清除能力下降,氧自由基通过链式自由基反应损伤细胞。机体清除氧自由基的过程主要包括超氧化物岐化酶(superoxide dismutase,SOD)将氧自由基通过歧化反应转变为H2O2,随后过氧化氢酶(CAT)将H2O2转变为O2和H2O[4]。SOD是一种广泛存在于生物体中的金属酶,根据其所结合的金属离子的不同,可分为MnSOD(SODA)、FeSOD(SODB)、Cu/ZnSOD(SODC)三种类型[1-3]。段学军等[5]的研究表明,机体总SOD活性会随着重金属的刺激而增加,然而具体是哪一种SOD起主导作用尚未能阐明。因此,本实验应用重组PCR技术,分别构建以SODA、SODB、SODC启动子驱动的红色荧光蛋白(RFP)报告基因载体,经不同浓度Cd2+刺激,诱导RFP基因表达,通过检测RFP荧光强度变化,从而研究不同SOD启动子在Cd2+诱导下对RFP的调控作用。
1 材料和方法
1.1 材料
1.1.1 RFP报告基因和菌种:含RFP基因的大肠杆菌BL21由浙江大学惠赠。
1.1.2 主要仪器及试剂:Tanon-1600R凝胶成像分析仪(上海天能科技有限公司),PCR梯度扩增仪(德国EPPENGERF,AG),Centrifuge 5415高速冷冻离心机(德国,EPPENDORF),RF-5301PC荧光分光光度计(SHIMADZU),FV1000激光共聚焦显微镜(Olympus),柱式琼脂糖凝胶DNA回收试剂盒(中国捷瑞生物工程有限公司),T4DNA连接酶(5 U/L)(宝生物工程有限公司),DNA ladder(中国捷瑞生物工程有限公司),pMD18-T克隆试剂盒(宝生物工程有限公司)。
1.2 方法
1.2.1 PCR引物:表1中的十个引物为自行设计,根据GenBank里大肠杆菌BL21(NC_012971)的SODA、SODB、SODC基因调控序列,及浙江大学惠赠的RFP基因,用Primer Premier 5.0软件完成引物设计,引物由上海捷瑞公司合成(见表1)。

表1 PCR引物及条件
1.2.2 SODA、SODB、SODC以及相应重叠区域的RFP基因的扩增:挑取一个含RFP基因质粒的大肠杆菌BL21菌落到100 mL ddH2O中悬浮混匀,100 ℃沸水浴10 min,冰浴3 min,12000 r/min离心5 min,取上清液为DNA模板,以引物SOA-RFP-1L和SODARFP-2R用于扩增SODA,SODA-RFP-3L和SOD-RFP-4R用于扩增RFP;SODB-RFP-1L和SODB-RFP-2R用于扩增SODB,SODB-RFP-3L和SOD-RFP-4R用于扩增RFP;SODC-RFP-1L和SODC-RFP-2R用于扩增SODC,SODCRFP-3L和SOD-RFP-4R用于扩增RFP。PCR反应体系:共30μL,10×Buffer 3μL,EX Taq酶1 U(0.2 μL),dNTP(Mg2+)1μL,模板DNA 2μL,ddH2O 22 μL,引物R 1μL,引物L 1μL。PCR反应条件:94 ℃ 5 min;94 ℃ 40 s,55℃ 40 s,72 ℃ 2 min,扩增35个循环;最后72 ℃ 20 min。PCR产物于1%琼脂糖凝胶中电泳。按DNA胶回收纯化试剂盒操作说明进行PCR产物的纯化。
1.2.3 SOD调控序列和报告基因RFP的连接:分别将上述PCR产物SODA、SODB、SODC和相应RFP纯化回收后等摩尔混合,作为扩增的模板,分别以SODARFP-1L和SOD-RFP-4R,SODB-RFP-1L和SOD-RFP-4R,SODC-RFP-1L和SOD-RFP-4R为引物,反应体系和条件均和前面的相同。PCR产物于1%琼脂糖凝胶中电泳,按DNA胶回收纯化试剂盒操作说明进行PCR产物的纯化。所得产物即:SODA-RFP、SODB-RFP、SODC-RFP融合基因。
1.2.4 重组产物与pMD18-T载体的连接及转化:以SOD-RFP的融合基因为control insert,参照TaKaRa pMD18-T Simple Vector 的使用说明书进行。将连接产物转入BL21感受态细胞。
1.2.5 SOD-RFP重组子的筛选、鉴定:菌落PCR鉴定:单个菌落的转化子在200 mL LB液体培养基中振荡培养1 h,然后直接进行PCR,琼脂糖凝胶电泳检测。低温诱导鉴定:挑取菌落PCR阳性的白色菌落,用接种针点种至加入Amp的普通LB平板中,37℃恒温培养过夜,放入4 ℃冰箱中低温诱导,观察是否有肉眼可见红色的菌落。将红色菌落涂片后,在激光共聚焦显微镜558 nm激发光下,观察红色荧光。基因测序鉴定:将低温诱导发红的菌种划板保存,并送至上海桑尼生物科技有限公司进行基因测序鉴定。
1.2.6 Cd2+诱导pMD18-T-SOD-RFP在宿主大肠杆菌BL21中表达:参考文献[5],取50 mL LB液体培养基加入50 mL的100 mg/mL Amp+,并接种阳性克隆菌,置于37 ℃,200 r/min,培养12 h(过夜)后,以1:25的比例分别接种到含Cd2+浓度为0.03、0.3、3.0 mg/mL的培养基中,全温振荡培养箱200 r/min,37 ℃培养,并于2、5、10、22、28、34、46、52 h八个时间点检测生长曲线OD值以及荧光发光强度。
2 结果
2.1 SODA、SODB、SODC及RFP片段的扩增 分别以SODA-RFP-1L和SODA-RFP-2R,SODB-RFP-1L和SODB-RFP-2R,SODC-RFP-1L和SODC-RFP-2R以及SODRFP-3L和SOD-RFP-4R为引物从含RFP基因的大肠杆菌BL21基因组DNA中分别进行扩增,1%琼脂糖凝胶电泳得到长度分别约为300、200、150、600 bp的条带(见图1),与预期SODA 284 bp、SODB 150 bp、SODC 165 bp、RFP 605 bp大小基本相符。
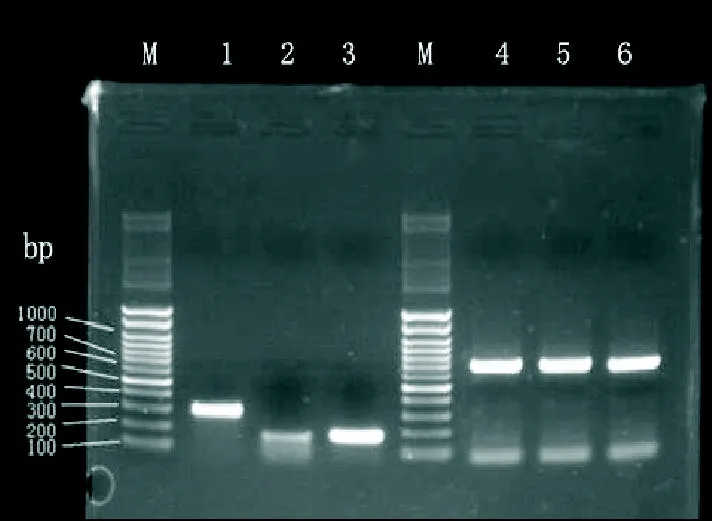
图1 SOD调控序列基因及RFP基因PCR产物电泳结果
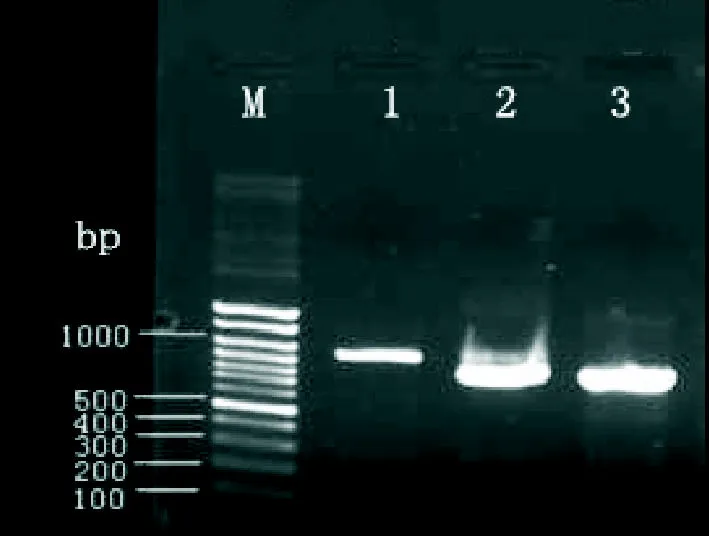
图2 SOD-RFP PCR产物结果
2.2 重组扩增SOD-RFP融合基因 以第一轮PCR产物纯化回收片段为模板,分别以SOD-RFP-1L和SODRFP-4R为引物进行扩增,琼脂糖电泳得到大小约为900、800、750 bp的条带(见图2),与预期SODARFP 889 bp、SODB-RFP 755 bp、SODC-RFP 770 bp大小基本相符。
2.3 T-A克隆重组产物的鉴定 将融合基因SODRFP扩增纯化产物与pMD-18 T载体连接后,转化入BL21感受态细胞,取白色单菌落,进行菌落PCR和低温诱导鉴定鉴定,结果表明重组载体中已插入融合基因SOD-RFP(见图3),挑取经低温诱导出现红色的菌落,涂片后,激光共聚焦显微镜558 nm激发光检测(×200),可以观察到明显的红色荧光(见图4)。从图4中可以知道,荧光表达强度以SODA为调控序列的最强,而以SODC为调控序列的最弱。
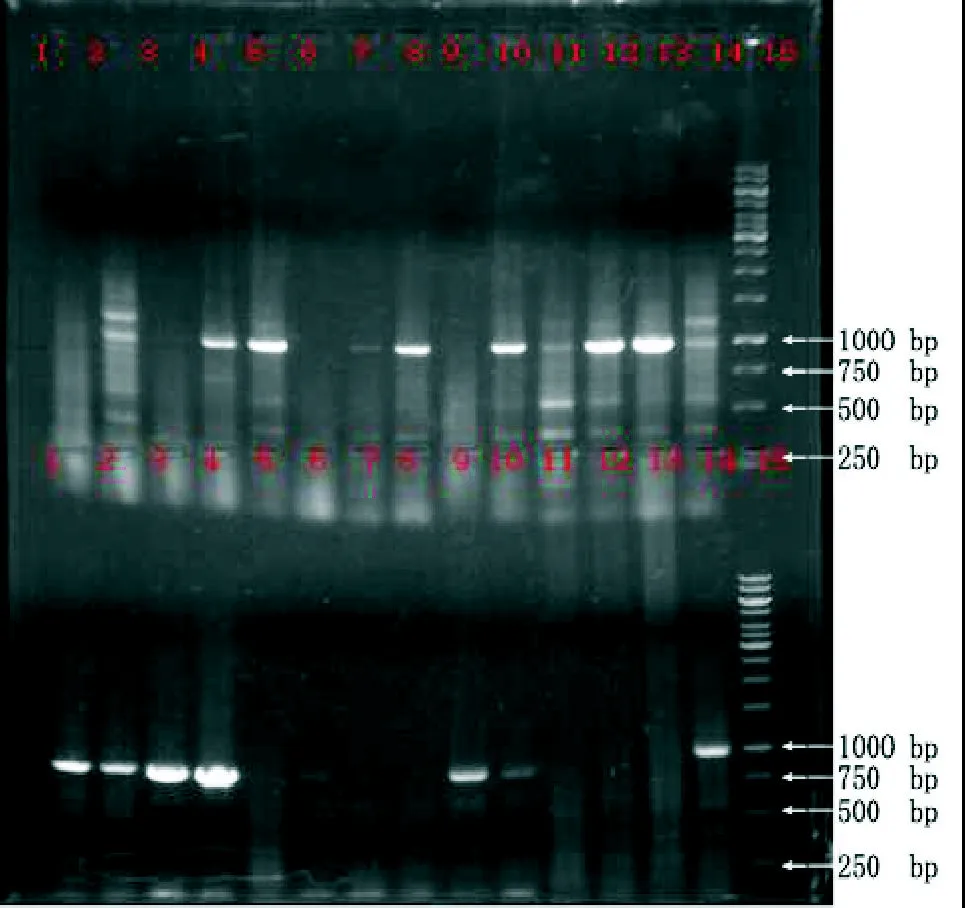
图3 菌落PCR鉴定

图4 以SODA(A)、SODB(B)、SODC(C)为调控序列分别在BL21 E.coli中表达红色荧光蛋白的情况
2.4 基因测序鉴定 将转入SODA-RFP、SODB-RFP、SODC-RFP的大肠杆菌送交上海桑尼生物科技有限公司,对其质粒DNA进行序列测定。测序结果通过在线BLAST(www.ncbi.nlm.nih.gov/Blast)和chromas软件进行序列分析,同源性为100%。
2.5 RFP诱导表达 分别用浓度为0.03、0.3、3.0 mg/mL的Cd2+及不加Cd2+相当量的纯净水进行诱导,并于2、5、10、22、28、34、46、52 h八个时间点检测生长曲线OD值以及荧光发光强度,以单位荧光强度(荧光强度与吸光度比值)为纵坐标,培养时间为横坐标,在室温静息状态下,可见微弱的红色荧光,经Cd2+诱导后,红色荧光亮度明显增强,其中0.03、0.3 mg/mL作用下SODA调控序列驱动的RFP表达最为明显,3.0 mg/mL作用下,3种SOD驱动的RFP表达全部受抑制(见图5)。

图5 SODA/B/C-RFP在4种不同Cd2+浓度下的单位菌量荧光强度
3 讨论
随着工农业生产的快速发展和人们生活水平的提高,人工合成化学物质的种类及用量急剧增加,农药、化肥、重金属、工业废水、生活垃圾等大量排入环境,对环境造成了严重污染[6-10]。恶劣环境的胁迫往往使自然界的生物体机体内活性氧代谢失衡,产生大量的超氧阴离子自由基,使机体内的酶失活,进而影响生物体生长。SOD作为生物机体防御过氧化损伤系统的关键酶之一,在真核及原核细胞中广泛存在分布,它对于清除氧自由基,防止氧自由基破坏细胞的组成、结构和功能,保护细胞免受氧化损伤具有十分重要的作用。
RFP是人们从珊瑚虫中克隆的一种与绿色荧光蛋白(GFP)同源的荧光蛋白。它无需底物存在,可在558 nm激发光下发射红光,具有灵敏度高、耐受多种苛刻环境条件等特点[11-12]。它作为一种新兴的、具有重要意义的分子标记,已经被广泛应用于生物传感器、生物发光材料和光化学等领域,有着巨大的应用价值[13]。我们以RFP作为报告基因,通过观察红色荧光强度来反映体内SOD的活性,了解三种SOD调控序列对RFP的调控作用。
本实验将含启动子和操纵子的Cu/ZnSOD、FeSOD、MnSOD基因调控序列与RFP报告基因通过重组PCR技术[14]进行融合,构建了一个有效地报告基因载体,较之传统的DNA重组技术,重组PCR不仅克服了小片段回收困难的问题,而且使重组体的形成过程完全不依赖于限制性内切酶和连接酶,大大简化了片段连接过程,从根本上摆脱了DNA 片段内部酶切位点的束缚。
构建的三种SOD-RFP融合基因在室温静息状态下有微弱的红色荧光,经不同浓度重金属镉诱导后,荧光强度均明显增加,表明其总SOD变化趋势与段学军等[5]的研究结果相符。本实验证实,SOD活性的变化主要是由SODA的表达变化引起的,SODA作为调控序列对后续报告基因的调控作用更加明显。该报告基因载体的成功构建,为研究SOD的基因表达调控机制提供重要的基础和工具。
目前,环境污染物的测定主要有理化分析方法和生物学方法[15]。利用SOD启动子的这种诱导性表达性能,通过对红色荧光强度的检测,我们能够把构建的报告基因载体转入大肠杆菌作为检测水体环境中刺激因子的微生物传感器,对重金属、可溶性有机物等污染物进行初筛。由于实验材料价廉易得,不需要昂贵的仪器设备和复杂的实验手段,简单快速,易操作,成本低,更易在欠发达地区或基层检测机构进行。
[1] 田春美,钟秋平.超氧化物歧化酶的现状研究进展[J]. 中国热带医学,2005,5(8):1730-1732.
[2] 陈淮扬,刘望荑.从超氧化物歧化酶的分布与结构看其分子进化[J].生物化学与生物物理进展,1996,23(5):408-412.
[3] Patel RN, Singh N,Shukla KK, et al. Synthesis, structure and biomimetic properties of Cu(II) - Cu(II) and Cu(II) - Zn(II)binuclear complexes:possible models for the chemistry of Cu-Zn superoxide dismutase[J].J Inorg Biochem,2005,99(2):651-663.
[4] Stefan IL,Irwin F.The effects of superoxide dismutase on H2O2formation[J]. Free Radic Biol Med,2007,42(10):1464-1469.
[5] 段学军,闵航. 稻田土壤细菌对重金属镉的氧化应激反应研究[J].安全与环境学报,2005,5(2):50-53.
[6] Zikic RV,Stain A, Saicic ZS, et al. The activities of superoxie dismutase, catalase and ascorbic acid content in the liver of goldfish(Carassius auratus gibelio Bloch) exposed to cadmium[J]. PhysiolRes,1996, 45(6):479-481.
[7] Caceresa T, Hea W, Naidu R,et al. Toxicity of chlorpyrifos and TCP alone and in combination to Daphnia carinata:The influence of microbial degradation in natural water [J].Water Res,2007,41 (19):4497-4503.
[8] Zaidi BR, Hinkey LM, RodriGuez NR, et al. Biodegradation of toxic chemicals in Guayanilla Bay, Puerto Rico[J]. Mar Pollut Bull,2003,46 (4):418-423.
[9] Erikssone E, Baun A, Scholes L, et al. Selected stormwater priority pollutants:a European perspective[J].Sci Total Environ,2007,383(1-3):41-51.
[10]Fatta D, Michael C, Michaelidou C, et al. Pesticides,volatile and semivolatile organic compounds in the inland surface waters of Cyprus[J].Desalination,2007,215(1-3):223-236.
[11]Baird GS,Zacharias DA,Tsien RY.Biochemistry,mutagenesis,and oligomerization of DsRed, a red fluorescent protein from coral[J]. Proc Natl Acad Sci USA,2000,97(22):11984-11989.
[12]Chalfie M,Tu Y, Eusk irchen G,et al.Green fluorescent protein as a maker for gene expression [J]. Science,1994,263(5148):802-805.
[13]Heim R,Cubitt AB,Tsien RY. Improved green fluorescence[J].Nature,1995,373(6516):663-664.
[14] 王皓,康现江,王琦,等.重组PCR技术研究进展和应用[J].中国生物工程杂志,2007,5(27):355-363.
[15]Ren S,Assessing wastewater toxicity to activated sludge:recent research and developments[J]. Environ Int,2004,30(8):1151-1164.
Construction and functional identification of red fluorescent protein reporter gene vector driven by different SOD regulatory sequence inE.coli
WENG Congcong*,FANG Yimin,HUANG
Runqiao,LIN Dongdong,ZHANG Hongqin,SHI Mengru.﹡
*Biology Department of Wenzhou Medical College,Wenzhou,325035
Objective:To construct red fluorescent protein(RFP)reporter gene vectors driven by different SOD regulatory sequence inE.colithrough fusing MnSOD,FeSOD,Cu/ZnSOD regulatory sequence and RFP gene in BL21E.coli.Methods:Red fluorescent protein(RFP)reporter gene and MnSOD,FeSOD,Cu/ZnSOD regulatory sequence were fused using recombinant PCR technology. And then the SOD-RFP genes connected with pMD18-T plasmid were transfered intoE.coli. The RFP expressed after stimulated by different concentrations Cd2+respectively.Results:Three SOD-RFP fusion genes were correctly constructed,which were proved by DNA sequencing;the reporter gene expressed a little of red flouresecent protein in the resting state at room temperature,but more red flouresecent intensity was obviously increased after Cd2+induction and SODA regulatory sequence was the most obviously RFP expression driven.Conclusion:The red fluorescent protein reporter gene vetors driven by different SOD regulatory sequence inE.colihave been constructed successfully with a sensitive response to Cd2+stimulation. This system provides an important basis and convenient tool for the further study of the regulatory mechanism of SOD gene expression and development of microbial sensor to detect the environmental pollutants.
superoxide dismutase;regulatory sequence;recombinant PCR;red fluorescent protein;reporter gene;E.coli
Q78
A
1000-2138(2012)01-0048-05
2011-05-03
翁丛丛(1988-),女,浙江瑞安人,本科生。
指导老师:施孟如,实验师,Email:dreamlike007@163.com。
丁敏娇)
·高教研究·

