线粒体D-环区单核苷酸多态性与食管癌患者预后关系的研究
范海燕,杨华,李胜棉,郭占军
线粒体D-环区单核苷酸多态性与食管癌患者预后关系的研究
范海燕,杨华,李胜棉,郭占军
目的探讨线粒体D-环区(D-loop区)单核苷酸多态性与食管癌患者预后的关系。方法选择2003—2004年在河北医科大学第四医院胸外科住院行食管癌根治性手术切除的食管癌患者55例,采用聚合酶链反应对其手术切除标本进行线粒体D-loop区测序。通过电话对患者进行随访并计算5年生存率,分析其与患者临床特征的关系。参照基因数据库AC_000021号从已完成的食管癌患者线粒体D-loop区1 000 bp序列中找出单核苷酸多态性位点,分析不同单核苷酸多态性位点与患者5年生存率的关系。结果本组55例患者均完成了5年的随访,共生存20例。不同性别、年龄(以55岁为界)及有无淋巴结转移食管癌患者5年生存率比较,差异均无统计学意义(P>0.05);不同TNM分期食管癌患者5年生存率比较,差异有统计学意义(P<0.05)。共找出88个单核苷酸多态性位点,采用Kaplan-Meier方法绘制生存曲线发现,仅16278和16399位点与食管癌患者的生存期有关。16278T型食管癌患者与16278C型患者、16399G型食管癌患者与16399A型患者5年生存率比较,差异均有统计学意义(χ2值分别为9.413和11.225,P<0.05)。采用Cox比例风险回归模型分析发现,16278T/C〔OR=3.638,95%CI(1.191,11.111)〕和16399G/A〔OR=5.096,95%CI(1.647,15.769)〕是食管癌患者预后的独立预测因子(P<0.05)。结论线粒体D-loop区单核苷酸多态性可作为食管癌患者预后的预测因子,有助于早期判断食管癌患者预后,更好地指导临床治疗,对估计生存期较短的术后患者可给予更积极的辅助治疗,以提高患者生存率。
多态性,单核苷酸;线粒体DNA;食管鳞状细胞癌;预后
食管癌是常见的恶性肿瘤之一,我国北方地区为食管癌高发区,年发病率>125/100 000,累积病死率男性约为20%,女性约为25%[1]。尽管近年来食管癌的诊断和治疗有了长足发展,但由于其侵袭性较高,患者的预后仍较差。研究发现,患者的一般状况、TNM分期和局部淋巴结转移等是食管癌患者预后的预测因子;一些分子机制的改变如P53基因突变和核因子κB(NF-κB)表达水平也可能影响食管癌患者的预后[2]。
线粒体DNA(mtNDA)处在活性氧自由基和其他自由基包围之中,缺乏组蛋白保护,且其内部缺乏完整的修复系统,与核内基因相比,mtDNA较容易发生突变[3]。线粒体D-环区(D-loop区)由1 122 bp组成(核苷酸16024-16569位点和1-576位点),含有复制起始点和转录启动子,多种肿瘤细胞中存在mtDNA广泛变异,近年来包括食管癌在内的多种肿瘤细胞中线粒体D-loop区已成为基因突变和多态性研究的热点[4-5]。本研究旨在探讨线粒体D-loop区单核苷酸多态性(SNPs)与食管癌患者预后的关系。
1 资料与方法
1.1 一般资料选择2003—2004年河北医科大学第四医院胸外科住院行食管癌根治性手术切除的食管癌患者55例,其中男34例,女21例;年龄39~76岁,平均(59.3±8.6)岁;患者ECOG评分(美国东部肿瘤协作组Eastern Cooperative Oncology Group制定的肿瘤活动状态评分)均为0分,术前均未进行任何临床药物治疗及放、化疗;患者均来自中国河北省食管癌高发地区,术后肿瘤切除标本经病理学检查证实均为鳞状细胞癌。
1.2 检测方法患者术前均空腹抽取外周静脉血,当天提取线粒体后置于-80℃恒温冰箱保存。采用聚合酶链反应(PCR)法提取mtDNA,全血mtDNA萃取试剂盒由美国Madison生产。迅速扩增mtDNA的D-loop区,扩增上游引物为5'-C CCCATGCTTACAAGCAAGT-3',下游引物为5'-GCTTTGAGGAGGTAAGCTAC-3',长度为1 000 bp,扩增参数按照剂盒说明。使用美国ABI PRISM 3100 DNA序列分析仪将PCR扩增产物纯化测序,同一标本采用双向重复测序。
1.3 观察指标通过电话对患者进行随访并计算5年生存率,分析其与患者临床特征的关系。参照基因数据库AC_000021号从已完成的食管癌患者线粒体D-loop区1 000 bps序列中找出SNPs位点,分析不同SNPs位点与患者5年生存率的关系。
1.4 统计学方法应用SPSS 11.5统计学软件进行分析,采用Kaplan-Meier方法绘制生存曲线,组间比较采用log-rank检验;采用Cox比例风险回归模型进行多因素分析,以相对危险度(OR)及其95%可信区间(95%CI)表示;以P<0.05为差异有统计学意义。
2 结果
2.1 食管癌患者临床特征与5年生存率的关系本组55例患者均完成了5年的随访,共生存20例。不同性别、年龄(以55岁为界)及有无淋巴结转移食管癌患者5年生存率比较,差异均无统计学意义(P>0.05);不同TNM分期食管癌患者5年生存率比较,差异有统计学意义(P<0.05,见表1)。

表1 食管癌患者临床特征与5年生存率的关系(n=55)Table 1 Relationship between clinical characteristics and 5-year survival rate in ESCC patients
2.2 线粒体D-loop区SNPs与患者5年生存率的关系共找出88个SNPs位点,绘制生存曲线发现,仅16278和16399位点与食管癌患者的生存期有关。16278T型食管癌患者与16278C型患者、16399G型食管癌患者与16399A型患者5年生存率比较,差异均有统计学意义(χ2值分别为9.413和11.225,P<0.05,见图1、2)。
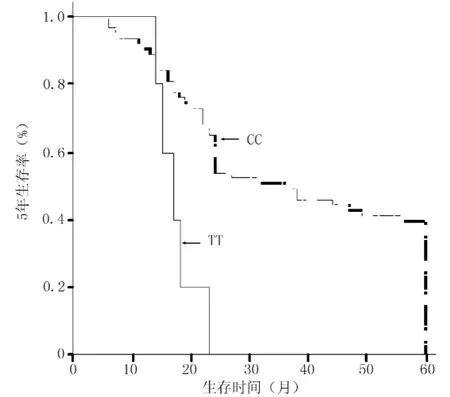
图1 多态位点16278CC与TT基因型患者5年生存率比较Figure 1 Comparison of 5-year survival rate between the patients with 16278CC or TT allele
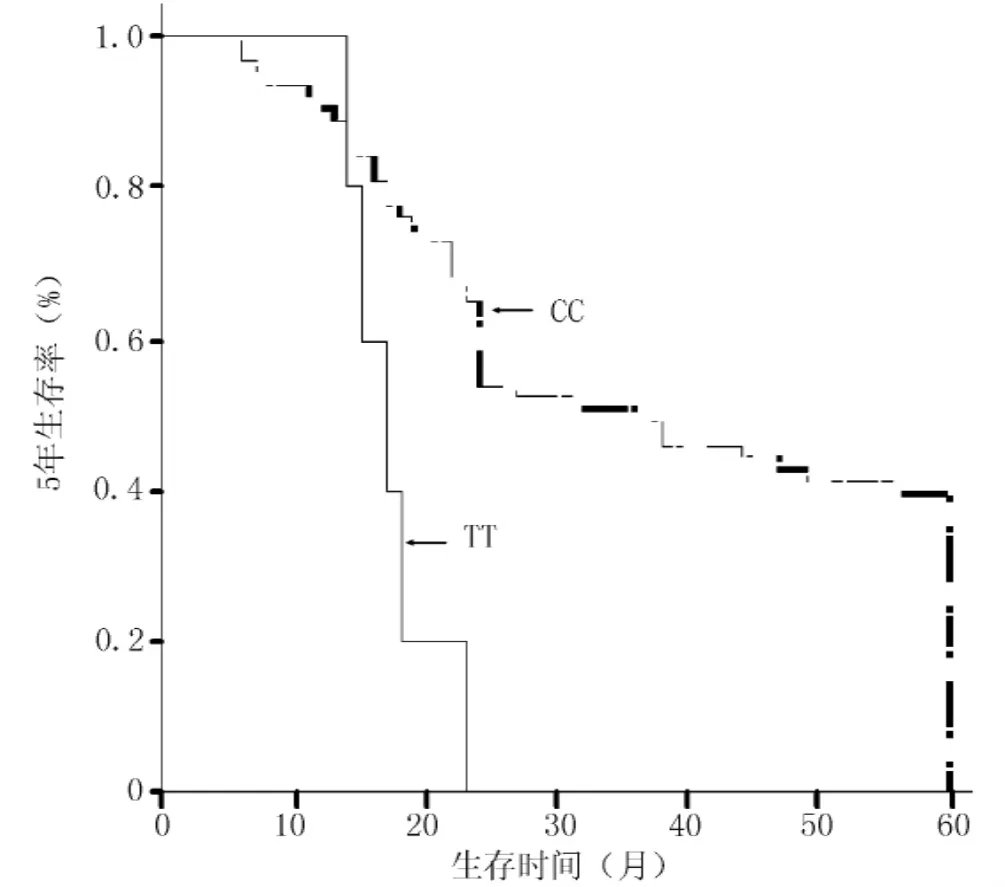
图2 多态位点16399AA与GG基因型患者生存期比较Figure 2 Comparison of 5-year survival rate between the patients with 16399AA or GG allele
2.3 多因素分析采用Cox比例风险回归模型分析TNM分期,16278T/C、16399G/A基因型与患者5年生存率的关系,结果发现,16278T/C、16399G/A是食管癌患者预后的独立预测因子(P<0.05,见表2)。
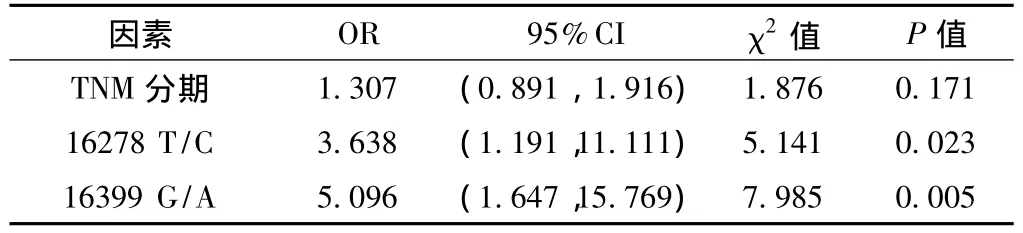
表2 食管癌患者Cox比例风险回归模型5年生存率多因素分析Table 2 Multivariate analysis of prognostic factors associated with 5-year survival rate in ESCC patients by Cox proportional hazards model
3 讨论
mtDNA是哺乳动物细胞内惟一的核外遗传物质。1981年Anderson等首次在《Nature》杂志上公布了人类mtDNA基因组的全部核苷酸序列,并绘制了详细的功能图谱。研究表明,mtDNA氧化损伤可能与人类衰老、神经退行性疾病、糖尿病以及肿瘤等疾病的发生有关[6]。近年来研究发现,很多实体恶性肿瘤存在高频率的mtDNA突变。D-loop区是mtDNA的非编码区,是mtDNA转录调控位点。对头颈部肿瘤、膀胱癌、乳腺癌等进行的研究发现,D-loop区是mtDNA突变的热点区域[7]。SNPs是指染色体基因组水平上单个核苷酸变异引起的DNA序列多态性,已成为继限制性片断长度多态性(RFLP)标志和微卫星标志后的第三代遗传标志,其在多基因疾病的易感基因研究领域应用较广。线粒体D-loop区SNPs位点与多种肿瘤的致癌风险和疾病预后有关[8-9]。
本研究结果发现,16278T型食管癌患者5年生存率低于16278C型患者,16399G型食管癌患者5年生存率低于16399A型患者;16278C/T和16399A/G基因型与食管癌患者预后相关,是食管癌患者预后独立的预测因子。16278T基因型患者死亡风险是16278C基因患者3.638倍,16399G基因型患者死亡风险是16399A基因型患者的5.096倍。提示mtDNA D-loop区含有这两个基因型多肽位点者有罹患食管癌的倾向,预后可能更差。
线粒体D-loop区在线粒体基因复制和表达过程中起着重要作用,这个区域的基因突变有可能影响mtDNA复制并导致氧化呼吸链的改变,导致大量活性氧自由基释放而损伤细胞核基因组,在肿瘤的发生、发展过程中起重要作用[10-11]。SNPs位点16278和16399位于线粒体D-loop高变区1(HV1),此序列具有高突变率,但其具体作用机制尚未明确[12],可能是使线粒体基因组转录发生改变,导致大量活性氧自由基产生,而氧自由基调节机制异常促进肿瘤的发生、发展,进而影响患者的预后。本研究同时发现,食管癌患者的性别、年龄、有无有淋巴结转移与其5年生存率无关,而TNM分期与5年生存率有关,但性别、年龄、有无淋巴结转移与患者生存时间的关系都显示有差异趋势,可能与本研究样本量较小、观察时间较短有关,有待进一步增加样本量,延长随访时间进行深入研究。
总之,线粒体D-loop区SNPs可作为食管癌患者预后的预测因子,有助于早期判断食管癌患者预后,更好地指导临床治疗,对估计生存期较短的术后患者可给予更积极的辅助治疗,以提高患者生存率。长远来看,应建立以mtDNA D-loop序列的SNPs分析为平台的食管癌预后评价系统,并推广应用于其他消化道恶性肿瘤分子研究。
1 Blot WJ,Li JY.Some considerations in the design of a nutrition intervention trial in Linxian,People's Republic of China[J].Natl Cancer Inst Monogr,1985(69):29-34.
2 Blanchard P,Quero L,Hennequin C.Prognostic and predictive factor of esophageal carcinoma[J].Bull Cancer,2009,96(4):379-389.
3 Shadel GS,Clayton DA.Mitochondrial DNA maintenance in vertebrates[J].Annu Rev Biochem,1997(66):409-435.
4 DiMauro S,Schon EA.Mitochondrial DNA mutations in human disease[J].Am J Med Genet,2001,106(1):18-26.
5 Wang L,McDonnell SK,Hebbring SJ,et al.Polymorphisms in mitochondrial genes and prostate cancer risk[J].Cancer Epidemiol Biomarkers Prev,2008,17(12):3558-3566.
6 Hibi K,Nakayama H.Mitochondrial DNA alteration in esophageal cancer[J].Int J Cancer,2001,92(3):319-321.
7 Fliss MS,Usadel H,Caballero OL,et al.Facile detection of mitochondrial DNA mutations in tumors and bodily fluids[J].Science,2000,287(5460):2017-2019.
8 Bai RK,Leal SM,Covarrubias D,et al.Mitochondrial genetic background modifies breast cancer risk[J].Cancer Res,2007,67(10):4687-4694.
9 Wang L,McDonnell SK,Hebbring SJ,et al.Polymorphisms in mitochondrial genes and prostate cancer risk[J].Cancer Epidemiol Biomarkers Prev,2008,17(12):3558-3566.
10 Suzuki M,Toyooka S,Miyajima K,et al.Alterations in the mitochondrial displacement loop in lung cancers[J].Clin Cancer Res,2003,9(15):5636-5641.
11 Richard SM,Bailliet G,Paez GL,et al.Nuclear and mitochondrial genome instability in human breast cancer[J].Cancer Res,2000,60(15):4231-4237.
12 Stoneking M.Hypervariable sites in the mtDNA control region are mutational hotspots[J].Am J Hum Genet,2000,67(4):1029-1032.
Relationship between Single Nucleotide Polymorphisms in Mitochondrial Displacement-loop and Prognosis of Esophage-al Squamous Cell Car
FAN Hai-yan,YANG-hua,LI Sheng-mian,et al.Department of Gastroenterology and Hepatology,the Fourth Hospital of Hebei Medical University,Shijiazhuang 050011,China
ObjectiveTo study the relationship between single nucleotide polymorphisms(SNPs)in mitochondrial DNA(mtDNA)displacement-loop(D-loop)region and prognosis of patients with esophageal squamous cell carcinoma(ESCC).Methods55 ESCC patients underwent radical operation in department of thoracic surgery of the Fourth Hospital of Hebei Medical University from 2003 to 2004 were selected.The D-loop region of mtDNA was sequenced by using polymerase chain reaction method.The patients were followed up by telephone for five years,and then the five year survival rate was calculated and its relationship with clinical features was analyzed.AC_000021 database was referred to in order to find SNPs from the 1 000-bp mitochondria D-Loop region,and the relationship between SNPs and five year survival rate was analyzed.ResultsAll the 55 patients completed the five-year following up and 20 cases survived.The survival rate between patients with different sex,age(55 as dividing line)and whether there was lymphatic metastasis or not showed no statistically significant differences(P>0.05).The five-year survival rate between patients with different TNM staging showed statistically significant difference(P<0.05).88 SNPs sites were detected in the 1 000-bp mitochondria D-Loop region.When drawing the survival curves with Kaplan-Meier method,only SNP sites of nucleotides 16278C/T and 16399A/G showed correlation with the survival of patients.The five-year survival rate between patients with 16278T and 16278C and patients with 16399G and 16399A both showed statistically significant differences(χ2=9.413 and 11.225,P<0.05).Cox proportional hazards regression models showed that 16278T/C〔OR=3.638,95%CI(1.191,11.111)〕and 16399G/A〔OR=5.096,95%CI(1.647,15.769)〕were independent risk factors for the prognosis of ESCC patients.ConclusionSNPs in the D-loop are independent prognostic factors for patients with ESCC and it is conducive to the early prediction of the prognosis of ESCC patients.Positive adjuvant therapy should be performed on patients with estimated shorter survival to improve their survival rate.
Polymorphism,single nucleotide;Mitochondrial DNA;Esophageal squamous cell carcinoma;Prognosis
R 735.1
A
1007-9572(2012)11-3839-03
10.3969/j.issn.1007-9572.2012.11.090
050011河北省石家庄市,河北医科大学第四医院消化内科
郭占军,050011河北省石家庄市,河北医科大学第四医院消化内科;E-mail:zjguo5886@yahoo.com.cn
2012-06-10:
2012-09-21)
(本文编辑:鹿飞飞)

