转基因玉米的多重PCR-毛细管电泳紫外检测技术研究
张春娇,许文涛,,程国灵,赵维薇,戴蕴青,罗云波,黄昆仑,,*
(1.中国农业大学食品科学与营养工程学院,北京100083;2.农业部转基因生物食用安全检验监督测试中心,北京100083)
转基因玉米的多重PCR-毛细管电泳紫外检测技术研究
张春娇1,许文涛1,2,程国灵1,赵维薇1,戴蕴青2,罗云波1,黄昆仑1,2,*
(1.中国农业大学食品科学与营养工程学院,北京100083;2.农业部转基因生物食用安全检验监督测试中心,北京100083)
建立了转基因玉米Ly038、Mon863和Mon810的品系特异性基因多重PCR产物的毛细管电泳-紫外检测方法。根据三种转基因玉米的基因序列设计多重PCR引物,优化PCR扩增体系和条件,以8.0g/L羟乙基纤维素为筛分介质,用毛细管电泳-紫外检测法同时检测出三种玉米的品系特异性基因,11.195min内即可完成检测。用Origin软件对电泳结果进行分析并得出不同范围的DNA曲线方程,样品出峰时间的相对误差在1.03%~5.02%之间。并对纯化前后的样品进行了毛细管电泳分离的对照,发现PCR产物纯化后更适于紫外检测的毛细管电泳。多重PCR具有节约模板、节省试剂和缩短检测时间的优点,毛细管相对于传统的琼脂糖凝胶电泳,具有高效、快速、灵敏且经济环保的优点,所以此方法可广泛地应用于食品安全检测和临床检验等领域中。
转基因玉米,多重PCR,毛细管电泳,紫外检测
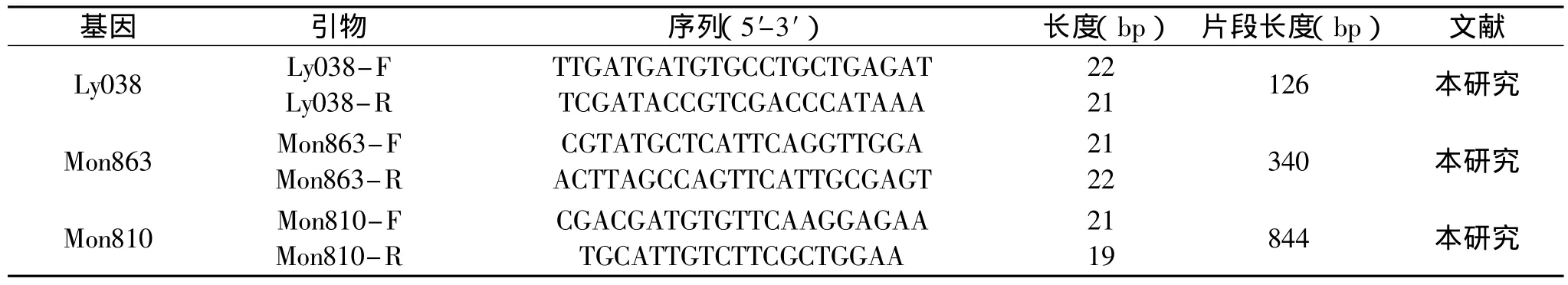
表1 多重PCR引物
1 材料与方法
1.1 材料与仪器
转基因玉米 Ly038、Mon863和Mon810为实验室自有品种;rTaq DNA聚合酶、dNTP(含 dATP、dGTP、dCTP和dTTP) TaKaRa公司;普通DNA产物纯化试剂盒 TIANGEN公司;羟乙基纤维素、Tris -base Amersco公司;引物 上海生物工程技术有限公司合成,PAGE纯化。
3DCE毛细管电泳仪 安捷伦;熔融石英毛细管长46.5cm(有效长度38cm),内径75μm,河北永年光导纤维厂。
1.2 实验方法
1.2.1 CTAB法提取转基因玉米基因组 将转基因玉米样品于液氮中研磨成粉末状,称取80mg加入到1.5mL的Eppendorf离心管中,加入1000μL CTAB溶液,65℃温育40min,其间混匀多次,4℃下12000r/min离心10min。取上清到一新管中,加入与上清等体积的Tris饱和酚∶三氯甲烷∶异戊醇(25∶24∶1),振荡均匀,4℃下12000r/min离心10min,并重复一次。取上清到一新管中,加入2/3体积的异丙醇[14],-20℃条件下静置20min,4℃下12000r/min离心10min。弃上清,加入适量的70%乙醇洗涤沉淀。吹干沉淀后用60μL无菌超纯水溶解,加入2μL RNA酶,37℃消化30min后,用1.0%的琼脂糖凝胶电泳对提取的基因组进行验证[15]。
1.2.2 引物设计 引物的选择是PCR反应能否成功进行的关键。选取三种品系的转基因玉米DNA序列,设计多重PCR产物,一条引物与玉米基因组匹配,另一条引物与转入的载体匹配。
本文设计了3对品系特异性引物,同时对三种转基因玉米Ly038、Mon863、Mon810的品系特异性基因进行扩增,一次性完成对三种玉米成分的筛选和鉴定。多重PCR引物的设计结果见表1。
社会实践的发展,不断提出需要解决的新课题,推动着人类的认识不断发展,人们在总结实践提供的新经验基础上,提出新理论,解决新问题。我国北方某县在20世纪80年代开始发展大田粮食作物喷灌,不仅有效缓解了当地农业用水紧缺状况,增加了城市供水,取得了实实在在的节水效益,而且在喷灌工程建设过程中充分调动多方积极性,做到了国家和地方、集体和农民共同投入,同时将土地经营规模适度集中,创造了喷灌技术的良好应用条件,加之良好的管理维护和浇地专业队等服务网络保障了喷灌系统的良好运行。正是因为对喷灌技术与设备的再认识,又把这些认识再次应用到具体的实践中去,在新的认识水平上开展农业节水喷灌工作,才取得了长久效益。
1.2.3 多重PCR反应体系和条件 PCR扩增体系如下(30μL):10×PCR buffer:3μL(10×PCR Buffer:100mmol/L Tris-HCl,500mmol/L KCl,15mmol/L MgCl2);2.5mmol/L dNTP:2.4μL;转基因玉米Ly038、Mon863和Mon810的品系特异性引物的终浓度依次为0.4、0.3和0.3μmol/L;Taq DNA聚合酶:0.4μL,提取的 DNA加入3μL作为模板,用 ddH2O水补足至30μL。
PCR的扩增条件如下:预变性:94℃,5min;变性:94℃,30s;退火:57℃,30s;延伸:72℃,30s;共设40个循环,终止延伸:72℃,7min。扩增结束后,用TIANGEN普通DNA产物纯化试剂盒对样品进行纯化。
1.2.4 毛细管电泳分离多重PCR产物 电泳开始之前要对新毛细管进行活化,前处理如下:1bar条件下(1bar=105pa)用0.1mol/L的盐酸冲洗毛细管5min;再用去离子水冲洗5min。活化后,在10bar的条件下填充8.0g/L的羟乙基纤维素5min。电泳缓冲液为TBE溶液(89mmol/L的 Tris,332mmol/L的硼酸,2mmol/L的EDTA,pH为7.4)。CE运行的参数如下:电泳温度为25℃,-5.0kV下电动进样20s,电泳的运行电压为-15.0kV,在Sig.260/8nm和Ref.350/ 80nm处检测。每次电泳结束后用8.0g/L的羟乙基纤维素冲洗毛细管3min,再进行下一次检测。
2 结果与分析
2.1 玉米样品的DNA提取
采用CTAB法提取了转基因玉米样品的基因组,CTAB法适用于一些深加工食物中的植物基因组提取[14]。1.0%琼脂糖凝胶电泳结果表明(见图1),玉米基因组的亮度良好,用Modulus多功能荧光计测定其浓度在10~15ng/μL之间,可以作为PCR反应的模板。
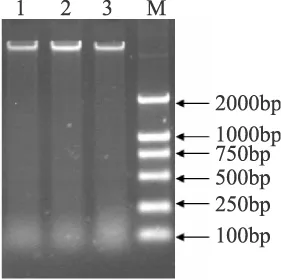
图1 转基因玉米基因组DNA琼脂糖电泳
2.2 多重PCR反应体系和条件的优化
影响多重PCR反应的因素有很多,其中退火温度、引物对的浓度和模板的量是比较关键的影响因素[16]。采用单因素实验分别对多重PCR体系的退火温度(56~62℃)、引物浓度(0.1~1.0μmol/L)、混合模板量(1~5μL)进行了实验。经过逐一优化,得到了三种转基因玉米的多重PCR扩增参数(详见1.2.3)。
2.3 毛细管电泳分离条件的优化
2.3.1 筛分介质浓度对DNA分离的影响 筛分介质是毛细管无胶筛分电泳能否达到分离效果的关键因素。本研究选取羟乙基纤维素为筛分介质。在一定的浓度范围内,筛分介质的浓度越大,DNA片段的分离效果越好,迁移时间逐渐变长,这是因为随着筛分介质浓度的提高,筛分孔径逐渐减小,DNA片段的迁移阻力也随之增大[17]。因此,要综合分离效果和迁移时间,确定一个最适的筛分介质浓度。本实验选用了分子生物学实验中最常见的 DNA Marker DL2000作为毛细管电泳的标记Marker,对筛分体系进行优化,考察了不同质量浓度(1.0~10.0g/L)的羟乙基纤维素对Marker的分离情况。由实验结果(图2)可知,当羟乙基纤维素的浓度小于5.0g/L时,DNA标准片段的电泳峰有所重叠;当浓度大于8.0g/L时,筛分介质的粘度过大,电泳时间变长。当筛分介质的浓度在5.0~8.0g/L之间时,随着筛分介质浓度的增大,Marker的各个条带的分离度越来越好。因此,本研究选取了质量浓度为8.0g/L的羟乙基纤维素作为CE的筛分介质。
2.3.2 电泳电压对DNA分离的影响 毛细管电泳的分离电压对DNA分子的迁移时间存在一定的影响[18]。一般而言,CE分离的电场强度在300~500V/cm的范围之间,综合考虑分析时间和分离效果,本文选择在320V/cm的场强下进行分离。体现到分离电压,压力为15kV,负压运行。
在优化好的毛细管电泳条件下,DNA Marker DL2000纯化前后的CE结果见图2。
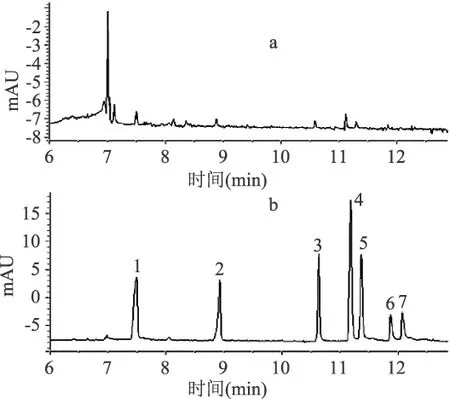
图2 纯化前(a)后(b)DNA Marker DL2000的毛细管电泳图
由图2可以看出,纯化前的DNA Marker的CE结果杂峰较多且峰形杂乱,无法对目的条带进行准确地判断。其原因可能是Marker的贮存液中含有某些在260nm处有紫外吸收的成分,从而导致在7min处出现杂质峰且目的条带的峰形不明显。
纯化后的DNA Marker的CE结果峰形完整,分离效果较好。DNA Marker的6个片段大小依次是100、250、500、750、1000、2000bp,而纯化后的 DNA Marker共出现7个峰,已知Marker中的750bp的浓度大概是其它条带浓度的两倍,因此可以确定前5个峰分别与100、250、500、750、1000bp相对应。6号峰和7号峰的峰形相似且峰面积较小,可能的原因有两点:此Marker专用于琼脂糖凝胶电泳,其灵敏度要比毛细管电泳低,在琼脂糖上无法看到的条带也可能会出现在CE的结果中,也许是Marker中混有微量的1000~2000bp之间的DNA片段所致;也有可能是2000bp的大片段DNA在纯化过程中出现了一定程度的降解而多出一个条带。因此,初步推测6号峰和7号峰均在2000bp左右。采用Origin软件对Marker的前5个峰进行分析并绘图(见图3),求得其曲线关系方程为:y=A2+(A1-A2)/[1+(x/x0)p],其中:A1=157.4062;A2=5145.82359;x0=12.69898;p=14.81059。6、7号峰的出峰时间依次为11.863min和12.063min,由公式计算出2000bp的出峰时间为12.2485min,6、7号峰与公式计算的出峰时间的相对误差分别为3.15%和1.51%,故初步判断7号峰为2000bp,6号峰是7号峰降解所致。一般而言,PCR产物的大小均在1000bp以内,所以6号峰的出现并不会影响对PCR产物大小的验证和判断,因此,采用DNA Marker DL2000作为毛细管电泳的标记Marker是可行的,不但可以降低实验成本,还可以提高实验进行的普遍性。
观察图2(b),可以看出前4个峰的出峰时间间隔较后面的时间间隔较大,这是由于每种浓度的筛分介质在不同范围内的分离效果有差异,可以推测出8g/L的筛分介质羟乙基纤维素对于100~750bp的分离效果要优于1000~2000bp的分离效果。因此,本文对于Marker的6个峰值(排除6号峰)做了两组曲线:1、2、3和4号峰为一组;3、4、5和7号峰为一组,以便对样品的CE结果进行有效的判断。采用Origin软件对以上两组数据进行分析并绘图(见图4和图5),其曲线关系方程分别为:y=A2+(A1-A2)/[1+(x/x0)p]和 y=A1+(A2-A1)/[1+10(logx0-x)p]第一组的参数为:A1=89.88261,A2=20148.47564,x0= 17.55886,p=7.53957;第二组的参数为:A1= 471.50899,A2= 2139.3661,Logx0= 11.52922,p=1.94856。
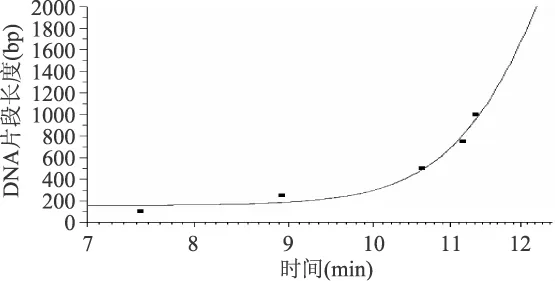
图3 Marker前5个条带的拟合曲线图
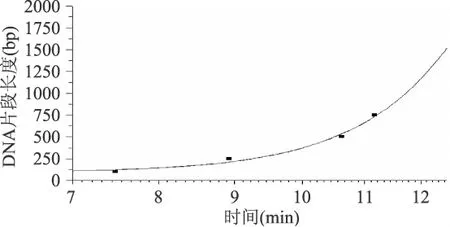
图4 Marker前4个条带的拟合曲线图
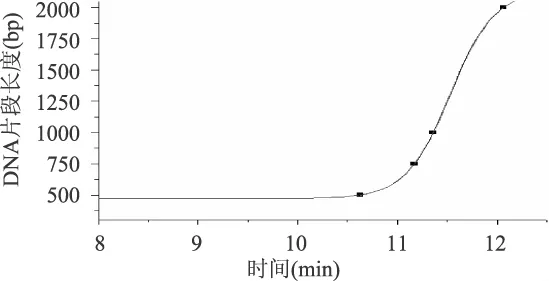
图5 Marker后4个条带的拟合曲线图
2.4 PCR产物纯化对毛细管电泳的影响
以单重PCR产物的原液和纯化后的单重PCR产物做为对照,在优化好的CE条件下进行分析分离。转基因玉米Ly038、Mon863和Mon810的品系特异性引物的单重PCR结果的纯化前后的CE结果分别见图6~图8。
从图6~图8中可以看出,3对品系特异性引物的单重PCR产物,纯化前的CE结果的杂峰很多,且杂质峰的出峰时间集中在6~7.5min之间。这说明PCR产物中含有一些除样品以外的具有紫外吸收物质,比如rTaq酶、Buffer、引物二聚体等等,由于其在紫外下有吸收而6~7.5min之间产生了杂峰。对3组样品纯化后的CE结果表明,纯化不但可以有效去除杂质峰,且在纯化中样品体积有所浓缩而增加了样品浓度,使目的条带的峰形更为突出。由此可见,PCR产物的纯化对于紫外检测的CE的分离具有很重要的作用和意义。由此可以推出,采用毛细管电泳分离多重PCR产物,纯化有着同样的重要性和必要性。
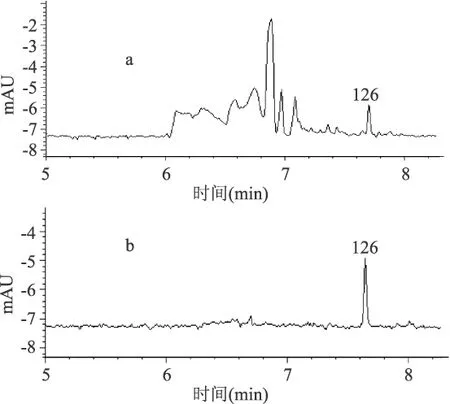
图6 纯化前(a)后(b)转基因玉米Ly038单重PCR产物的毛细管电泳图
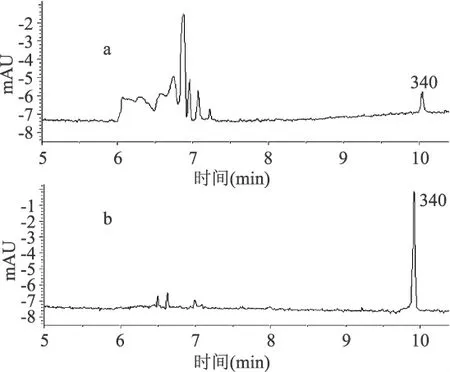
图7 纯化前(a)后(b)转基因玉米Mon863单重PCR产物的毛细管电泳图
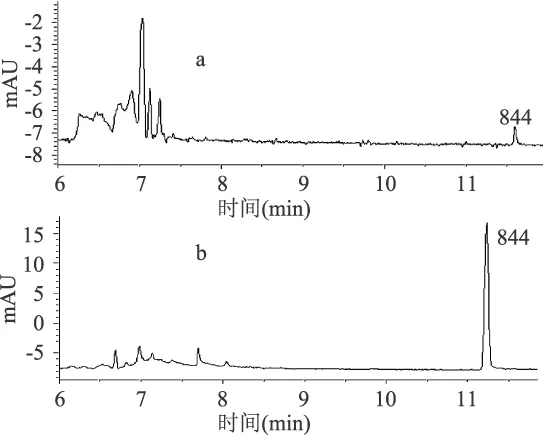
图8 纯化前(a)后(b)转基因玉米Mon810单重PCR产物的毛细管电泳图
2.5 多重PCR产物的毛细管电泳分离
在验证了样品的纯化对毛细管电泳的积极作用之后,对多重PCR产物进行了同样的纯化操作,在优化好的毛细管电泳条件下,对多重PCR产物进行分离。CE的分离结果见图9。
由图9可见,纯化后的多重PCR产物,PCR反应体系中的一些有紫外吸收的杂质得到了有效地去除,多重PCR产物得到了有效的分离,其峰形明显,分离效果良好。样品的出峰时间依次为 7.674、9.901、11.195min,由图4的曲线方程计算出1号样品和2号样品的出峰时间依次为 7.59584min和9.8325min,图5中的曲线方程计算出3号样品的出峰时间为11.25143min。样品出峰时间的相对误差依次为1.03%、0.70%和5.02%,均在10%以内,可见此方法的准确度良好,在11.195min即可实现对多重PCR产物的分离。
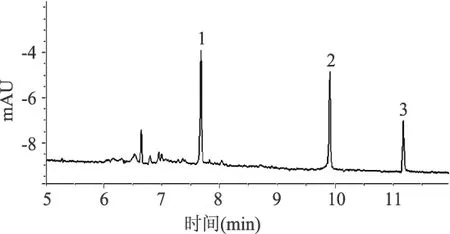
图9 多重PCR产物的毛细管电泳图
3 结论
本文成功地建立了紫外检测器下的无胶筛分毛细管电泳对多重PCR产物进行快速分离的方法,实现了对加工食物中是否含有转基因玉米品系Ly038、Mon863和Mon810的快速检测。采用了分子生物学实验中常见的DNA Marker DL2000作为毛细管电泳的标记Marker,不但降低了实验成本,而且具有简便性、普遍性和可行性。通过进行样品纯化前后的毛细管电泳对照实验,充分证明了纯化样品对于紫外检测器下进行的毛细管电泳的必要性。采用优化的毛细管电泳体系和条件,仅需11.195min即可完成对三种品系的转基因玉米的检测,并且将Origin绘图软件应用于CE结果的分析,通过建立不同范围的DNA曲线方程来对样品的CE结果进行有效的判断,验证了检测方法的准确度。可以作为一种检测手段,为今后检测更多品系的转基因玉米以及转基因作物奠定了基础,也为食品安全检测和临床检验等领域提供了一种可靠的检测和监测手段。
[1]Xu WT,Bai WB,Luo YB,et al.Research process in detection technique for genetically modified organisms[J].Journal of Agricultural Biotechnology,2008,16(4):714-722.
[2]Hemmer W,Pauli U.Labelling of food products derived from genetically engineered crops[J].A Survey on Detection Methods. European Food Research and Technology,1998(8):27-38.
[3]Onishi M,Matsuoka T,Kodama T,et al.Development of a multiplex polymerase chain reaction method for simultaneous detection of eight events of genetically modified maize[J].Journal of Agricultural and Food Chemistry,2005,53:9713-9721.
[4]Xu W T,Yuan Y F,Luo Y B,et al.Event-specific detection of stacked genetically modified maize Bt11×GA21 by UP-MPCR and real-Time PCR[J].Journal of Agricultural and Food Chemistry,2009,57(2):395-402.
[5]Yuan Y F,Xu W T,Zhai Z F,et al.Universal primermultiplex PCR approach for simultaneous detection of Escherichia coli,Listeria monocytogenes,and Salmonella spp.in food samples[J].Journal of Food Science,2009,74:446-452.
[6]Miguel H,Virginia G,Carolina S,et al.Recent advances in the application of capillary electromigration methods for food analysis and Foodomics[J].Electrophoresis,2008,29:294-309.
[7]Owens C V,Davidson Y Y,Kar S,et al.High-Resolution separation of DNA restriction fragments using capillary electrophoresis with near-IR,diode-based,laser-induced fluorescence detection[J].Analytical Chemistry,1997,69:1256 -1261.
[8]Hjerten S J.High-Performance electrophoresis elimination of electroendosmosis and solute adsorption[J].Journalof Chromatography,1985,347:191-198.
[9]Han F,Huynh B,Ma Yinfa,et al.High-Efficiency DNA separation by capillary electrophoresis in a polymer solution with ultralow viscosity[J].Analytical Chemistry,1999,71:2385-2389.
[10]Mejia E,Ding Y S,Mora M F,et al.Determination of banned sudan dyes in chili powder by capillary electrophoresis[J].Food Chemistry,2007,102:1027-1033.
[11]Chen H,Wu Y H,Song D Y,et al.On-line preconcentration and UV determination of DNA fragments by dynamic coating capillary electrophoresis and its application to detection of genetically modified oilseed rape based on PCR[J]. Microchemical Journal,2007,86(1):17-22.
[12]Ana J G,Yolanda P,Guillermina F.Capillary electrophoresis for analyzing pesticides in fruits and vegetables using solid-phase extraction and stir-bar sorptive extraction[J].Journal of Chromatography A,2005,1073:229-236.
[13]Mao HX,Li YQ,Pei XF,et al.Rapid detection of three foodborne pathogenic bacteria by multiplex polymerase chain reaction- capillary electrophoresis with Laser- Induced Fluorescence detector[J].Chinese Journal of Chromatography,2007,25(4):473-477.
[14]Xu W T,Huang K L,Guo F,et al.Postharvest grapefruit seed extract and chitosan treatments of table grapes to control Botrytis cinerea[J].Postharvest Biology and Technology,2007,46(1):86-94.
[15]Yang R,Xu WT,Luo YB,et al..Event-specific qualitative and quantitative polymerase chain reaction detection of roundup ready event GT73 based on the 3’-transgene integration sequence[J].Plant Cell Reports,2007,26:1821-1831.
[16]Shrestha HK,Hwu KK,Wang SJ,et al.Simultaneous detection of eight genetically modified maize lines using a combination of event-and construct-specific multiplex-PCR technique[J].Journal of Agricultural and Food Chemistry,2008,56:8962-8968.
[17]Swerdlow H,Zhang JZ,Chen DY,et al.Three DNA sequencing methods using capillary gel electrophoresis and laserinduced fluorescence[J].Analytical Chemistry,1991,63:2835 -2841.
[18]Liang DH,Chu B.High speed separation of DNA fragments by capillary electrophoresis in poly(ethylene oxide)-poly(propylene oxide)-poly(ethylene oxide)triblock polymer[J]. Electrophoresis,2005,19:2447-2453.
Study on genetically modified maize by multiplex polymerase chain reaction-capillary electrophoresis with UV detection
ZHANG Chun-jiao1,XU Wen-tao1,2,CHENG Guo-ling1,ZHAO Wei-wei1,DAI Yun-qing2,LUO Yun-bo1,HUANG Kun-lun1,2,*
(1..College of Food Science and Nutritional Engineering,China Agricultural University,Beijing 100083,China;2.The Supervision,Inspectionamp;Testing Center of Genetically Modified Orginisms Food Safety,Ministry of Agriculture,Beijing 100083,China)
Event-specific qualitative detection of three kinds of genetically modified(GM)maize by multiplex polymerase chain reaction-capillary electrophoresis(CE)with UV detection was established.Three sets of primers were designed to amplify the event-specific fragments of GM maize events(Ly038,Mon863 and Mon810). Multiplex PCR and the separation of CE were systematically optimized,8.0g/L hydroxyethylcellulose in trisphosphate-EDTA(TBE)buffer as sieving matrix,three event-specific multiplex PCR products were detected by CE in 11.195min.Using Origin program to analyze the CE results and also get the fitting curves of different DNA range,the relative error was between 1.03%and 5.02%.Unpurified and purified normal PCR products were also detected by CE which suggested purification of PCR products was important for CE with UV detection.Compared with normalPCR and agarose gelelectrophoresis,themultiplexpolymerasechain reaction-capillary electrophoresis method in this study was efficient,rapid,sensitive and low-cost.This event-specific detection method could be widely applied in the fields of food safety and clinical tests.
genetically modified maize;multiplex polymerase chain reaction;capillary electrophoresis;UV detection
TS207.3
A
1002-0306(2011)02-0328-05
2010-01-07 *通讯联系人
张春娇(1986-),女,硕士研究生,研究方向:食品安全检测技术。
国家高技术研究发展计划(863)项目(2006AA10Z440);国家自然基金(30800770);转基因生物重大专项(2008ZX08012-001)。

