基于12S rRNA部分序列分析的中国8种笛鲷科鱼类系统发育初探
唐优良, 章 群, 余帆洋, 周佳怡, 司丛利, 黄小彧, 马 奔
(暨南大学 水生生物研究所, 广东 广州 510632)
基于12S rRNA部分序列分析的中国8种笛鲷科鱼类系统发育初探
唐优良, 章 群, 余帆洋, 周佳怡, 司丛利, 黄小彧, 马 奔
(暨南大学 水生生物研究所, 广东 广州 510632)
测定了中国近海8种笛鲷鱼类线粒体12S rRNA基因部分序列, 结合GenBank中下载的2种笛鲷鱼类序列, 以鲈形目(Perciformes)中的单列齿鲷(Monotaxis grandoculis)、黄带裸颊鲷(Lethrinus obsoletus)、尖嘴蓝子鱼(Siganus unimaculatus)、褐蓝子鱼(Siganus fuscescens)和金眼鲷目(Peryciformes)中的点鳍棘鳍鱼(Sargocentron rubrum)、大鳞锯鳞鱼(Myripristis berndti)、红金眼鲷(Beryx splendens)以及鲤形目(Cypriniformes)中的印度斑马鱼(Danio rerio)为外类群, 构建最大似然树和贝叶斯推断树, 发现在895bp的同源序列中, 共有170个变异位点, 其中简约信息位点126个, 转换/颠换值为2.39; 最大似然树和贝叶斯推断树显示梅鲷鱼类(Caesioninae)镶嵌在笛鲷科(Lutjanidae)鱼类中, 支持将梅鲷科并入笛鲷科的观点, 但是梅鲷属在笛鲷科中的系统位置需要进一步研究; 马拉巴笛鲷(Lutjanus malabaricus)与千年笛鲷(Lutjanus sebae)聚为一支, 支持根据形态学特征的分类处理; 但形态相似的约氏笛鲷(Lutjanus johni)和紫红笛鲷(Lutjanus argentimaculatus)的遗传差异较大, 表明形态特征的相似与亲缘关系的远近并无直接联系。
笛鲷科(Lutjanidae); 马拉巴笛鲷(Lutjanidae malabaricus); mtDNA 12S rRNA; 系统发育
笛鲷科(Lutjanidae)隶属鲈形目(Perciformes), 是广泛分布于全球热带和亚热带海洋中的重要经济鱼类, 目前全世界有17属103种[1], 国内记录有12属37种[2]。笛鲷鱼类的早期分类和系统发育分析主要基于形态学特征[3-4], 不少种类因具有较为相似的形态性状和体色而常常被人们当作同一物种[5], 如红笛鲷(Lutjanidae sanguineus)、马拉巴笛鲷(L.malabaricus)和红鳍笛鲷(L. erythopterus)分类地位和物种有效性一直存在争议[2,4-8]。Johnson[3]和Carpenter[9]将梅鲷属(Caesio)分别归入笛鲷科和梅鲷科(Caesionidae), 而 Johnson[10]和 Reader等[11]则认为梅鲷属应隶属于笛鲷科梅鲷亚科(Caesioninae), 表明主要基于形态特征的笛鲷鱼类系统发育分析上存在不少分歧和困难。
线粒体DNA(Mitochondrial DNA, 简称mtDNA)进化速度较核基因快, 母系遗传, 有效群体数量仅为核基因的 1/4, 是动物种群遗传和进化研究的良好分子标记[12]。Miller等[13]用线粒体16S rRNA和cyt b基因序列研究了印度-太平洋的 27种笛鲷的系统发育, 支持将梅鲷科并入笛鲷科, 但是梅鲷类在笛鲷科中的分类地位因其较低的支持率而不确定。Guo等[14]用线粒体cyt b和COⅡ研究了中国南海12种笛鲷, 认为中国南海存在马拉巴笛鲷。线粒体 12S rRNA基因具有相对保守的进化速率, 可以很好地区分种间和属间的亲缘关系, 已经广泛地应用于鱼类等脊椎动物的分子系统学研究[15-17]。本研究基于线粒体12S rRNA基因序列分析, 构建中国沿海几种常见笛鲷鱼类的系统发育关系, 旨在为笛鲷鱼类的起源演化与种质资源的保护和开发利用提供基础资料。
1 材料和方法
1.1 样品来源
测序样品采自中国南海至台湾海峡, 用 95%酒精固定、保存, 依据 FishBase并参照《中国鱼类系统检索》、《南海鱼类志》和《东海鱼类志》等鉴定。 样品采集和自GenBank下载的序列信息见表1。
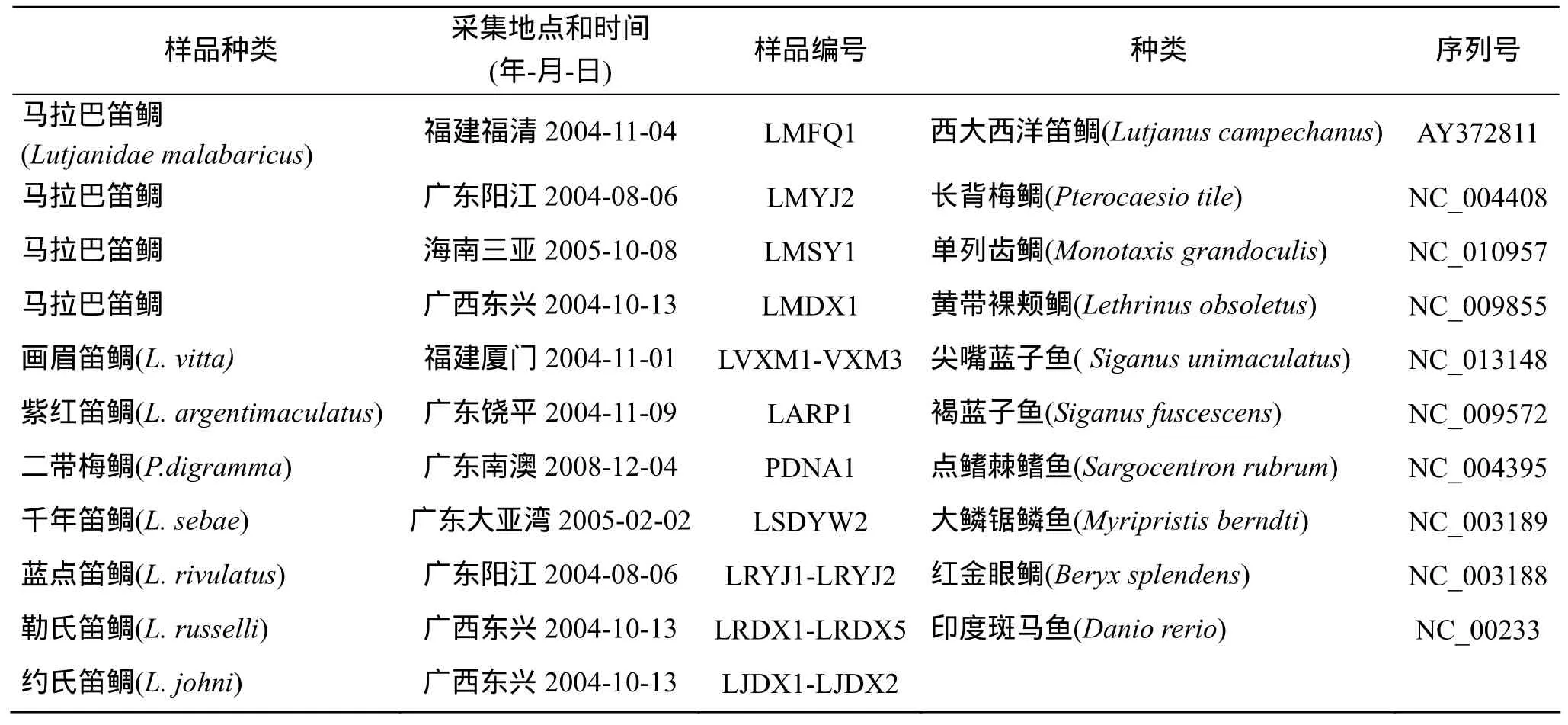
表1 本研究测序的笛鲷鱼类采样信息与GenBank下载的12S rRNA笛鲷和外类群序列Tab. 1 Sampling locations, time, identifiers of lutjanids sequenced in the present study (left) and 12S rRNA sequences of lutjanids and outgroups downloaded from GenBank (right)
1.2 DNA提取与PCR扩增和测序
取适量背部肌肉, 采用高盐法抽提总DNA[18]。引物 12SF:5'-cccgnargcacaaaggcttggtcc和12SR: 5'-tcagctttcccttgcggtac用于PCR扩增和测序。PCR反应与测序条件见文献[19]。
1.3 数据处理
测定序列校对后与GenBank中下载序列合并,用Clustal2软件比对[20]。采用MEGA5计算碱基组成、变异位点、简约信息位点、转换/颠换值, 并以Kimura双参数模型计算遗传距离; 用最大似然法构建系统发育树, 自举检测(Bootstrap)1000次计算各分枝置信度[21]; 使用 MrBayes3.1.2软件包进行贝叶斯推断树构建[22], 其节点支持率为后验概率, 采用jModeltest 0.01软件, 按hLRTs标准选择最适合的替代模型[23]。
2 结果与分析
2.1 线粒体12S rRNA基因序列的特点
比对后截取的笛鲷鱼类 12S rRNA基因部分序列长度为 895bp, 碱基 T、C、A、G 的平均含量为20.5%、26.8%、30.6%、22.2%。序列中共有变异位点170个, 其中简约信息位点126个, 插入/缺失位点20个, 转换/颠换值为 2.39, 表明表明 12S基因的进化尚未达到饱和, 适合进行系统发育分析[21]。
2.2 基于 12S rRNA基因部分序列的笛鲷鱼类系统发育关系
以鲈形目(Perciformes)中单列齿鲷、黄带裸颊鲷、尖嘴蓝子鱼、褐蓝子鱼和金眼鲷目(Peryciformes)中点鳍棘鳍鱼、大鳞锯鳞鱼、红金眼鲷以及鲤形目(Cypriniformes)中的印度斑马鱼为外类群, 最大似然树和贝叶斯推断树见图1。
在最大似然树和贝叶斯推断树中, 笛鲷属与梅鲷属鱼类聚成一个大支(自举检测支持率和后验概率分别为60%和88%)。勒氏笛鲷是画眉笛鲷的姐妹群(自举检测支持率为 95%, 后验概率为 100%), 二者与蓝点笛鲷聚为一个大支, 再与西大西洋笛鲷聚类。马拉巴笛鲷是千年笛鲷的姐妹群(自举检测支持率为92%, 后验概率为 100%), 二者与约氏笛鲷聚为一支;梅鲷属中长背梅鲷和二带梅鲷聚类的自举检测支持率和后验概率均为100%。
3 讨论
3.1 马拉巴笛鲷的鉴定及其分类地位
不同学者对于形态鉴定和参考标准的不同会产生不同鉴定结果。《中国鱼类系统检索》记载红笛鲷分布于中国南海和东海[2], 《南海鱼类志》和《东海鱼类志》只将此种笛鲷记录为红鳍笛鲷, 并认为红笛鲷是红鳍笛鲷的同物异名[7,24]。据FishBase记录, 红笛鲷分布于西印度洋、红海东部至阿拉伯海和纳塔尔南部, 原记录于东印度洋和西太平洋中心的红笛鲷是马拉巴笛鲷的错误鉴定[4]; 红鳍笛鲷分布于印度洋至西太平洋: 阿曼湾至东南亚, 北达日本南部,南至澳大利亚北部; 马拉巴笛鲷分布于印度洋至西太平洋: 波斯湾和阿拉伯海至斐济, 北达日本南部,南至澳大利亚北部。王以康[6]认为中国分布有红笛鲷和马拉巴笛鲷。朱世华等[8]支持红笛鲷和红鳍笛鲷为同物异名的观点。本研究根据所采鱼样尾柄上有黑色鞍状斑且镶以珍珠似的白色边缘, 判定为马拉巴笛鲷, 支持 Guo等[14]中国南海存在马拉巴笛鲷的结论。至于红笛鲷和红鳍笛鲷在中国沿海是否存在以及二者物种有效性和地理分布则有待进一步研究。
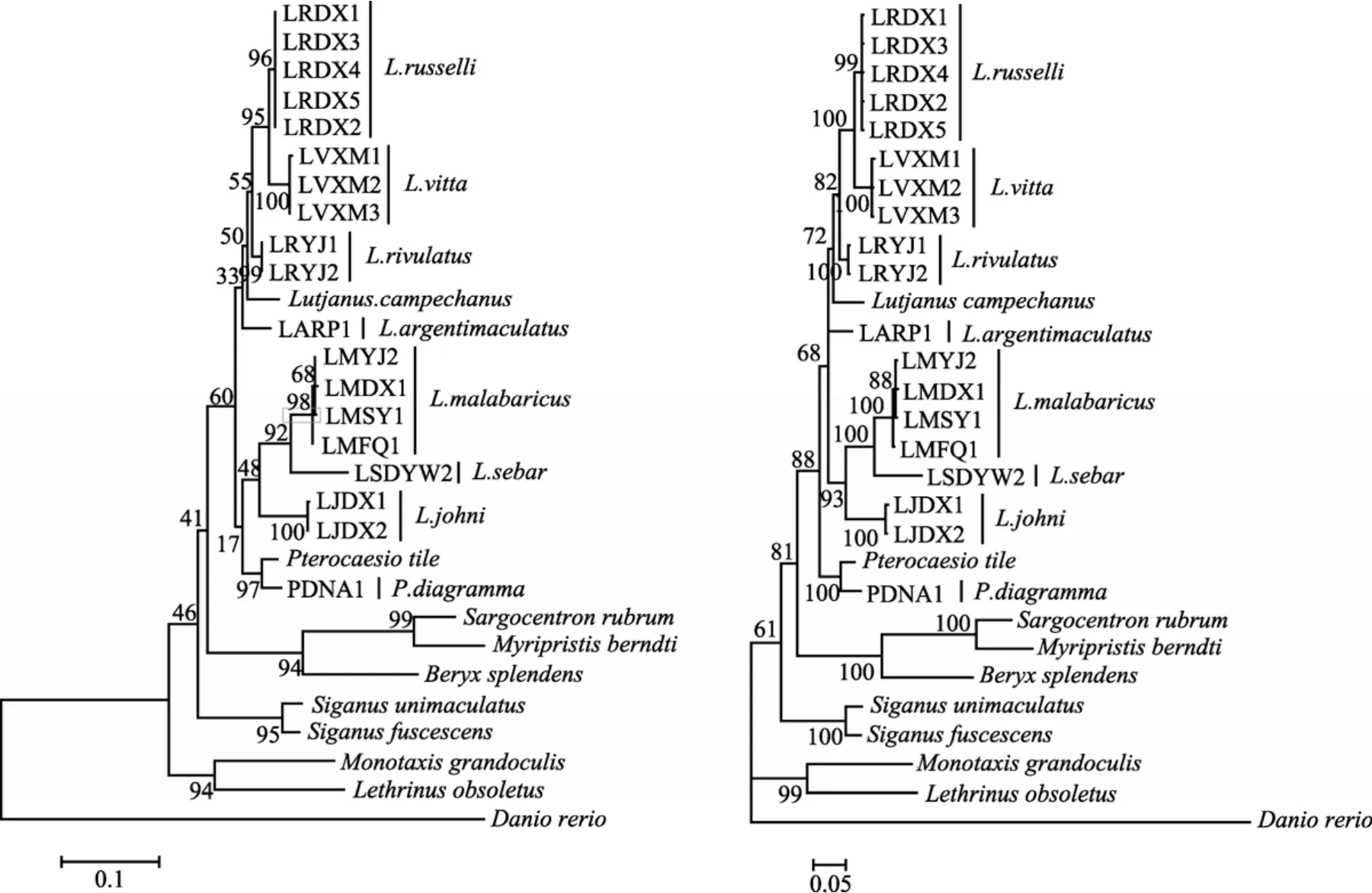
图1 基于12S rRNA基因部分序列的笛鲷鱼类最大似然树(左)和贝叶斯推断树(右)Fig. 1 Phylogenetic trees based on partial 12S rRNA sequences of snappers inferred from maximum likelihood method (left) and Bayesian inference (right)
在分子系统树上, 马拉巴笛鲷与千年笛鲷聚为一支, 后验概率为 100%, 表明二者亲缘关系较近,说明形态学上根据笛鲷侧线上下方鳞片的排列方式来划分是可靠的。
3.2 紫红笛鲷和约氏笛鲷的亲缘关系
紫红笛鲷和约氏笛鲷因具有“全部或大部分与侧线平行的侧线上方鳞列”特征而较其他笛鲷更为密切[2], 但在分子系统树上的系统位置却较为疏远,说明仅根据形态学特征不一定能够正确反映物种间的亲缘关系。由于形态特征的趋同或趋异以及不同性状的独立起源可能造成物种外部形态相似而亲缘关系甚远或形态差异较大而亲缘关系较近的情况[25],因此, 外部形态特征的相似并不一定表明亲缘关系的密切, 仅根据形态特征构建的分类系统仍然需要分子数据的证明。同时, 由于海洋中近缘类群间可能存在的杂交现象[26], 而线粒体仅为母系遗传, 二者间的关系仍需要核基因数据加以证实。
3.3 梅鲷的分类地位
在最大似然树中, 梅鲷属鱼类镶嵌在笛鲷属中,但支持率比较低, 不足以明确肯定梅鲷属鱼类的分类地位; 在贝叶斯推断树中, 梅鲷属与笛鲷属鱼类分别属于不同的支系, 梅鲷属与笛鲷属各自聚类,前者的后验概率为 100%, 而后者聚类的后验概率(68%)则相对较低; 但 2个分支间的遗传距离短, 共同构成一个后验概率为88%的单系分支。
Miller等[13]用16S rRNA和cyt b基因序列研究了印度-太平洋27种笛鲷, 认为梅鲷科应该并入笛鲷科。本研究中12S rRNA基因系统树上梅鲷属鱼类与笛鲷属聚为一支, 支持将梅鲷科并入笛鲷科的分类处理。值得注意的是, 梅鲷属鱼类在笛鲷科中的具体位置随不同分析方法而有所不同。在前者16S rRNA和cyt b基因序列联并分析的贝叶斯推断树和最大简约树中, 梅鲷属鱼类镶嵌在笛鲷属鱼类之中, 支持率不高[13]; 本研究12S rRNA基因最大似然树中梅鲷属鱼类同样以较低的支持率镶嵌在笛鲷属鱼类之中,而在贝叶斯推断树中, 梅鲷鱼类却单独成支, 并且支持率较高, 表明梅鲷属在笛鲷科与其他属种的亲缘关系仅仅从线粒体12S rRNA, 16S rRNA和cyt b基因的角度来分析可能是不够的, 仍需要结合线粒体其他基因或核基因或增加所分析的属种数量来确定。
[1] Froese R, Pauly D. FishBase[EB]. version(09/2010).http://www.fishbase. org/2010-12-25.
[2] 成庆泰, 郑葆珊. 中国鱼类系统检索[M]. 北京: 科学出版社, 1987: 327-332.
[3] Johnson G D. The limits and relationships of the Lutjanidae and associated families[J]. Bull Scripps Inst Ocean, 1980: 24: 1-114.
[4] Allen G R. FAO species catalogue, snappers of the world. An annotated and illustrated catalogue of lutjanid species known to date[J]. FAO Fisheries Synopsis,1985, 6(125): 1-103.
[5] Marko P B. Fisheries: mislabelling of a depleted reef fish[J]. Nature, 2004, 430: 309-310.
[6] 王以康. 鱼类分类学[M]. 上海: 科技卫生出版社,1958: 299-303.
[7] 朱元鼎, 张春霖, 成庆泰. 东海鱼类志[M]. 北京: 科学出版社, 1963: 303-304.
[8] 朱世华, 杨迎春, 沈锡权, 等. 从细胞色素 b 基因序列探讨笛鲷属的分子系统发生关系[J]. 动物学报,2006, 52(3): 514-521.
[9] Carpenter K E. A phylogenetic analysis of the Caesionidae (Perciformes: Lutjanoidea)[J]. Copeia, 1990, 3:692-717.
[10] Johnson G D.Percomorph phylogeny:progress and problems[J]. Bulletin of Marine Science, 1993, 52(1): 3-28.
[11] Reader S E, Leis J M. Larval development in the lutjanid subfamily Caesioninae (Pisces): The genera Caesio, Dipterygonotus, Gymnocaesio and Pterocaesio[J].Bulletin of Marine Science, 1996, 59(2): 310-369.
[12] Harrison R G. Animal mitochondrial DNA as a genetic marker in population and evolutionary biology[J].Trends in Ecology & Evolution, 1989, 4(1): 6-11.
[13] Miller T L. Cribb T H. Phylogenetic relationships of some common Indo-Pacic snappers (Perciformes: Lutjanidae) based on mitochondrial DNA sequences, with comments on the taxonomic position of the Caesioninae[J]. Molecular Phylogenetics and Evolution, 2007,44: 450-460.
[14] Guo Y, Wang Z, Liu C, et al. Phylogenetic relationships of South China Sea snappers(genus Lutjanus; family Lutjanidae) based on mitochondrial DNA sequences[J].Mar Biotechnol(NY), 2007, 9(6): 682-688.
[15] Sarver S K, Freshwater D W, Walsh P J. Phylogenetic relationships of Western Atlantic snappers (family:Lutjanidae) based on mitochondrial DNA sequences[J].Copeia, 1996, 3: 715-721.
[16] Wang H Y, Tsai M P, Dean J, et al. Molecular phylogeny of gobioid fishes (Perciformes: Gobioidei) based on mitochondrial 12S rRNA sequences[J]. Molecular Phylogenetics and Evolution, 2001, 20(3): 390-408.
[17] Zhang J, Liu X. The phylogenetic relationship of the family Lutjanidae based on analyses of AFLP and mitochondrial 12s rRNA sequences[J]. Chinese Science Bulletin, 2006, 51: 143-148.
[18] Aljanabi S M, Martinez I. Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques[J]. Nucleic Acids Res, 1997, 25: 4 692-4 693.
[19] 陈艺燕, 章群, 任岗, 等. 10种石斑鱼系统发育的线粒体细胞色素 b 基因序列分析[J]. 海洋科学, 2006,30(6): 12-15.
[20] Larkin M A, Blackshields G, Brown N P, et al. Clustal W and clustal X version 2.0[J]. Bioinformatics, 2007,23: 2 947-2 948.
[21] Tamura K, Dudley J, Nei M, et al. MEGA4: molecularevolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24:1 596-1 599.
[22] Huelsenbeck J P, Ronquist F.MRBAYES: Bayesian inference of phylogenetic trees[J]. Bioinformatics,2001, 17(8): 754-755.
[23] Posada D. jModelTest: phylogenetic model averaging[J]. Molecular Biology and Evolution, 2008, 25:1 253-1 256.
[24] 中国科学院动物研究所, 中国科学院海洋研究所, 上海水产学院. 南海鱼类志[M]. 北京: 科学出版社,1962: 463-485.
[25] Sturmbauer C, Meyer A. Mitochondrial phylogeny of the endemic mouthbrooding lineages of cichlid fishes from Lake Tanganyika in Eastern Africa[J]. Molecular Biology and Evolution, 1993, 10(4): 751-768.
[26] Gardner J P A. Hybridization in the sea[J]. Adv Mar Biol, 1997, 31: 1-78.
Received: Oct., 15, 2010
Key words:Lutjanidae;Lutjanidae malabarius; mtDNA 12s rRNA; phylogeny
Abstract:Partial sequences of 12S rRNA gene of eight snappers collected in coastal waters of china determined in the present study and two lutjanid sequences downloaded from GenBank were combined to reconstruct Maximum likelihood and Bayesian inference trees withSiganus unimaculatus,S. fuscescens, Monotaxis grandoculis,Lethrinus obsoletus(perciformes),Myripristis berndti,Beryx splendens, Sargocentron rubrum(Beryciformes), andDanio rerio(Cypriniformes) as outgroups. Totally 170 variable sites and 126 parsimony-informative sites existed in the 895 bp sequences, and the ts/tv ratio was 2.39. Phylogenetic trees showed that the Caesioninae were nested within the Lutjaninae, supporting the recent opinion that the Caesionidae shoud be regarded as a synonym of the Lutjanidae, but their phylogenetic relationships to other lutjanid species remained to be further studied.L.malabariusandL.sebaeformed a well-supported clade, supporting the taxonomical treatment ofL.malabariusandL. sebaebased on morphological characters; however morphologically similar speciesL. johniandL. argentimaculatuswere genetically quite different, indicating that morphologically similar species were not necessarily genetically closely related.
(本文编辑:谭雪静)
Phylogeny of eight snappers (Lutjanidae) in Chinese coastal waters inferred from partial mtDNA 12S rRNA Sequences
TANG You-liang, ZHANG Qun, YU Fan-yang, ZHOU Jia-yi, SI Cong-li,HUANG Xiao-yu, MA Ben
(Institute of Hydrobiology, Jinan University, Guangzhou 510632, China)
Q959.483
A
1000-3096(2011)02-0022-05
2010-10-15;
2010-12-15
国家自然科学基金资助项目(30770415、41071034); 留学归国人员科研启动基金项目(2005); 暨南大学科研培育与创新基金资助项目(21609710)
唐优良(1986-), 男, 湖南人, 硕士, 主要从事分子生态学研究, E-mail: tangyouliang123@163.com; 章群, 通信作者, E-mail:
tqzhang@jnu.edu.cn

