乌苏里拟鲿野生群体和人工养殖群体遗传多样性的比较研究
(苏州大学 医学部基础医学与生物科学学院,苏州大学 水产研究所,江苏 苏州 215123)
乌苏里拟 (Pseudobagrus ussuriensis)曾用名乌苏里,俗称牛尾巴,隶属于硬骨鱼纲(Dsteichthyes)、辅鳍亚纲(Actinopteri)、鲇形目(Silurformes)、科(Bagridae)、拟 属(Pseudobagrus)。因其杂食性、生长快、肉质鲜等特点,成为优良的养殖品种[1]。同其他许多鱼类一样,乌苏里拟 的养殖群体是依靠采捕野生亲鱼进行大量繁殖而来的,随着人工繁殖技术的突破以及天然野生亲鱼群体数量的严重不足,难免会导致近亲繁殖的发生。多代乌苏里拟 自交养殖群体可能会产生基因库萎缩和经济性状衰退等问题,这将严重影响乌苏里拟 种质资源保护及其养殖业的进一步发展。目前,有关于乌苏里拟 的研究主要集中在人工繁育、生化遗传学等领域[2-4],至于其分子方面的研究尚未见到报道。相关序列扩增多态性(sequence-related amplified polymorphism,SRAP)具有简便、稳定、中等产率和容易得到选择条带序列等优点[5-6],已越来越多地被应用于植物和动物包括鱼类基因组的分析中[7-9]。本文在确定该技术对乌苏里拟 遗传多样性分析适用性的基础上,通过比较乌苏里拟 野生群体和人工养殖群体间的遗传结构差异,首次从分子水平探讨乌苏里拟 遗传多样性的变化,为制定乌苏里拟 的种质标准提供基础资料,也为该鱼人工养殖群体的遗传结构是否产生遗传变异提供理论依据。从而为其珍贵的野生资源的可持续地利用以及养殖业的可持续发展提供保障。
1 材料与方法
1.1 材料
乌苏里拟 野生群体采自江苏省洪泽湖(HL,32尾);淮安市水产研究所的人工繁殖群体(HA,34尾,F2代)为2006年人工繁殖的乌苏里拟 ,其亲本(F1代)是 2000年由洪泽湖捕获的野生成鱼繁殖而来的;苏州市东山水产养殖场的人工繁殖群体(SZ,31尾,F3代) 为 2009年人工繁殖的乌苏里拟 ,其亲本(F2代)是2006年底从淮安市水产研究所购进的1万尾乌苏里拟 夏花培育而成的。本实验从以上 3个群体中随机取样的情况见表1。
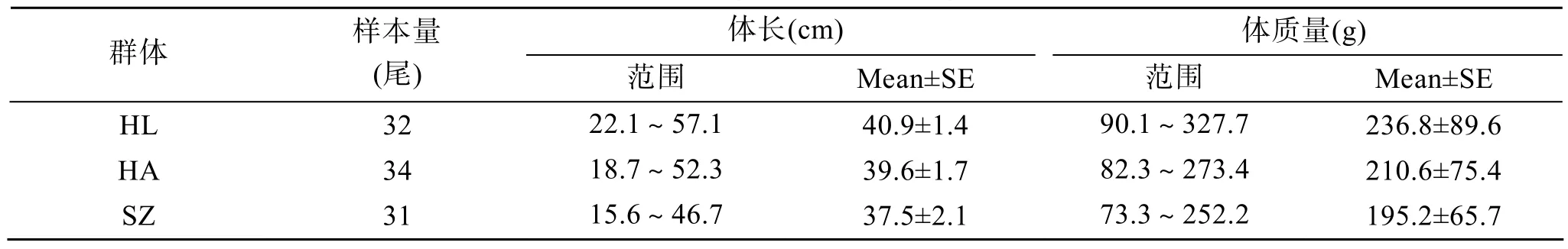
表1 3个乌苏里拟鲿群体的样本量、体长及体质量范围Tab.1 Amount of samples,body lengths,and body weights of Pseudobagrus ussuriensis
1.2 基因组DNA的提取
剪取鳍条0.1 g,加200 µLSTE缓冲液(10 mmol/L Tris-Cl,10 mmol/L NaCl,1 mmol/L EDTA),20µLSDS,5µL蛋白酶K(20 g/L),于55℃下消化过夜,之后用酚-氯仿抽提和乙醇沉淀的方法提取其基因组DNA,经 l%琼脂糖凝胶电泳检测,分光光度法定量分析,将DNA稀释至0.05 g/L。
1.3 SRAP引物
参照文献[10]和文献[11]设计引物,引物由上海生物工程公司合成,其序列见表2。上下有交叉组合得到100对引物。Taq酶、dNTP、DNA Maker等均为上海生工产品。

表2 SRAP引物Tab.2 Primers and sequences of SRAP
1.4 SRAP-PCR反应及电泳检测
PCR反应总体积为25 µL,包括PCR Buffer 2.5µL,Mg2+2 µL,dNTPs 0.2 mmol/L,DNA 模板(0.05 g/L)1 µL,1.0 µmol/L 引物 1 µL,Taq 酶 1.0 U。反应的程序为:94℃预变性5 min,94℃变性1 min,35℃退火1 min,72℃延伸2 min,5个循环;94℃变性1 min,53℃退火1min,72℃延伸2 min,35个循环;72℃下延伸7 min。扩增产物经8%的聚丙烯酰胺凝胶电泳分离,银染法染色,数码相机拍摄保存。
1.5 数据处理
用Gel-Pro analyzer对显色后的SRAP电泳图谱进行统计分析,每一条带视为一个位点,有带记为“1”,无带记为“0”。转换成数字矩阵再进行分析,选择基因频率作为比较指标,用 PopGene软件进行分析,并统计相应的数据。相关计算公式如下:
(1) 群体多态位点Pi=该群体的多态位点数/位点总数×100%
(2) 隐性等位基因a的基因频率q是根据群体中隐性纯合个体(即对应某一扩增位点,不出现该扩增带的个体) 频率的平方根计算而来,对应的显性等位基因A 的基因频率p= 1-q,文中提到的基因频率均为每位点显性等位基因的基因频率。
(3) 遗传相似系数Sij=Nij/ (Ni+Nj+Nij)[12]
其中,Sij为任意两个个体间的遗传相似系数,Nij为i个体和j个体共享位点数,Ni、Nj分别为i个体和j个体各自扩增的位点总数。群体间的遗传相似系数为两群体任意两个体遗传相似系数的平均数。Sij为两群体间的遗传相似系数;Si、Sj分别表示群体i和群体j的遗传相似系数。
2 结果与分析
2.1 SRAP扩增结果

图1 部分引物对乌苏里拟 3个群体的扩增结果Fig.1 PCR amplification in the three populatins
用筛选出的12对SRAP引物组合对3个乌苏里拟 群体共扩增出202个清晰稳定的位点(图1),每组引物对乌苏里拟 3群体扩增均产生 11~17个扩增位点,片段大小在 572~111bp,其中多态位点有130个,多态位点比例为64.36%。对各引物组合扩增的位点总数及多态位点比例的统计结果发现,不同引物组合在 3个群体中的扩增位点总数和多态位点数均有显著差异(表3)。但是,除 F8R8引物组合中,野生群体的多态位点数少于两个养殖群体的多态位点数外,其他各引物组合在野生群体中所扩增出的多态位点数均多于2个养殖群体,野生HL群体的多态位点率为 63.13%,养殖 HA群体的多态位点率为56.54%,而养殖SZ群体的多态位点率为54.88%。
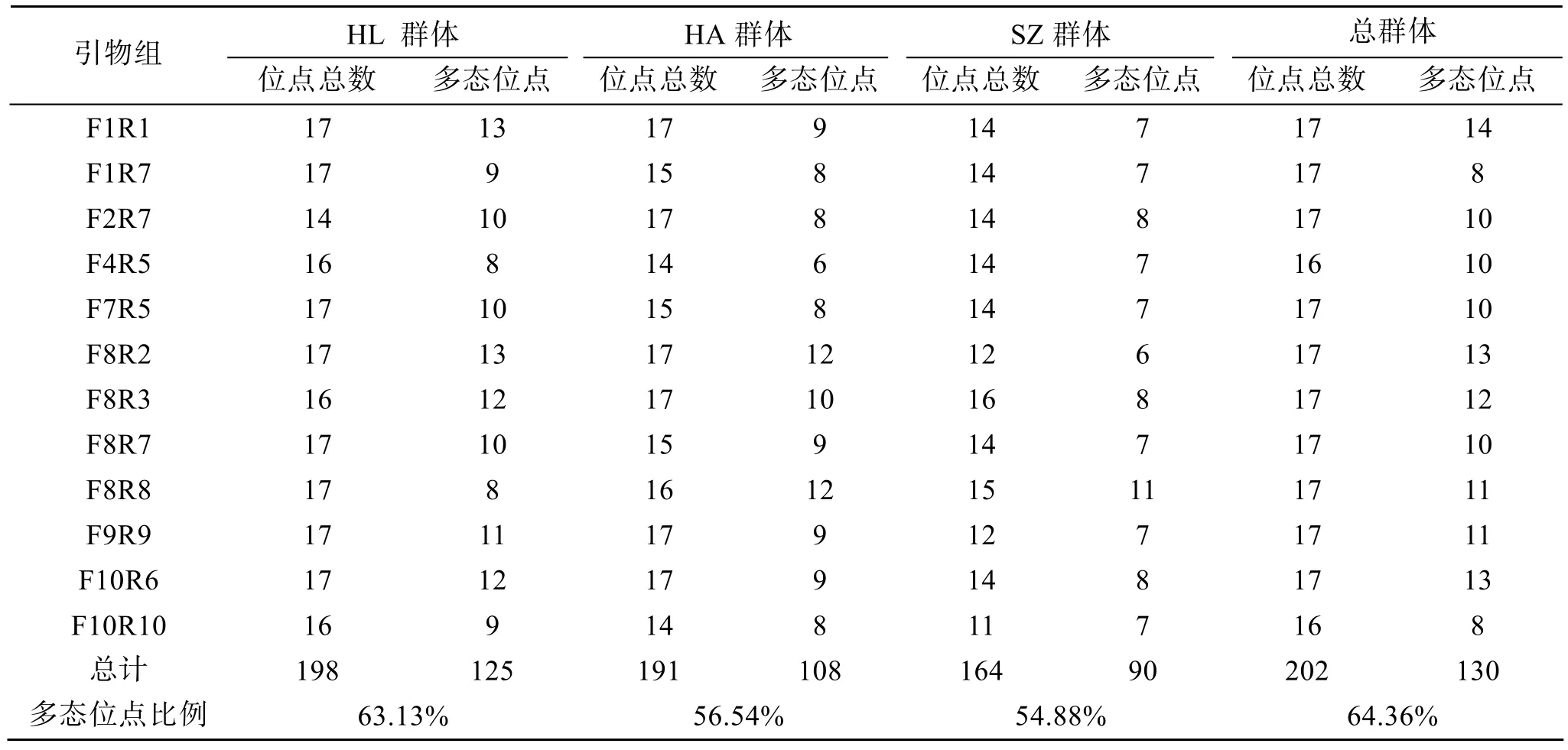
表3 3个群体扩增位点数及多态位点比例Tab.3 Number of ampliphied loci and the percentages of polymorphic loci in the three populations
2.2 三个群体扩增位点数在不同显性基因型频率的分布
参照张志伟等[14]和张全启等[15]的方法,将 202个扩增位点的显性基因型频率以 10%为单位划分区间,0和 100%分别设为单独的区间,这样共划分为12个区间,统计显性基因型频率在各区间内的位点数。结果表明,3个群体在不同显性频率区间内的扩增为点数呈现一定的分布规律,反映了 3个群体的遗传结构特点。2个养殖群体在不同区间内的分布规律基本相同,在 100%区间位点数较多,其他区间位点数则相对较少,在不同区间位点数的变化规律则基本一致,这表明两个养殖群体的遗传结构非常相近;与野生群体的比较显示,在 1%~9%区间内野生群体的多态位点显著多于两个养殖群体,位点数分别为野生群体53个、养殖群体21个、养殖群体24个,在100%区间野生群体位点数显著少于养殖群体,其他区间野生群体与养殖群体的位点数分布规律则无显著差异(图2)。这表明养殖群体中的隐形纯合位点明显高于野生群体,养殖群体中的低频位点显著减少,养殖群体的遗传多样性水平有所降低。
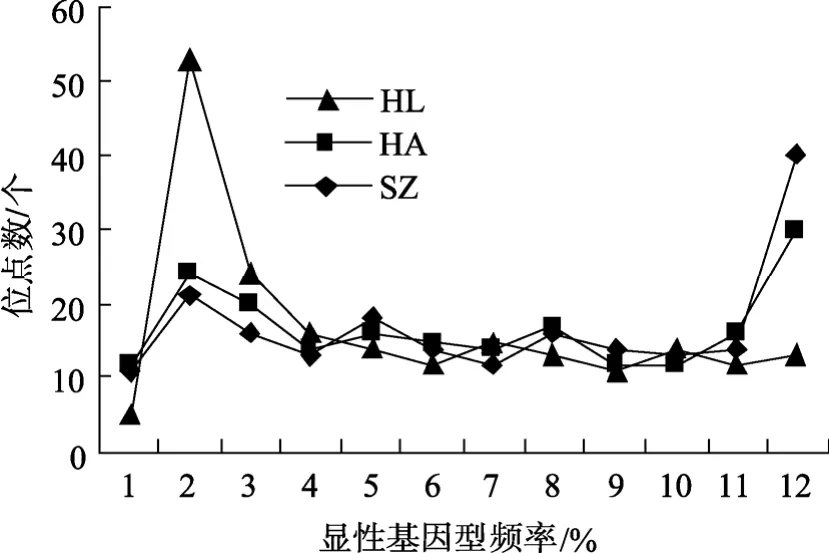
图2 乌苏里拟 3群体扩增位点数在不同显性基因型频率区间内的分布Fig.2 Distributions of amplified loci of the three populations in different frequency intervals
2.3 3个群体的遗传多样性指数及遗传距离
Nei’s基因多样性指数(H)和 Shannon’s信息指数(I)是衡量群体遗传多样性的重要参数。Nei’s基因多样性指数(H)反映的是群体在数个基因座位上的遗传变异,是最适合衡量群体遗传变异的一个参数,其值的大小可以反映群体遗传变异的高低。从表4中可以看出,乌苏里拟 野生群体的Nei’s基因多样性指数和Shannon’s信息指数(I)更高,两个养殖群体的Nei’s基因多样性指数和 Shannon’s信息指数(I)则相对较低,这表明野生群体的遗传多样性水平高于养殖群体。从表5中可以看出,HL野生群体与HA养殖群体间的遗传距离最大为 0.1730,两个养殖群体间的遗传距离最小为0.1087,HL野生群体与SZ养殖群体间的遗传距离居中。这表明养殖群体存在较为严重的遗传分化,且遗传多样性水平降低。

表4 乌苏里拟鲿3群体的遗传多样性指数Tab.4 Genetic diversity indices of each population

表5 乌苏里拟鲿3群体的遗传相似系数及群体间的遗传距离Tab.5 Intra-population genetic similarity and relative genetic distances among three populations
3 讨论
3.1 SRAP在乌苏里拟 遗传多样性研究中的适用性
SRAP呈显性标记,一条带代表一等位基因[16]。其主要是利用独特的引物设计,对开放式阅读框架进行扩增。正向引物长 17bp,5′端的前10bp是一段填充序列,紧接着是 CCGG,它们组成核心序列,核心序列及3′端的3个选择性碱基,能对外显子进行特异扩增。反向引物长18bp,5′端的前10-1lbp是一段填充序列,紧接着是 AATT,它们组成核心序列,在3′端3个选择碱基的协同作用下,对内含子区域、启动子区域进行特异性扩增[17]。SRAP由于其独特的引物设计原则以及其成本低、重复性强、稳定等优点,已广泛应用于作物及水产动物种质资源方面的研究[18-20]。郑翠芳等[21]采用 153对引物对爱玉子的性别及品系进行了研究,得到一 200bp的特异条带将雌雄株分开,并用8对引物对24个雌性品系和16个雄性品系进行比较扩增,显示较高的多态性。张志伟等[14]采用88对引物对草鱼的3个群体进行SRAP分析,结果表明草鱼人工繁殖群体的遗传多样性水平有所下降。辛文婷[22]采用 100对引物构建了基于SRAP分子标记的黄颡鱼遗传图谱。傅洪拓等[23]采用SRAP分子标记对海南沼虾的5个中群体进行遗传多样性分析,为海南沼虾的种质资源保护和利用提供了理论依据。本研究构建了适用于乌苏里拟 分子标记的 SRAP反应体系,并筛选出合适的引物组合,对乌苏里拟 1个野生群体和2个人工养殖群体的遗传结构进行了比较研究,表明 SRAP分子标记同样适用于乌苏里拟 遗传多样性的分析。
3.2 鱼类野生群体与人工养殖群体间的遗传多样性分析
作者采用稳定性强、产率高的 SRAP分子标记首次对乌苏里拟 1个野生群体和2个人工养殖群体的遗传结构进行比较,3个群体(HL群体、HA群体、SZ群体)的多态位点比例分别为63.13%、56.54%、54.88%;Nei’s基因多样性指数(H)为 0.2641、0.2546、0.2469;Shannon’s 信息指数(I)为 0.4118、0.4050、0.3861。与野生群体相比,2个养殖群体的多态位点比例、Nei’s 基因多样性指数(H)、Shannon’s 信息指数(I)有着不同程度的下降,这说明养殖群体遗传多样性水平较低,且存在较为明显的遗传分化趋势。不同学者在其他物种的研究中同样发现类似现象,如:张志伟等[14]采用 SRAP分子标记技术对草鱼野生群体和人工繁殖群体遗传结构的比较研究发现:草鱼养殖群体期望杂合度平均值显著低于野生群体,部分等位基因丢失,推测主要是亲本数量少以及养殖环境发生改变,使得养殖群体出现中指上的同质化,致使部分等位基因丢失;张全启等[15]采用 AFLP分子标记技术在对牙鲆野生群体和养殖群体的遗传多样性分析时不仅检测到养殖群体的遗传杂合度降低,还检测到由于低频位点的丢失造成总数目位点的减少,推测可能是由于在人工繁殖过程中一些不合理的因素(如繁殖亲鱼数量少、遗传漂变等)造成的。潘伟志等[4]采用聚丙烯酰胺凝胶电泳方法对黑龙江水系乌苏里拟 心、肝、肾、眼、肌肉和性腺共 6种组织进行了 9种同工酶生化遗传学分析,结果表明,乌苏里拟 EST、GDH、IDH、POD同工酶谱中存在不同程度的组织特异性,EST表现出明显的性别差异性。他们还通过电泳酶谱带命名和遗传学参数计算的方法,共记录了 19个基因位点,多态位点百分数P=21.05%,种群遗传偏离指数d=-1,该乌苏里拟 种群内部杂合子缺失较严重,偏离 Hardy-Weinberg平衡,养殖群体遗传多样性较低,这也与本文的研究结果相吻合。潘伟志等[3]对乌苏里拟 繁殖生物学的研究结果表明,体长 225~468mm 个体的绝对怀卵数为(2684±1029)粒,发现比野生群体自然繁殖期中的怀卵数量少,这可能与养殖群体遗传结构的改变有关。我们对F3代进行人工繁殖时也发现了类似现象,是由于养殖群体的遗传多样性降低还是环境和养殖条件等因素改变导致的繁殖力下降,有待于进一步研究。
本研究采用 SRAP分子标记技术检测到养殖群体的遗传多样性有所降低,还发现在养殖群体中低频位点显著减少,而隐性纯合基因显著增加。推测其原因,可能是养殖群体的亲本数量比较少,尤其在人工繁殖时由于雄鱼个体远大于雌性个体,常常出现雌雄亲鱼数量比例过高的现象,导致人工养殖群体遗传结构发生改变,遗传漂变的结果使得后代中一些稀有位点丢失、低频位点显著减少,而隐性纯合基因显著增加。但从遗传距离及遗传分化的角度分析,人工养殖群体还没有形成独立的遗传结构。因此,在未来乌苏里拟 的保种或人工繁殖和养殖工作中,应该注意亲本的更新,并选用足够大的、有代表性的亲本群体,尤其在人工繁殖时要保证雄性亲鱼的使用量,从而在保持优良性状的同时最大限度的保留群体的遗传多样性,以促进乌苏里拟 种质资源保护和养殖产业的可持续发展。
[1]薛淑群,尹洪滨.乌苏里拟 的染色体组性研究[J].水产学杂志,2008,25(2):75-78.
[2]王鹏,赵春刚,叶继丹,等.乌苏里拟 人工繁育技术及开发利用——乌苏里拟 仔幼鱼食性与生长的初步研究[J].水产学杂志,2001,14(2):4-6.
[3]潘伟志,王鹏,赵春刚.乌苏里拟 人工繁育技术及开发利用——乌苏里拟 繁殖生物学及人工催产初步研究[J].水产学杂志,2001,14(2):1-3.
[4]潘伟志,尹洪滨,孙中武,等.乌苏里拟 同功酶分析[J].东北林业大学学报,2006,34(6):66-69.
[5]海燕,何宁,康明辉等.新型分子标记 SRAP及其应用[J].河南农业科学,2006,9:9-11.
[6]杨迎花,李先信,曾柏全,等.新型分子标记 SRAP的原理及其研究进展[J].湖南农业科学,2009,(5):15-17.
[7]林忠旭,张献龙,聂以春,等.棉花 SRAP遗传连锁图构建[J].科学学报,2003,48(15):1676-1679.
[8]丁炜东,曹丽萍,曹哲明.草鱼种质相关 SRAP及SCAR的分子标记[J].动物学报,2008,54(3):475-481.
[9]田明礼,吴孝兵.扬子鳄SRAP-PCR反应体系的优化及引物筛选[J].安徽师范大学学报(自然科学版).2008,31(2):163-167.
[10]Li G,Quiros C F.Sequence-related amplified polymorphism (SRAP),a new marker system based on a simple PCR reaction its application to mapping and gene tagging inBrassica[J].Theor Appl Genet.2001,103:455-461.
[11]辛文婷,孙中武,尹洪滨,等.黄颡鱼雌雄差异的SRAP标记[J].东北林业大学学报,2009,37(5):112-113.
[12]You Feng,Xiang Jianhai,Song Linsheng,et al.Genetic variation in natural and cultured stocks of SHANDONGParalichthys olivaceusas revealed by RAPD[J].Studia Marina Sinica,2002,44:28-234.
[13]You Feng,Zhang Peijun,Wang Keling,et al.Genetic variation of natural and cultured stocks ofParalichthys olivaceusby allozyme and RAPD[J].Chinese Journal of Oceanology and Limnology,2007,25(1):78-84.
[14]张志伟,仲霞铭,张志勇,等.草鱼野生群体和人工繁殖群体遗传结构的比较研究[J].中国水产科学,2007,14(5):721-725.
[15]张全启,徐晓斐,齐洁,等.牙鲆野生群体和养殖群体的遗传多样性分析[J].中国海洋大学学报,2004,34(5):816-820.
[16]李弛.利用SRAP、SSR分子标记检测分析29份棉花种质遗传完整性[J].植物遗传资源学报.2007,8(1):21-25.
[17]姚建华,傅洪拓,龚永生,等.SRAP分子标记技术及其在水产育种中的应用前景[J].安徽农学通报.2009,15(08):53-55.
[18]程宁宁,杨爱国, 刘志鸿,等.栉孔扇贝(♀)×虾夷扇贝(♂)子一代杂种优势的SRAP分析[J].海洋科学,2009,10:107-111.
[19]王守现,刘宇,耿小丽,等.应用 SRAP标记对六个鸡腿菇菌株的多态性分析[J].江西农业大学学报 ,2006,05:753-757.
[20]李巧燕,林瑞庆,朱兴全.SRAP分子标记及其应用概述[J].热带医学杂志.2006,6(4):467-469.
[21]郑翠芳,朱晓东,方丽娜,等.爱玉子性别与品系的SRAP分析[J].热带作物学报.2009,30(12):1 740-1 745.
[22]辛文婷.黄颡鱼 SRAP遗传图谱的构建[D].哈尔滨:东北林业大学,2009.
[23]傅洪拓,乔慧,姚建华,等.基于 SRAP分子标记的海南沼虾种群遗传多样性[J].生物多样性,2010,18(2):150-154.
——乌苏里白鲑

